耐有机溶剂氨基酸脱氢酶基因挖掘与非天然氨基酸的非水相合成
2021-07-24段凌暄姚光晓江亮王世珍
段凌暄,姚光晓,江亮,王世珍
(厦门大学化学化工学院化学工程与生物工程系,福建厦门361005)
引 言
非水相酶催化在手性中间体药物合成、精细化学品和生物能源生产中广泛应用[1-2]。脂肪酶、酯酶等水解酶非水相体系催化研究已经广泛开展[3],而针对耐有机溶剂氧化还原酶的研究较少。氧化还原酶结构复杂,往往由多个亚基组成,并具有辅酶和底物等多个结合位点,以及催化、调控等多个功能模块,因此在有机溶剂体系中易失活。开发耐有机溶剂的氧化还原酶不仅可用于非水相体系制备手性化合物、开发体外诊断试剂和有机相酶传感器[4],也可为构建非水相体系的体外合成生物学平台提供支撑。对耐有机溶剂的氧化还原酶性能的挖掘、开发及其对溶剂体系的应答有待进一步研究。由于自然界缺乏大量有机溶剂存在的生境,通过筛选获得耐受有机溶剂的氧化还原酶仅有少数报道[5]。
海洋、盐湖等高盐、高渗透压等极端生境与有机溶剂体系中的低水活度相似。因此,部分嗜盐、耐盐菌株来源的耐盐酶具有耐有机溶剂的特性,可用于非水相酶催化[6]。Alsafadi 等[7]报道了来源于Haloferax volcanii的嗜盐醇脱氢酶在高盐浓度下才能正确折叠并保持酶活性,二甲基亚砜(DMSO)和甲醇可以取代部分盐以维持酶的结构并提高酶稳定性。Munawar 等[8]报道了来源于Halobacterium salinarum的谷氨酸脱氢酶分别在含10%的甲醇、乙醇、乙腈和二甲基亚砜反应体系中,室温下保存8 d基本保持活性。Timpson 等[9]发现来源于死海极端嗜盐菌Haloarcula marismortui的乙醇脱氢酶对高盐、高碱、有机溶剂具有良好的耐受性,并且最适温度为60℃,在工业生物催化中具有应用优势。极端氧化还原酶具有与陆地来源酶迥异的底物特异性和催化性能,无疑会给众多的生物催化领域增添新的活力[10-11]。来自极端环境的大多数微生物不可培养,即便获得少数培养菌株,对培养条件要求也比较特殊,例如低温、含盐。因此研究极地等特殊环境中的微生物应该采取特殊的培养技术以及基因组学数据挖掘与酶学性质考察等研究方法相结合[12-15]。
氨基酸脱氢酶(amino acid dehydrogenase,AaDH)催化不对称还原胺化,可合成系列手性天然氨基酸、非天然氨基酸和手性胺等。这些手性化合物均为医药、农药、食品添加剂等精细化学品的重要手性砌块[16-17]。L-高苯丙氨酸可用于合成血管紧张素转换酶抑制剂,包括苯那普利、依那普利、赖诺普利和奎那普利,已被广泛用于治疗高血压和心脏病[18]。目前氨基酸脱氢酶的研究主要基于催化位点和结合位点的修饰提高活性、拓展底物谱和提高稳定性,而对耐有机溶剂性能报道较少[19]。获得高效稳定、耐盐、耐有机溶剂的具有自主知识产权的氨基酸脱氢酶对于我国工业应用和临床检测、摆脱对国外进口试剂依赖有着极大意义。
本文针对耐有机溶剂的氨基酸脱氢酶难以通过点突变改造获得的难题,基于极端微生物的基因组挖掘与分析,获得耐盐氨基酸脱氢酶特征序列并利用热力学数据分析,进一步模拟对接筛选获得目标酶。通过基因合成、表达和分离纯化,并进行实验验证。基于生物信息学与实验验证相结合的方法,阐明耐有机溶剂酶的机制,发展催化效率提升策略。
1 实验材料和方法
1.1 基因挖掘
基于NCBI和Microscope[20]基因数据库分析平台的资源,针对网络数据库中已有的极端环境来源的菌株基因组序列,挖掘获得潜在的极端氨基酸脱氢酶编码基因。主要采用特征结构域挖掘的方法,以与NAD(P)H 结合的NAD(P)-binding domain (结构域ID:IPR016040)作为指针挖掘氨基酸脱氢酶,以避免未被明确标注功能的脱氢酶序列的漏检。
1.2 序列比对与同源模建
对已筛选获得的氨基酸脱氢酶进行分析,利用Blast 等[21]进行基因序列比对,选择亲缘性较远的具有代表性的酶分别进行同源模建。采用iTasser 网络服务器进行同源模建[22],获得蛋白三维结构图,利用SWISS-MODEL 获得拉氏构象图[23],对所获得的立体结构进行Ramachandran 构象评价,获得的结构模型在允许区和最大允许区的氨基酸残基占整个蛋白质的比例高于90%,认为该模型的构象符合立体化学的规则,结构合理。
1.3 热力学参数模拟计算
将同源模建获得的酶的结构模型提交Scoop 网络服务器(http://babylone.ulb.ac.be/SCooP),通过计算获得表征酶热稳定性的热力学参数,包括焓变(ΔHm), Gibbs 自由能变化(ΔGr),蛋白质热容量变化(ΔCp)和蛋白溶解温度(Tm)[24]。
1.4 结构比对与底物对接
将酶与天然底物L-苯丙氨酸和非天然氨基酸底物L-高苯丙氨酸进行对接。小分子模型由ChemBioDraw 绘制2D 结构式,随后转化为3D 结构,并且对3D 结构进行能量最小化操作来优化结构。对接工作通过AutoDock4.0 完成。输入配体文件后首先进行加氢、加电荷等准备工作,并保存为pdbqt格式。随后在AutoDock 中载入NT2349 蛋白结构作为受体。进行去除水分子,氨基酸残基加氢、加电荷等准备工作,同样转化为pdbqt 文件。在准备工作完成后,对受体进行活性空腔的扫描,确定对接区域。并设置对接参数,利用AutoDock中的Vina程序完成对接工作。
1.5 酶的表达和分离纯化
所用的菌株为重组表达菌株E.coliBL21(pET28a-AaDH),氨基酸脱氢酶(NT2349)的制备经过了基因合成、质粒导入、菌株活化、诱导培养等步骤。
表达NT2349 的重组大肠杆菌活化[25]:从-20℃保存的甘油管中取出保存的NT2349 菌种,以1%的比例接种。LB 培养基中加入卡那霉素至终浓度为40µg/ml,37℃,200 r/min下过夜培养。
转接扩大培养:取2 ml 上述活化后的菌液加入到200 ml经灭菌的LB培养基中,加入200µl卡那霉素,37℃、200 r/min下培养至OD600为0.6~0.8。
诱导表达:待培养基OD600为0.6~0.8 后,加入200 µl 乳糖诱导剂异丙基-β-D-硫代半乳糖苷(IPTG)至其终浓度为0.1 mol/L,25℃条件下,200 r/min继续诱导培养12~14 h。
纯化:收集细胞并将其重悬于上样缓冲液(20 mmol/L Tris,0.25 mol/L NaCl,pH7.4),通过细胞破碎仪进行超声破碎、离心获得上清液即为粗酶液,将粗酶液通过膜滤器过滤并加入Ni-NTA His Bind Resin(重力预装柱)上进行结合,用15 倍柱体积的洗杂缓冲液(20 mmol/L 咪唑,20 mmol/L Tris(pH7.4),0.25 mol/L NaCl)冲洗柱子,然后用15 倍柱体积的洗脱缓冲液(200 mmol/L 咪唑,20 mmol/L Tris (pH7.4),0.25 mol/L NaCl)洗脱目的蛋白。最后采用Macrosep超滤管(截留分子量10×103)进行浓缩和脱盐,得到纯酶。
1.6 酶活性测定
NT2349 属于辅酶NAD+/NADH 依赖型,酶活测定方法是检测NADH 吸光值的变化。酶活力测定定义为:在30℃下,每分钟消耗氧化1 µmol NADH所需的酶量为1 个酶活力单位。在Gly-NaOH (pH 10)缓冲液体系中催化底物L-苯丙氨酸(L-Phe)氧化脱氨生成苯丙酮酸,同时辅酶NAD+转化为NADH。在NH4Cl-NH3·H2O(pH 8.5)缓冲液体系中催化2-氧代-4-苯基丁酸乙酯(EOPB)还原胺化生成L-高苯丙氨酸(L-HPA),同时将NADH 转化为NAD+。检测由NADH 的生成而导致的340 nm 处吸光度的变化,即可测定酶活。
1.7 圆二色谱表征方法
在Gly-NaOH 缓冲液体系中加入有机溶剂(乙醇,正丁醇,环己烷,甲基叔丁基醚)。在含30%有机溶剂的缓冲溶液中加入AaDH,4℃下放置6 h,测定190~250 nm 的远紫外区的圆二色谱,以不添加有机溶剂的缓冲液体系中酶的圆二色谱数据作为对照。
2 结果与讨论
2.1 基因挖掘
针对NCBI、Microscope 等网络数据库中已有的嗜热、嗜盐,以及双嗜极的嗜热嗜碱、嗜盐嗜碱等菌株基因组序列,挖掘获得潜在的耐有机溶剂、耐热的极端氨基酸脱氢酶编码基因。以NAD(P)H 结合的domain(IPR016040)作为探针,采用结构域比对的方法进行挖掘,避免未被明确标注功能的脱氢酶序列的漏检。 获得了来源于嗜热(Geobacillus kaustophilus、Natranaerobius thermophilus、Thermotoga maritima)、嗜 盐(Halobacterium salinarum、Haloferax volcanii)以及嗜热嗜碱(Anaerobranca gottschalkii、Halothermothrix orenii)、嗜 盐 嗜 碱(Euhalothecenatronophila、Ferroplasma acidiphilum)等嗜极微生物的16种脱氢酶的基因序列(表1)。

表1 筛选获得的来源于极端微生物的氨基酸脱氢酶Table 1 Screening of amino acid dehydrogenases from extremophiles
2.2 亲缘分析与同源模建
对上述来源于嗜盐、嗜热等极端微生物的氨基酸脱氢酶进行亲缘关系分析(图1)。利用Blast等进行基因序列与氨基酸序列比对,从基因序列新颖性、进化关系等角度进行分析,并结合菌株所在生境确定后续研究的目标基因。
进化树种垂直方向可以看出不同物种之间进化关系的远近,距离接近说明亲缘关系接近。对嗜盐酶的研究可得知,嗜盐酶中含有较多的天冬氨酸和谷氨酸是蛋白嗜盐的关键因素之一[26]。从序列分析中可以看出在嗜热、嗜盐蛋白中也存在较多的酸性氨基酸(Asp、Glu)。选择符合嗜盐酶特征,且亲缘性较远的8个具有代表性的氨基酸序列酶进行后续分析。
2.3 酶的热力学参数筛选
经过上述筛选获得的8个氨基酸脱氢酶序列采用iTasser 网络服务器进行同源模建,获得蛋白三维结构图,利用iTasser 中ModRefiner 将同源模建获得的结构模型进行能量优化,获得平衡态下的蛋白三维结构模型。利用SWISS-MODEL 对所获得的立体结构进行Ramachandran 构象评价,获得了拉氏构象图。基于Scoop 模拟计算来源于不同生境的极端菌株的氨基酸脱氢酶的热力学参数,包括蛋白熔点(Tm)、失活自由能变化(ΔGr)以及蛋白热容量变化(ΔCp)(表2)。基于这些热力学参数进一步优选具有良好稳定性能的氧化还原酶。
反应体系为催化制备高苯丙氨酸,因此优选苯丙氨酸脱氢酶。由于嗜热酶的热稳定性优于嗜盐酶,因此选择标准不能仅以Tm等热力学参数作为唯一判断标准。嗜盐酶具有较密集的氢键和盐桥等相互作用网络,综合考察其表面电荷分布和带电性能以及酶表面酸碱性氨基酸的分布。综合考虑酶所来源的极端菌株的嗜盐性能,优选来源于嗜热菌的Natranaerobius thermophiles苯丙氨酸脱氢酶NT2349进行酶结构特征研究(图2)。

图1 筛选获得极端微生物的氨基酸脱氢酶的亲缘关系分析图Fig.1 Phylogenetic analysis of genetic relationship among screened AaDHs
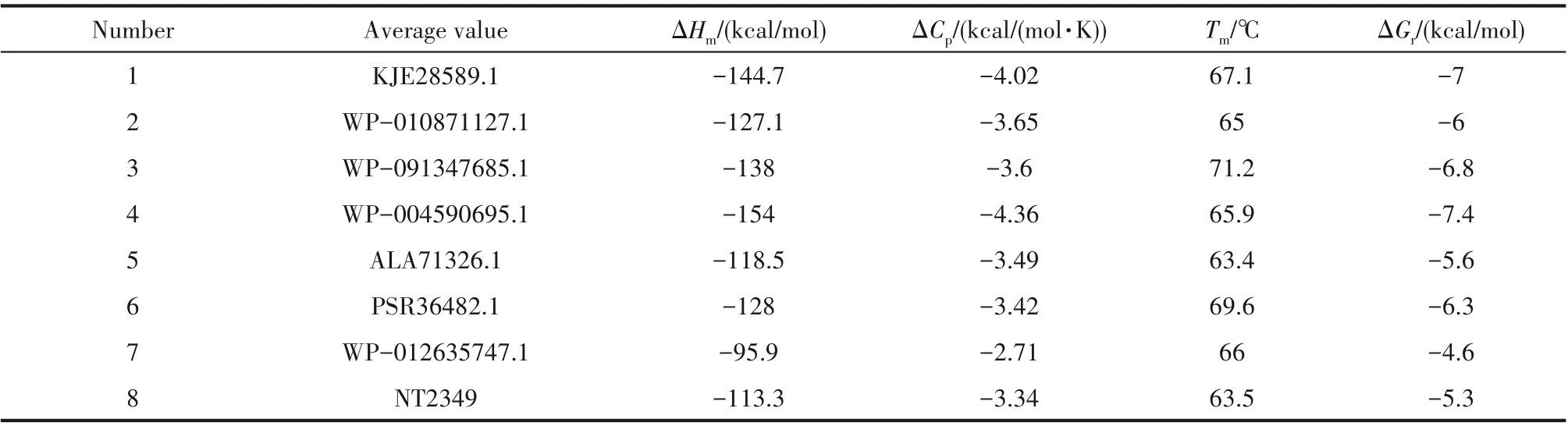
表2 不同极端菌株来源的氨基酸脱氢酶的热力学参数比较Table 2 Comparison of thermodynamic parameters of amino acid dehydrogenases from extremophiles
2.4 氨基酸脱氢酶与底物相互作用
利用Protein Contacts Atlas(https://www.mrc-lmb.cam.ac.uk/rajini/index.html)进行底物结合位点的相互作用分析[27],如图3 所示。图3(a)显示与底物结合的活性位点氨基酸残基,图3(b)显示NT2349 的活性位点上的重要的氨基酸残基与底物苯丙氨酸的相互作用的强度比较。从相互作用热点图(Heatmap图)图3(b)可以看出,底物与活性位点的氨基酸残基N264、N265、Y287 等具有多层次的相互作用。因此,在后续DOCK对接中,选择这些具有较高相互作用的结合位点进行对接。
2.5 底物对接
将酶与天然底物L-苯丙氨酸和非天然底物L-高苯丙氨酸进行对接,活性位点选择上述研究中发现的具有较高相互作用的残基进行与底物的对接,分析底物结合位点与底物相互作用的差异。首先对NT2349与天然底物苯丙氨酸对接,AutoDock 对接完成后,将对接结果按照对接能量由低到高排序。选取能量最低的构象,保存为PDB 结构。在Discover Studio 中进行作图,选定配体后查看配体与蛋白的作用力。
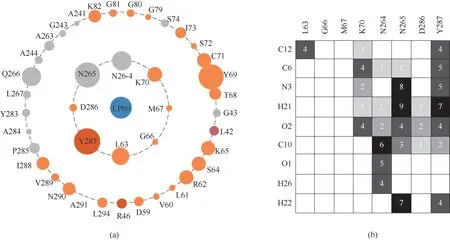
图3 NT2349与底物的二级结构相互作用图(a)NT2349的氨基酸残基与苯丙氨酸相互作用图;(b)NT2349活性位点与苯丙氨酸相互作用的Heatmap图Fig.3 Interactions of secondary structure of NT2349 with L-phenylalanine(a)key residues interact with L-phenylalanine;(b)interaction Heatmap of active site with L-phenylalanine
从三维结构空间查看对接结果,L-苯丙氨酸(phenylalanine)和L-高苯丙氨酸(homophenylalanine)位于同一个对接区域,都为两结构域之间的空腔,说明对接位点选取位置合理。NT2349 与L-高苯丙氨酸的结合能为-3.82 kcal/mol,在合理的结合能范围内。图4 可见,底物L-高苯丙氨酸与酶的结合以氢键等作用力为主。底物的酮基与Asn265 残基形成氢键;底物的氨基与Asn265、Tyr287 形成氢键;底物的酮基与Asn264 形成了碳氢键。底物中的苯环与Met67、Leu63、Lys70、Tyr287 的氨基酸残基形成较弱的π相互作用力。
2.6 苯丙氨酸脱氨氧化反应
在上述的模拟筛选的基础上,择优选择来源于Natranaerobius thermophilus的氨基酸脱氢酶(NT2349)进行基因合成和表达,分离纯化获得酶。通过实验验证酶催化性能和抗逆性能。分别考察了温度、pH 及有机溶剂对NT2349 催化氧化脱氨和还原胺化的活性的影响。在Gly-NaOH 缓冲液中催化L-苯丙氨酸的氧化脱氨生成苯丙酮酸的反应(图5)。
研究NT2349 在不同温度下的氧化脱氨催化活性。如图6 所示,NT2349 在30~80℃下均保持85%以上的高催化活性。NT2349 在60℃下活性达到最大值,之后随温度升高,活性下降。
对于氧化脱氨活性反应体系的pH,如图7 所示,随着pH 递增,酶活力呈上升趋势,在pH 12时酶活力达到最大值,之后随pH 继续升高,酶活力迅速下降。因此,氧化脱氨活性反应体系的最适pH 为12,说明该酶可适应高碱性条件。高碱性条件可改变酶结构中氨基酸残基的带电状态和质子化状态,易造成酶失活。目前鲜有报道可适应如此高pH 的氨基酸脱氢酶。
测定含有30%的有机溶剂的体系对NT2349 氧化脱氨活性的影响。本实验选择8 种有机溶剂,以不添加有机溶剂的缓冲液体系中测得酶活作为100%计算相对酶活,结果如图8。研究发现,在30%的二甲亚砜溶剂中,酶表现出最好的氧化脱氨活性,相较于不含有机溶剂的缓冲液体系,其活性提高了20.0%。在30%的甲基叔丁基醚和30%的环己烷溶剂中,酶的活性分别提高了13.5%和11.5%。而30%的乙醇、正丁醇、乙腈、异丙醇和丙酮有机溶剂均对酶的氧化脱氨活性产生了抑制作用。
考察NT2349 在30%有机溶剂下的催化氧化脱氨反应的酶活稳定性。从图9 看出,随着时间的延长,酶在30%有机溶剂下酶活性下降。NT2349 在30%甲基叔丁基醚和环己烷中保温24 h,分别保留93.2%和91.7%的酶活。在含有30%二甲亚砜和丙酮的体系中保温24 h,仅保持64.6%和22.4%的残余酶活。实验表明,酶在含有30%的甲基叔丁基醚(MTBE)和环己烷有机溶剂下催化氧化脱氨反应时,具有良好的有机溶剂耐受性能。二甲亚砜和丙酮的极性比甲基叔丁基醚强,易造成酶失活。氢键和盐桥作用力是维持蛋白结构的关键作用力。氨基酸残基与水分子形成氢键,从而形成蛋白表面的水合层[28]。极性有机溶剂可影响酶表面的结合水层,进一步破坏酶的空间结构,使蛋白质发生变性而失活,并可能渗透到酶活性中心影响酶与底物的结合[29]。

图4 NT2349与高苯丙氨酸对接结果(a)NT2349与高苯丙氨酸的对接;(b)3D相互作用力;(c)2D相互作用力示意图Fig.4 Docking of NT2349 with L-homophenylalanine
2.7 还原胺化制备高苯丙氨酸
考察酶催化底物2-氧代-4-苯基丁酸乙酯(EOPB)还原胺化高选择性制备L-高苯丙氨酸(图10)。
研究NT2349 在不同温度下的还原胺化催化活性。如图11 所示,NT2349 在30~80℃下都保持着90%以上的高催化活性,且在70℃下酶活达到最大值,而后随温度升高酶活有所下降。
测定不同pH 的缓冲液中还原胺化反应的酶活,如图12所示。NT2349催化EOPB 的还原氨化反应在pH 为8.5 时酶活力达到最大值;pH 大于8.5,酶活性呈下降趋势。因此,该酶在不同催化体系中的最适pH有较大差异。
测定有机溶剂对NT2349 还原胺化活性的影响。在本实验中选择8 种有机溶剂作为反应溶剂(图13)。研究发现,在含有30%的甲基叔丁基醚和二甲亚砜的反应体系中,相较于在NH4Cl-NH3·H2O缓冲液中,酶的还原胺化活性分别保持99.2%和101.3%。而30%的乙醇、正丁醇、乙腈、异丙醇和丙酮均大幅度抑制了酶的活性。
总体来看,在30%的二甲亚砜溶剂中,酶催化还原胺化活性最高。有机溶剂中酶与底物接触在一定程度上受到扩散限制的影响。二甲亚砜是极性溶剂,与水互溶,可提高难溶于水的底物EOPB 的溶解度[30]。
研究酶在30%有机溶剂下的催化还原胺化反应的稳定性。从图14 看出,NT2349 在30%二甲亚砜中保温,酶活性呈先下降后上升再下降最后达到稳定的趋势,24 h 后剩余77.8%酶活。在30%环己烷中酶活性呈逐渐下降趋势,24 h 后仅剩余20.0%酶活。在30%甲基叔丁基醚中24 h 后酶仅剩余25.1%的活性。有机溶剂对酶稳定性的影响与溶剂极性关系较大。一般而言,蛋白的柔性在含非极性有机溶剂的疏水环境中下降,增加了结构稳定性,而强极性有机溶剂易造成酶失活。
2.8 二级结构变化
酶在一定比例的有机溶剂体系中构象会发生改变,α-螺旋、β-折叠和无规Loop 区的构象均可能改变,导致酶活发生变化[30-31]。运用圆二色谱研究酶在有机溶剂中二级结构变化。该酶的α-螺旋结构在192 nm 处有一正谱带;β-折叠在195~200 nm有一正谱带;β-转角在205 nm 处有一正谱带,而在220~230 nm处有一个弱的负谱带。
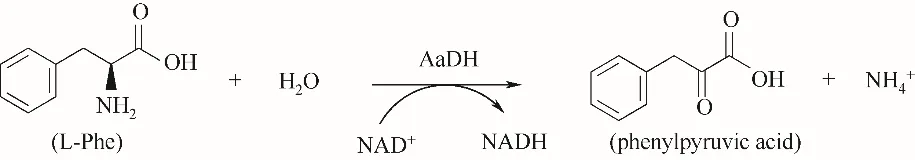
图5 AaDH 催化L-苯丙氨酸(L-Phe)的氧化脱氨反应Fig.5 AaDH catalyzes the oxidative deamination of L-phenyalanine
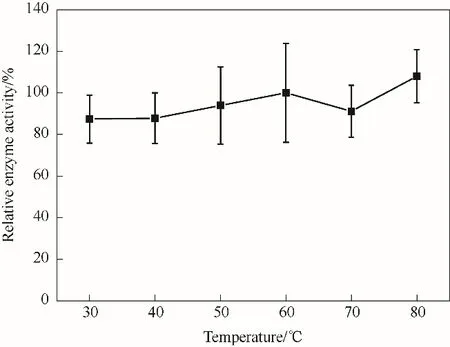
图6 温度对NT2349的氧化脱氨活性影响Fig.6 Effect of temperature on oxidative deamination activity of NT2349
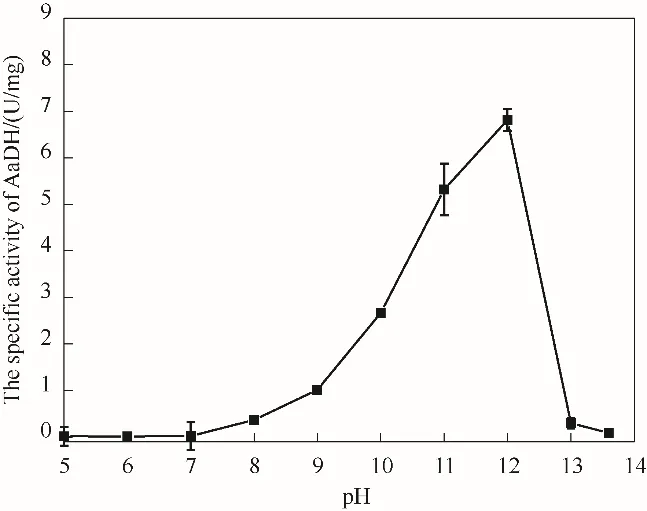
图7 pH对NT2349的氧化脱氨活性影响Fig.7 The effect of pH on oxidative deamination activity of NT2349
从图15 可以看出,NT2349 在水溶液中,在192 nm处有一正谱带,为α-螺旋结构;在197 nm处有一正谱带,为β-折叠结构;在205 nm 处有一正谱带,在222 nm 处有一个弱的负谱带,为β-转角结构。在四种有机溶剂中,192 nm 处的正谱带峰都减弱,α-螺旋结构受到了影响。与水溶液中的NT2349 相比,在乙醇和甲基叔丁基醚中,β-折叠结构在195 nm处的正谱带位置左移了约2 nm;在正丁醇和环己烷中β-折叠结构的谱图位置没有偏移。说明乙醇和甲基叔丁基醚对β-折叠有一定影响。
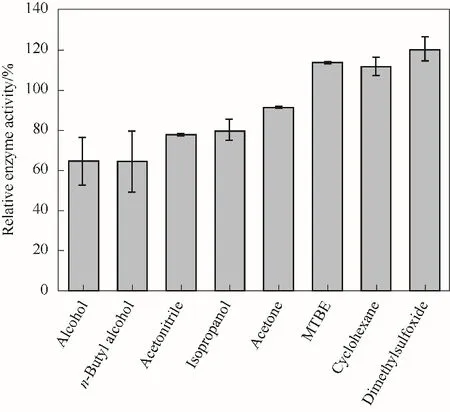
图8 有机溶剂对NT2349的氧化脱氨反应酶活影响Fig.8 Effect of organic solvent on oxidative deamination activity of NT2349

图9 有机溶剂体系中酶催化氧化脱氨的酶活稳定性Fig.9 Enzyme activity stability of oxidative deamination of NT2349 in organic solvents

图10 NT2349 催化EOPB的还原胺化反应制备高苯丙氨酸Fig.10 NT2349 catalyzes the synthesis of L-homophenylalanine by reductive amination of EOPB
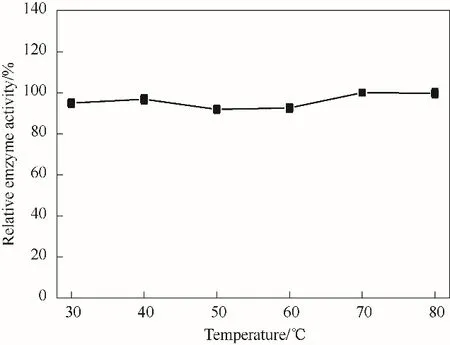
图11 温度对NT2349的还原胺化活性影响Fig.11 The effect of temperature on the reductive amination activity of NT2349

图12 pH对NT2349的还原胺化活性影响Fig.12 The effect of pH on the reductive amination activity of NT2349
3 结 论
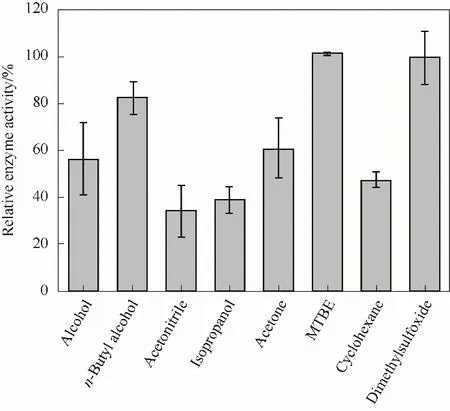
图13 有机溶剂对NT2349的还原胺化反应酶活影响Fig.13 Effect of organic solvent on the activity of reductive ammoniation reactivity
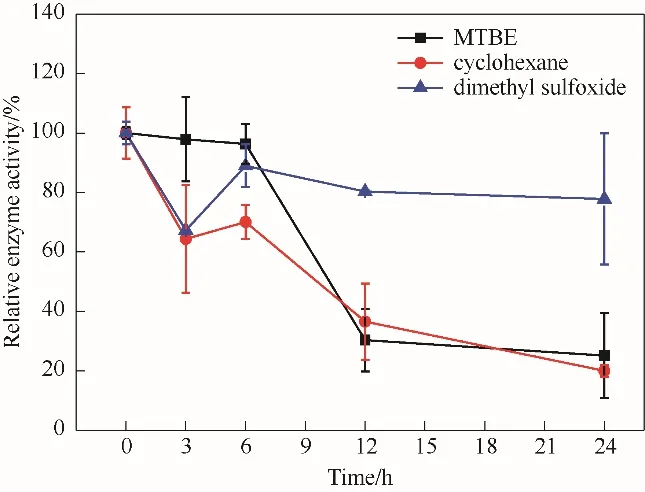
图14 有机溶剂中NT2349催化还原胺化的酶活稳定性Fig.14 Enzyme activity stability of NT2349 reductive amination in organic solvents
(1)挖掘和改造获得抗逆酶以适应工业生物催化所需的极端pH、高温、高盐和非水相等反应体系是使酶从细胞走向生物反应器,实现绿色生物制造的关键。本文研究了极端微生物来源的耐盐氨基酸脱氢酶。
(2)首先,通过基因挖掘,获得了17 个来源于极端微生物的氨基酸脱氢酶基因。进而基于进化分析、热力学参数计算和底物对接等,筛选获得来源于Natranaerobius thermophilus的苯丙氨酸脱氢酶。考察了有机溶剂体系中该酶催化L-苯丙氨酸氧化脱氨和还原胺化制L-高苯丙氨酸的反应特性。
(3)实验结果表明,该酶具有良好的有机溶剂耐受性,且可耐受较高的温度和高pH 的碱性环境。目前的研究仅仅针对一个非天然氨基酸底物进行了氨基酸突变研究,可进一步拓展耐有机溶剂苯丙氨酸脱氢酶的底物谱。
(4)该研究可为新型抗逆氧化还原酶资源的开发应用和生物催化新途径开发与催化性能提升提供方法借鉴。
