传统发酵牦牛酸乳宏基因组DNA三种提取方法比较研究
2015-10-21陈炼红索化夷
谢 婕,赵 欣,骞 宇,陈炼红,李 键,索化夷,*
(1.西南大学食品科学学院,重庆400715;2.西南大学国家食品科学与工程实验教学中心,重庆400715;3.重庆第二师范学院生物与化学工程系,重庆400067;4.西南民族大学青藏高原研究院,四川成都610041)
传统发酵牦牛酸乳宏基因组DNA三种提取方法比较研究
谢婕1,2,赵欣3,骞宇3,陈炼红4,李键4,索化夷1,2,*
(1.西南大学食品科学学院,重庆400715;2.西南大学国家食品科学与工程实验教学中心,重庆400715;3.重庆第二师范学院生物与化学工程系,重庆400067;4.西南民族大学青藏高原研究院,四川成都610041)
微生物区系复杂,自然环境中约95%是无法用常规的培养方法获得的,为了对未能培养或不可培养的微生物进行研究,本实验选用传统发酵牦牛酸乳,分别采用CTAB-SDS-冻融法、液氮研磨法、玻璃珠吸附法来提取宏基因组DNA,然后测得三种方法所提取的DNA的浓度、纯度和片段分布状况,并对总DNA进行16S rRNA基因片段PCR扩增。实验结果表明,本实验所采取的三种DNA提取方法均能提取到适于后续研究质量的DNA,但其中液氮研磨法同时具有提取的总DNA质量较好和步骤简单、成本低的优点,是提取牦牛酸乳宏基因组DNA最合适的方法。
微生物,传统发酵牦牛酸乳,宏基因组,DNA
牦牛在我国主要分布于四川省以及青藏高原地区,是高原地区牧民主要经济来源[1]。传统发酵牦牛酸乳中不仅含有丰富的蛋白质、必需氨基酸、脂肪、乳糖和矿物质[2],还含有大量有益微生物,它们不仅能够提高食品营养价值、延长保存期,还可以改善食品的功能性质[3],对人类健康起着至关重要的作用,正日益引起人们的重视。传统上,一般采用分离、纯化、培养、鉴定的方法来分析微生物的多样性及其群落结构,但由于自然环境中的微生物约90%~99%是无法用常规的培养方法获得的[4],因此通过纯培养的方法并不能真正反映环境中微生物的多样性。为了克服传统研究方法的不足,充分挖掘微生物的巨大潜能,直接以环境中微生物宏基因组DNA[5-7]为基础,应用各种分子生物学的研究方法应运而生,为研究自然环境中微生物的多样性及其功能提供了更全面、快速、可靠的信息,成为一条寻找新基因和基因产物的新途径。
宏基组DNA是进行高通量测序技术、变性梯度凝胶电泳(DGGE)、单链构象多态性(SSCP)[8]等非培养手段研究微生物多样性技术的基础,因此找到一种有效的宏基因组提取方法尤为重要。目前对土壤、水体、污泥等微生物宏基因组的提取方法已有较多报道[9],而牦牛酸乳宏基因组提取方法的研究较少。本实验采用CTAB-SDS-冻融法、玻璃珠吸附法、液氮研磨法三种方法来提取四川省红原县牧民自然发酵耗牛酸奶中微生物宏基因组DNA,通过比较DNA纯度、浓度和片段分布情况来分析哪种提取方法更好,或哪种方法提取的DNA更适合后续的研究。
1 材料与方法
1.1材料与仪器
牦牛酸乳采自四川省红原县,冰盒保藏,运输到实验室,4℃保存;Tris-HCl、RNA酶、蛋白酶K、玻璃珠、V(酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1北京索莱宝生物科技有限公司;EDTA、CTAB、NaCl、Na2HPO4、NaAc成都市科龙化工试剂厂;SDS北京鼎国昌盛生物技术有限责任公司;其他试剂均为分析纯。
H-205OR台式高速冷冻离心机长沙湘仪仪器有限公司;MiNi紫外分光光度计岛津制作所;S1000PCR扩增仪BIO-RAO;SYNGENE凝胶成像系统Gene Company Lim ited。
1.2实验方法
1.2.1宏基因组DNA提取方法
1.2.1.1玻璃珠吸附法玻璃珠吸附法参照Martin-Laurent等[10]报道的方法进行。取1.0m L发酵乳样品于10m L离心管中,加1g酸化玻璃珠(一定量的玻璃珠,用10mmol/L的盐酸浸泡,再放入电热恒温干燥箱中烘干,即得酸化玻璃珠),再加入3m L的DNA提取液(100mmol/L Tris-HCl,100mmol/L EDTA,100mmol/L Na2HPO4,1.5mol/L NaCl,1%CTAB,pH8.0),涡旋振荡3m in,加0.3m L SDS(10%)和10μL蛋白酶K(10mg/m L),50℃恒温摇床100r/m in振荡2h。20℃10000r/m in离心10m in,取上清,用等体积V(酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1抽提一次,水相中加0.1倍体积的NaAc和1倍体积的冰异丙醇,20℃14000r/m in离心5m in,收集沉淀DNA,1.5m L 70%乙醇洗涤两次,风干后用90μL TE、10μL RNA酶溶解。
1.2.1.2液氮研磨法液氮研磨法参照Murray等[11]的方法进行。取一定量发酵乳样品加液氮冻结,研磨,直至约为100目的粉末。取1g粉末于10m L离心管中,加3m L的DNA提取液,涡旋振荡3m in,加0.3m L SDS(10%)和10μL蛋白酶K(10mg/m L),50℃恒温摇床100r/m in振荡2h。20℃10000r/m in离心10m in,取上清,用等体积V(酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1抽提一次,水相中加0.1倍体积的NaAc和1倍体积的冰异丙醇,20℃14000r/m in离心5m in,收集沉淀DNA,1.5m L 70%乙醇洗涤两次,风干后用90μL TE、10μL RNA酶溶解。
1.2.1.3CTAB-SDS-冻融法CTAB-SDS-冻融法参照Bourrain等[12]的方法进行。取1.0m L发酵乳样品加3m L的DNA提取液,在液氮、65℃水浴中反复冻融三次。加0.3m L SDS(10%)和10μL蛋白酶K(10mg/m L)50℃恒温摇床100r/m in振荡2h,20℃10000r/m in离心10m in,取上清。将沉淀重悬于0.9m L DNA、0.1m L 20%SDS中涡旋10s,在液氮、65℃水浴中反复冻融三次,20℃12000r/m in离心10m in,取上清。收集两次上清与等体积的氯仿混合后10000r/m in离心10min。继续收集上清用等体积V(酚)∶V(氯仿)∶V(异戊醇)= 25∶24∶1抽提一次,水相中加0.1倍体积的NaAc和1倍体积的冰异丙醇,20℃14000r/min离心5min,收集沉淀DNA,1.5m L 70%乙醇洗涤两次,风干后用90μL TE、10μL RNA酶溶解。
1.2.2DNA相关指标的测定
1.2.2.1DNA的纯度及浓度将提取DNA用紫外分光光度计测量OD260和OD280,并根据OD260/OD280比值估算纯度。DNA含量(ng/m L)=50ng/m L×(OD260的校正值)×稀释的倍数[13]。
1.2.2.2DNA片段的分布将提取出的总DNA样品进行琼脂糖凝胶电泳,浓度为0.8%,电压80V,电泳1h,溴化乙锭染色后,在凝胶成像系统中观察凝胶。
1.2.316S rRNA基因片段PCR扩增在0.5m L无菌离心管中加入2.5μL 10×PCR Buffer、1μL dNTP、1μL正向引物27F(F)5’-AGAGTTTGATCCTGGCTCAG-3’、1μL反向引物1495R(R)5’-GGCTACCTTGTTACG ACTT-3’[14]、0.2μL Taq酶(2U/μL)、1μL DNA模板(50ng/μL~1μg/μL)、13.3μL ddH2O。将上述混合液稍加混匀、离心,置PCR仪上扩增。扩增循环程序为:95℃,5m in;94℃,1m in;58℃,1m in;72℃,1m in,共30个循环;72℃,7min。反应结束后,所得产物用1.0%琼脂糖凝胶80V电压电泳1h,在凝胶成像系统中检测。
2 结果与分析
2.1DNA的OD值及浓度
应用三种提取方法得到宏基因组DNA,分别进行OD值检测分析,结果测得OD260、OD280数值和DNA浓度见表1。
对于DNA而言,当OD260/OD280的范围在1.7~1.9之间时,表明DNA纯度好。小于1.7说明有蛋白质污染,大于2.0说明有RNA污染或DNA已经降解。本实验中每个样品的4个平行实验结果比较接近,这表明因实验操作造成的检测误差比较小。三种方法提取出的总DNA浓度均在200ng/μL以上,其中玻璃珠吸附法得到的DNA浓度较高,其次是液氮研磨法,最低的是CTAB-SDS-冻融法提取出的总DNA浓度。采用液氮研磨法提取基因组DNA的OD均值(OD260/OD280)都在1.8左右,CTAB-SDS-冻融法所得到DNA的OD均值(OD260/OD280)都在1.85左右,表明以上两种方法提取的总DNA纯度好。只有玻璃珠吸附法所得DNA的OD均值(OD260/OD280)在1.7及以下,原因可能是玻璃珠吸附法对乳酸菌细胞壁破碎不完全,或者提取出的DNA不纯,有蛋白质污染。

表1 DNA的浓度和纯度Table 1 DNA of concentration and purity
2.2总DNA电泳分析
三种方法提取到的总DNA凝胶电泳图如图1所示。

图1 总DNA0.8%琼脂糖凝胶电泳图Fig.1 Total DNA 0.8%agarose gel electrophoresis figure
从图1的琼脂糖凝胶电泳检测结果中可以看出,3种提取方法所得到的DNA片段都在23kb以上,表明这些方法都能够提取到DNA。但是3、4号条带较亮,特别是4号条带明显亮于其他条带,这表明液氮研磨法提取总DNA效果优于其他两种方法。
2.316SrRNA基因PCR结果
总DNA16S rRNA基因PCR,如图2所示。
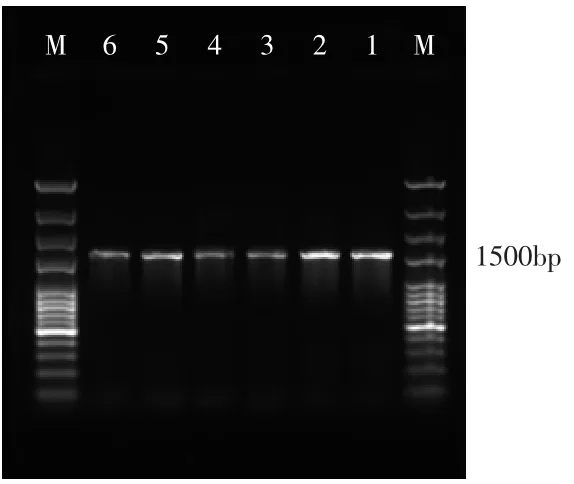
图2 PCR扩增产物的1%琼脂糖凝胶电泳图Fig.2 Amplified PCR product 1%agarose gel electrophoresis figure
从图2可以看出,3个样品均在1500bp左右处出现清晰明亮的条带,这证明了本实验中所提取得到的总DNA提取方法均适宜于后续的研究。
3 结论与讨论
传统培养法研究微生物的多样性存在很多不足,并不能正确反映微生物的多样性,分子生物学方法克服了传统培养法的缺陷,在微生物多样性方面得到了越来越多的应用。在微生物分子生物学研究中,样品总DNA的提取是最基本也是最重要的实验技术之一。但由于微生物的复杂多样以及环境样品的性质多变,没有一种提取DNA的方法能适用于所有的环境样品[15]。目前,国内外许多学者对各种微生物多样性进行了研究,提取了各种微生物的总DNA。熊开容等[16]用多种方法的组合对活性污泥中的微生物DNA进行了提取,结果表明用冻融+玻璃珠+溶菌酶+SDS方法获得的DNA最适合于酶解和PCR扩增的要求。植物总DNA的提取通常采用液氮冷冻研磨破碎细胞壁的方法提取基因组DNA[17-18]。淡瑞芳等[19]通过对植物的CTAB法进行改进,建立了一种简单快速的提取瘤胃内容物总DNA的方法,且获得的瘤胃总DNA符合分子生物学的要求。Leser等[20]用氧化锆-二氧化硅微珠破碎细胞法提取猪肠道微生物总DNA。张春林等[21]研究西藏地区传统发酵乳宏基因组DNA的提取并对微生物群落多样性进行分析,结果发现经过改良的CTAB-SDS-冻融法对DNA的提取效果较佳,可直接用于后续的微生物多样性分析,运用细菌16S rRNA-RFLP和真菌ITS-PCR相结合的方法能够全面快速地比较分析发酵乳中的微生物多样性。这些方法都为提取牦牛酸乳微生物总DNA提供了参考。
本实验运用三种方法提取来自四川省红原县牧民家中的传统牦牛发酵酸乳的宏基因组DNA,并对提取结果进行比较研究,结果发现,尽管玻璃珠吸附法得到的DNA浓度较高,但提取纯度较差。CTABSDS-冻融法提取出的总DNA虽然纯度好,但提取浓度相比其他方法较低,且操作比较复杂;液氮研磨法提取的总DNA纯度好,浓度较高,适合后续的研究,且操作简单、成本低,是提取发酵牦牛酸乳宏基因组DNA最合适的方法。
[1]Sheng Q,Li J,Alam M S,et al.Gross composition and nutrient profiles of Chinese yak(Maiwa)milk[J].International Journal of Food Science&Technology,2008,43(3):568-572.
[2]LiH,Ma Y,LiQ,etal.The chemical composition and nitrogen distribution of Chinese yak(Maiwa)milk[J].International Journalof Molecular Sciences,2011,12(8):4885-4895.
[3]尹胜利,杜鉴,徐晨.乳酸菌的研究现状及其应用[J].食品科技,2012(9):25-29.
[4]李红,周友兵,陈炳耀,等.分子生物学技术在微生物多样性研究中的应用[J].河北大学学报:自然科学版,2004,23(4):450-454.
[5]Gewin V.Genomics:Discovery in the dirt[J].Nature,2006,439(7075):384-386.
[6]Oh H J,Cho K W,Jung I S,et al.Expanding functional spaces of enzymes by utilizing whole genome treasure for library construction[J].Journal of Molecular Catalysis B:Enzymatic,2003,26(3):241-250.
[7]Brady SF,Clardy J.Long-chain N-acylamino acid antibiotics isolated from heterologously expressed environmental DNA[J]. Journal of the American Chemical Society,2000,122(51):12903-12904.
[8]冯美琴.宏基因组学的研究进展[J].安徽农业科学,2008,36(2):415-416.
[9]微生物非培养技术原理与应用[M].北京:科学出版社,2010.
[10]Martin-Laurent F,Philippot L,Hallet S,etal.DNA extraction from soils:old bias for new microbial diversity analysismethods[J].Applied and Environmental Microbiology,2001,67(5):2354-2359.
[11]Murray M G,Thompson W F.Rapid isolation of high molecular weight plant DNA[J].Nucleic Acids Research,1980,8(19):4321-4326.
[12]Bourrain M,Achouak W,Urbain V,et al.DNA extraction from activated sludges[J].Current Microbiology,1999,38(6):315-319.
[13]黄永莲.琼脂糖凝胶电泳实验技术研究[J].湛江师范学院学报,2009,30(6):83-85.
[14]Aquilanti L,Mannazzu I,Papa R,et al.Amplified ribosomal DNA restriction analysis for the characterization of Azotobacteraceae:a contribution to the study of these free-living nitrogen-fixing bacteria[J].Journal of Microbiological Methods,2004,57(2):197-206.
[15]滕齐辉,曹慧,崔中利,等.PCR-RFLP分析[J].生物多样性,2006,14(4):345-351.
[16]熊开容,张智明,张敏.活性污泥总DNA提取方法的研究[J].生物技术,2005,15(4):43-45.
[17]刘小勇,田素忠.提取植物和微生物DNA的SDS-CTAB改进法[J].北京林业大学学报,1997,19(3):100-103.
[18]潘和平,晋玲,高天鹏,等.沙拐枣DNA提取方法改进及PCR扩增检测[J].草业科学,2006,23(10):28-31.
[19]淡瑞芳,龙瑞军,杨予海.瘤胃微生物总DNA快速提取法研究[J].家畜生态学报,2006,27(1):18-21.
[20]Leser TD,Lindecrona R H,Jensen TK,etal.Changes in bacterial community structure in the colon of pigs fed different experimentaldietsandafterinfectionwithBrachyspira hyodysenteriae[J].Applied and EnvironmentalMicrobiology,2000,66(8):3290-3296.
[21]张春林,刘文俊,张彦斌,等.西藏地区传统发酵乳宏基因组DNA的提取及微生物群落多样性分析[J].食品与发酵工业,2010(4):15-20.
Study on com parison of three extraction methods of traditional fermented yak yogurtmacro genom ic DNA
XIE Jie1,2,ZHAO Xin3,QIAN Yu3,CHEN Lian-hong4,LI Jian4,SUO Hua-yi1,2,*
(1.College of Food Science,Southwest University,Chongqing 400715,China;2.Engineering Technology Research Center for Characteristic Food of Chongqing,Chongqing 400715,China;3.Departmentof Biologica and Chemical Engineering,Chongqing University of Education,Chongqing 400067,China;4.Institute of Qinghai-Tibetan Plateau,Southwest University for Nationalities,Chengdu 610041,China)
Microflora was so com p lex that around which 95%could not be obtained by culture in conventional way.This paper selected the traditional fermented yak yogurt,and extracted the macro genom ic DNA by three methods respectively,inc luding CTAB-SDS-frozen-thawed,liquid nitrogen trituration and g lass bead adsorp tive p rocess.Then the DNA concentration,purity and fragment d istribution,and the total DNA of 16S rRNA gene PCR amp lification was tested.The result showed that all the three methods could extract the DNA suitab le for the follow-up study.But liquid nitrogen trituration method not only had the p riority of a better DNA quality,but also was sim p lest and lowest costed way com pared to the other two methods.So it was the most suitab le method to extract yak yogurtmacro genom ic DNA.
m ic roorganism;traditional fermented yak yogurt;macro genom ic;DNA
TS252
A
1002-0306(2015)06-0175-04
10.13386/j.issn1002-0306.2015.06.031
2014-05-12
谢婕(1988-),女,硕士研究生,研究方向:发酵微生物。
索化夷(1978-),男,博士,副教授,研究方向:发酵微生物。
国家公益性行业(农业)科研专项(201203009);中央高校基本业务费项目(XDJK2013B010);重庆市基础与前沿研究计划项目(CSTC2013JCYJA80006)。
