大豆品种北农103矮秆基因定位和候选基因克隆
2021-09-18唐铮灏
唐铮灏,刘 洁,陈 巧,谢 皓
(北京农学院植物科学技术学院/农业应用新技术北京市重点实验室,北京102206)
株高是大豆育种中一个重要的农艺性状,矮秆表型对于选育具有理想株型的高产品种起到重要作用[1],矮秆能够提高抗倒伏和光能利用率,实现大豆产量的提高[2-3]。近年来,随着分子生物学技术的发展,矮秆基因不断被发掘出来,并应用于培育矮秆品种[4]。例如,HWANG等[5]利用辐照发现一个大豆矮秆突变体,培育出Hobbit 87、Charleston、Apex和Strong等半矮秆品种,显著提高大豆的产量。据分析[6-7],大豆株高的遗传力较高,约85.6%,受主效基因和多对微效基因共同控制,同时环境对大豆表型影响也较大。何平等[8]对矬大豆、早丰5、九交7601等品种的研究表明,株高由隐性主效单基因及若干修饰基因控制;目前在SoyBean数据库中收集约200多个与株高有关的数量性状位点(quantitative trait loci,QTL)[9-10]。除发现调控株高的主效基因外,DONG等[11]研究认为,大豆品种Jimidou-1和Gongjiao 9112杂交后代的半矮性状受一个隐性基因控制;LI等[12]用EMS诱变大豆品种中品661,获得一个矮秆突变体dw,发现dw的矮秆性状由1对隐性核基因控制。
本课题组在大豆新品种选育过程中,从高秆品种海系13与矮秆品种北农103杂交的分离后代中,发现高秆和矮秆性状极端分离的情况。经对该杂交组合F2群体的遗传分析和分子标记初步定位,北农103携带1对调控矮秆性状的隐性核基因,位于大豆19号染色体(L连锁群)上[13]。本研究在此基础上,对该基因进行精细定位和克隆,为进一步研究和利用北农103及其矮秆基因奠定基础。
1 材料与方法
1.1 供试材料
大豆矮秆品种北农103、高秆品种海系13和以北农103为父本、海系13为母本杂交构建的688个F5代重组自交系(342个矮秆系和346个高秆系)。其中,北农103为北京农学院选育、北京市农作物品种审定委员会审定的夏播品种,为有限生长型品种,株高55 cm;海系13为南京品种,半有限生长型,北京夏播株高90 cm。试验材料由本课题组保存和提供,种植在北京农学院大豆试验地。
1.2 试验方法
1.2.1 株高鉴定与田间取样 大豆植株开花结束后,株高不再生长时,鉴定各个自交系的株高,矮秆系50 cm左右,高秆系90 cm左右。分别采取矮秆系和高秆系幼嫩叶片于-80 ℃冰箱保存备用。
1.2.2 BSA-Seq混池的构建、重测序及矮秆基因初步定位 选取矮秆和高秆重组自交系叶片各30份,提取DNA,将DNA等量混合构建矮秆和高秆混池,并以矮秆父本北农103和高秆母本海系13为对照,利用高通量重测序技术,分析株高在大豆全基因组变异情况及相关区域。测序及数据基本分析由上海美吉生物医药科技有限公司完成。
1.2.3 分子标记的开发 在高通量重测序的基础上,参照大豆数据库Soybase(https://soybase.org/)提供的SSR引物序列合成引物,同时利用测序数据设计插入缺失位点标记引物。引物由北京擎科新业生物科技有限公司合成。
1.2.4 DNA提取和PCR体系 CTAB法提取叶片DNA。参照袁鹰等[13]的方法,PCR反应体系10 μL,其中,1 μL引物(2 μmol/L),5 μL 2×T5 Super PCR Mix,2 μL样品DNA(15~25 ng/μL),2 μL ddH2O;PCR反应程序,95 ℃预变性5 min;95 ℃变性45 s;55~60 ℃退火45 s;72 ℃延伸30 s;35次循环, 72 ℃延伸10 min。利用8%非变性聚丙烯酰胺凝胶电泳检测扩增产物。
1.2.5 遗传连锁图谱的构建及目标基因的定位 以(海系13×北农103) F5重组自交系为定位材料,根据688个重组自交系产生的分子标记和对应自交系的田间株高表型,利用Mapmaker 3.0和MapDraw V2.1软件进行遗传连锁距离分析;根据标记与株高性状的紧密连锁程度,参照大豆遗传连锁图谱(https://soybase.org/)定位矮秆基因。
1.2.6 基因克隆 根据矮秆基因定位结果,在NCBI(https://www.ncbi.nlm.nih.gov/)上分析定位区域内的已知基因,推测可能与矮秆性状相关的候选基因,并根据其序列设计引物。以海系13、北农103和(海系13×北农103) F5重组自交系中矮秆、高秆各3株幼叶提取的DNA为模板,进行PCR扩增,扩增结果连接至pEASY-BluntZero载体上,培养后挑取单克隆及重组体菌液PCR鉴定。取300 μL阳性克隆菌液上游引物、下游引物各20 μL,在北京擎科新业生物技术有限公司进行测序。pEASY-BluntZeroCloning Kit试剂盒购自北京全式金生物公司。
1.2.7 株高发育期候选基因表达量分析 采用实时荧光定量分析法,分别在苗期、花芽分化期、开花期、成熟前期摘取北农103和海系13顶部相同位置的叶片,利用试剂盒TransZol Up,提取RNA;以Goldenstar RT6 cDNA Synthesis Kit试剂盒反转录RNA。根据候选基因序列设计引物进行实时荧光定量PCR,检测候选基因表达量,参照刘洁等[14]的方法,PCR体系为25 μL:上游引物和下游引物各1 μL(10 μmol/L),12.5 μL SYBR GREEN PCR Premix HS Taq,2 μL模板cDNA,8.5 μL H2O(DNase free)。PCR反应程序,94 ℃预变性30 s;94 ℃变性30 s;60 ℃退火30 s;72 ℃延伸30 s,40次循环;熔解曲线,95 ℃ 10 s,65 ℃ 60 s,97 ℃ 1 s。试剂购自北京鼎国昌盛生物技术公司。
1.3 数据分析
实时荧光定量分析试验均重复3次,经仪器自动分析获得基因的Ct值,用Microsoft Excel软件作图分析,SPSS软件分析显著性,用3次重复的平均值比较高秆基因和矮秆基因的相对表达量,相对表达量的计算采用2-△△Ct法[15]。
2 结果与分析
2.1 株高性状高通量测序分析
株高性状高通量测序分析见表1。全基因组高通量重测序共获得178.48G数据量,原始数据总序列数为539.24M,Q30达到93.84%,亲本样品平均测序深度为35X,矮秆系和高秆系混池样品平均测序深度为36X。测样与参考基因平均比对率为99.72%,平均覆盖深度为36X,基因组覆盖度为93%。当置信度为0.99时,控制株高性状的基因可能位于大豆10号、19号、2号、4号和8号染色体的5个区域中,共有1 461个候选基因,301个单核苷酸多态性位点和57个插入缺失位点。
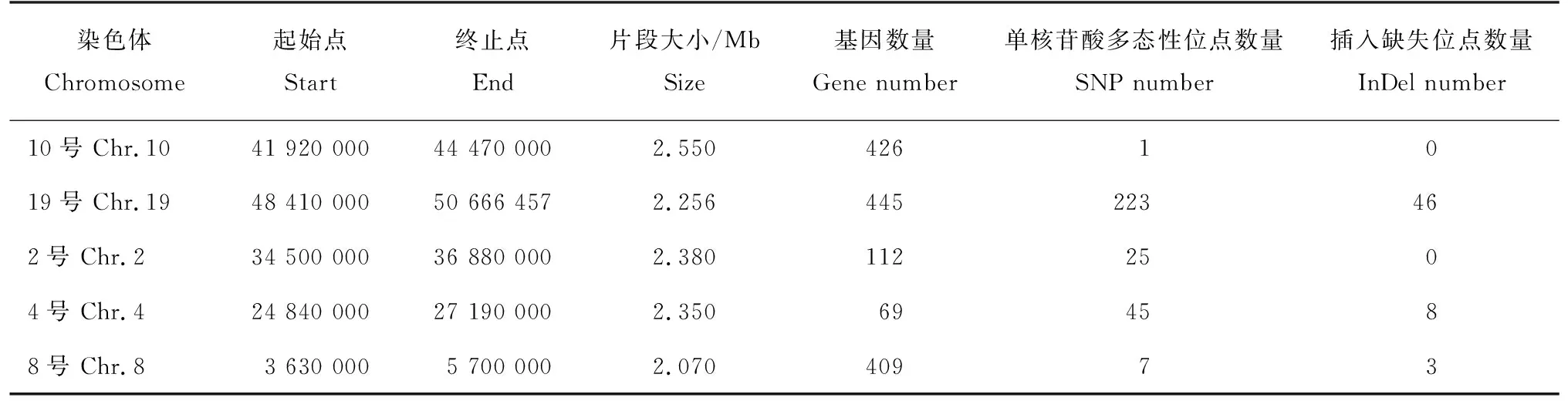
表1 矮秆性状基因和高秆性状基因全基因组高通量重测序结果Tab.1 Whole genome high throughput resequencing of dwarf/tall trait genes
2.2 矮秆基因精细定位
根据初步定位结果,选取10号、19号、2号、4号和8号染色体上SSR引物,从(海系13×北农103) F5重组自交系群体中选取矮秆系和高秆系各4份,采用集团分离分析法,建立矮秆和高秆2个近等基因池,通过PCR扩增,检测引物产生的多态性。在19号染色体(L)上的56对SSR引物中有12对引物在混池扩增出多态性,而在10号、2号、4号和8号染色体上的SSR引物的扩增结果未检测出多态性。将产生多态性的12对SSR引物,分别对群体中346个高秆系和342个矮秆系的DNA进行PCR检测,12对引物中有10对引物产生的多态性标记与矮秆基因紧密连锁。为增加标记的数量,根据全基因组重测序结果中19号染色体(L)48 410 000到50 666 457区域内DNA的序列,设计50对插入/缺失标记,通过PCR多态性检测,发现缺失标记XH-8和XH-23与矮秆基因紧密连锁。最终,共发现12对引物(表2)扩增产生的标记与矮秆基因紧密连锁。其中,引物Satt373的部分PCR结果如图1。

表2 12对SSR引物的序列信息及位置Tab.2 Information of 12 pairs of SSR primers used in this study
利用Mapmaker3.0和MapDraw V2.1软件对12对引物与688个自交系的DNA扩增的标记基因型数据和688个自交系的表型数据进行遗传连锁分析,绘制出遗传连锁图谱(图2)。根据袁鹰等[13]的建议,将矮秆基因暂命名为Gmd1。遗传连锁分析表明标记Satt373、XH-8、Sat_245、Satt513、XH-23、Satt229、Satt527、Satt166、Satt481、Satt418 、Sat_195和Satt278与Gmd1的遗传距离分别为0.8、1.0、1.8、2.3、4.4、8.3、11.4、12.3、15.6、22.2、25.4和28.4 cM,从遗传连锁图谱可以看出Gmd1位于标记Satt373与XH-8之间,与12个标记的连锁位置为Satt278-Sat_195-Satt418-Satt481-Satt166-Satt527-Satt229-XH-23-Satt513-Satt373-Gmd1-XH- 8-Sat_245。结合大豆品种Williams 82的物理图谱(https://www.soybase.org),分子标记Satt373至XH-8区间对应于19号染色体上195 kb的物理区间。
2.3 候选基因的序列分析
检索NCBI(https://www.ncbi.nlm.nih.gov/)的大豆基因组,发现在引物Satt 373和XH-8区间包含25个基因,根据功能对这些基因进行初步筛选,2个候选基因GRF1和IAA16与矮秆性状相关,参照GRF1和IAA16的序列设计引物(表3),以海系13、北农103及其后代高秆系和矮秆系为材料,对候选基因进行克隆和序列分析。候选基因GRF1全长1 100 bp,在海系13、北农103、高秆系和矮秆系中的GRF1序列完全相同;候选基因IAA16全长2 760 bp,在海系13和高秆系中IAA16序列相同,而北农103和矮秆系的IAA16序列相同,海系13与北农103相比,IAA16在1 564 bp处海系13碱基为A,而北农103为C;在1 571 bp处海系13碱基为G,而北农103为C(图3)。分析IAA16序列,编码序列全长为4 35 bp,共编码144个氨基酸,海系13和北农103中因碱基的不同出现两处氨基酸变化(图4)。北农103候选基因IAA16在第132个氨基酸,由酪氨酸突变成丝氨酸;在第134个氨基酸,由丝氨酸突变成苏氨酸。经蛋白结构分析推测,北农103的候选基因IAA16中氨基酸变异在可引起蛋白结构变化的结构域范围之内。
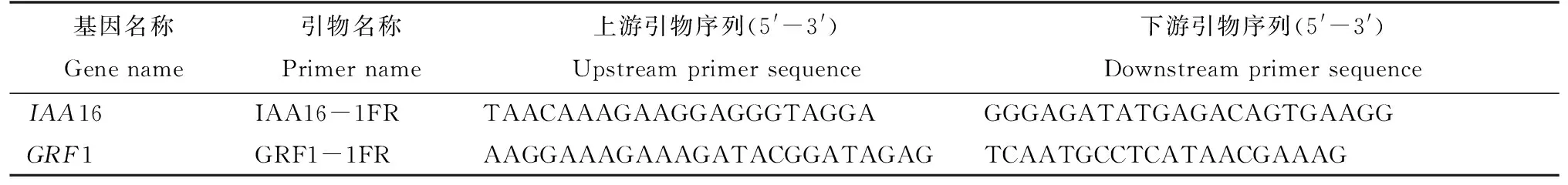
表3 候选基因克隆引物序列Tab.3 Sequence information of candidate gene clone primers
2.4 株高和生育期候选基因表达量分析
为验证候选基因IAA16对株高的影响,在矮秆品种北农103和高秆品种海系13的苗期、花芽分化期、开花期和成熟前期,分别取幼叶,提取RNA反转录为cDNA,利用实时荧光定量分析法对候选基因IAA16在不同生育期的表达量进行分析(图5)。苗期和花芽分化期IAA16表达量相对较高,海系13中IAA16表达量略高于北农103,但差异不明显(P>0.05);开花期海系13中IAA16表达量较高,而北农103较低,相差6倍,差异极显著(P<0.01);成熟前期IAA16表达量都较低,虽然海系13的IAA16表达量也比北农103高,但差异不明显(P>0.05)。
3 讨 论
目前,对大豆矮秆基因的定位区间多位于6号、7号、13号和18号染色体上,且多为QTL定位[14],而QTL在实际应用上有一定的难度。本研究以矮秆品种北农103、高秆品种海系13及其杂交F2分离群体衍生的F5重组自交系群体为研究材料,通过全基因组高通量重测序技术初步定位,F5重组自交系群体的精细定位,筛选与克隆候选基因,并对候选基因进行生育期表达量分析,以确定候选基因的可靠性。大豆品种北农103携带有1对控制矮秆性状的隐性基因,该基因暂定名为Gmd1,位于大豆19号染色体(L连锁群)上,与12对引物产生的分子标记紧密连锁,在标记Satt373与XH-8之间,遗传距离分别为0.8 cM和1.0 cM;区间内北农103候选基因IAA16发生两处单碱基变异,分别发生在1 564 bp和1 571 bp处,导致氨基酸变化,从而蛋白结构受影响;海系13在开花期候选基因IAA16的表达量高于北农103,相差6倍,差异极显著,作为高秆和半有限生长型品种的海系13,开花期仍是株高生长的快速期,而北农103为矮秆和有限生长型品种,开花期株高的生长基本停止,表明IAA16开花期在海系13中的高表达量与其对株高的调控有关。结合IAA16在海系13和北农103中的DNA序列和蛋白结构分析,推测IAA16的等位突变基因可能是调控北农103矮秆性状的基因,即Gmd1。
据报道,在大豆上已发现的矮秆基因dw[12],位于8号染色体;半矮基因sdf-1[11]位于19号染色体45 201 612 bp到45 250 751 bp之间。本研究发现Gmd1定位在19号染色体49 191 336 bp到49 386 333 bp之间,与前人的研究不在同一定位区间,推测Gmd1可能是一个新的矮秆基因。目前与株高相关的基因中,大多与植物激素有关,并通过调控植物激素的合成或其信号传导途径,以此控制植物株高[16-17]。尽管本研究获得的候选基因IAA16的功能有待进一步确定,但是为研究北农103及其携带的矮秆基因提供重要的信息。此外,刘洁等[14]的研究表明北农103还携带有1对调控早花的隐性基因e1,因此,北农103作为改良大豆株高和成熟期的品种资源具有一定的利用价值。
