基于生物信息学分析黑素瘤中hTERT共表达基因及基因调控网络
2021-02-03白志艺李青
白志艺 李青










[摘要] 目的 通過生物信息学的方法对hTERT基因网络及其相互作用的基因进行解密,研究hTERT共表达基因潜在的预后价值。方法 使用LinkedOmics数据库鉴定黑素瘤中hTERT共表达基因,使用GSEA对激酶-靶标富集、miRNA-靶标富集和转录因子-靶标富集进行分析。DAVID在线数据库对180个共表达基因进行GO和KEGG分析。STRING数据库、Cytoscape软件用于构建180个共表达基因的PPI网络图,并识别PPI网络中最重要的模块化基因。c-BioPortal分析模块化核心基因的生存分析结果,Oncomine数据库分析核心基因在黑素瘤组织和正常组织中的差异表达。 结果 LinkedOmics数据库挖掘结果提示hTERT与几种肿瘤相关的激酶(如ATR)和转录因子(E2F家族)特异相关,这些激酶和转录因子调节基因组稳定性、有丝分裂和细胞周期。hTERT表达与癌症通路相关的功能网络有关。筛选出共表达基因180个,网络节度分析得到了8个核心基因,依次为TERT、ANLN、CDC25B、CDK4、CEP55、E2F7、KIF23、OIP5。蛋白-蛋白网络互作图(PPI)显示,hTERT与CDK4在蛋白互作关系上是直接关联。CDK4基因突变与扩增是黑素瘤患者DFS、OS的独立预后因素(P=0.0163、6.843E-3)。结论 数据挖掘结果提示hTERT在黑素瘤中潜在的调控网络信息,与hTERT共表达的CDK4基因可能是黑素瘤预后标志物,为进一步研究hTERT在黑素瘤发生、发展中的作用奠定了基础。
[关键词] 生物信息学;黑素瘤;hTERT;共表达基因
[中图分类号] R735.7 [文献标识码] A [文章编号] 1673-9701(2021)35-0037-07
Analysis of hTERT co-expressed genes and gene regulatory network in melanoma based on bioinformatics
BAI Zhiyi1 LI Qing2
1.Department of Dermatology, First Affiliated Hospital of Xiamen University, Xiamen 361000, China; 2.Department of Dermatology, Longgang Maternal and Child Health Hospital, Shenzhen 518038, China
[Abstract] Objective To decrypt the hTERT gene network and its interacting genes through bioinformatics methods and study the potential prognostic value of hTERT co-expressed genes. Methods The LinkedOmics database was used to identify hTERT co-expressed genes in melanoma. GSEA was used to analyze kinase-target enrichment, miRNA-target enrichment, and transcription factor-target enrichment. The DAVID online database was used to conduct GO and KEGG analysis on 180 co-expressed genes. STRING database and Cytoscape software were used to construct a PPI network diagram of 180 co-expressed genes and identify the most important modular genes in the PPI network. The survival analysis results of modular core genes were analyzed using c-BioPortal. The Oncomine database was used to analyze the differential expression of core genes in melanoma tissues and normal tissues. Results The result of LinkedOmics database mining suggested that hTERT was specifically related to several tumor-related kinases (such as ATR) and transcription factors (E2F family). These kinases and transcription factors regulated genome stability, mitosis, and cell cycle. The expression of hTERT was related to the functional network related to cancer pathways. A total of 180 co-expressed genes were screened, and 8 core genes were obtained by network node degree analysis, which were TERT, ANLN, CDC25B, CDK4, CEP55, E2F7, KIF23, and OIP5. The protein-protein network interaction map (PPI) showed that hTERT and CDK4 were directly related to the protein interaction relationship. CDK4 gene mutation and amplification were independent prognostic factors of DFS and OS in melanoma patients (P=0.0163, 6.843E-3). Conclusion The data mining results suggest the potential regulatory network information of hTERT in melanoma. The CDK4 gene co-expressed with hTERT may be a prognostic marker of melanoma, which lay the foundation for further research on the role of hTERT in the occurrence and development of melanoma.
[Key words] Bioinformatics; Melanoma; hTERT; Co-expressed genes
85%~90%人类癌症和70%以上永生人类细胞系的端粒酶活性高度升高[1]。目前有多个关于hTERT在黑素瘤中的预后研究[2-6]。研究证实,hTERT对黑素瘤发生发展过程起到重要作用,是黑素瘤的预后因素。然而,hTERT的生物学功能和意义尚未被充分阐明。不同的基因和通路参与了对hTERT的调控[7]。然而,hTERT与这些基因的相互作用机制并没有被充分挖掘,为更好地理解端粒酶在黑素瘤中的功能,有必要对hTERT基因网络及其相互作用的基因进行解密。研究hTERT共表达基因潜在的预后价值。因此,本研究使用数据挖掘技术,通过多个生物信息学数据库来探讨黑素瘤中hTERT共表达基因和功能网络。进一步了解端粒酶的其他生物学功能。筛选hTERT共表达基因群中潜在预后标志物。
1 材料与方法
1.1 LinkedOmics数据库分析hTERT mRNA相关的差异表达基因
LinkedOmics数据库[8](http://www.linkes.org/login.php)可用于分析32种收录入TCGA数据库中癌症相关的多维数据集。使用LinkedOmics的LinkFinder模块研究TCGA数据库黑素瘤中hTERT共表达基因。使用GSEA对激酶-靶标富集、miRNA-靶标富集和转录因子-靶标富集进行了分析。检验标准P值<0.05,模拟500次。
1.2 预测分析MiR-497-5p靶基因
Hodi等[15]课题组前期工作中,发现hTERT是 miR-497-5p的直接靶向基因。miRNA与mRNA具有一对多及多对一的对应关系,同样靶向miR-497-5p的基因群可能与hTERT富集于相同生物功能及信号通路。故本研究中倒转过来,通过Targetscan,ENCORI数据库来预测miR-497-5p的靶基因。寻找这组靶基因与hTERT共表达基因群之间的交集基因。
1.3 GO与KEGG通路分析
使用DAVID在线数据库对180个取交集的基因组进行GO和KEGG分析,P<0.05为差异有统计学意义。
1.4 PPI蛋白网络构建与模块分析
利用STRING数据库建立180个交集基因之间的功能蛋白-蛋白相互作用网络(PPI)。使用Cytoscape将这180个交集基因形成可视化网络图。通过MCODE识别PPI网络中最重要的模块。MCODE设置标准为:评分>5分,分度cut-off=2分,节点cut-off=0.2分,最大深度=100分,k-score=2分。
1.5 c-Bioportal數据库进行生存分析
使用c-BioPortal分析模块化核心基因的生存分析结果。
1.6 Oncomine数据库分析差异表达
使用Oncomine数据库比较生存分析中有预后价值的核心基因在黑素瘤组织和正常组织中的差异表达,作为生存分析结果的第二佐证。以P<0.05为差异有统计学意义。
2 结果
2.1 LinkedOmics数据库分析黑素瘤中与hTERT mRNA相关的差异表达基因
使用LinkedOmics功能模块分析TCGA数据库中黑素瘤患者的hTERT共表达基因数据。火山图(封三图2)显示,1402个基因(暗红点)与hTERT mRNA呈显著正相关,1518个基因(暗绿点)与hTERT mRNA呈显著负相关(P<0.05)。50个重要的基因与hTERT mRNA呈正相关,50个与hTERT mRNA呈负相关。见封三图3。这个结果表明hTERT基因对转录组有广泛的影响。与hTERT mRNA相关性最高的前3个基因分别是TROAP、IQGAP3、SKA。与TROAP表达呈正相关(皮尔森相关系数=0.61,P=2.53E-07),IQGAP3(皮尔森相关系数=0.48,P=3.17E-07),和SKA3(皮尔森相关系数=0.47,P=5.75E-07)。
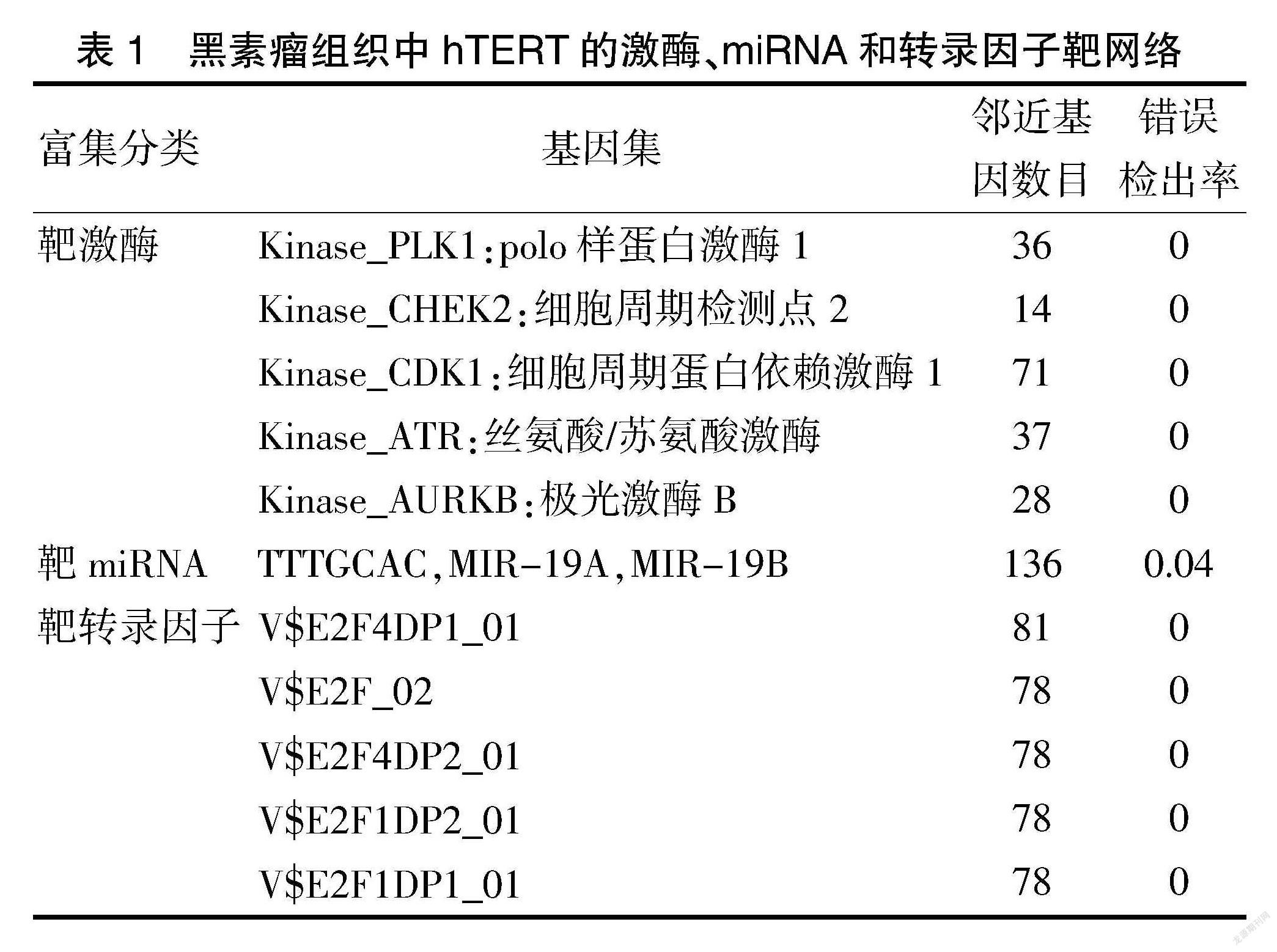
为进一步探讨hTERT在黑素瘤中的靶点,分析GSEA产生的与hTERT正相关差异表达基因集的激酶、miRNA和转录因子靶点网络。miRNA靶点只有一个MIR-19A,MIR-19B。激酶靶点网络主要相关:polo样激酶(PLK1),细胞周期检测点激酶(CHEK2),细胞周期蛋白依赖性激酶(CDK1),丝氨酸/苏氨酸激酶(ATR),极光激酶B(AURKB)。转录因子-靶标网络主要与E2F转录因子(E2F)家族相关,包括V[S] E2F4 DP1_01,V[S] E2F_02,V[S] E2F4DP2_01,V[S] E2F1DP2_01,V[S] E2F1DP1_01。见表1。
2.2 miR-497-5p靶基因
Targetscan,ENCORI数据库预测miR-497-5p靶基因,取交集共鉴定出1515个靶基因。将miR-497-5p的靶基因与hTERT共表达基因群(2920个)取交集得到180个基因,见封三图4。
2.3 交集基因的GO富集与KEGG通路富集分析
利用DAVID数据库分析了180个交集基因的GO功能和KEGG通路富集分析。GO分析结果显示,生物过程(BP)变化显著富集于细胞周期、调控细胞增殖、细胞周期阶段、细胞周期过程。细胞成分(CC)的变化主要集中在非细胞膜固定性细胞器、胞内细胞膜固定性细胞器、细胞骨架。分子功能(MF)的变化主要体现在核苷酸绑定,嘌呤核苷酸结合。KEGG 通路富集于癌症中的通路、细胞周期。见表2。
2.4 PPI蛋白网络构建与模块分析
使用STRING数据库分析180个共表达基因的相互作用网络,预测hTERT与这180个基因关系及hTERT潜在功能。通过 Cytoscape软件将其可视化,利用 MCODE插件计算 PPI 网络中每个基因的节点度(degree)(图1),筛选 PPI 网络中核心蛋白质的编码基因(核心基因),共得到8个模块化核心基因(图2)。
2.5 核心基因在黑素瘤中的生存分析
利用c-Bioportal数据库对核心基因进行总体生存分析(OS)及无病生存分析(DFS)。结果发现,有CDK4、E2F7改变的黑素瘤患者总体生存期(OS)较差,差異有统计学意义(P=6.843E-3、0.0128)。见图3。黑素瘤患者伴有OIP5、CDK4 的改变显示更差的无病生存(DFS)(P=0.038、0.0163)。见图4。综合分析,与hTERT表达密切相关的基因CDK4、E2F7、OIP5在黑素瘤的发生或进展中或许起到了重要作用,尤其是CDK4。因此,在下一步使用Oncomine数据库挖掘CDK4在黑素瘤中的表达情况,作为其生存分析结果的佐证。
2.6 CDK4在黑素瘤组织中的差异表达
使用Oncomine数据库来比较CDK4在黑素瘤组织和正常组织中的mRNA差异表达水平。发现CDK4在3个数据集中均表现为高表达,且表达差异有统计学意义(P=2.80E-5、0.009、0.003),其中1个数据表达为差异无统计学意义(P=0.568)。见图5。合并4组数据进行meta分析。见封三图5。结果显示CDK4在黑素瘤中高表达,表达差异有统计学意义(P=0.004)。
3讨论
黑素瘤是起源于表皮黑素细胞的高度恶性肿瘤,多发生于皮肤、黏膜[9]。发病率与死亡率逐年升高。发生发展机制尚不清楚。端粒存在于真核生物,是一种帽状结构,位于线性染色体末端。端粒保护染色体末端不受降解、融合和重组的影响[10-11]。端粒是5-TTAGGG-3 DNA重复序列的长链,是由端粒酶(TERT)合成的,人类核心端粒酶由至少两个基本亚基组成,即蛋白质亚基、人类端粒酶逆转录酶(hTERT)和RNA亚基、人类端粒酶RNA(hTR)[12]。hTERT位于5号染色体,由16个外显子15个内含子组成,位于5号染色体的短臂上(5p.15:33),长度超过40 kb,与5p端之间有一个兆比特距离。hTERT启动子区被认为是端粒酶表达非常重要的调控元件[10,13-14]。
黑素瘤是一种侵袭性肿瘤,具有不可预测的生物学行为。虽然各种治疗方法已经用于治疗黑素瘤,但存活率在过去几十年里没有显著提高[15-16]。端粒酶逆转录酶(hTERT)是端粒酶全酶复合体的催化亚基,在细胞衰老和肿瘤发生中起重要作用。端粒酶调控的分子机制影响转录、转录后、翻译后和亚细胞定位[14]。
hTERT诱导/端粒酶激活通过稳定端粒长度使癌细胞具有无限的增殖潜能,而最近的观察显示,其多种致癌活性独立于其对端粒功能增强作用,包括其对线粒体和泛素-蛋白酶体功能的影响,DNA损伤修复,基因转录、microRNA表达等。hTERT被发现直接与β-catenin及增强其转录调控效应,从而刺激上皮间充质转化(EMT)[17-18]。近期有研究评估了mRNA转录的交替剪接作用。hTERT可以从多个不同的剪接转录本翻译,只有最长的变体具有逆转录酶活性[19]。其他没有催化功能的转录本在乳腺癌细胞系的过表达已被证明可以减少细胞凋亡,从而获得生存优势[20]。这表明hTERT具有超越端粒延伸的新功能。因此本研究的生信分析可能为进一步探讨hTERT激活端粒酶多样的机制提供线索。
本次生信分析中,GO功能富集分析及KEGG通路富集分析,主要涉及到细胞周期信号转导,调控细胞周期,调控细胞的增殖、分裂。这与hTERT的功能是一致的。值得注意的是,本研究在分析核心基因的网络互作时,发现hTERT与CDK4是直接相关的,而这两个基因富集在癌症通路中。
利用GSEA对靶基因进行富集分析,有助于揭示靶激酶、miRNA和转录因子的重要网络。本研究结果表明,hTERT的功能网络主要参与细胞周期。基因组不稳定性和诱变是癌细胞的基本特征,激酶及其相关的信号通路有助于稳定和修复基因组DNA[21-22]。本研究发现黑素瘤中的hTERT与包括ATR、AURKB和CDK1在内的激酶网络相关。这些激酶调节基因组稳定性、有丝分裂和细胞周期[23-24]。事实上,ATR是基因组稳定性的核心激酶调节因子之一;它启动细胞对基因组不稳定和修复的反应,直接磷酸化1000多种重要的底物,包括肿瘤抑制基因p53蛋白和细胞周期调节蛋白[25]。ATR激酶抑制剂可杀伤肿瘤细胞,并与放化疗的杀伤细胞协同作用[26]。在黑素瘤中,hTERT可能通过ATR激酶调节DNA复制、修复和细胞周期进展。
E2F2和E2F4是E2F家族中主要的转录因子,在增殖正常黑素细胞、黑素瘤细胞和新分离的黑素瘤中起作用。E2F1是细胞周期调控网络的关键环节之一,可导致细胞凋亡、生长停滞或两者同时发生[27]。E2F1异常表达参与了黑素瘤的发生发展。本研究分析表明E2F1是hTERT的一个重要靶点,hTERT通过该因子调节黑素瘤的细胞周期和增殖能力。
8个核心基因的网络互作图中,hTERT与CDK4是直接相关的。CDKs是一种蛋白激酶,参与驱动细胞周期进展、控制转录过程和调节细胞增殖。在黑素瘤中,超过90%的病例中CDK4通路与激活基因组改变相关[28-29]。通过文献回顾,发现CDK基因在黑素瘤发生发展过程中起到了关键作用。本研究生信分析结果也验证了这个观点。Oncomine数据库中挖掘结果发现其在黑素瘤组织与正常组织比较,表达差异有统计学意义,在c-Bioportal数据库中的数据挖掘结果显示,CDK4基因突变与扩增是黑素瘤患者DFS、OS的独立预后因素。此外,PPI网络互作图显示,hTERT与CDK4在蛋白互作关系上是直接关联的。
在机制上,对黑素细胞CDK4通路的系统研究表明,该通路的激活,加上端粒酶逆转录酶(hTERT)的引入,可以导致这些细胞的永生,但不足以转化。缺乏p16INK4A/p14ARF等位基因失活突变的人黑素细胞衰老延迟,但可以通过引入hTERT而不朽[30]。此外,hTERT结合CDK4过表达或抑制RB1,使细胞不朽[31]。黑素细胞的独特之处在于,除了hTERT的表达外,CDK4的激活是永生所必需的,而在大多数细胞系中单独引入hTERT就足够了。这阐明了hTERT与CDK4共表达对于黑素瘤细胞永生和抑制凋亡起到的独有而关键的作用。
本研究通过数据挖掘提示了hTERT在黑素瘤中潛在的调控网络信息。并找到与hTERT共表达的CDK4基因可能是黑素瘤的潜在预后标记物。本研究使用基于最流行的生物信息学理论的在线工具,对公共数据库中的肿瘤数据进行目标基因分析。与传统的芯片筛选方法比较,该方法具有样本量大、成本低、操作简单等优点。这使大规模的黑素瘤基因组学研究和随后的功能研究成为可能。
[参考文献]
[1] Marco DV,Francesco B,Antonella S. Telomere length maintenance in cancer:At the crossroad between telomerase and alternative lengthening of telomeres(ALT)[J]. Int J Mol Sci,2018,19(2):606.
[2] Schwaederle M,Krishnamurthy N,Daniels GA,et al. Telomerase reverse transcriptase promoter alterations across cancer types as detected by next-generation sequencing:A clinical and molecular analysis of 423 patients[J]. Cancer,2018,124(6):1288-1296.
[3] Ofner R,Ritter C,Heidenreich B,et al. Distribution of TERT promoter mutations in primary and metastatic melanomas in Austrian patients[J]. J Cancer Res Clin Oncol,2017,143(4):613-617.
[4] Roh MR,Park KH,Chung KY,et al. Telomerase reverse transcriptase(TERT) promoter mutations in Korean mela-noma patients[J]. Am J Cancer Res,2017,7(1):134-138.
[5] Hugdahl E,Kalvenes MB,Mannelqvist M,et al. Prognostic impact and concordance of TERT promoter mutation and protein expression in matched primary and metastatic cutaneous melanoma[J]. Br J Cancer,2018,118(1):98-105.
[6] Nagore E,Heidenreich B,Rachakonda S,et al. TERT promoter mutations in melanoma survival[J]. Int J Cancer,2016,139(1):75-84.
[7] Im E,Yoon JB,Lee HW,et al. Human telomerase reverse transcriptase(hTERT)positively regulates 26s proteasome activity[J].J Cell Physiol,2017,232(8):2083-2093.
[8] Vasaikar SV,Straub P,Wang J,et al. LinkedOmics:Analyzing multi-omics data within and across 32 cancer types[J]. Nucleic Acids Res,2018,46(D1):D956-D963.
[9] Osella-Abate S,Bertero L,Senetta R,et al. TERT promoter mutations are associated with visceral spreading in melanoma of the trunk[J]. Cancers(Basel),2019,11(4):452.
[10] Shay JW,Wright WE. Telomeres and telomerase:Three decades of progress[J]. Nat Rev Genet,2019,20(5):299-309.
[11] Liu T,Yuan X,Xu D. Cancer-specific telomerase reverse transcriptase (TERT) promoter mutations:Biological and clinical implications[J]. Genes(Basel),2016,7(7):38.
[12] Yuan X,Kronström M,Hellenius ML,et al. Longitudinal changes in leukocyte telomere length and mortality in elderly swedish men[J]. Aging(Albany NY),2018,10(10):3005-3016.
[13] Lai AG,Pouchkina-Stantcheva N,Di Donfrancesco A,et al. The protein subunit of telomerase displays patterns of dynamic evolution and conservation across different metazoan taxa[J]. BMC Evol Biol,2017,17(1):107.
[14] Kim W,Ludlow AT,Min J,et al. Regulation of the human telomerase gene TERT by telomere position effect-over long distances(TPE-OLD):Implications for aging and cancer[J]. PLoS Biol,2016,14(12):e2000 016.
[15] Hodi FS,Chiarion-Sileni V,Gonzalez R,et al. Nivolumab plus ipilimumab or nivolumab alone versus ipilimumab alone in advanced melanoma(CheckMate 067):4-year outcomes of a multicentre,randomised,phase 3 trial[J]. Lancet Oncol,2018,19(11):1480-1492.
[16] Mohammadpour A,Derakhshan M,Darabi H,et al. Melanoma:Where we are and where we go[J]. J Cell Physiol,2019,234(4):3307-3320.
[17] Liu Z,Li Q,Li K,et al. Telomerase reverse transcriptase promotes epithelial-mesenchymal transition and stemcell-like traits in cancer cells[J]. Oncogene,2013,32(36):4203-4213.
[18] Zhang K,Guo Y,Wang X,et al. WNT/β-catenin directs self-renewal symmetric cell division of hTERThigh prostate cancer stem cells[J]. Cancer Res,2017,77(9):2534-2547.
[19] Saebøe-Larssen S,Fossberg E,Gaudernack G. Characterization of novel alternative splicing sites in human telomerase reverse transcriptase(hTERT):Analysis of expression and mutual correlation in mRNA isoforms from normal and tumour tissues[J]. BMC Mol Biol,2006,7:26.
[20] Listerman I,Sun J,Gazzaniga FS,et al. The major reverse transcriptase-incompetent splice variant of the human telomerase protein inhibits telomerase activity but protects from apoptosis[J]. Cancer Res,2013,73(9):2817-2828.
[21] Karimian A,Ahmadi Y,Yousefi B. Multiple functions of p21 in cell cycle,apoptosis and transcriptional regulation after DNA damage[J]. DNA Repair(Amst),2016,42:63-71.
[22] Yogosawa S,Yoshida K. Tumor suppressive role for kinases phosphorylating p53 in DNA damage-induced apoptosis[J]. Cancer Sci,2018,109(11):3376-3382.
[23] Fang H,Niu K,Mo D,et al. RecQL4-Aurora B kinase axis is essential for cellular proliferation,cell cycle progression,and mitotic integrity[J]. Oncogenesis,2018,7(9):68.
[24] Herrera MC,Chymkowitch P,Robertson JM,et al. Cdk1 gates cell cycle-dependent tRNA synthesis by regulating RNA polymerase III activity[J]. Nucleic Acids Res,2018, 46(22):11 698-11 711.
[25] Cortez D,Guntuku S,Qin J,et al. ATR and ATRIP:Partners in checkpoint signaling[J]. Science,2001,294(5547):1713-1716.
[26] Hafsi H,Dillon MT,Barker HE,et al. Combined ATR and DNA-PK inhibition radiosensitizes tumor cells independently of their p53 status[J]. Front Oncol,2018,8:245.
[27] Polager S,Ginsberg D. E2F-at the crossroads of life and death[J]. Trends Cell Biol,2008,18(11):528-535.
[28] Walker GJ,Flores JF,Glendening JM,et al. Virtually 100% of melanoma cell lines harbor alterations at the DNA level within CDKN2A,CDKN2B,or one of their downstream targets[J]. Genes Chromosomes Cancer,1998, 22(2):157-163.
[29] Curtin JA,Fridlyand J,Kageshita T,et al. Distinct sets of genetic alterations in melanoma[J]. N Engl J Med,2005, 353(20):2135-2147.
[30] Sviderskaya EV,Gray-Schopfer VC,Hill SP,et al. p16/cyclin-dependent kinase inhibitor 2A deficiency in human melanocyte senescence,apoptosis,and immortalization:Possible implications for melanoma progression[J]. J Natl Cancer Inst,2003,95(10):723-732.
[31] Gray-Schopfer VC,Cheong SC,Chong H,et al. Cellular senescence in naevi and immortalisation in melanoma:A role for p16?[J]. Br J Cancer,2006,95(4):496-505.
(收稿日期:2021-09-27)
