水稻落粒性的研究进展
2019-11-18苟亚军杨维丰林少俊高彦昊栾鑫
苟亚军 杨维丰 林少俊 高彦昊 栾鑫,*
水稻落粒性的研究进展
苟亚军1,2杨维丰1,2林少俊1,2高彦昊1,2栾鑫1,2,*
(1广东省植物分子育种重点实验室,广州 510642;2亚热带农业生物资源保护与利用国家重点实验室,广州 510642;*通讯联系人,E-mail: luanxin@scau.edu.cn)
落粒性是野生稻适应自然环境和保持群体繁衍的重要性状,落粒性的丧失是水稻驯化过程中的一个关键事件。落粒程度既直接影响水稻的产量,也影响其对现代化机械收割方式的适应性。本文综述了近年来水稻落粒性的研究进展,包括水稻落粒的生理基础、落粒性基因的定位与克隆,以及调控落粒性的分子机理。
水稻;落粒性;离层;驯化
早在10000年以前,人类社会就开始从狩猎和采集过渡到农耕;4000年前完成了主要作物由野生种到栽培种的驯化,包括水稻、小麦、高粱和玉米等[1]。在水稻的驯化过程中,人类对穗粒数、育性、株型、粒形、抽穗期、种皮颜色、落粒性等重要性状进行了选择[2]。与野生稻相比,栽培稻一般直立生长、穗密、穗粒数增多、无芒、种子休眠期短、不落粒或难落粒[3]。落粒性的丧失是水稻驯化过程中的关键事件之一,最开始的选择可能是无意识的,因为不易掉落的籽粒更容易被收获和种植。随着不落粒等位基因的频率增加,最终在驯化过程中取代了落粒等位基因[4]。从进化的观点来看,野生物种成熟后落粒是一种适应性特征,能够使植物将更多的种子传播到周围环境中,有利于自我繁殖和保存种群;然而在农业生产方面,降低落粒性能促进种子成熟后的有效收集,避免落粒造成的产量损失[5, 6]。
在稻属中,落粒性一般是野生稻>籼稻>粳稻[7]。水稻的落粒程度对其收割方式和产量均有重要影响,容易落粒的品种往往在尚未完全成熟时谷粒便脱落,在生产上会造成严重的损失[8]。难落粒或不落粒的品种适合用小型收割机,落粒程度中等的品种则适合用大型收割机或手工脱粒[9]。因此,在生产上培育中等落粒程度的水稻品种,有助于满足现代化农业生产方式的需求。
定位和克隆水稻落粒性相关基因,阐明其遗传基础和内在的分子作用机理,不仅可以为培育落粒性适中的水稻品种提供理论指导,也对阐明水稻的驯化过程具有重要意义。
1 水稻落粒的生理基础
脱落是指植物的营养器官或生殖器官由于细胞分离而脱离母体,器官脱落的组织区域及邻近的数层细胞被称为离区(abscission zone, AZ)。在脱落时,离区会分化出一到几层薄壁细胞形成的离层(abscission layer, AL),离层相邻的细胞层具有厚实和木质化的细胞壁,有助于提供分离时所需要的机械力[5, 10]。
a-水稻的离区;b-完整离层;c-不完整离层;RA-小穗轴;SL-护颖;AZ-离区;AL-离层;PE-花梗;VB-维管束;d~f-花和花梗交界处纵切面的荧光图像[15];d-完整离层;e~f-不完整离层;白色箭头所指处为离层,bar = 50 μm。
a, Abscission zone of rice; b, Complete abscission layer; c, Incomplete abscission layer; RA-Rachila; SL-Sterile lemma; AZ-Abscission zone; AL-Abscission layer; PE-Pedicel; VB-Vascular bundle; d-f, Fluorescence images of a longitudinal section of the flower and pedicel junction[15]; d-Complete abscission layer; e-f, Incomplete abscission layer, white arrows indicate abscission layer, bar = 50 μm.
图1 水稻离层图解
Fig. 1. Schematic of rice abscission layer.
水稻落粒也与护颖和花梗间离层的形成密切相关[11],如图1所示。脱落过程主要分为四个步骤[5, 12-14]:1)在离区分化出离层细胞。水稻枝梗组织中离层的形成发生在抽穗前16~20 d,即配子体细胞在幼穗中开始分化的时期。离层由1~2层形态较小、胞质稠密并且呈扁平状排列的薄壁细胞组成,而周围的枝梗和颖片细胞由大的厚壁细胞组成。2)当种子逐渐成熟,离层细胞开始响应各种信号发生降解反应。植物激素是最主要的调控信号,通常乙烯、脱落酸、茉莉酸促进脱离过程,而生长素、赤霉素、油菜素内酯抑制脱离过程。3)由于特定信号和环境因素的影响,离层细胞中纤维素酶、多聚半乳糖醛酸酶、β-内切葡聚糖酶等水解酶的活性被激活,然后降解离层细胞的胞间层和细胞壁导致谷粒从植株上脱离。4)在离层的分离面形成保护层。
不同的水稻品种具有不同的离层形态,难以落粒的品种通常没有离层或具有不降解的离层。中等落粒程度的品种中有些具有薄壁细胞和厚壁细胞共存的不规则离层;有些具有在枝梗的内稃侧部分发育的离层;有些品种在内稃侧具有完整的离层,而在外稃侧是不规则的离层。易落粒的品种具有从表皮到接近维管束的发育良好的离层[14]。
2 落粒性QTL的鉴定
对于落粒性的鉴定常用的方法有揉搓法、下落法和仪器测量法。揉搓法是指用手握住成熟的稻穗,轻轻揉搓,观察落粒情况[16]。该方法最为简单高效,但结果容易受人为因素的影响,适用于大群体质量性状基因和主效QTL的定位[17]。下落法是将稻穗从一定高度自由坠落到平铺在地面的铁板上,以落粒数占该穗总粒数的百分率作为落粒率[18]。仪器测量法是利用数字测力仪测量使谷粒从枝梗脱落的作用力,根据作用力大小判断落粒难易程度[14]。仪器测量法测量结果准确,能够避免人为因素引起的实验误差,但操作较为繁琐,费时费力,适用于多基因控制的性状或微效QTL的鉴定[19]。
随着分子生物学的发展,研究者利用不同的作图群体和定位方法,确定了许多控制水稻落粒性的QTL,目前在http://www.gramene.org/上注册的水稻落粒性QTL有29个,但已定位的落粒性QTL数量远远多于Gramene上公布的数值,共计95个,广泛分布于水稻的12条染色体上[2, 3, 7, 9, 14, 15, 17, 18, 20-39],如图2所示。其中第1、4染色体上鉴定到的QTL最多,均有18个,QTL集中在染色体长臂末端区域;在第9、12染色体上鉴定到的QTL较少,分别只有2个和1个。
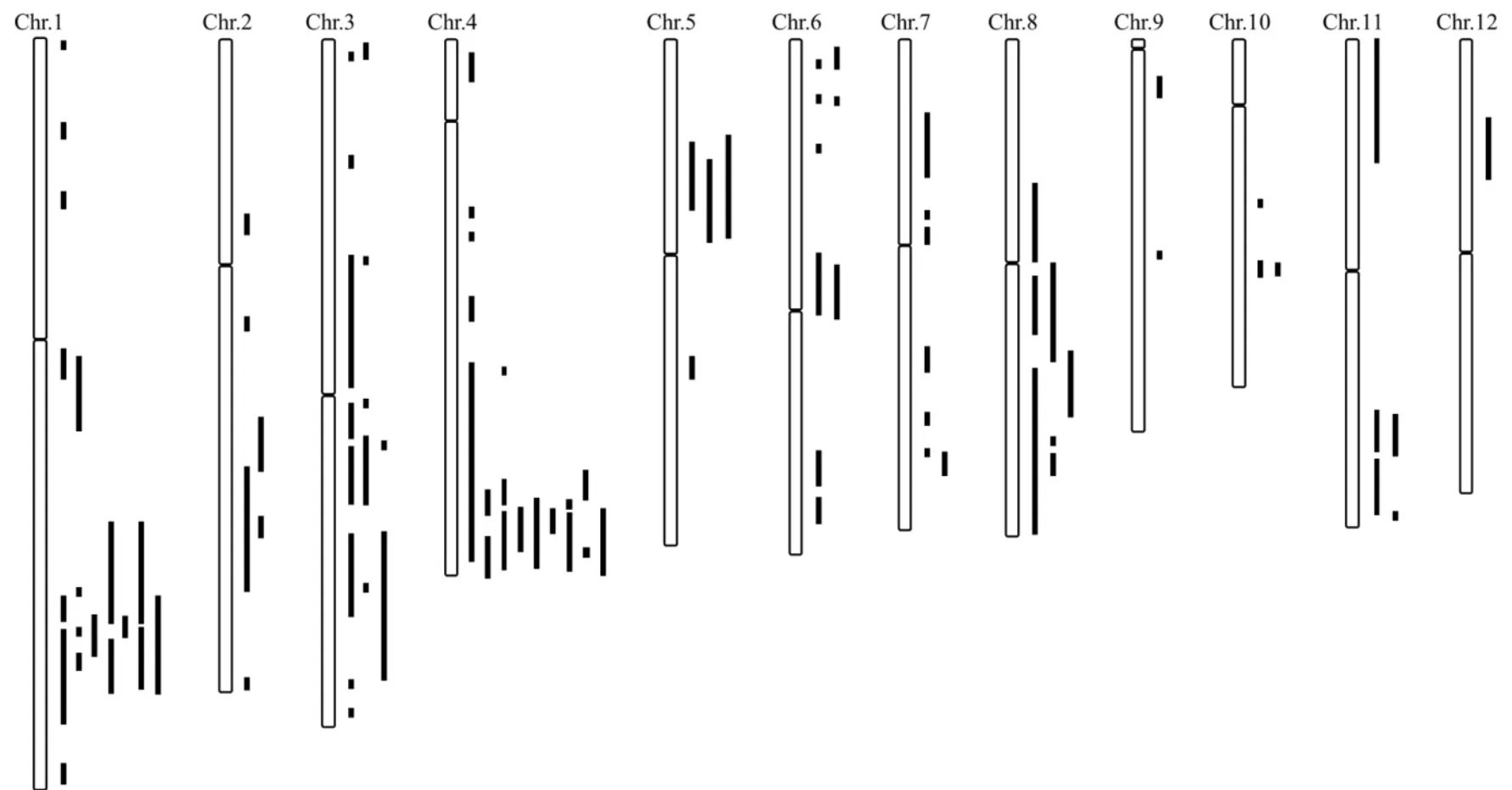
图2 落粒性QTL在水稻染色体上的分布
Fig. 2. Shattering related QTLs on chromosomes in rice.
在这些QTL的基础上,精细定位并克隆相关基因,研究基因的功能变异位点及作用机理,是阐明水稻落粒性调控的重要途径。
3 落粒性相关基因的克隆
目前已克隆的与水稻落粒相关的基因有12个(表1),其中、、、/、、、和是控制落粒的主效基因,/、、、和属于多效基因,在控制落粒性的同时对其他的农艺性状也具有显著的效应。这些基因编码转录因子或激酶,主要通过影响离层的发育或降解来调控落粒性。
3.1 编码转录因子的落粒基因
转录因子也称反式作用因子,是一种具有特殊结构、行使调控基因表达功能的蛋白质分子,通过与启动子区域中顺势作用元件发生特异性相互作用激活或抑制转录[48]。在植物中分离鉴定到的转录因子种类繁多,例如-、、、、、等转录因子家族,它们能够调控植物对各种环境信号的响应及生长发育进程,对于植物器官的脱落也具有重要的作用。
3.1.1基因
同源异型盒基因(genes)编码同源异型蛋白,以转录因子的方式对靶基因进行调控,参与植物的多种生长发育过程。拟南芥中的基因属于基因家族,能够维持长角果上假隔膜的发育,影响果实开裂和种子脱落[49]。同源基因在不同的物种中有着相似的功能,都能影响离层的发育进而调控器官脱落。
在水稻中,位于第1染色体上的能够解释不落粒的粳稻品种日本晴和易落粒的籼稻品种Kasalath之间68.8%的表型变异,是基因的直系同源基因。日本晴中的基因在5’端调节区存在一个单碱基突变(G/T),导致其小穗基部将要形成脱离层的区域中表达量降低,离层无法正常形成而引起落粒性丧失[2]。位于第5染色体上,与高度同源,同样编码BEL1型同源异型蛋白,在离层中的表达量很高,对离层的发育非常重要。在Kasalath中抑制的表达会抑制离层的形成,导致落粒性降低;在中等程度落粒的粳稻品种Dongjin中过表达能够促进离层的发育,增强落粒性;此外,在不落粒的粳稻品种IIpum中过表达能够在离层未发育时降低小穗基部木质素的含量,使其更易落粒[45]。
油菜中的基因也通过控制假隔膜的发育影响落粒,其上游调控区顺式元件中的一个核苷酸的变化,使得易落粒表型变成了不易落粒的表型[50]。Zhang等[51]等利用同源克隆的方法获得了在小麦中的直系同源基因,对离区的发育也至关重要。将导入到拟南芥中既影响果实开裂又影响花器官的脱落,过表达后的拟南芥植株矮化,花瓣离区细胞的发育延迟,长角果上的假隔膜也变窄。
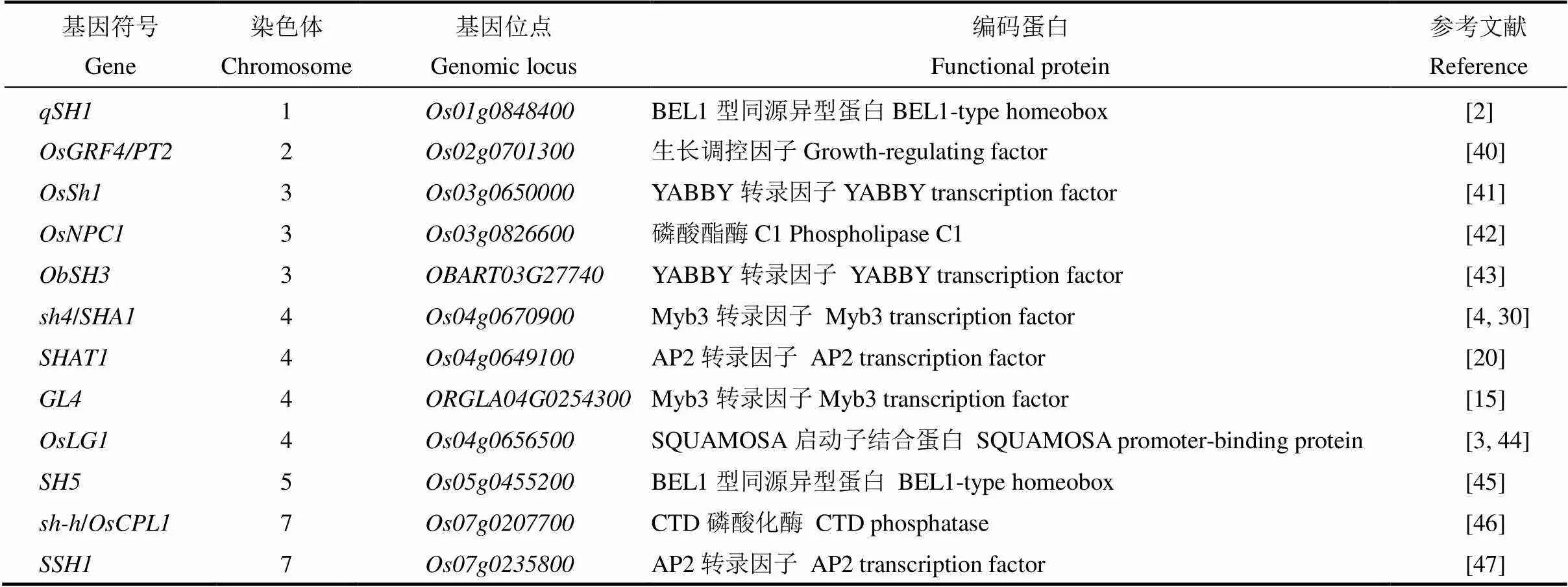
表1 已克隆的落粒性相关基因
3.1.2转录因子
亚洲栽培稻的祖先是普通野生稻[52],非洲栽培稻的祖先是短舌野生稻[53]。两种栽培稻是独立驯化而来的,在驯化过程中两种栽培稻对一些相同基因做出了平行选择。
Li等[29]利用亚洲栽培稻和尼瓦拉野生稻构建F2群体,定位了多个影响水稻关键农艺性状的QTL,包括三个控制落粒性的QTL,分别是、、,其中位于第4染色体上的能解释69%的表型变异。编码一个与同源的转录因子,在离层的早期发育中起重要作用,能够增加落粒性。亚洲栽培稻中的等位基因第一个外显子上的第237位碱基突变(G/T)导致转录因子DNA结合域上的一个天冬氨酸变成丝氨酸,使得离层不能发育完全,从而使落粒变为不落粒[4]。Lin等[30]利用易落粒的元江野生稻导入系IL105与其轮回亲本特青构建分离群体,定位并克隆了基因。与的氨基酸序列具有98%的一致性,其编码区第237位碱基也存在G到T的变异。但小穗离层组织学分析和基因转化实验表明并不参与水稻离层的形成,而是控制离层细胞的降解,从而影响水稻的落粒性。与相比,第152位有一个碱基变化,第158到163位有一个6 bp的插入,这两处变异可能导致了两个基因拥有不同的功能。
在非洲栽培稻中,基因第237位碱基并未发生突变,而是第一个外显子上的另一个突变(C/T),使得谷氨酰胺变成终止密码子导致翻译提前终止,从而引起落粒性的丧失[54]。Wu等[15]还发现亚洲栽培稻中的不影响粒形大小,而非洲栽培稻中的/能够通过调控外颖和内颖纵向细胞的伸长,控制非洲栽培稻的粒长。
3.1.3转录因子
转录因子对于植物的果实发育和脱落有着重要作用,拟南芥中的基因负向调节和基因的表达,抑制假隔膜的发育,促进果荚开裂[55]。小麦中的基因影响小麦穗轴中离层的发育,是控制小麦落粒的主效基因[56]。在水稻中也存在两个控制落粒的家族基因,和[20, 47]。
Zhou等[20]利用籼稻品种GLA4和普通野生稻W1943构建含有已克隆基因和的染色体片段代换系SL4,通过辐射诱变SL4,筛选在和同时存在时仍不落粒的突变体,分离并鉴定了两个不落粒突变体和。其中,落粒性下降是受一个编码转录因子的基因控制,落粒性下降是由已知落粒基因的突变造成的。突变体中的第一个外显子中一个碱基缺失导致移码突变,造成表达量的下降,从而引起该突变体的离区出现部分缺失,落粒性降低。
Jiang等[47]利用具有强落粒性的野生稻渗入系YIL100构建EMS突变体库,并从中筛选出了一个落粒性降低的突变体。结合 MutMap定位和遗传转化实验,鉴定到控制落粒性的基因,是的等位基因,编码植物特异性转录因子。第9内含子中的一个单碱基突变(C/A)影响了该基因 mRNA 的正常剪接,进而改变了离层和维管束的发育,导致突变体的落粒性降低。与野生型相比,突变体的粒长变长、千粒重增加。将突变型等位基因导入到优良籼稻品种93-11中,可使粒长增加9.5%,千粒重增加7.7%,表明在控制落粒性的同时可以提高水稻产量。
3.1.4转录因子
是控制高粱落粒性的重要基因,编码转录因子。玉米中的和是的两个同源基因,也与玉米的不落粒性状相关。在水稻中的同源基因是,通过对不落粒的突变体和易落粒的Nanjing 11进行全基因组比较和转录表达分析,发现突变体的上第3个内含子中有一个超过4 kb的插入,降低了的转录水平,影响离层的发育从而导致不落粒表型[41]。
来源于非洲野生稻,位于第3染色体上,同样编码一个转录因子。非洲栽培稻的基因组的区段缺失,导致种子内外颖两侧离层不对称性发育且发育不完整,引起落粒性的丧失[43]。
3.1.5 其他转录因子
来源于籼稻品种Chuandali的/是一个能够调控水稻粒形、穗长和落粒性的多效基因,位于第2染色体上,编码一个生长调控因子,Chuandali中的基因编码区中结合区域的一个突变导致表达量增加,使得籽粒变大、穗长增加、落粒性降低[40]。
Ishii等[3]利用密穗的日本晴与散穗的普通野生稻W630作为亲本构建回交群体,发现一个同时影响穗型和落粒性的QTL,并将定位在第4染色体上标记S-K3-S-G4之间9.3 kb的区域内。该区域没有编码序列,但在该区域下游10 kb处有一个已克隆的调控水稻叶舌和叶耳发育的基因。编码家族转录因子,在中含有能够调节表达的序列,位点的突变使得日本晴中表达量降低,植株的散穗变为密穗,且落粒性降低。
3.2 编码激酶的落粒基因
Ji等[14]利用不落粒的粳稻品种Hwacheong诱导突变得到易落粒的突变体,在第7染色体上定位到一个控制落粒性的QTL,位于标记RM8262-RM7161,是一个隐性基因,影响离层的发育。进一步研究发现编码CTD磷酸酶,在穗发育期间抑制离层细胞分化,基因突变后功能失活促进离层的形成,导致易落粒[46]。
位于第3染色体上,编码一个磷酸脂酶。过表达导致茎节中硅含量降低、颖壳中的硅含量增加,茎节的厚壁组织厚度减小、维管束纤维素细胞减少使得其受力后更易弯曲,穗节点变得更脆,种子容易脱落[42]。
3.3 落粒性基因之间的调控关系
已克隆的落粒相关基因中,是的等位基因[30];与是同源基因[43];与是同源基因[15];与也是同源基因[45]。通过影响离层细胞的降解来控制落粒性[30]。通过影响穗型的变化调控落粒性,散穗的植株更容易落粒[3]。通过介导茎节和籽粒中硅的分布以及次生细胞壁的沉淀影响茎干机械强度和种子落粒[42]。调控落粒的机理尚不清楚。其余基因都是通过影响离层的形成与发育来调控落粒性(图3)。
部分基因之间存在相互作用。Zhou等[20]通过原位杂交的方法,鉴定了和及的调控关系,基因正向调节的表达,反过来也起到维持在离层表达的作用,两者共同促进离层早期的分化,在下游维持和的持续表达,而在离区的表达受和正调控,最终形成有功能的离层。能够诱导和的表达[45]。能够正向调控和的表达,影响离区木质素沉积和离层发育,进而调控水稻落粒性[47]。
4 落粒与驯化
植物驯化是一个漫长而复杂的进化过程,人类对植物的野生种进行栽培和改良,使得其形态和生理均发生改变从而满足生活和生产需要。驯化植物通常具有区别于其野生祖先的一系列特征,被称为“驯化综合征”[57]。不落粒或落粒性降低对于栽培作物的有效收割具有重要意义,是作物驯化中最典型的性状,对于落粒性基因的解析有助于更好地了解水稻人工驯化的分子基础。
Zhang等[58]通过对31份亚洲栽培稻和44份普通野生稻进行序列分析,发现经过长期的人工选择,非落粒性等位基因在籼粳亚种中得到固定。但是亚洲栽培稻中控制落粒性状的关键基因变异位点(G/T)与非洲栽培稻中的变异位点(C/T)不一样[54],两个基因在驯化过程中受到了平行选择,说明这两个栽培种是独立驯化而来的。Lv等[43]利用93份非洲栽培稻和94份短舌野生稻的基因组重测序数据对和的基因型和地理分布进行分析,发现基因的地理分布更为广泛,说明在非洲栽培稻中受到的选择时间可能更长。
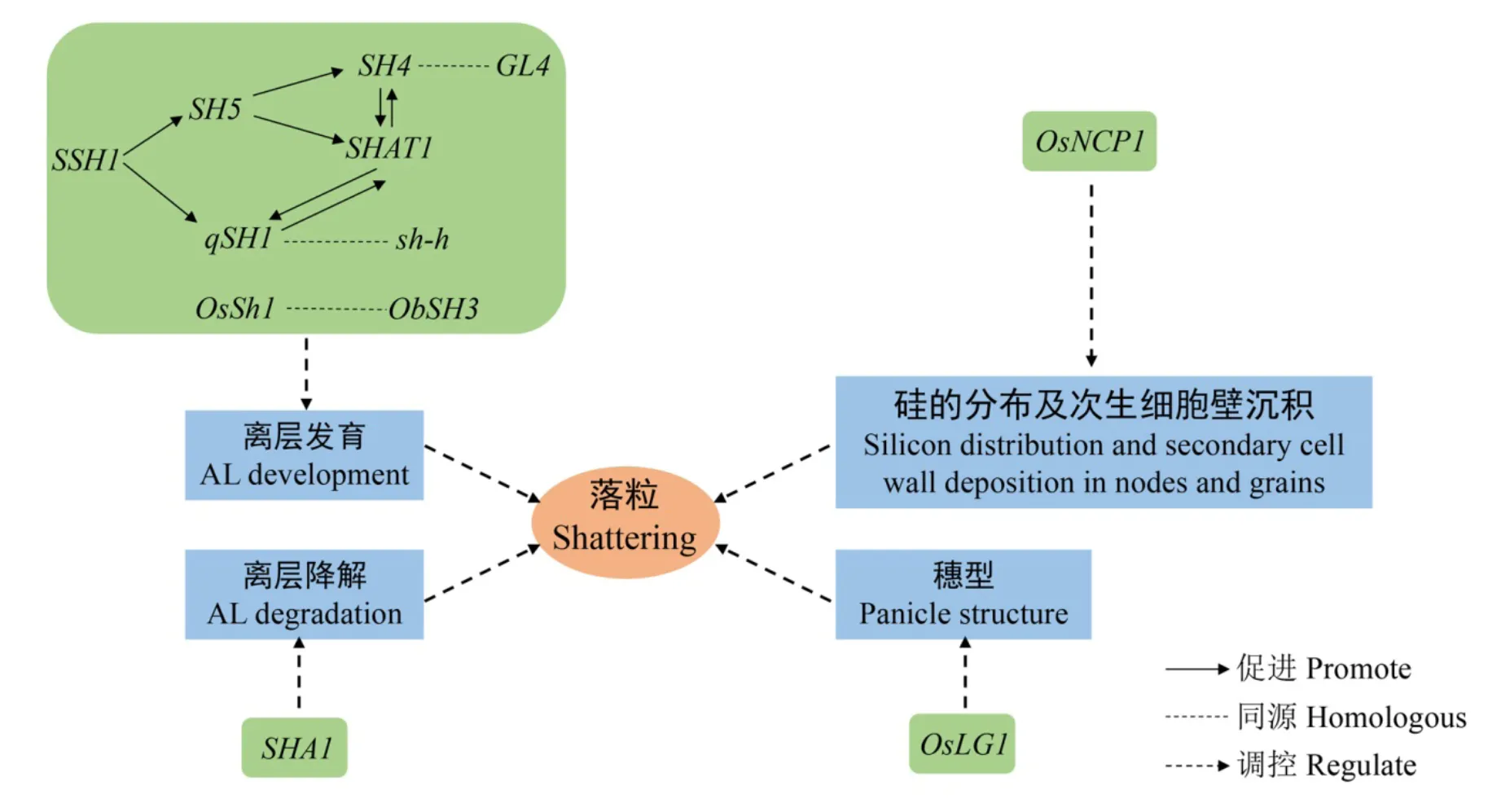
AL-Abscission layer
图3 落粒性基因的调控解析
Fig. 3. Regulatory pathways of grain shattering genes in rice.
Konishi等[2]对75份栽培稻进行单倍型分析发现,在水稻落粒品种中基因都具有与Kasalath相同的SNP位点,而不落粒的品种中都具有与日本晴一样的SNP位点,表明在驯化过程中该位点受到了强烈的人工选择。Lin等[30]对92份粳稻和108份籼稻的检测都发现了不落粒等位基因,但在24份野生稻材料中没有发现,说明在水稻驯化过程中对不落粒基因的选择要早于籼粳分化。
高粱中的,玉米中的与和水稻中的是不同物种中的同源基因,均能通过影响离层的发育控制落粒性[41]。油菜中的基因与拟南芥中的基因均通过控制角果中假隔膜的发育影响落粒[50]。水稻中的和小麦中的都是的直系同源基因,也与落粒性状密切相关[51],这些说明了控制落粒性状的基因在驯化过程中的保守性。
5 总结与展望
水稻是世界上最重要的粮食作物之一,也是单子叶植物的模式生物[59]。水稻的基因组较小,对于其驯化的遗传分析特别是相关基因的克隆,不仅能够揭示水稻的发育和进化过程,用于遗传改良;也可能应用到其他禾本科作物的改良中[29, 60]。随着分子生物学和基因组学研究的深入发展,对于水稻驯化相关性状的研究和调控机理解析日趋简单快速。高密度遗传图谱和物理图谱的构建、全基因组测序的完成、大规模的基因功能分析和基因组重测序等积累了大量的数据[61,62];各种作图群体的发展以及分子标记的多样化提高了基因定位准确性;CRISPR/Cas9系统的应用和改良可以高效靶向编辑基因组[63, 64],为后基因组时代的水稻研究提供了巨大便利。
落粒性的丧失是水稻驯化过程中的关键事件,落粒性相关基因的鉴定和克隆可以为作物的起源进化提供理论依据;同时,落粒程度对水稻的产量和收割方式具有重要影响,培育落粒程度适中的水稻品种既有利于高效收割又能避免产量损失。已克隆的水稻落粒相关基因多数编码转录因子且具有相对保守性。在独立驯化的不同物种中,落粒相关的性状多表现出趋同进化的现象。来自不同物种中落粒性相关的同源基因,往往具有相同或者相似的生物学功能,因此通过反向遗传学的方法快速克隆落粒性相关的基因,将为落粒性状研究提供新的思路。这也可以扩展到其他非模式作物落粒性状的研究中去,以解决目前非模式作物农业生产上面临的实际问题,并且加深我们对作物驯化的认识。
定位和克隆水稻落粒性相关基因,不仅可以揭示单个基因在水稻驯化中的作用和机理;而且可以研究它们之间的分子调控网络及它们在进化过程中的相互作用关系,对深入了解水稻的进化过程、水稻的传播和品种多样性以及人工再驯化遗传改良具有重要意义。虽然目前已经定位到许多控制落粒的QTL,但已克隆的基因非常有限,其中一些基因的作用机理尚不清楚。因此,克隆落粒性相关的基因,解析其调控落粒的分子机理,以及阐释落粒基因之间的互作网络仍将是水稻落粒性研究的主要方向。
[1] Doebley J F, Gaut B S, Smith B D. The molecular genetics of crop domestication., 2006, 127(7): 1309-1321.
[2] Konishi S, Izawa T, Lin S Y, Ebana K, Fukuta Y, Sasaki T, Yano M. An SNP caused loss of seed shattering during rice domestication., 2006, 312: 1392-1396.
[3] Ishii T, Numaguchi K, Miura K, Yoshida K, Thanh P T, Htun T M, Yamasaki M, Komeda N, Matsumoto T, Terauchi R, Ishikawa R, Ashikari M.regulates a closed panicle trait in domesticated rice., 2013, 45(4): 462-465.
[4] Li C, Zhou A, Sang T. Rice domestication by reducing shattering., 2006, 311(5769): 1936-1939.
[5] Estornell L H, Agustí J, Merelo P, Talón M, Tadeo F R. Elucidating mechanisms underlying organ abscission., 2013, 199-200: 48-60.
[6] Purugganan M D, Fuller D Q. The nature of selection during plant domestication., 2009, 457(7231): 843-848.
[7] 李仕贵, 马玉清, 何平, 黎汉云, 陈英, 周开达, 朱立煌. 水稻籼粳杂交落粒性的遗传分析和基因定位. 西南农业学报, 1999, 12(S2): 77-80.
Li S G, Ma Y Q, He P, Li H Y, Chen Y, Zhou K D, Zhu L H. Genetic analysis and mapping the shattering habit in rice (L.)., 1999, 12(S2): 77-80. (in Chinese with English abstract)
[8] 朱立宏, 顾铭洪. 水稻落粒性的遗传. 遗传, 1979, 1(4): 17-19.
Zhu L H, Gu M H. Inheritance of shattering in rice., 1979, 1(4): 17-19. (in Chinese)
[9] Lee G H, Kang I K, Kim K M. Mapping of novel QTL regulating grain shattering using doubled haploid population in rice (L.)., 2016, 2016: 1-8.
[10] Balanzà V, Roig-Villanova I, Marzo M D, Masiero S, Colombo L. Seed abscission and fruit dehiscence required for seed dispersal rely on similar genetic networks., 2016, 143(18): 3372-3381.
[11] Oba S, Sumi N, Fujimoto F, Yasue T. Association between grain shattering habit and formation of abscission layer controlled by grain shattering genein rice (L.)., 1995, 64(3): 607-615.
[12] Roberts J A, Whitelaw C A, Gonzalez-Carranza Z H, Mcmanus M T. Cell separation processes in plants-models, mechanisms and manipulation., 2000, 86(2): 223-235.
[13] Patterson S E. Cutting Loose. Abscission and dehiscence in., 2001, 126(2): 494-500.
[14] Ji H S, Chu S H, Jiang W Z, Cho Y, Hahn J H, Eun M Y, McCouch S R, Koh H J. Characterization and mapping of a shattering mutant in rice that corresponds to a block of domestication genes., 2006, 173(2): 995-1005.
[15] Wu W G, Liu X Y, Wang M H, Meyer R S, Luo X J, Ndjiondjop M N, Tan L B, Zhang J W, Wu J Z, Cai H W, Sun C Q, Wang X K,Wing R A, Zhu Z F. A single- nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication., 2017, 3(6):17064.
[16] 应存山. 中国稻种资源. 北京: 中国农业科技出版社, 1993.
Ying C S. Rice Germplasm Resources in China. Beijing: China Agricultural Science and Technology Press, 1993. (in Chinese)
[17] 朱子超, 王楚桃, 何永歆, 蒋刚, 欧阳杰, 黄乾龙, 李贤勇. 水稻落粒性的遗传分析和基因定位. 杂交水稻, 2014, 29(1): 62-66.
Zhu Z C, Wang C T, He Y X, Jiang G, Ouyang J, Huang Q L, Li X Y. Genetic analysis and molecular mapping of seed shattering in rice., 2014, 29(1): 62-66. (in Chinese with English abstract)
[18] 沈圣泉, 庄杰云, 王淑珍, 包劲松, 郑康乐, 舒庆尧, 夏英武. 籼稻落粒性QTL定位与环境互作效应检测. 分子植物育种, 2004, 2(5): 627-632.
Shen S Q, Zhuang J Y, Wang S Z, Bao J S, Zheng K L, Shu Q Y, Xia Y W. Mapping QTLs for rice shattering trait and analysis major effects., 2004, 2(5): 627-632. (in Chinese with English abstract)
[19] 朱文银, 朱镇, 杨德卫, 林静, 赵凌, 张亚东, 陈涛, 王才林. 一个水稻落粒性基因的SSR标记定位. 中国农学通报, 2008, 24(8): 84-87.
Zhu W Y, Zhu Z, Yang D W, Lin J, Zhao L, Zhang Y D, Chen T, Wang C L. Mapping of, a dominant gene controlling seed shattering using SSR markers., 2008, 24(8): 84-87. (in Chinese with English abstract)
[20] Zhou Y, Lu D F, Li C Y, Luo J H, Zhu B F, Zhu J J, Shangguan Y Y, Wang Z X, Sang T, Zhou B, Han B. Genetic control of seed shattering in rice by the APETALA2 transcription factor., 2012, 24(3): 1034-1048.
[21] Fukuta Y, Yagi T. Mapping of a shattering resistance gene in a mutant line SR-5 induced from anrice variety, Nanjing 11., 1998, 48: 345-348.
[22] Xiong L Z, Liu K D, Dai X K, Xu C G, Zhang Q F. Identification of genetic factors controlling domestication -related traits of rice using an F2population of a cross betweenand., 1999, 98(2): 243-251.
[23] Cai H W, Morishima H. Genomic regions affecting seed shattering and seed dormancy in rice., 2000, 100(6): 840-846.
[24] Bres-Patry C, Lorieux M, Clément G, Bangratz M, Ghesquière A. Heredity and genetic mapping of domestication-related traits in a temperateweedy rice., 2001, 102(1): 118-126.
[25] Thomson M J, Tai T H, McClung A M, Lai X H, Hinga M E, Lobos K B, Xu Y, Martinez C P, McCouch S R. Mapping quantitative trait loci for yield, yield components and morphological traits in an advanced backcross population betweenand thecultivar Jefferson., 2003, 107(3): 479-493.
[26] Gu X Y, Kianian S F, Hareland G A, Hoffer B L, Foley M E. Genetic analysis of adaptive syndromes interrelated with seed dormancy in weedy rice ()., 2005, 110(6): 1108-1118.
[27] Lee S J, Oh C S, Suh J P, McCouch S R, Ahn S N. Identification of QTLs for domestication-related and agronomic traits in an×BC1F7population., 2005, 124(3): 209-219.
[28] 许旭明, 周元昌, 吴为人. 利用分子标记定位籼稻落粒性QTL. 福建农林大学学报: 自然版, 2005, 34(3): 11-18.
Xu X M, Zhou Y C, Wu W R. Mapping of QTLs underlying shattering habit inrice using molecular markers., 2005, 34(3): 11-18. (in Chinese with English abstract)
[29] Li C, Zhou A, Sang T. Genetic analysis of rice domestication syndrome with the wild annual species,., 2006, 170(1): 185-193.
[30] Lin Z W, Griffith M E, Li X R, Zhu Z F, Tan L B, Fu Y C, Zhang W X, Wang X K, Xie D X, Sun C Q. Origin of seed shattering in rice (L.)., 2007, 226(1): 11-20.
[31] Onishi K, Horiuchi Y, Ishigoh-Oka N, Takagi K, Ichikawa N, Maruoka M, Sano Y. A QTL cluster for plant architecture and its ecological significance in Asian wild rice., 2007, 57(1): 7-16.
[32] 朱文银, 杨德卫, 林静, 赵凌, 张亚东, 朱镇, 陈涛, 王才林. 利用染色体片段置换系定位水稻落粒性主效QTL. 植物学通报, 2008, 25(4): 443-448.
Zhu W Y, Yang D W, Lin J, Zhao L, Zhang Y D, Zhu Z, Chen T, Wang C L. Substitution mapping of quantitative trait loci for seed shattering in chromosome segment substitution lines of rice (L.)., 2008, 25(4): 443-448. (in Chinese with English abstract)
[33] Qin Y, Kim S M, Zhao X H, Jia B Y, Lee H S, Kim K M, Eun M Y, Jin I D, Sohn J K. Identification for quantitative trait loci controlling grain shattering in rice., 2010, 32(2): 173-180.
[34] Subudhi P K, Singh P K, DeLeon T, Parco A, Karan R, Biradar H, Cohn M A,Sasaki T. Mapping of seed shattering loci provides insights into origin of weedy rice and rice domestication., 2013, 105(2):276-287.
[35] Htun T M, Inoue C, Chhourn O, Ishii T, Ishikawa R. Effect of quantitative trait loci for seed shattering on abscission layer formation in Asian wild rice., 2014, 64: 199-205.
[36] Yao N, Wang L, Yan H X, Liu Y S, Lu B R. Mapping quantitative trait loci (QTL) determining seed-shattering in weedy rice: Evolution of seed shattering in weedy rice through de-domestication., 2015, 204(3): 513-522.
[37] Cheng J P, He Y Q, Zhan C F, Yang B, Xu E S, Zhang H S, Wang Z F. Identification and characterization of quantitative trait loci for shattering in rice landrace jiucaiqing from Taihu Lake Valley, China., 2016, 9(3): 1-9.
[38] 郑丽媛, 魏霞, 周可, 向佳, 李燕, 刘宝玉, 何光华, 凌英华, 赵芳明. 携带主效落粒基因的水稻染色体片段代换系Z481的鉴定及定位. 科学通报, 2016, 61(7): 748-758.
Zheng L Y, Wei X, Zhou K, Xiang J, Li Y, Liu B Y, He G H, Ling Y H, Zhao F M. Identification of a rice chromosome segment substitution line Z481 carrying a major gene for seed shattering and mapping of., 2016, 61(7): 748-758. (in Chinese with English abstract)
[39] Li F, Numa H, Hara N, Sentoku N, Ishii T, Fukuta Y, Nishimura N, Kato H. Identification of a locus for seed shattering in rice (L.) by combining bulked segregant analysis with whole-genome sequencing., 2019, 39: 36. https://doi.org/10.1007/s11032- 019-0941-3.
[40] Sun P Y, Zhang W H, Wang Y H, He Q, Shu F, Liu H, Wang J, Wang J M, Yuan L P, Deng H F.controls grain shape, panicle length and seed shattering in rice., 2016, 58(10): 836-847.
[41] Lin Z W, Li X R, Shannon L M, Yeh C T, Wang M L, Bai G H, Peng Z, Li J R, Trick H N, Clemente T E, Doebley J, Schnable P S, Tuinstra M R, Tesso T T, White F, Yu J M. Parallel domestication of thegenes in cereals., 2012, 44(6): 720-724.
[42] Cao H S, Zhuo L, Su Y, Sun L X, Wang X M. Non- specific phospholipase C1 affects silicon distribution and mechanical strength in stem nodes of rice., 2016, 86(4): 308-321.
[43] Lv S W, Wu W G, Wang M H, Meyer R S, Ndjiondjop M N, Tan L B, Zhou H Y, Zhang J W, Fu Y C, Cai H W, Sun C Q, Wing R A, Zhu Z F. Genetic control of seed shattering during African rice domestication., 2018, 4: 331-337.
[44] Lee J, Park J J, Kim S L, Yim J, An G. Mutations in the rice liguleless gene result in a complete loss of the auricle, ligule, and laminar joint., 2007, 65(4): 487-499.
[45] Yoon J, Cho L H, Kim S L, Choi H, Koh H J, An G. The BEL1-type homeobox geneinduces seed shattering by enhancing abscission-zone development and inhibiting lignin biosynthesis., 2014, 79(5): 717-728.
[46] Ji H, Kim S R, Kim Y H,Kim H, Eun M Y, Jin I D, Cha Y S, Yun D W, Ahn B O, Lee M C, Lee G S, Yoon U H, Lee J S, Lee Y H, Suh S C, Jiang W Z, Yang Z II, Jin Ping, McCouch S R, An G, Koh H J. Inactivation of the CTD phosphatase-like geneenhances the development of the abscission layer and seed shattering in rice., 2010, 61(1): 96-106.
[47] Jiang L Y, Ma X, Zhao S S, Tang Y Y, Liu F X, Gu P, Fu Y C, Zhu Z F, Cai H W, Sun C Q, Tan L B. The APETALA2-Like transcription factorcontrols rice seed shattering and seed size., 2019, 31(1): 17-36.
[48] 刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用. 科学通报, 2000, 45(14): 1465-1474.
Liu Q, Zhang G Y, Chen S Y. Structure and regulation of plant transcription factors., 2000, 45(14): 1465-1474. (in Chinese)
[49] Roeder A H K, Ferrándiz C, Yanofsky M F. The role of the REPLUMLESS homeodomain protein in patterning thefruit., 2003, 13(18): 1630- 1635.
[50] Arnaud N, Lawrenson T, Østergaard L, Sablowski R. The same regulatory point mutation changed seed-dispersal structures in evolution and domestication., 2011, 21(14): 1215-1219.
[51] Zhang L, Liu D M, Wang D, Zhang R Z, Geng S F, Wu L, Li A L, Mao L. Over expression of the wheat BEL1-like geneaffects floral organ abscission in Arabidopsis thaliana., 2013, 56(2): 98-105.
[52] Wei X, Qiao W H, Chen Y T, Wang R S, Cao L R, Zhang W X, Yuan N N, Li Z C, Zeng H L, Yang Q W. Domestication and geographic origin ofin China: insights from multilocus analysis of nucleotide variation ofand., 2012, 21(20): 5073-5087.
[53] Meyer R S, Choi J Y, Sanches M, Plessis A, Flowers J M, Amas J, Dorph K, Barretto A, Gross B, Fuller D Q, Bimpong I K, Ndjiondjop M N, Hazzouri K M, Gregorio G B, Purugganan M D. Domestication history and geographical adaptation inferred from a SNP map of African rice., 2016, 48(9): 1083-1088.
[54] Win K T, Yamagata Y, Doi K, Uyama K, Nagai Y, Toda Y, Kani T, Ashikari M, Yasui H, Yoshimura A. A single base change explains the independent origin of and selection for the nonshattering gene in African rice domestication., 2017, 213(4): 1925-1935.
[55] Ripoll J J, Roeder A H K, Ditta G S, Yanofsky M F. A novel role for the floral homeotic geneduring Arabidopsis fruit development., 2011, 138(23): 5167-5176.
[56] Simons K J, Fellers J P, Trick H N, Zhang Z C, Tai Y S, Gill B S, Faris J D. Molecular characterization of the major wheat domestication gene., 2006, 172(1): 547-555.
[57] Hammer K. Das Domestikationssyndrom. Die Kulturp flanze. 1984, 32(1): 11-34.
[58] Zhang L B, Zhu Q H, Wu Z Q, Ross-Ibarra J, Gaut B S, Ge S, Sang T. Selection on grain shattering genes and rates of rice domestication., 2009, 184(3): 708-720.
[59] Causse M A, Fulton T M, Cho Y G, Ahn S N, Chunwongse J, Wu K, Xiao J, Yu Z, Ronald P C, Harrington S E. Saturated molecular map of the rice genome based on an interspecific backcross population., 1995, 138: 1251-1274.
[60] 区树俊, 汪鸿儒, 储成才. 亚洲栽培稻主要驯化性状研究进展. 遗传, 2012, 34(11): 1379-1389.
Ou S J, Wang H R, Chu C C. Major domestication traits in Asian rice., 2012, 34(11): 1379-1389. (in Chinese with English abstract)
[61] Nagamura Y, Antonio B A, Sato Y, Miyao A, Namiki N, Yonemaru J I, Minami H, Kamatsuki K, Shimura K, Shimizu Y, Hirochika H. Rice TOGO Browser: A platform to retrieve integrated information on rice functional and applied genomics., 2011, 52(2): 230-237.
[62] International Rice Genome Sequencing Project. The map-based sequence of the rice genome., 2005, 436: 793-800.
[63] Li X T, Xie Y Y, Zhu Q L, Liu Y G. Targeted genome editing in genes andregulatory regions improves qualitative and quantitative traits in crops., 2017, 10(11): 1368-1370.
[64] Hu X X, Wang C, Fu Y P, Liu Q, Jiao X Z, Wang K J. Expanding the range of CRISPR/Cas9 genome editing in rice., 2016, 9(6): 943-945.
Research Progress on Rice Shattering
GOU Yajun1,2, YANG Weifeng1,2, LIN Shaojun1,2, GAO Yanhao1,2, LUAN Xin1,2,*
(Guangdong Provincial Key Laboratory of Plant Molecular Breeding,,;State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresources,,;Corresponding author,:)
Seed shattering is one of the most important traits in wild rice. As an adaptation to the natural environment and the propagation of their offspring, the loss of seed shattering in rice has been a main target during plant selection and domestication. The seed shattering has a direct impact on the yield of rice and the way of its mechanical harvest. The review covers the current advances that have substantially deepen our understanding on seed shattering, such as the physiologic basis responsible for the seed shattering, the identification of crucial genes controlling this trait and the genetic mechanism underlying seed shattering in rice.
rice; shattering; abscission layer; domestication
S511.01; S511.032
A
1001-7216(2019)06-0479-10
10.16819/j.1001-7216.2019.9038
2019-04-01;
2019-07-18。
国家重点研发计划资助项目(2016YFD0100406);广东特支计划资助项目(2016TX03N224)。
