水稻抗稻瘟病分子机制研究进展
2019-11-18曹妮陈渊季芝娟曾宇翔杨长登梁燕
曹妮 陈渊 季芝娟 曾宇翔 杨长登 梁燕
水稻抗稻瘟病分子机制研究进展
曹妮 陈渊 季芝娟 曾宇翔 杨长登*梁燕*
中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006;*通讯联系人, E-mail: ycd311400@163.com; ly318318@126.com)
稻瘟病是危害世界水稻生产最严重的真菌病害之一。稻瘟病菌生理小种变异快,水稻品种的抗性一般仅能维持3~5年。培育和种植抗性品种是目前最经济有效的措施。近年来,对稻瘟病菌致病机制和抗性基因分子机理的系统研究,加深了对该病原菌-宿主系统中病原相关分子模式诱导的免疫反应机制和病原菌效应蛋白诱导的免疫反应机制的了解。本文综述了水稻抗稻瘟病的两种天然免疫机制研究的最新进展,并对目前水稻抗稻瘟病分子机制研究中急需解决的问题和挑战进行探讨和展望。
水稻;稻瘟病;抗病基因;无毒基因;分子机制
水稻是最重要的粮食作物之一,全球有近一半的人口以水稻为主食,水稻生产在解决粮食安全问题中有着举足轻重的地位[1]。到2035年,世界水稻产量需增加26%(2010同比)才能满足因人口快速增长而增加的粮食需求[2],但水稻的高产、稳产、优质一直受到病虫害的制约。稻瘟病是一种由子囊真菌引起的世界性的水稻病害,为植物十大真菌病害之一[3]。每年在全球各水稻种植区都会有不同程度的发生,且因此造成的产量损失达11%~30%[4],经济损失高达660亿美元,足够养活6千万人口[5]。2013−2017年我国水稻稻瘟病年平均危害面积在7500万hm2左右(数据来源于全国农业技术推广服务中心网站,https://www.natesc.org.cn/sites/ MainSite/)。在水稻品种审定中,稻瘟病抗性是不可或缺的关键条件。发掘抗性基因以培育抗性品种是目前控制稻瘟病最经济有效、安全健康与环境友好的策略。水稻与稻瘟病菌互作系统是植物-微生物互作研究的模式系统之一。本文总结了水稻抗稻瘟病分子机制的研究进展,以期通过对基础理论的认知,一方面对该领域未来的研究难点和热点进行讨论;另一方面为新的病害防控措施的提出提供新思路,为育种新技术加持的抗病育种奠定理论基础。
1 水稻免疫反应分子机制
植物在与病原菌长期协同进化过程中形成了多种免疫机制抵抗病原物的入侵。目前研究较透彻的有两种[6]:病原相关分子模式(pathogen-associated molecular patterns, PAMPs)诱导的免疫反应机制(PAMP-triggered immunity, PTI)和病原菌效应蛋白(effector)诱导的免疫反应机制(effector-triggered immunity, ETI)。PTI是由植物细胞表面的模式识别受体(pattern-recognition receptors, PRRs)识别病原菌分泌的PAMPs分子而激发的非特异性免疫反应。病原菌为了克服植物PTI反应,进化出能抑制植物PTI反应的效应蛋白,该蛋白能诱导植物感病(ETS)。植物为了克服病原菌的ETS反应,进化出了第二种防御机制ETI,即通过编码抗性蛋白识别病原物效应蛋白并诱发更激烈的免疫反应。ETI只对与植物长期协同进化中形成“基因对基因”关系的病原菌有效。但这两种免疫机制并不能完全解释寄主与病原物之间的免疫关系[7]。植物RNAi在识别病原菌双链RNA后使之沉默,从而达到抑制病原菌入侵的目的。该机制与PTI相似,病原菌为了克服这一机制也进化出抑制子来抑制植物RNA沉默反应,这一过程类似ETS。随后抑制子被植物抗性蛋白识别,激发了类似ETI的抗性反应[8]。
1 水稻抗稻瘟病PTI防御机制
植物PTI防御机制中,植物模式识别受体PRRs分为两类,一是胞外富含亮氨酸重复序列与胞内激酶的类受体蛋白激酶(receptor-like kinases, RLKs);二是胞内无激酶的类受体蛋白(receptor-like proteins, RLPs),且该类受体蛋白(RLPs)因缺乏胞内激酶的特性,需要与其他含有胞内激酶的蛋白互作从而诱导下游免疫反应[9]。水稻基因组具有超过1131个类受体蛋白激酶(RLKs)和90个无激酶的类受体蛋白(RLPs)基因[10],且编码蛋白可通过识别植物细胞表面的各种信号参与调控植物生理生化过程[11]。植物PRRs可识别病原菌PAMPs,PAMPs在病原菌中是一种保守的结构分子,如延伸因子(elongation factor Tu, EF-Tu)、几丁质、鞭毛蛋白(flagellin peptide, flg22)、脂多糖(lipopolys- accharides, LPS)、肽聚糖(peptidoglycan, PGN)等[12]。
拟南芥中的PRR受体FLS2(Flagellinsensing 2)是一个丝氨酸/苏氨酸类受体蛋白激酶,可特异性识别细菌鞭毛蛋白N端的一段含22个氨基酸残基的保守性多肽flg22,并激活下游抗病反应[13-14]。水稻中的FLS2同源蛋白OsFLS2可直接识别flg22,激发水稻的抗病反应。过表达可增强水稻对flg22的应答。将水稻转入拟南芥突变体可使其缺陷表型恢复[15]。OsFLS2还可识别FLS2所不能识别的flg22衍生物,表明该蛋白特异性识别在不同物种间存在差异。Lu等[16]鉴定了一个富含亮氨酸的受体蛋白激酶BIK1(botrytis-introduced kinase 1),是丝裂原活化蛋白(mitogen-activated protein, MAP)信号转导通路中的必需组分,BIK1将微生物相关分子模式(micro-associated molecular pattern, MAMP)受体复合物与下游胞内信号传导联系起来。水稻中,的表达可被BTH、SA、ACC和HA等抗病信号分子激活,在水稻与稻瘟病菌的非亲和反应中上调表达,而过表达的转基因后代对稻瘟病的抗性显著提高[17],表明在水稻免疫应答方面有一定作用。Sun等[18]发现FLS2可与BAK1(BR11-associated receptor kinase 1)结合并异构化,且FLS2与共受体BAK1以及BIK1一起形成动态复合物来识别鞭毛蛋白并启动免疫信号,由此说明FLS是以异源二聚化形式来识别病原菌PAMPs的。
几丁质是真菌细胞壁的重要组成成分,也是激活植物免疫反应的一类PAMPs。研究表明,在拟南芥中,一类含有细胞外溶解素基序(lysin motif- containing proteins, LysM)结构域的类受体蛋白激酶CERK1(chitin elicitor receptor kinase 1)能够识别几丁质和肽聚糖等PAMPs,激发植物免疫反应[19]。在水稻中,几丁质先与能够编码含有溶解素基序(LysM)结构域受体蛋白的几丁质壳寡糖激发子结合蛋白OsCEBiP(Chitin elicitor binding protein)结合,OsCEBiP再与OsCERK1形成复合体激活下游抗病反应[20]。水稻敲除突变体抑制了几丁质激发的免疫反应,对稻瘟病的抗性减弱。RNAi水稻植株也表现为对稻瘟病抗性减弱[21],说明和在对水稻抗稻瘟病反应中是必需的,缺一不可。Liu等[22]报道含有细胞外溶解素基序(LysM)结构域的类受体蛋白LYP4和LYP6也参与了识别几丁质的过程。OsCERK1作为LYP4和LYP6结合的衔接子,在水稻先天免疫机制中的几丁质和肽聚糖信号传导中发挥双重作用。此外,受体细胞质激酶OsRLCK185和OsRLCK176在几丁质和肽聚糖信号通路中的OsCERK1下游起作用,表明几丁质和肽聚糖共享细胞内信号组分[23]。OsCERK1与OsRLCK185互作,识别几丁质后,OsCERK1磷酸化OsRLCK185,引发水稻细胞内的免疫应答。同源蛋白OsRLCK57、OsRLCK107和OsRLCK118 RNAi水稻抑制了水稻几丁质和肽聚糖介导的免疫反应,包括防御基因表达、活性氧积累等,表明在水稻中,OsRLCK57、OsRLCK107和OsRLCK118正向调节几丁质和肽聚糖介导的抗病反应[24]。在拟南芥中,当宿主细胞识别几丁质时,细胞膜上的AtCERK1将通过其胞外LysM二聚化,进而自身磷酸化来激活下游防卫反应[25]。由此可见,无论是AtCERK1的同源二聚化还是FLS2与BAK1的异源二聚化,模式识别受体PRRs被激活都需要多个激酶相互作用,以复合体形式识别病原菌PAMPs。
2 水稻抗稻瘟病ETI防御机制
ETI是由植物中的特异性抗病蛋白专化性识别病原菌特定效应子蛋白,从而激活植物免疫反应。本文分两方面介绍稻瘟病ETI防御机制,一是稻瘟病抗性基因与无毒基因的发掘与克隆;二是水稻抗性蛋白与稻瘟病菌无毒蛋白之间的互作相关机制。
2.1 水稻稻瘟病抗性基因
近年来,科学家就水稻稻瘟病抗性基因进行了较为深入的系统研究,利用分子标记手段定位到100多个稻瘟病主效抗性基因,已克隆了36个[26](表1)。这些基因编码蛋白分为四类:1)核苷酸结合位点(NBS)-富含亮氨酸重复序列(LRR)蛋白(NBS-LRR),如和。2)受体蛋白激酶(RLK),如克隆自水稻品种地谷,编码细胞外富含B-凝集素结构域和细胞内丝氨酸/苏氨酸激酶结构域的跨膜受体蛋白激酶[27],为组成型表达的单拷贝显性基因,对生理小种ZB15具有特异性抗性,其蛋白质定位于细胞质膜。的第441位单个氨基酸差异区分抗感等位基因。3)富含脯氨酸结构域蛋白,如为隐性基因[28],其编码蛋白结构域共有5个富含脯氨酸的区域,抗性品种Owarihatamochi中的基因与感病品种Aichiasahi中的等位基因相比,第一个与第二个脯氨酸结构域分别有21 和48 bp的缺失,使得抗性提高。4)富含ARM重复序列蛋白,如近期鉴定的编码一个非典型的广谱抗性蛋白,该蛋白包含4个Armadillo重复区,的抗病性与两个编码NBS-LRR蛋白的抗病基因和2有关,并且可以编码定位于细胞质的两种异构体。同源分析结果表明,基因是单子叶所独有的,这也说明单子叶植物存在特有的抗病系统[29]。5)富含四肽重复序列(Tetratricopeptide repeats, TPRs)蛋白,如Chen等[30]鉴定并克隆的是对水稻稻瘟病和白叶枯病均具有广谱抗性的隐性基因。Bsr-k1蛋白可以结合与免疫应答相关的基因家族中的大多数mRNA,使得水稻体内木质素合成减少,削弱免疫应答;第2447位点单碱基G突变为A后导致编码蛋白Bsr-k1功能丧失,基因家族中的mRNA积累,木质素合成增多,功能丧失赋予了水稻对稻瘟病菌和黄单胞杆菌的广谱抗性。的免疫反应相对温和,对水稻的主要农艺性状没有明显影响,该研究证明了通过点突变提升水稻广谱持久抗病性路径的可行性,为水稻广谱抗性育种提供了新基因和新策略。
目前已定位的稻瘟病抗性基因多集中位于第6、11染色体上,少量抗性基因定位于第1、9、12染色体上,在第2、4、8染色体上各有一个抗性基因。大多数广谱抗性基因以多基因或基因簇的形式存在,如第6和12染色体的着丝粒处以及第11染色体的长臂处,多为复等位基因或紧密连锁基因。第6染色体短臂靠近着丝粒处的抗性基因有等[31]。来自野生稻,抗来自13个国家或地区的43个菌株[32];抗来自我国13个水稻栽培区的792个菌株[33];来源于我国水稻品种谷梅4号,抗来自世界各地的50个菌株[34];克隆自日本品种TKM1,对7个菌株表现出抗性[35];第11染色体短臂上的抗性基因簇,如39不仅抗中国广东、江苏、贵州、吉林、云南5个省的475个菌株,且对华南稻区的菌株表现高抗[36];对ZA、ZB、ZC等生理小种都表现出高抗[37];来源于野生稻,对来自云南省的16个菌株表现高抗[38];第11染色体的长臂上存在抗性基因簇位点,/54是从越南品种Tetep中鉴定出来的,对来自印度的稻瘟病菌株表现出高抗[39];来自西非水稻品种LAC23,对来自中国8个稻区的稻瘟病菌株表现出抗性,对华南稻区的菌株表现显著抗性[40];第12染色体的抗稻瘟病主效基因与其对应的无毒基因Avr-互作[41],并且与2和形成基因簇,对稻瘟病表现出显著的广谱抗性[29]。
2.2 稻瘟病无毒基因
植物抗性基因与病原菌无毒基因之间的互作关系符合经典基因对基因学说:植物抗性蛋白能够识别病原菌分泌的无毒蛋白,从而激发下游植物抗性反应。在病原菌中鉴定了与抗性基因相对应的无毒基因24个,其中有12个已被克隆,如、、、、、等(表1)。已被克隆的无毒基因中除和之外都编码少于200个氨基酸的分泌蛋白,而编码分泌中性锌指蛋白酶[41],不分泌但编码次级代谢物并产生杂合蛋白[45]。
2.3 水稻抗性蛋白与稻瘟病菌无毒蛋白之间的互作机制
目前已成对的克隆抗性基因和无毒基因有/,/,/,/,/,/,/,/和/。其中,除/和/之外,其余7对抗性蛋白-无毒蛋白的分子互作关系已被详细解析。该7对水稻抗性蛋白与稻瘟病菌无毒蛋白之间的互作关系可分为两类,一类是两者直接互作:分别是Pita/AVR-Pita、Pik/AVR-Pik、Pia/AVR-Pia、Pi-CO39/ AVR-PiCO39;另一类是两者间接互作,分别是Piz-t/AvrPiz-t和Pii/AVR-Pii。
抗性蛋白-无毒蛋白直接互作有三种方式:一是一种抗病蛋白对应一种无毒蛋白,符合经典基因对基因学说,Pi-ta与AVR-Pita是最早被报道的植物抗性蛋白,两者可直接互作,从而激活抗病反应,且植物抗性蛋白Pi-ta中LRR结构域突变后会导致Pi-ta与AVR-Pita之间互作关系丧失[41],说明LRR结构域对与之间互作是必需的;与Pi-ta/AVR-Pita类似,Pi54/AVR-Pi54可能直接互作。AVR-Pi54编码一个在N端有信号肽的分泌蛋白。模拟实验表明,无毒蛋白AVR-Pi54与抗性蛋白Pi54直接互作[72]。与不同的是,只在病原菌侵染时诱导应答,表明赋予的防御反应具有诱导性。二是两种抗病蛋白以成对形式出现,且只有一个与无毒蛋白互作,如Pik由两个NBS-LRR蛋白Pik-1、Pik-2组成,实验证明只有Pik-1作为AVR-Pik的受体,与其直接互作,但Pik-2也可与Pik-1结合,以复合体的形式参与调控宿主防御反应[73]。值得注意的是,位点有7个等位基因(、、、、、和),有5个等位基因(、、、和),作为祖先型等位基因,可特异性识别、、、和;相应地可特异性识别、和;可特异性识别和,说明抗性等位基因的出现伴随着无毒等位基因的出现,揭示了抗病基因与无毒基因之间的协同进化机制;三是两种抗病蛋白以成对形式出现,但只有一个与多个无毒蛋白互作,如是由和组成,RGA4和RGA5在参与调控AVR-Pia或AVR1-CO39的抗稻瘟病反应时,只有作为AVR蛋白受体的RGA5-A直接与AVR-Pia或AVR1-CO39互作,且解除了RGA5对RGA4的抑制作用,激发细胞凋亡,促进超敏反应的发生。进一步的研究发现,RGA5中的RATX1域在AVR与其识别过程中扮演着极其重要的作用[74]。
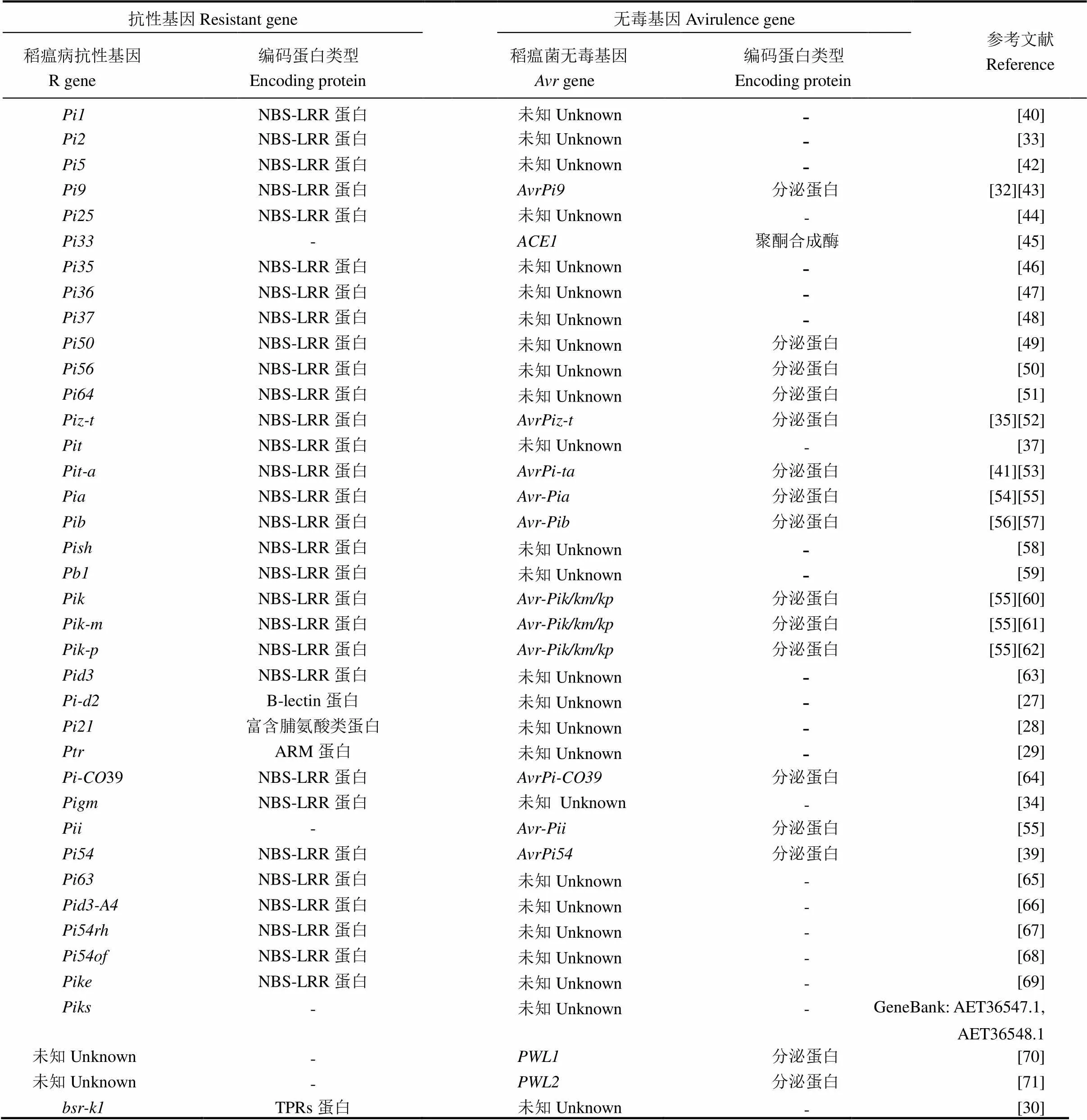
表1 已克隆的稻瘟病抗性基因和无毒基因信息
3 病原菌效应子与寄主靶标蛋白
更多的水稻抗性蛋白与稻瘟病菌无毒蛋白之间不存在直接互作关系,而是通过植物蛋白或激素等信号分子来间接互作,从而调控免疫应答。研究发现,抗性蛋白Piz-t与其相对应的无毒蛋白AvrPiz-t之间间接互作。编码一个N端包含信号肽,长度为108个氨基酸的分泌蛋白,当把异源表达于含有的水稻中,会诱发激烈的感病反应,并显著抑制几丁质介导的PTI免疫反应,说明Piz-t与AvrPiz-t之间存在某种特殊的识别机制[52]。Park等[75]鉴定到12个与AvrPiz-t存在互作的水稻蛋白APIPs(AvrPiz-t interacting protein),对调控寄主抗性具有重要作用。其中蛋白APIP6编码环指型E3泛素连接酶,而AvrPiz-t能破坏APIP6的E3泛素连接酶活性并使APIP6降解,从而抑制了APIP6介导的PTI反应,表明稻瘟病菌通过干扰寄主泛素蛋白降解系统来抑制植物抗病性,并且APIP6对水稻PTI防御机制起正调控作用。此外,APIP10作为一种E3泛素连接酶,可靶向性连接稻瘟病菌中的AvrPiz-t和水稻中的。APIP10是的负调节因子,可通过26S蛋白酶体系统促进Piz-t的降解[76]。水稻中抑制蛋白APIP5的表达可导致水稻细胞死亡,当Piz-t不存在时,AvrPiz-t与靶标蛋白APIP5在细胞质中互作,特异性抑制APIP5蛋白转录活性和蛋白积累,水稻细胞死亡,ETN(effector-triggered necrosis)反应发生;而当Piz-t存在时,Piz-t与APIP5互作,能够稳定APIP5蛋白积累,水稻细胞死亡被抑制,阻止了ETN (Effector-Triggered Necrosis)反应发生。同时,APIP5正调控Piz-t蛋白积累,稳定Piz-t的正常积累水平,表明APIP5对水稻免疫反应起正调控作用[77]。
4 基因天然变异调控水稻抗性
Chen等[78]采用全基因组关联分析的方法,鉴定到一个与广谱抗病表型高度相关的SNP位点,位于编码C2H2类转录因子基因的启动子区,启动子在该区域天然变异,可以提高水稻广谱持久稻瘟病抗性,并且对水稻产量性状和品质性状没有显著影响。该转录因子上游受MYB转录因子负调控,下游正调控过氧化物酶基因的表达,进而影响水稻体内过氧化氢积累,这一新机制极大丰富了水稻广谱抗病性的分子机理。具有一般抗性基因所不具备的优势,即无稻瘟病菌生理小种特异性,应用前景十分广泛。全球收集的3000种质资源中,仅313份水稻材料含有抗性变异位点,说明该位点在水稻育种中已有一定程度的定向选择。
5 表观遗传修饰调控水稻稻瘟病抗性
蛋白磷酸化和甲基化修饰等表观遗传修饰在植物抵抗病原菌入侵过程中发挥重要作用。研究发现稻瘟病抗性蛋白PID2与E3泛素连接酶OsPUB15间接互作,具有激酶活性的PID2K使OsPUB15磷酸化,磷酸化形式的OsPUB15具有E3泛素连接酶活性。过表达OsPUB15的转基因水稻受侵染时可诱发细胞死亡,过氧化氢过量积累,病程相关基因表达量上调,稻瘟病抗性增强,证明OsPUB15正向调控植物抗病反应[79]。水稻理想株型建成的核心基因,不仅能增加水稻产量,还可以提高水稻对稻瘟病的抗性,其编码蛋白IPA1(Ideal Plant Architecture 1)的磷酸化修饰是平衡产量和抗性的关键枢纽,IPA1在稻瘟病菌侵染诱导下被磷酸化,进而改变IPA1与DNA的结合特性,不是结合()等穗发育相关基因的启动子来建成水稻理想株型,而是结合抗性相关基因的启动子来提高免疫应答,表明单个基因可以同时实现增产与抗病,为高产高抗育种提供了重要理论基础和新策略[80]。抗稻瘟病基因既调控稻瘟病广谱持久抗性又不影响产量。在位点存在多个NBS-LRR类抗病基因的基因簇,但只有具有生物学功能。PigmR蛋白自身形成同源二聚体,调控水稻稻瘟病抗性,同时会导致千粒重降低,产量下降。受表观遗传(甲基化水平)调控,仅在水稻的花粉中特异高表达,可提高水稻的结实率,抵消对产量的影响;而在叶片、茎秆等病原菌侵染的组织表达量很低,且PigmS可与PigmR竞争形成异源二聚体抑制介导的广谱抗病性,可为病原菌提供“避难所”,病原菌的进化选择压力变小,减缓了病原菌对PigmR的致病性进化,因此具有持久抗病性。这一机制也表明表观遗传参与调控位点两个抗性基因的表达水平,从而实现产量与抗性的平衡,为利用该基因进行抗性改良和品种选育提供参考[81]。
6 营养元素调控水稻稻瘟病抗性
钾元素在植物生长发育和抗病方面都发挥着重要的作用[82],稻瘟病菌无毒蛋白AvrPiz-t与钾离子通道蛋白OsAKT1作用,能抑制定位于质膜的蛋白OsAKT1介导的钾离子电流。敲除OsAKT1后,钾离子含量和稻瘟病抗性均降低,稻瘟病抗性与外界环境中钾离子含量正相关。该研究为我们提供了一种新机制,即病原菌可通过调节寄主的钾离子通道来破坏植物免疫系统[83]。
7 结论与展望
自1992年第一个抗玉米圆斑病()基因被克隆,截至2017年相继有314个抗病基因被鉴定,其中128个基因给出了可能的抗病机制。有研究将R蛋白诱导疾病抗性的分子机制把这些R基因分为九大类[84],水稻-稻瘟病互作系统只是庞大植物-病原物互作系统的冰山一角。迄今为止,克隆和鉴定了超过50个PRR、R基因和一系列稻瘟病菌无毒基因,无毒基因与R基因的分子互作等方面也取得显著进展,这些基因为稻瘟病抗性育种提供了新思路、理论基础和育种中间材料。如Deng等利用基因培育出既有稻瘟病广谱抗性又高产的水稻品种隆两优3189[81];向聪等[85]也利用基因改良两系不育系C815S的稻瘟病抗性,获得了3个携带纯合抗性基因的改良不育株系;刘文强等[86]将导入优质但易感稻瘟病品种湘晚籼13号,获得3个与亲本相比稻瘟病明显增强的导入系,为培育优质高产抗稻瘟病品种提供中间材料;以下几方面还需要深入研究:
1)抗性蛋白与无毒蛋白互作激活的下游信号途径及其信号途径之间的交叉互作(crosstalk):尽管已鉴定到一系列稻瘟病抗性基因和无毒基因,阐明了相互之间的作用机制,但关于抗性基因与无毒基因互作后激活的下游信号通路的研究甚少。AvrPiz-t与APIP6、APIP5、APIP10和OsAKT1之间的作用机制预示着抗性基因与无毒基因互作后激活的下游信号通路是一个极其复杂的网络系统,各信号通路之间的交叉互作是未来研究的难点和热点。
2)水稻广谱抗稻瘟病的分子机理及其与产量和品质等的平衡机制:具有广谱抗稻瘟病的抗源材料是目前抗稻瘟病育种急需资源,而对广谱抗病的分子机制仍将是后续研究的热点,以介导的广谱抗病性为例,除稻瘟病菌侵染前后和相互作用机制不清楚外,在水稻和稻瘟病菌的亲合和非亲和反应中,二者如何互作以及如何调控下游的抗病反应仍未知。除此之外,如何打破抗病基因位点与较差的产量和品质之间的连锁效应也是抗病育种中急需解决的问题。
3)表观遗传修饰参与调控水稻免疫机制:表观遗传修饰实现了抗性和产量的平衡,其在病原菌入侵前后的调控机制还有待阐明;其他已克隆抗性基因的表达是否受到病原菌入侵引起的全基因组/特异位点表观遗传修饰的诱导仍需进一步研究。
4)利用不断增加的水稻-稻瘟病系统先天免疫的基础理论,发掘新的抗稻瘟病基因,利用双单倍体技术、转基因技术、分子标记辅助育种技术、基因组编辑技术、智能不育技术、杂种优势固定技术等作物育种技术,提高育种效率,培育具有广谱抗稻瘟病的新品种,早日实现绿色防控稻瘟病。
[1] 杜轶威. 水稻开花相关RING蛋白1(FRRP1)基因的克隆和开花功能分析. 北京: 中国农业大学, 2016.
Du Y W. Molecular cloning and functional analysis of() in rice. Beijing: China Agriculture University, 2016. (in Chinese with English abstract)
[2] Seck PA, Diagne A, Mohanty S, Wopereis M C S. Crops that feed the world 7: Rice., 2012, 4(1): 7-24
[3] Dean R, van Kan J A, Pretorius Z A, Hammond-Kosack K E, Di Pietro A, Spanu P D, Rudd J J, Dickman M, Kahmann R, Ellis J, Foster G D. The top 10 fungal pathogens in molecular plant pathology., 2012, 13(4): 414-430.
[4] Skamnioti P, Gurr S J. Against the grain: Safeguarding rice from rice blast disease., 2009, 27(3):141-150.
[5] Pennisi E. Armed and dangerous., 2010, 327(5967): 804-805
[6] Nakahara K, Masuta C. Interaction between viral RNA silencing suppressors and host factors in plant immunity., 2014, 20: 88-95.
[7] Akerley B J, Cotter P A, Miller J F. Ectopic expression of the flagellar regulon alters development of the Bordetella-host interaction., 1995, 80(4): 611-620.
[8] Dow M, Newman M A, von Roepenack E. The induction and modulation of plant defense responses by bacterial lipopolysaccharides., 2000, 38: 241-261.
[9] Zipfel C. Pattern-recognition receptors in plant innate immunity., 2008, 20(1): 10-16.
[10] Shiu S, Karlowski W, Pan R, Tzeng Y H, Mayer K F, Li W H. Comparative analysis of the receptor-like kinase family inand rice., 2004, 16(5): 1220-1234.
[11] Chen X, Ronald P. Innate immunity in rice., 2011, 16(8): 451-459.
[12] Liu B, Li J, Ao Y, Qu J, Li Z, Su J, Zhang Y, Liu J, Feng D, Qi K, He Y, Wang J, Wang H B. Lysin motif- containing proteins LYP4 and LYP6 play dual roles in peptidoglycan and chitin perception in rice innate immunity., 2012, 24(8): 3406-3419.
[13] Felix G, Duran JD, Volko S, Boller T. Plants have a sensitive perception system for the most conserved domain of bacterial flagellin., 1999, 18(3): 265-276.
[14] Delphine C, Martin R, Boller T. Thereceptor kinase fls2 binds flg22 and determines the specificity of flagellin perception., 2006, 18(2): 465-476.
[15] Shinya T, Osada T, Desaki Y, Hatamoto M, Yamanaka Y, Hirano H, Takai R, Che F S, Kaku H, Shibuya N. Characterization of receptor proteins using affinity cross-linking with biotinylated ligands., 2010, 51(2): 262-270.
[16] Lu D, Wu S, Gao X, Zhang Y, Shan L, He P. A receptor- like cytoplasmic kinase, BIK1, associates with a flagellin receptor complex to initiate plant innate immunity., 2010, 107(1): 496-501.
[17] 张慧娟. 磷酸-1-鞘氨醇在植物抗病反应中的作用及水稻和拟南芥BIK1在逆境反应中的功能分析. 杭州: 浙江大学, 2009.
Zhang H J. Role of sphingosine-1-phosphate in plant defense response and functional analysis ofand ricein stress responses. Hangzhou: Zhejiang University, 2009. (in Chinese with English abstract)
[18] Sun Y, Li L, Macho AP, Han Z, Hu Z, Zipfel C, Zhou J M, Chai J. Structural basis for flg22-induced activation of theFLS2-BAK1 immune complex., 2013, 342(6158): 624-628.
[19] Miya A, Albert P, Shinya T, Desaki Y, Ichimura K, Shirasu K, Narusaka Y, Kawakami N, Kaku H, Shibuya N. CERK1, a LysM receptor kinase, is essential for chitin elicitor signal in in., 2007, 104(49): 19613-19618.
[20] Shimizu T, Nakano T, Takamizawa D, Desaki Y, Ishii-Minami N, Nishizawa Y, Minami E, Okada K, Yamane H, Kaku H, Shibuya N. Two LysM receptor molecules, CEBiP and OsCERK1, cooperatively regulate chitin elicitor signaling in rice., 2010, 64(2): 204-214.
[21] Kaku H, Nishizawa Y, Ishii-Minami N, Akimoto- Tomiyama C, Dohmae N, Takio K, Minami E, Shibuya N. Plant cells recognize chitin fragments for defense signaling through a plasma membrane receptor., 2006, 103(29): 11 086-11 091.
[22] Liu B, Li J F, Ao Y, Qu J, Li Z, Su J, Zhang Y, Liu J, Feng D, Qi KB, He Y M, Wang J F, Wang H B. Lysin motif-containing proteins LYP4 and LYP6 play dual roles in peptidoglycan and chitin perception in rice innate immunity., 2012, 24(8): 3406-3419.
[23] Ao Y, Li Z Q, Feng D R, Xiong F, Liu J, Li J F, Wang J, Liu B, Wang H B. OsCERK1 and OsRLCK176 play important roles in peptidoglycan and chitin signaling in rice innate immunity.2014, 80: 1072-1084.
[24] Li Z, Ao Y, Feng D, Liu J, Wang J, Wang H B, Liu B. OsRLCK 57, OsRLCK107 and OsRLCK118 positively regulate chitin- and PGN-induced immunity in rice., 2017, 10(1): 6.
[25] Kawasaki T, Yamada K, Yoshimura S, Yamaquchi K. Chitin receptor-mediated activation of MAP kinases and ROS production in rice and., 2017: e1361076
[26] WangBH, Ebbole DJ, WangZH. The arms race betweenand rice: Diversity and interaction ofandgenes.,2017, 16: 2746-2760.
[27] Chen X, Shang J, Chen D, Lei C, Zou Y, Zhai W, Liu G, Xu J, Ling Z, Cao G. A B-lectin receptor kinase gene conferring rice blast resistance., 2006, 46(5): 794-804.
[28] Fukuoka S, Saka N, Koga H, Ono K, Shimizu T, Ebana K, Hayashi N, Takahashi A, Hirochika H, Okuno K, Yano M. Loss of function of a proline-containing protein confers durable disease resistance in rice., 2009, 325: 998-1001.
[29] Zhao H, Wang X, Jia Y, Minkenberg B, Wheatley M, Fan J, Jia M H, Famoso A, Edward J D, Wamishe Y, Valent B, Wang G L, Yang Y. The rice blast resistance geneencodes an atypical protein required for broad-spectrum disease resistance., 2018, 9(1): 2039.
[30] Zhou X, Liao H, Chern M, Yin J, Chen Y, Wang J, Zhu X, Chen Z, Chen Z, Yuan C, Zhao W, Wang J, Li W , He M, Ma B, Wang J, Qin P, Chen W, Wang Y, Liu J, Qian Y, Wang W, Wu X, Li P, Zhu L, Li S, Ronald P C, Chen X. Loss of function of a rice TPR-domain RNA-binding protein confers broad-spectrum disease resistance., 2018, 115(12): 3174-3179.
[31] Wu Y, Yu L, Pan C, Dai Z, Li Y, Xiao N, Zhang X, Ji H, Huang N, Zhao B. Development of near-isogenic lines with different alleles oflocus and analysis of their breeding effect under Yangdao 6 background., 2016, 36(2): 12.
[32] Qu S, Liu G, Zhou B, Bellizzi M, Zeng L, Dai L, Han B, Wang GL. The broad-spectrum blast resistance geneencodes a nucleotide-binding site-leucine-rich repeat protein and is a member of a multigene family in rice., 2006, 172 (3): 1901-1914.
[33] Chen D H, Zeigler R S, Ahn S W, Nelson R J. Phenotypic characterization of the rice blast resistance gene(t)., 1996: 80.
[34] Deng Y, Zhu X, Shen Y, He Z. Genetic characterization and fine mapping of the blast resistance locus(t) tightly linked toandin a broad-spectrum resistant Chinese variety., 2006, 113(4): 705-713.
[35] Zhou B, Qu S H, Liu G F, Dolan M, Sakai H, Lu GD, Bellizzi M, Wang G L. The eight amino-acid differences within three leucine-rich repeats betweenandresistance proteins determine the resistance specificity to., 2006, 19 (11): 1216-1228.
[36] Hua L X, Liang L Q, He X Y, Wang L, Zhang W S, Liu W, Liu X Q, Lin F. Development of a marker specific for the rice blast resistance genein the Chinese cultivar Q15 and its use in genetic improvement., 2015, 29 (3): 448-456.
[37] Hayashi K, Yoshida H. Refunctionalization of the ancient rice blast disease resistance geneby the recruitment of a retrotransposon as a promoter.2009, 57: 413-425.
[38] Xu X, Hayashi N, Wang CT, Fukuoka S, Kawasaki S, Takatsuji H, Jiang C J. Rice blast resistance gene(t), a member of a resistance gene cluster on chromosome 4, encodes a nucleotide-binding site and leucine-rich repeat protein., 2014, 34 (2): 691-700.
[39] Sharma T R, Rai A K, Gupta S K, Singh N K. Broad-spectrum blast resistance genecloned from rice line Tetep designated as., 2010, 19(1): 87-89.
[40] Hua L, Wu J Z, Chen C X, Wu W H, He X Y, Lin F, Wang L, Ashikawa I, Matsumoto T, Wang L, Pan Q H. The isolation of Pi1, an allele at thelocus which confers broad spectrum resistance to rice blast., 2012, 125(5): 1047-1055.
[41] Orbach M J, Farrall L, Sweigard J A, Chumley F G, Valent B. A telomeric avirulence gene determines efficacy for the rice blast resistance gene.2000, 12: 2019-2032.
[42] Lee S K, Song M Y, Seo Y S, Kim H K, Ko S, Cao P J, Suh J P, Yi G, Roh J H, Lee S, An G, Hahn T R, Wang G L, Ronald P, Jeon J S. Rice-mediated resistance torequires the presence of two coiled-coil-nucleotide-binding-leucine-rich repeat genes.,2009, 181(4): 1627-1638.
[43] Wu J, Kou Y, Bao J, Li Y, Tang M, Zhu X, Ponaya A, Xiao G, Li C, Song M Y, Cumagun C J, Deng Q, Lu G, Jeon J S, Naqvi N I. Comparative genomics identifies theavirulence effectorthat triggers-mediated blast resistance in rice., 2015, 206: 1463-1475.
[44] Chen J, Shi Y F, Liu W Z, Chai R Y, Fu Y, Zhuang J Y, Wu J L.Aallele from rice cultivar Gumei 2 confers resistance to., 2011, 38: 209-216.
[45] Bohnert HU, Fudal I, Dioh W. A putative polyketide synthase/peptide synthetase fromsignals pathogen attack to resistant rice.2004, 16: 2499-2513.
[46] Fukuoka S, Yamamoto S I, Mizobuchi R, Yamanouchi U, Ono K, Kitazawa N, Yasuda N, Fujita Y, Nguyen TTT, Koizumi S, Sugimoto K, Matsumoto T, Yano M. Multiple functional polymorphisms in a single disease resistance gene in rice enhance durable resistance to blast., 2014: 4.
[47] Liu X, Lin F, Wang L, Pan Q. The in silico map-based cloning of, a rice coiled-coil nucleotide-binding site leucine-rich repeat gene that confers race specific resistance to the blast fungus., 2007, 176: 2541-2549.
[48] Lin F, Chen S, Que Z, Wang L, Liu X, Pan QThe blast resistance geneencodes a nucleotide binding site leucine-rich repeat protein and is a member of a resistance gene cluster on rice chromosome 1.,2007, 177: 1871-1880.
[49] Su J, Wang W, Han J, Chen S, Wang C, Zeng L, Feng A, Yang J, Zhou B, Zhu X. Functional divergence of duplicated genes results in a novel blast resistance geneat thelocus.,2015, 128: 2213-2225.
[50] Liu Y, Liu B, Zhu X, Yang J, Bordeos A, Wang G, Leach J E, Leung H. Fine-mapping and molecular marker development for(t), a NBS-LRR gene conferring broad-spectrum resistance toin rice., 2013, 126(4): 985-998.
[51] Ma J, Lei C, Xu X, Hao K, Wang J, Cheng Z, Ma X, Ma J, Zhang X, Guo X, Wu F, Lin Q, Wang C, Zhai H, Wang H, Wan J., encoding a Novel CC-NBS-LRR protein, confers resistance to leaf and neck blast in rice., 2015, 28: 558-568.
[52] Li W, Wang B, Wu J, Lu G, Hu Y, Zhang X, Zhang Z, Feng Q, Zhang H, Wang Z, Wang G, Han B, Wang Z, Zhou B. Theavirulence geneencodes a predicted secreted protein that triggers the immunity in rice mediated by the blast resistance gene.2009, 22: 411-420.
[53] Bryan G T, Wu K S, Farrall L, Jia Y, Hershey H P, McAdams S A, Faulk K N, Donaldson G K, Tarchini R, Valent B. A single amino acid difference distinguishes resistant and susceptible alleles of the rice blast resistance gene., 2000, 12: 2033-2046.
[54] Okuyama Y, Kanzaki H, Abe A ,Yoshida K, Tamiru M, Saitoh H, Fujibe T, Matsumura H, Shenton M, Galam D C, Undan J, Ito A, Sone T, Terauchi R. A multifaceted genomics approach allows the isolation of the rice-blast resistance gene consisting of two adjacent NBS-LRR protein genes.2011, 66: 467-479.
[55] Yoshida K, Saitoh H, Fujisawa S, Kanzaki H, Matsumura H, Tosa Y, Chuma I, Takano Y, Win J, Kamoun S, Terauchi R. Association genetics reveals three novel avirulence genes from the rice blast fungal pathogen., 2009, 21: 1573-1591.
[56] Zhang S, Wang L, Wu W, He L, Yang X, Pan Q. Function and evolution ofavirulence gene AvrPib responding to the rice blast resistance gene., 2015, 5: 11642.
[57] Wang Z X, Yano M, Yamanouchi U, Iwamoto M, Monna L, Hayasaka H, Katayose Y, Sasaki T. Thegene for rice blast resistance belongs to the nucleotide binding and leucine-rich repeat class of plant disease resistance genes., 1999, 19: 55-64.
[58] Takahashi A, Hayashi N, Miyao A, Hirochika H. Unique features of the rice blast resistancelocus revealed by large scale retrotransposon-tagging., 2010, 10(1): 175.
[59] Hayashi N, Inoue H, Kato T, Funao T, Shirota M, Shimizu T, Kanamori H, Yamane H, Hayano S Y, Matsumoto T, Yano M, Takatsuji H. Durable panicle blast-resistance geneencodes an atypical CC-NBS- LRR protein and was generated by acquiring a promoter through local genome duplication., 2010, 64: 498-510.
[60] Zhai C, Lin F, Dong Z, He X, Yuan B, Zeng X, Wang L, Pan Q. The isolation and characterization of, a rice blast resistance gene which emerged after rice domestication., 2011, 189: 321-334.
[61] Ashikawa I, Hayashi N, Yamane H, Kanamori H, Wu J, Matsumoto T, Ono K, Yano M. Two adjacent nucleotide- binding site-leucine-rich repeat class genes are required to confer-specific rice blast resistance.2008, 180: 2267-2276.
[62] Yuan B, Zhai C, Wang W, Zeng X, Xu X, He X, Lin F, Wang L, Pan Q. Theresistance toin rice is mediated by a pair of closely linked CC-NBS-LRR genes.2011, 122: 1017-1028.
[63] Shang J, Tao Y, Chen X, Liu W, Chai R, Fu Y, Zhuang J, Wu J. Identification of a new rice blast resistance gene,, by genome wide comparison of paired nucleotide- binding site leucine-rich repeat genes and their pseudogene alleles between the two sequenced rice genomes.2009, 182: 1303-1311.
[64] Ribot C, Cesari S, Abidi I. Theeffector AVR1-CO39 is translocated into rice cells independently of a fungal-derived machinery.2013, 74: 1-12.
[65] Xu X, Hayashi N, Wang C T, Fukuoka S, Kawasaki S, Takatsuji H, Jiang C. Rice blast resistance gene(t), a member of a resistance gene cluster on chromosome 4, encodes a nucleotide-binding site and leucine-rich repeat protein., 2014, 34: 691-700.
[66] Lü Q, Xu X, Shang J, Jiang G, Pang Z, Zhou Z, Wang J, Liu Y, Li T, Li X, Xu J, Cheng Z, Zhao X, Li S, Zhu L. Functional analysis of, an ortholog of rice blast resistance generevealed by allele mining in common wild rice., 2013, 103: 594-599.
[67] Das A, Soubam D, Singh P K, Thakur S, Singh NK, Sharma R. A novel blast resistance gene,cloned from wild species of rice,confers broad spectrum resistance to., 2012, 12: 215-228.
[68] Devanna NB, Vijayan J, Sharma TR. The blast resistance geneof cloned frominteracts withthrough its novel non-LRR domains., 2014, 9: e104840.
[69] Chen J, Peng P, Tian J, He Y, Zhang L, Liu Z, Yin D, Zhang Z., a rice blast resistance allele consisting of two adjacent NBS-LRR genes, was identified as a novel allele at thelocus., 2015, 35: 117.
[70] Kang S, Sweigard J A, Valent B. The PWL host specificity gene family in the blast fungus., 1995, 8(6): 939-948.
[71] Sweigard J A, Carroll A M, Kang S, Farrall L, Chumley F G, Valent B. Identification, cloning, and characterization of, a gene for host species specificity in the rice blast fungus., 1995, 7: 1221-1233.
[72] Ray S, Singh P K, Gupta D K, Mahato A K, Sarkar C, Rathour R, Singh N K, Sharma T R. Analysis ofgenome reveals a fungal effector, which is able to induce resistance response in transgenic rice line containing resistance gene,., 2016(7): 1140.
[73] Kanzaki H, Yoshida K, Saitoh H, Fujisaki K, Hirabuchi A, Alaux L, Fournier E, Tharreau D, Terauchi R. Arms race co-evolution ofand ricegenes driven by their physical interactions., 2012, 72(6): 894-907.
[74] Ortiz D, de Guillen K, Cesari S, Chalvon V, Gracy J, Padilla A, Kroi T. Recognition of theeffector AVR-Pia by the Decoy domain of the rice NLR immune receptor RGA5., 2017, 29(1): 156-168.
[75] Park C H, Chen S, Shirsekar G, Zhou B, Khang C H, Songkumarn P, Afzal A J, Ning Y, Wang R, Bellizzi M. Theeffector AvrPiz-t targets the RING E3 ubiquitin ligase APIP6 to suppress pathogen- associated molecular pattern-triggered immunity in rice., 2012, 24 (11): 4748-4762.
[76] Park C H, Shirsekar G, Bellizzi M, Chen S, Songkumarn P, Xie X, Shi X, Ning Y, Zhou B, Suttiviriya P, Wang M, Umemura K, Wang G L. The E3 ligase APIP10 connects the effector AvrPiz-t to the NLR receptorin rice., 2016, 12(3): e1005529.
[77] Wang R, Ning Y, Shi X, He F, Zhang C, Fan J, Jiang N, Zhang Y, Zhang T, Hu Y, Bellizzi M, Wang G L. Immunity to rice blast disease by suppression of effector- triggered necrosis., 2016, 26(18): 2399-2411.
[78] Li W, Zhu Z, Chern M, Yin J, Yang C, Ran L, Cheng M, He M, Wang K, Wang J, Zhou X, Zhu X, Chen Z, Wang J, Zhao W, Ma B, Qin P, Chen W, Wang Y, Liu J, Wang W, Wu X, Li P, Wang J, Zhu L, Li S, Chen X. A natural allele of a transcription factor in rice confers broad-spectrum blast resistance., 2017, 170 (1): 114-126.
[79] Wang J, Qu B, Dou S, Li L, Yin D, Pang Z, Zhou Z, Tian M, Liu G, Xie Q, Tang D, Chen X, Zhu L. The E3 ligase OsPUB15 interacts with the receptor-like kinase PID2 and regulates plant cell death and innate immunity., 2015, 15(1): 49.
[80] WangJ, Zhou L, Shi H, Chern M, Yu H, Yi H, He M, Yin J, Zhu X, Li Y, Li W, Liu J, Wang J, Chen X, Qing H, Wang Y, Liu J, Wang W, Li P, Wu X, Zhu L, Zhou J M, Ronald P C, Li S, Li J, Chen X. A single transcription factor promotes both yield and immunity in rice., 2018, 361(6406): 1026.
[81] Deng Y, Zhai K, Xie Z, Yang D, Zhu X, Liu J, Wang X, Qin P, Yang Y, Zhang G. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance., 2017, 355(6328): 962-965.
[82] Wang Y, Wu W H. Genetic approaches for improvement of the crop potassium acquisition and utilization efficiency., 2015, 25: 46-52.
[83] Shi X, Long Y, He F, Zhang C, Wang R. The fungal pathogensuppresses innate immunity by modulating a host potassium channel., 2018, 14(1): e1006878.
[84] Kourelis J, van der Hoorn R A L. Defended to the nines: 25 years of resistance gene cloning identifies nine mechanisms for R protein function., 2018, 30(2): 285-299.
[85] 向聪, 任西明, 雷东阳, 陈英. 分子标记辅助选择改良C815S的稻瘟病抗性. 湖南农业大学学报: 自然科学版, 2018, 44(1): 62-65.
Xiang C, Ren X M, Lei D Y, Chen Y. Improvement of rice blast resistance of C815S through molecular marker-assisted selection., 2018, 44(1): 62-65. (in Chinese with English abstract)
[86] 刘文强, 李小湘, 黎用朝, 潘孝武, 盛新年, 段永红. 分子标记辅助选择改良优质稻湘晚籼13号的稻瘟病抗性. 分子植物育种, 2017, 15(8): 3063-3069.
Liu W Q, Li X X, Li Y C, Pan X W, Sheng X N, Duan Y H. Improvement of rice blast resistance of Xiangzaoxian No. 13 with high quality by molecular marker-assisted selection., 2017, 15(8): 3063-3069. (in Chinese with English abstract)
Recent Progress in Molecular Mechanism of Rice Blast Resistance
CAO Ni, CHEN Yuan, JI Zhijuan, ZENG Yuxiang, YANG Changdeng*, LIANG Yan*
(,,,;Corresponding author, E-mail: ycd311400@ 163.com;)
Rice blast disease, caused by, threatens global food security. Owning to the rapid evolution ofisolates, resistant cultivars always become susceptible in 3-5 years. Breeding and planting durable resistant cultivars is the most effective method. Recent advances in understanding the pathogenesis ofand rice resistance mechanisms led to a deeper understanding of PAMPs- and effector- triggered immunity in this pathosystem. This review summarizes the recent progresses for PTI, the cloned rice blast R genes, cloned Avr genes ofand the interaction between them. We also discussed some of the major unanswered questions for this pathosystem and the opportunities for future investigations.
rice; rice blast; disease resistance gene; avirulence gene; molecular mechanism
S435.111.4+1; S511.034
A
1001-7216(2019)06-0489-10
10.16819/j.1001-7216.2019.8126
2018-11-22;
2019-03-23。
农业部西南作物有害生物综合治理重点实验室开放基金资助项目(XNYH2016-1);国家自然科学基金资助项目(31801681);浙江省自然科学基金青年基金资助项目(LQ17C130005)。
