基于双荧光素酶报告基因系统的人源SREBP1基因启动子的活性研究
2019-10-11杨念李琛李洪亮郑燕房树华汪选斌赵雅玮
杨念,李琛,李洪亮,郑燕,房树华,汪选斌△,赵雅玮△
脂代谢重新编程是肿瘤发生发展的重要机制之一。固醇调节元件结合蛋白1(SREBP1)是含有bHLH-Zip 结构的转录因子[1],可调控内源性胆固醇、脂肪酸、三酰甘油和磷脂合成所需酶的表达,以维持血脂动态平衡[2]。研究发现,SREBP1的过度表达与某些恶性肿瘤的发生发展密切相关,在脂肪酸合成活跃的组织器官,如肝脏[3]、乳腺[4]、前列腺[5],其发生肿瘤之后的恶性程度与SREBP1的表达增加有关。本研究利用生物信息学预测、分析并构建SREBP1基因启动子双荧光素酶报告基因载体,以期为研究针对SREBP1靶点的天然活性抑制剂筛选奠定基础。
1 材料与方法
1.1 材料 限制性内切酶KpnⅠ和XhoⅠ、RPMI 1640 培养基、Opti-MEM 和lipofectamine 3 000 转染试剂购自美国Thermo Fisher Scientific 公司,DNA marker 和感受态细胞Trans1-T1购自北京全式金生物公司,KOD-Plus-Neo 购自日本TOYOBO 公司,Gibson Assembly Master Mix 购自美国NEB公司,细胞基因组提取试剂盒、胶回收试剂盒和去内毒素质粒抽提试剂盒购自美国Omega公司,双荧光素酶报告基因检测试剂盒购自美国Promega 公司,新生牛血清购自杭州天杭四季青公司,人肝癌细胞系HepG2由湖北医药学院附属人民医院肿瘤中心中药药理实验室保存,质粒pGL3-Basic 和pRL-SV40 由湖北医药学院郭兴荣副教授惠赠,大黄素购自上海陶素生化公司(批号T2869),引物委托上海生工生物工程公司合成,序列测定委托武汉擎科创新生物科技有限公司完成。
1.2 方法
1.2.1 分析软件 选取SREBP1(Ensembl:ENSG00000072310)基因起始密码子上游5 000 bp 序列,利用在线软件NNPP、Prometer SCAN、TSSG、TSSW 和Nsite 在默认条件下对SREBP1基因启动子序列及其转录因子结合位点进行预测分析。 分别在CpGFinder、MethPrimer、MethPrimer 2.0 和CpGPlot 四个软件上传获取的基因序列,按照默认条件进行CpG 岛预测分析。将Cister 软件阈值设置为0.1,对SREBP1基因顺式元件进行预测,并得出相应结果。本研究使用的在线软件见表1。
1.2.2 HepG2 细胞培养 HepG2 细胞株用含10%胎牛血清的RPMI 1640 培养基在37 ℃、5%CO2及饱和湿度条件下培养,根据细胞生长情况进行适当换液传代。
1.2.3 pGL3-SREBP1-pro 启动子载体构建 培养HepG2 细胞,按照试剂盒说明书抽提HepG2 细胞基因组。以基因组DNA 为模板,PCR 扩增SREBP1基因启动子,扩增片断约2 000 bp(引物序列见表2)。50 μL PCR 反应体系:10×PCR Buffer for KOD-Plus-Neo 5 μL、2 mmol/L dNTPs 5 μL、25 mmol/L MgSO43 μL、上下游引物(10 μmol/L)各1.5 μL、KODPlus-Neo 1 μL、基因组DNA模板5 μL、ddH2O 28 μL。PCR反应条件:94 ℃预变性2 min;98 ℃变性10 s,63 ℃退火30 s,68 ℃延伸1 min,35个循环;68 ℃延伸10 min。PCR产物电泳检测,用DNA 凝胶回收试剂盒纯化回收后,-20 ℃保存。用KpnⅠ、XhoⅠ双酶切质粒pGL3-Basic,50 μL 酶切反应体系:10×Buffer 5 μL、质粒5 μg、KpnⅠ0.5 μL、XhoⅠ0.5 μL、ddH2O补足至50 μL。37 ℃酶切1.5 h后终止反应,酶切产物用DNA凝胶试剂盒纯化回收后,-20 ℃保存。Gibson Assembly 是一种基于双链DNA 分子同源序进行无缝拼接的DNA 连接方式。其中克隆的DNA片段与预处理好的pGL3-Basic载体两末端具有相互重叠的核酸序列,以保证DNA片段按正确的顺序进行拼接,再将这些片段直接加入混合酶(T5核酸外切酶、DNA合成酶、DNA连接酶)中,一步完成无缝拼接,流程图见图1。连接体系为10 μL:载体2 μL,克隆片段2 μL,Gibson Assembly Mix 6 μL,50 ℃反应1 h。连接产物经转化Trans1-T1 感受态细胞,挑阳性菌落接种于20 mL 氨苄抗性的LB 培养基中,37 ℃、250 r/min振荡培养过夜后,提取质粒进行酶切鉴定,测序正确后命名为pGL3-SREBP1-pro。

Tab.1 Bioinformatics online software used in this study表1 本研究使用的生物信息学在线软件

Tab.2 The amplification primer of SREBP1 promoter表2 SREBP1基因启动子扩增引物
1.2.4 双荧光素酶报告基因的检测 常规培养肝癌HepG2细胞,设置空载组(pGL3-Basic+pRL-SV40)、携带报告基因载体的对照组(pGL3-SREBP1-pro+pRL-SV40)及添加天然活性抑制剂大黄素组(大黄素+pGL3-SREBP1-pro+pRLSV40)。转染前1 d 将5×105/mL 的HepG2 细胞接种至6 孔板培养过夜,第2 天细胞密度达70%~90%时,在转染前约1 h换新鲜培养基,天然活性抑制剂大黄素组加入含大黄素的培养基(大黄素终浓度100 μmol/L)。按照Lipofectamine 3 000试剂盒要求配制转染液,每孔加2.5 μg质粒(2 475 ng目的载体和25 ng 内参载体pRL-SV40)。室温孵育转染混合液5 min 后加入含有细胞的培养板培养24 h,用PBS 洗2 次,加入1×PLB(passive lysis buffer)室温摇动培养板裂解15 min,取上清,13 000 r/min 离心15 s;在96 孔酶标板中加入50 μL LARⅡ试剂,再加入10 μL细胞裂解液上清,立即放入酶标仪混匀并读取萤火虫荧光酶活性读数,随后立刻向反应液中加入50 μL Stop & GoBuffer(萤火虫荧光素酶终止液和海肾荧光素酶的底物)立即放入酶标仪混匀并读取海肾荧光素酶活性读数。每个实验组重复3次,取平均值,每个样品的萤火虫荧光酶数值与海肾荧光素酶数值的比值即为荧光素酶表达强度。
1.3 统计学方法 应用SPSS 21.0 统计学软件进行数据分析,符合正态分布的计量资料以均数±标准差(±s)表示,多组间比较采用单因素方差分析,组间多重比较采用LSD-t检验。以P<0.05为差异有统计学意义。
2 结果
2.1 SREBP1生物信息分析
2.1.1SREBP1启动子区特征序列及其转录因子结合位点预测结果 在SREBP1基因起始密码子上游5 000 bp 序列,NNPP 和Prometer SCAN 两个软件均预测存在5 个启动子特征序列,见表3。TSSG 结果显示,有2 个转录因子结合位点,分别位于1 810 bp处和4 955 bp 处。TSSW 结果显示,有3 个转录起始位点在1 838 bp、2 587 bp 和4 936 bp 处,2 555 bp 处有TATA 盒。Nsite 预测有17 个转录因子结合位点,见表4。
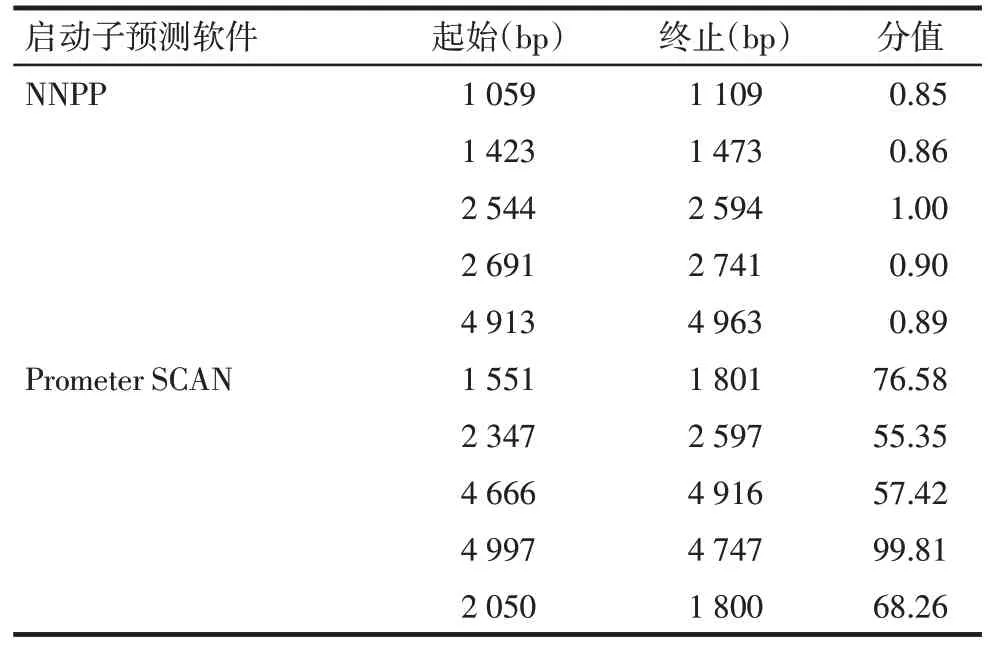
Tab.3 The analysis results of the NNPP and Prometer SCAN predicting SREBP1 promoter表3 NNPP和Prometer SCAN软件预测SREBP1基因启动子分析结果
2.1.2SREBP1基因CpG 岛分析 CpGFinder、MethPrimer、MethPrimer 2.0 和CpGPlot4 个软件预测SREBP1基因分别有1 个、4 个、3 个、2 个CpG 岛,结果见表5。
2.1.3SREBP1可能的功能性顺式元件 Cister软件对SREBP1基因顺式元件的预测结果见图2。共有49 个可能的功能性顺式元件,主要集中在SREBP1上游5´端约1 000~2 000 bp 和4 000~5 000 bp 的位置。
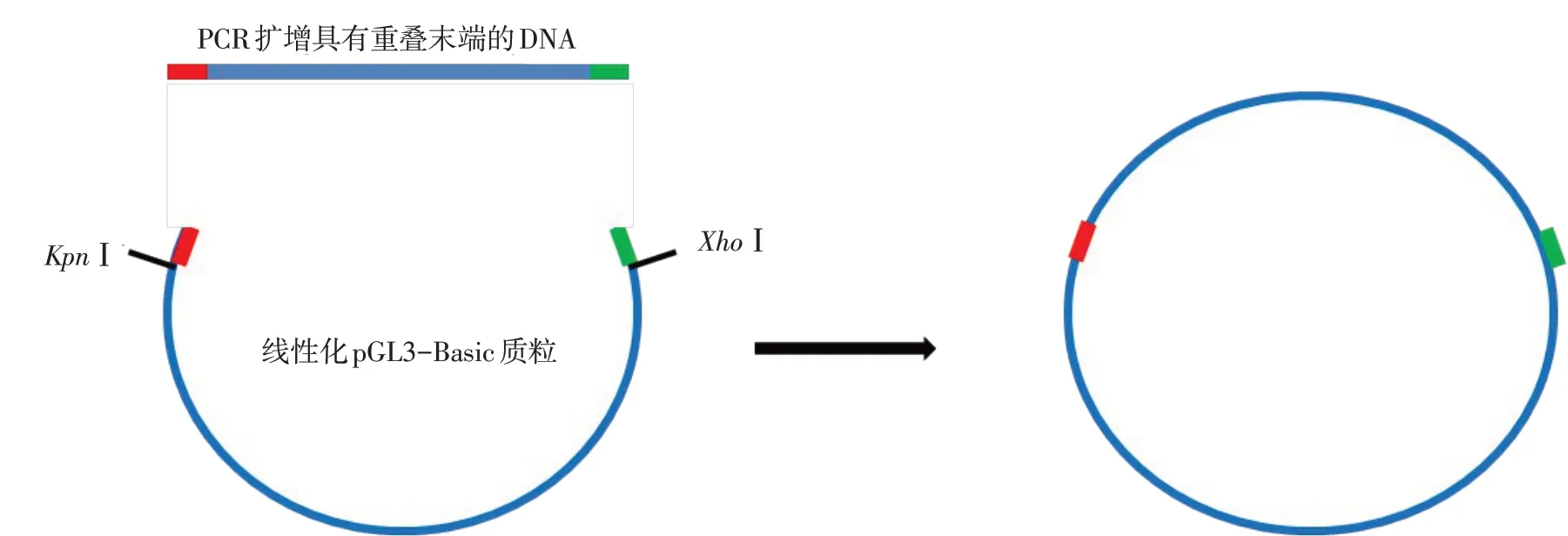
Fig.1 The flowchart of pGL3-SREBP1-pro vector constructed by Gibson Assembly图1 Gibson Assembly构建pGL3-SREBP1-pro载体流程图

Tab.4 The analysis results of the Nsite predicting SREBP1 transcription factor binding sites表4 Nsite软件预测SREBP1基因转录因子结合位点分析结果
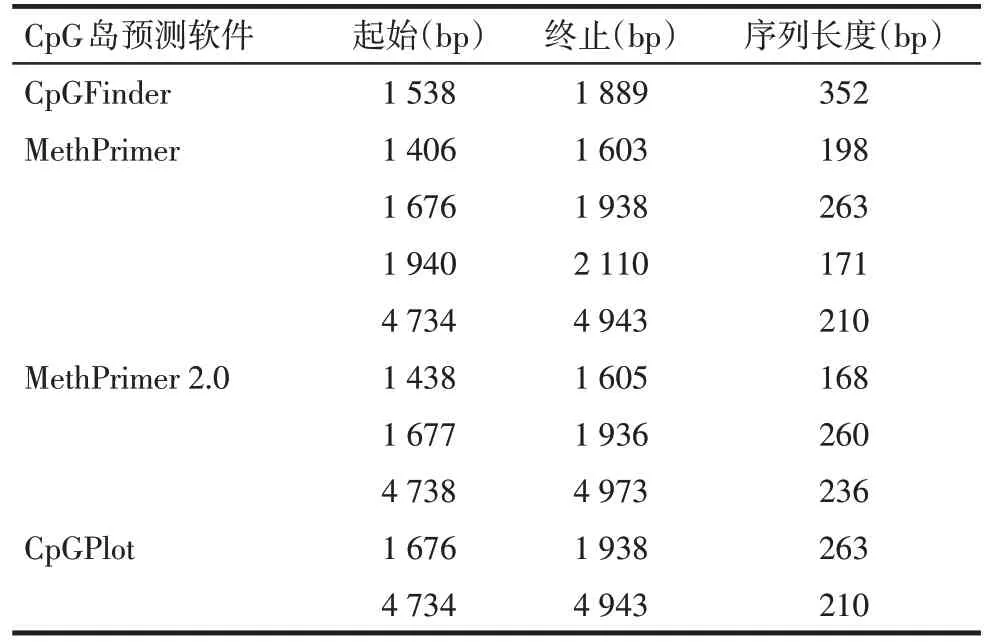
Tab.5 The prediction results of SREBP1 CpG Island表5 SREBP1基因CpG岛预测结果
2.2SREBP1启动子双荧光素酶报告基因载体的构建结果 根据生物信息学的分析结果,发现SREBP1启动子的转录结合位点,CpG 岛及顺式作用元件主要集中在前2 000 bp的位置,因此选取长度为2 036 bp的SREBP1启动子开展后续试验。以HepG2细胞基因组DNA为模板,扩增SREBP1的启动子,扩增片断约2 000 bp(图3)。Gibson Assembly 拼接PCR 片断和线性化pGL3-Basic 载体,构建pGL3-SREBP1-pro启动子载体,pGL3-SREBP1-pro酶切鉴定结果见图4。构建载体测序后证实序列与人源性SREBP1启动子区序列相一致。
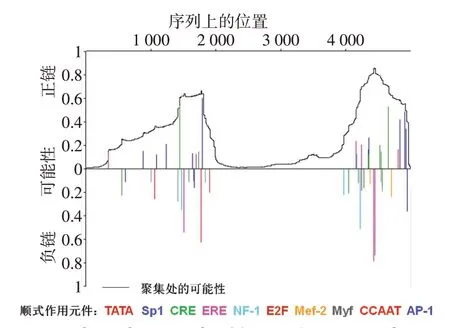
Fig.2 The prediction results of Cister to SREBP1 cis-element图2 Cister对SREBP1基因顺式元件的预测结果
2.3 大黄素对SREBP1启动子双荧光素酶报告基因转录活性的影响 结果显示,3组间荧光素酶活性差异有统计学意义(n=3,F=488.647,P<0.001),pGL3-SREBP1-pro组荧光素酶活性显著高于pGL3-Basic 组(3.124 5±0.206 3vs.0.020 3±0.001 9,P<0.001),所构建的SREBP1双荧光素酶报告基因具有启动子活性。大黄素给药组荧光素酶活性(1.452 6±0.043 7)显著低于pGL3-SREBP1-pro 组(P<0.001),大黄素能明显抑制SREBP1的转录活性。
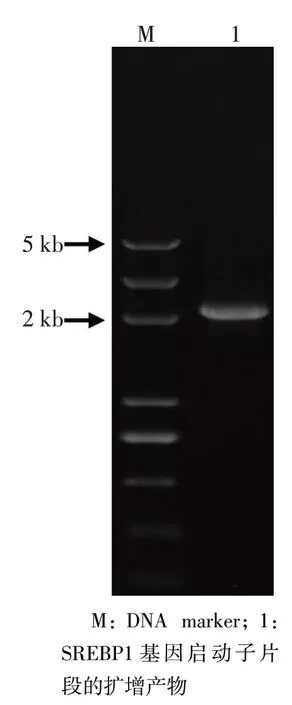
Fig.3 The PCR product of SREBP1 promoter图3 SREBP1启动子PCR产物
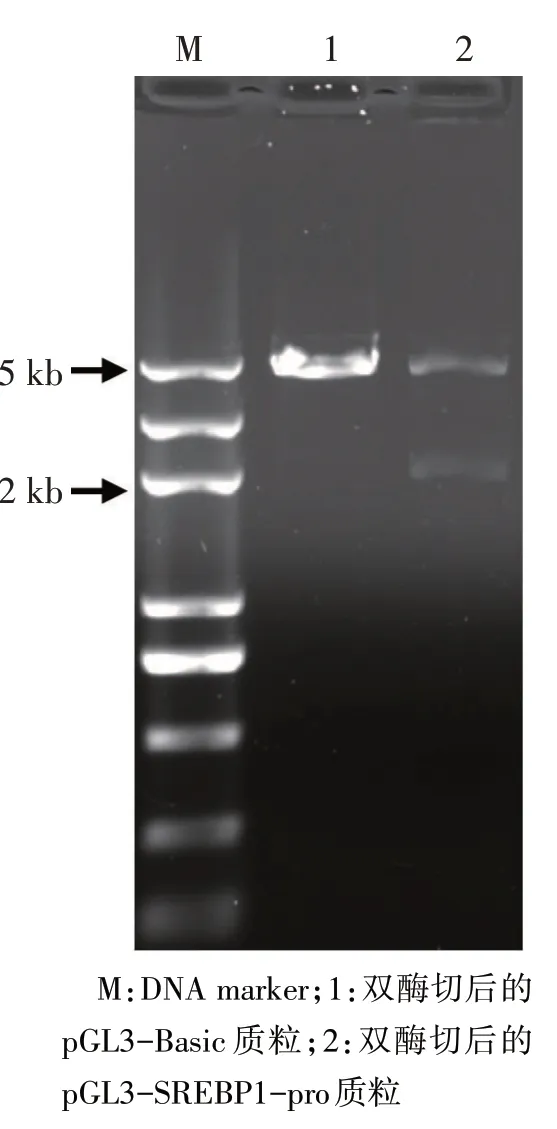
Fig.4 The double enzyme cutting identification result of recombinant plasmid图4 重组质粒双酶切鉴定
3 讨论
3.1 SREBP1是潜在的癌症治疗新靶点 脂代谢的改变是影响肿瘤生长、生存和对抗药性治疗的关键因素。肿瘤病人组织SREBP1 表达水平增高,SREBP1 所调节的脂肪生成基因转录激活。因此,SREBP1是理想的潜在癌症治疗靶点。
3.2 生物信息学方法是预测SREBP1启动子的重要手段 启动子是调控基因表达的重要组成部分,利用生物信息学软件对启动子进行分析和预测,可为后续研究指明方向。为搜寻启动子区域,本研究分别采用5 种不同的启动子分析软件对SREBP1基因启动子区域进行预测,结果发现该基因至少存在2个启动子,且存在TATA 盒。虽然由于各预测软件的原理不同可能导致预测结果有一定偏差,但基本可以确定启动子存在于SREBP1基因的5´非编码区上游。CpG岛通常位于基因启动子的核心序列和转录起始点附近。本研究应用在线软件CpGFinder、MethPrimer、MethPrimer 2.0 和CpGPlot 在SREBP1上游启动子区域2 000 bp的位置均发现CpG岛。转录因子亦称为反式作用因子,通过与顺式作用元件(即转录因子结合位点)结合才能有效激活基因转录,启动子调控元件的鉴定有助于寻找相应的转录因子。Cister 软件分析出SREBP1基因启动子转录区5 000 bp片段中有49个可能的功能性顺式元件,因此后续构建系列截短荧光素酶表达载体时要充分参考此结果,不能中断这些转录因子结合位点。本研究运用生物信息学预测SREBP1启动子,有效地从基因组中查找已知及末知的转录调控元件,为研究基因的结构和功能提供了可靠依据。
3.3 双荧光素酶报告基因可有效检测SREBP1活性 双荧光素酶报告基因是研究基因表达调控和基因工程的重要手段之一,荧光素酶的表达量与荧光的强度相关,可作为真核细胞中基因表达量及启动子活性的测定手段。笔者根据SREBP1基因生物信息学分析得到的结果,采用新型无缝克隆技术Gibson Assembly 方法成功构建了SREBP1启动子双荧光素酶报告基因载体,此方法较传统的酶切连接、同源重组方法更为高效、简单、多变,提高了构建成功率,为进一步研究SREBP1基因启动活性及转录调控元件、探讨基因表达调控机制提供了实验依据。
3.4SREBP1转录活性检测为寻找肿瘤脂代谢新靶点提供技术平台 目前,中药、天然产物抗肿瘤己经成为国内外研究的热点。已有研究表明,中药能够抑制肿瘤脂代谢和SREBP1转录活性[7]。利用SREBP1转录活性检测,可为寻找肿瘤脂代谢新靶点提供技术平台。同时,大黄素作为天然活性抑制剂抑制了SREBP1转录活性,与笔者前期发现的大黄素能够抑制SREBP1mRNA 和蛋白水平表达的结论一致[6],对相应的SREBP1天然抑制剂的开发和利用具有重要意义。
