基于Oncomine数据库及生物信息学方法挖掘UBE2C基因在卵巢癌中的表达及作用机制
2019-07-17殷雪琴倪娜张琴冷天艳杨丽华
殷雪琴,倪娜,张琴,冷天艳,杨丽华
卵巢癌是妇科常见的恶性肿瘤之一,其死亡率高居女性生殖道恶性肿瘤之首,病理类型/分子类型均复杂,病理类型以上皮性卵巢癌为主[1]。目前研究发现上皮性卵巢癌的发生和发展与多种基因的差异 表 达 有 关[2]。 泛 素 结 合 酶 E2C(Ubiquitin-Conjugating Enzyme E2C,UBE2C)基因是一种蛋白质编码基因,其相关通路与细胞周期、有丝分裂等有关,并可能参与癌症的恶化。UBE2C 基因在健康组织中表达较低,而在多种癌组织中呈高表达,包括胃癌[3]、乳腺癌[4]、甲状腺癌[5]、肺癌[6]、结直肠癌[7]和食管癌[8]等。此外,已有文献表明UBE2C 的高表达也与肿瘤高度恶性的表型和较差的存活率有关[3-4,6-9],然而目前少见其在卵巢癌中的作用及机制研究。
Oncomine 数据库是目前全球最大的癌基因芯片数据库和整合数据挖掘平台,旨在挖掘癌症基因信息[10]。本研究通过收集Oncomine 数据库中的公共数据集,挖掘UBE2C 基因在卵巢癌组织中的表达,通过Kaplan-Meier 分析了解UBE2C 表达与卵巢癌患者生存的关系,并结合Genecard 数据库,分析UBE2C相关蛋白网络图,探讨其作用机制。
1 资料与方法
1.1 Oncomine 数据库基因信息的提取 登陆在线数据库Oncomine(http://www.oncomine.org),进行用户注册,随后在数据库中设置用来筛选和提取目的基因相关数据的筛选条件。其筛选条件依次设定为(1)Gene:UBE2C。(2)Analysis Type:Ovarian Cancervs.Normal Analysis。(3)Threshold By:P<0.05;Fold Change:All;Gene Rank:All。挖掘数据集中关于UBE2C基因的表达信息(临界值设定:P<0.05),结果以三线表展示。
1.2 患者生存分析 利用Kaplan-Meier Plotter 数据库(http://kmplot.com/analysis/)对UBE2C在卵巢癌患者数据信息中进行生存分析。进入数据库主页,设置限定条件为(1)Cancer:Ovarian cancer。(2)Gene:UBE2C(Affymetrix ID:202954_at)。(3)Survival:PFS、OS。
1.3 使用Genecards 网站分析UBE2C 基因相关蛋白功能及途径富集 利用Genecards(https://www.genecards.org)查找UBE2C 基因相关信息和相关蛋白,通过STRING(https://string-db.org/cgi/input.pl)构建UBE2C 基因的相关蛋白网络图,挖掘出该基因相关蛋白的富集分析。
1.4 统计学方法 生存分析采用Kaplan-Meier 和Log-rank检验法,P<0.05为差异有统计学意义。
2 结果
2.1 UBE2C 基因在卵巢癌中的表达情况 通过在Oncomine数据库页面设置限定条件后,对UBE2C进行分析,共有14 项研究,包括2 212 例标本,分别发表 于 Br J Cancer、Cancer Res、Clin Cancer Res、Cancer Sci,Proc Natl Acad Sci 等。分析显示,UBE2C基因在卵巢癌组织相对于正常组织呈现高表达,差异有统计学意义(P<0.05)。继续挖掘Oncomine 数据库中各卵巢癌数据集中UBE2C基因表达情况,发现在不同的基因芯片数据集中,卵巢癌组织中的UBE2C 基因较正常卵巢组织均呈高表达,差异有统计学意义(P<0.05),见表1。
2.2 Kaplan-Meier Plotter 数据库分析结果 UBE2C基因表达水平与卵巢癌患者预后密切相关,高表达组总生存期(OS)、无病生存期(PFS)均明显短于低表达组(P<0.05),见图1。
2.3 UBE2C 基因主要相关蛋白网络图 通过Genecards 分析UBE2C 基因主要相关蛋白有RLIM、VFLI、TCEB2、ASB2、UBE2Q2、FBXW9、UBR1、FBXL18、CUL7、FBX06、S1AH2、TRIM39、FBXO9、RNF19A、BTRC、CBX4、HIST1H2AJ、HISST1H2BD、CCNB1、CCNA2、PSMD7、SHFM1、PSMD11、PSMD14、PSM1311。通过STRING 构建获得UBE2C 相关蛋白网络图,见图2。将以上相关蛋白进行富集分析,主要富集在泛素依赖蛋白的分解代谢过程、蛋白质分解代谢过程、蛋白质泛素化等生理过程,见表2。
3 讨论
泛素依赖性蛋白的水解与不同的细胞进程密切相关,包括细胞周期过程、信号转导和分化。在这个系统中,底物蛋白通过3种不同的酶进行降解,其中包括泛素激活酶(E1)、泛素共轭酶(E2)和泛素连接酶(E3)。而UBE2C(也称UbcH10)是E2基因家族的一员,编码19.6 ku 的蛋白,参与泛素依赖性蛋白的水解。有研究报道UBE2C 是后期促进复合物/环状体(APC/C)的成员之一,可促进中期/后期转换期间沿细胞周期进程的几个靶蛋白的降解,特别是有丝分裂周期蛋白(如Cyclin B),从而参与调控细胞周期M 阶段[8]。Okamoto 等[11]证明 UBE2C 在许多正常组织中表达水平极低,但在胃[3]、甲状腺[5]、乳腺[4]、
肺[6]、食管[8]、胰腺[9]等多种人类实体癌症中UBE2C表达均异常增高。UBE2C 的表达与某些肿瘤的侵袭深度和肿瘤淋巴结转移期呈正相关,表明UBE2C具有促进细胞增殖和侵袭的能力[3]。有研究证明UBE2C 转基因小鼠容易诱发肺部肿瘤和广泛的自发性肿瘤,提示UBE2C在人类多种实体肿瘤中可能发挥着癌基因的作用[12]。但目前关于UBE2C 基因在卵巢癌中的作用机制尚不清楚。
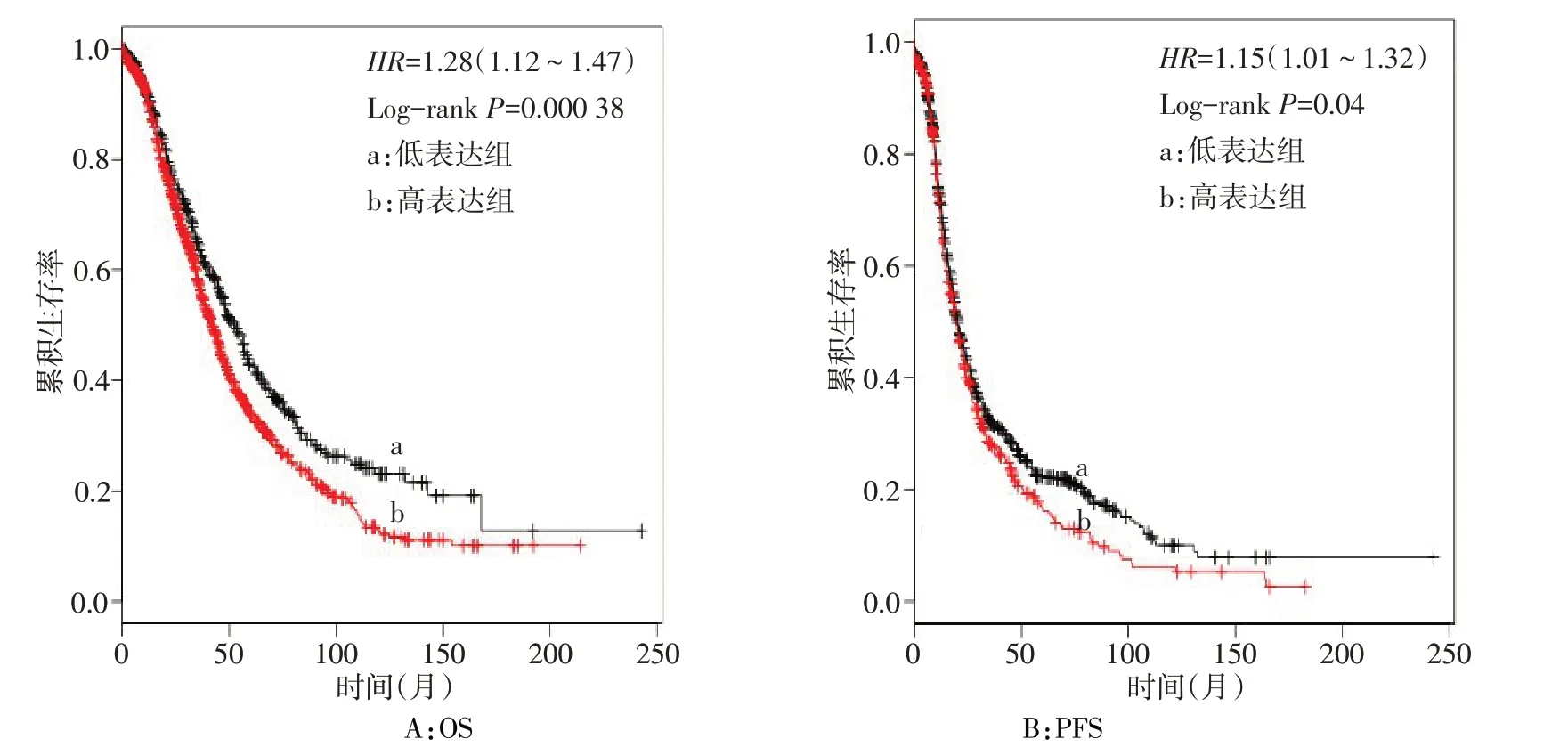
Fig.1 The relationship between the prognosis of ovarian cancer and UBE2C expression图1 人UBE2C基因表达水平与卵巢癌患者的预后关系
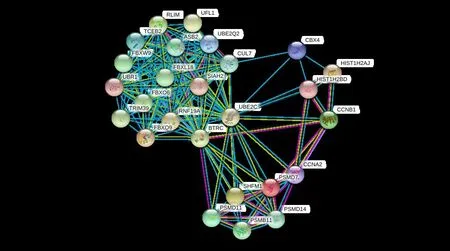
Fig.2 Network diagram of UBE2C related proteins图2 UBE2C相关蛋白网络关系图

Tab.2 Analysis of biological process enrichment of UBE2C related proteins表2 UBE2C相关蛋白参与的富集过程分析
本研究通过挖掘Oncomine 数据库中多个数据集组成的大样本分析UBE2C 在卵巢癌中的表达情况,以此避免因样本量过小、人群种族差异和研究方法等其他因素而导致结论的偏颇。本研究显示UBE2C 基因在卵巢癌组织中的表达明显高于正常组织,通过Kaplan-Meier 法分析了UBE2C 基因的生存数据,发现UBE2C基因表达水平对卵巢癌患者的总生存期存在明显的影响,UBE2C 基因高表达组无病生存期、总生存期均明显缩短,UBE2C 基因低表达患者预后可能会更好。在此基础上进一步通过Genecards挖掘到该基因的相关蛋白,并对这些相关蛋白进行富集分析,发现主要富集在泛素依赖蛋白的分解代谢过程、有丝分裂细胞周期的调控等生理过程。综合以上研究结果和文献报道,笔者推测高表达的UBE2C基因通过调节泛素依赖蛋白代谢,在卵巢癌细胞中破坏有丝分裂细胞周期蛋白(如Cyclin B),导致细胞退出有丝分裂,迅速进入下一周期,加速癌细胞的增殖,从而发挥癌基因的作用。UBE2C 基因高表达可能为提示卵巢癌患者预后较差的一个指标,靶向UBE2C可能是一个潜在的肿瘤诊断和治疗工具。
总之,通过对Oncomine、Kaplan-Meier Plotter 和Genecards在线数据库的挖掘和STRING等软件的应用来对大量的标本数据进行分析的方法,相较于传统分析方法,可以减少数据下载和处理的繁杂程序,更高效、便捷地为研究者利用大数据探索研究方向提供生物学证据,也为进一步探索UBE2C基因与卵巢癌的关系奠定了基础。
