网络药理学研究策略在组合药物研究中的应用
2018-04-26李玮婕郭秋岩张彦琼卢建秋
李玮婕,毛 霞,郭秋岩,张彦琼,卢建秋,林 娜
(1中国中医科学院中药研究所,北京100700;2北京中医药大学中药学院,北京100102)
0 引言
组合药物(drug combination)通常是为了达到综合治疗某一疾病的目的,将两个或多个作用机制明确、作用靶点不同的药物或药效成分按照合理的剂量配伍后一起或顺次给患者服用的药物[1]。研究[1]表明,通过单一靶点治疗疾病的传统治疗方法,作用靶点相对明确,药理机制相对清晰,但药理效应指标相对单一,忽略了人体的整体性,导致其毒副作用大,长期服用出现抗药性等问题。随着人们对医药卫生要求的提高,复杂疾病发病率的升高,以及单一药物的治疗弊端的出现,使得组合药物发展更为迫切。幸运的是,近年来组合药物发展并应用于多种复杂疾病领域,尤其在癌症、艾滋病、白血病、糖尿病、复杂脑部疾病等。
1.1 组合药物在西药研究中的应用 西药多数是通过单一靶点作用于人体从而达到治疗疾病的目的,其作用机制相对明确,药效物质清晰,但毒副作用较大,对复杂疾病的治疗有一定的局限性。组合药物的开发和应用有效地弥补了西药在临床应用中的缺陷。食品药品监督管理局(the food and drug administration,FDA)在过去5年中已经批准了越来越多组合药物作为成分明确剂量固定的商业产品进行销售,尤其针对心血管疾病、Ⅱ型糖尿病、艾滋病、呼吸系统疾病、神经退行性疾病、癌症等多种复杂疾病。临床研究表明,组合药物的疗效与药物单独使用相比,具有较好的治疗效果,并且毒副作用较少。
在艾滋病治疗(HIV)领域,2006年7月FDA批准Atripla作为治疗艾滋病病毒感染“鸡尾酒疗法”(the cocktail therapy)组合药物上市销售,是由分别在1998年、2001年和2003年被FDA批准上市的依发韦仑(efavirenz, Sustiva)、替诺福韦酯(tenofovir disoproxil fumarate, Viread)和恩曲他滨(emtricitabine,Emtriva)三种药物成分组成[2]。 在临床试验中,244名患者中80%的人在使用Atripla后HIV病毒显著减少,且正常CD4细胞的数量明显增加;组合药物Epzicom中的两种成分拉米夫定和阿巴卡韦都是逆转录酶抑制剂,有抑制 HIV增加、抗免疫力减弱的作用[3];由Viread和Emtriva组成的组合药物特鲁瓦达(Truvada)为抗逆转录病毒药,通过抑制病毒逆转录酶达到抑制HIV复制的目的[3]。
在癌症治疗领域,曲妥珠单抗与渥曼青霉素协同治疗乳腺癌[4];由Palbociclib与曲来唑组合而成的IBRANCE显著提高了Palbociclib的抗肿瘤活性[5];FDA于2014年1月对BRAF V600E或V600K突变的黑色素瘤进行了第一次组合治疗[6],采用达拉非尼(Dabrafenib)和曲美替尼(Trametinib)的组合药物安全性显著提高,尚未观察到典型的MEK抑制剂毒性,同时与BRAF抑制剂相关的毒性很弱。特别地,早期患者的治疗结果表明,达拉非尼和曲美替尼的组合药物可以抑制与皮肤鳞状细胞癌相关的病变。到目前为止,FDA已经批准了适当的药物组合,以不同的剂量治疗不同的癌症亚型。此外,FDA特批了由阿托品和普拉德肟组合而成的DuoDote,治疗由有机磷神经毒剂和有机磷杀虫剂引起的中毒[7]。
以上临床研究显示,组合药物并不是简单的药效增加,而是发挥药物的协同性,减轻单独使用的毒副作用,增加安全性,在一定程度上减轻患者的病痛,提高临床治愈率。
1.2 组合药物在中药研究中的应用 中医基本理论以辨证论治和整体观念为主要内容,认为人体是一个有机的整体,与自然界有着密切的联系。中药在中医理论的指导下,具有多途径、多靶点的作用特点,这与组合药物的治疗原理相一致。因此,组合药物在中药领域也取得了一定的进展。
脑缺血[8]是由多个途径相互作用引起的高度复杂的疾病,但以往的研究局限于单个基因或单个途径层面。彭世陆等[9]探究了小檗碱、黄芩苷和栀子苷的组合药物及黄连解毒汤复方对大鼠脑组织小檗碱药动学的影响,利用UPLC-MS法测定小檗碱在脑组织中的浓度,发现与小檗碱单体给药相比,组合药物促进小檗碱进入脑组织,黄连解毒汤则抑制小檗碱进入脑组织。这一结果表明复方在治疗脑部疾病时并非各单味药主要药效成分的简单加和,这一发现为后期针对脑部疾病的中药复合粒子剂型设计和新药研发奠定基础。Zhang等[10]基于药代动力学和独创性通路分析(the ingenuity pathway analysis,IPA)系统揭示黄芩苷和栀子苷的组合药物治疗脑缺血的加性机制(addition mechanism),研究表明其组合效果优于黄芩苷或栀子苷单药。临床用人参皂甙Rb1,红细胞生成素和五味子素的药物组合治疗心血管疾病具有很好的疗效[11]。 梁晨等[12]分析粉防己碱、三氧化二砷联合作用于MDA-MB-435S及HCC1937乳腺癌细胞;两药联合使用后细胞形态发生改变,细胞凋亡率增加,Caspase-3、bax、bid相关基因的表达升高,同时Bcl-2、suivivin调控基因的表达降低(表1)。
尽管组合药物临床疗效显著,在医药界有一定的发展,但由于组合药物的一些限制,例如组合药物的作用机制不明,如何设计和研发有效的协同药物组合等,使得目前已获得FDA批准的组合药物数量尚不多。
近年来,网络药理学的提出和发展为组合药物的研制和开发提供了强有力的理论和技术支撑。接下来,本文拟从网络药理学可用的数据资源、算法和软件,以及现有的组合药物研究方法等方面入手,简要概述网络药理学研究策略在组合药物发展中的应用。
2 网络药理学常用的数据资源、算法和软件
网络药理学是由Andrew L.Hopkins于2007年首次系统阐述,是一种建立在高通量组学数据分析、计算机虚拟计算及网络数据库检索基础上的新药发现、药物药效及作用机制挖掘的研究方法[33]。网络药理学基于“疾病-基因-靶点-药物”相互作用网络,系统综合地观察药物对疾病网络的干预与影响,从而揭示药物系统作用于人体的机制[34]。这一研究策略不仅与中医辩证的整体观念相一致,符合中医药对疾病整体性的认识,同时与组合药物多途径、多靶点的治疗原理不谋而合。网络药理学通过同时调控基因网络中的多个成分来模拟药物作用的影响[35],很好地揭示组合药物的作用机制。近年来,随着越来越多的国内外学者研究,网络药理学已成为药物机制研究、新药研发和组合药物个性化设计的新方法。多种相关数据库的构建、计算方法和软件的开发更是为组合药物作用机制研究和协同药物组合的发现提供了重要的支撑。
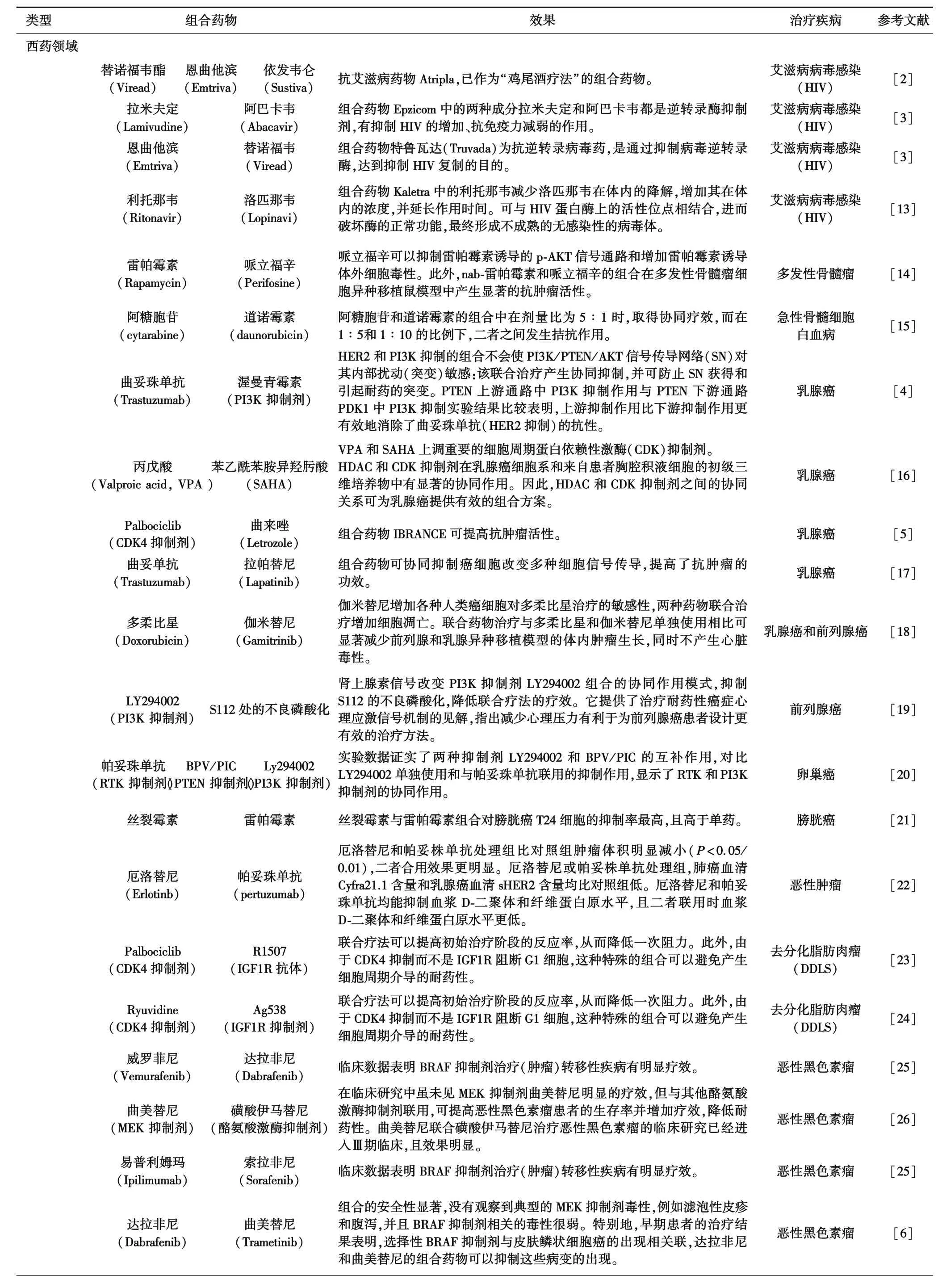
表1 组合药物在中西医临床中的应用
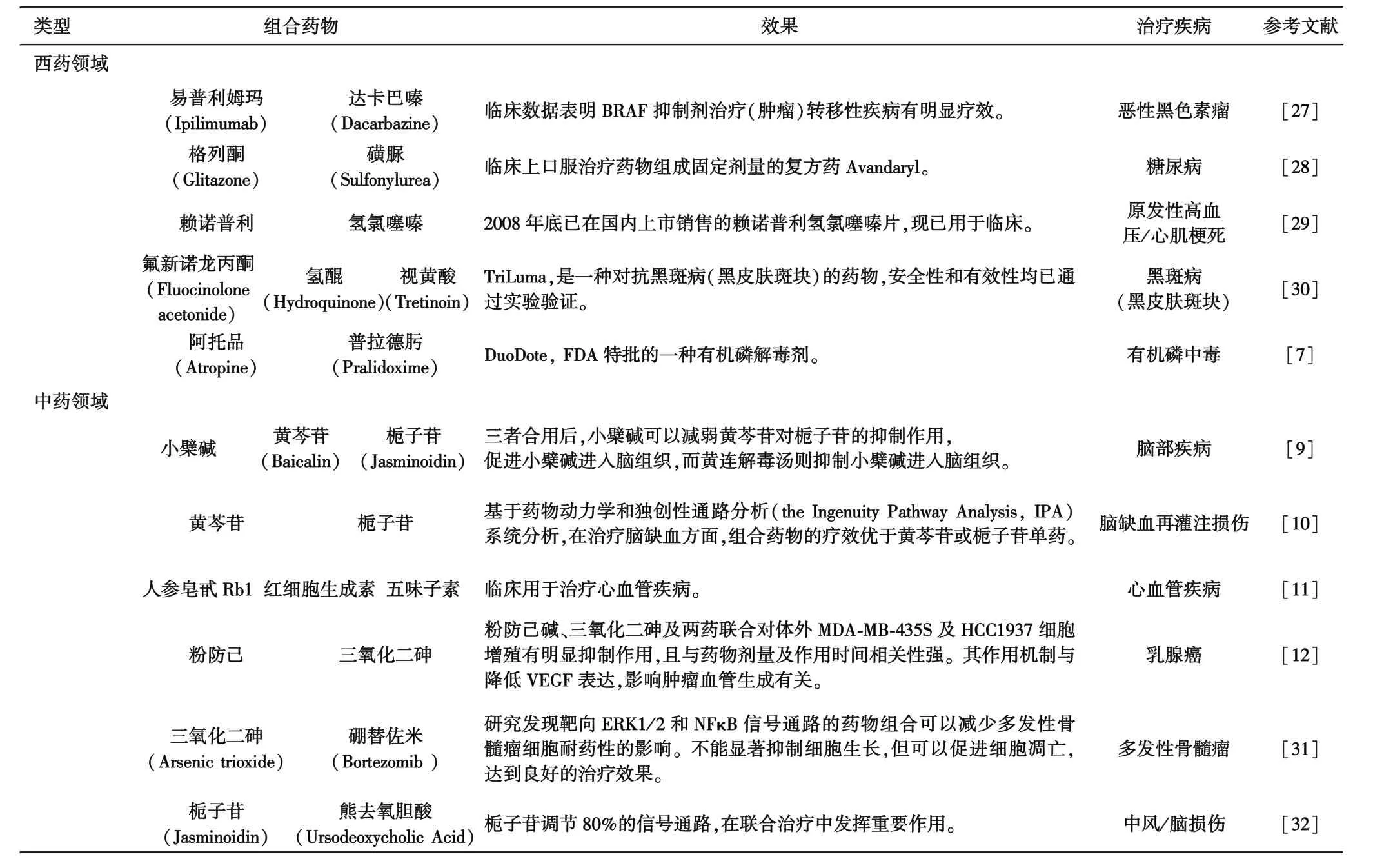
表 1(续)
2.1 网络药理学常用的数据资源 网络药理学常用的数据库包括药物化学成分数据库(DCDB,ATC/DDD,ChEMAL和PubChem)、中药化学数据库(TCM Database@ Taiwan,TCMID,HIT,TCMSP和 CHEMTCM)、药物靶标-靶标相互作用关系数据库(TTD,Drugbank和PubMed文献数据)、中医药生物信息数据库(HerbBioMap)、生物分子相互作用数据库(Biogrid, Dialogue for Reverse Engineering Assessments and Method,pharmgkb和String)、生物分子通路数据库(KEGG)、化合物蛋白质相互作用数据库(STITCH)、蛋白质相互作用信息数据库(HAPPI,Reactome, OPHID, InAct, HPRD, MINT, DIP 和PDZBase)、与疾病相关的信息数据库(ASDCD,Euresist project,GDSC,OMIM,dbNEI和SIDER)、与药物代谢动力学/药物效用动力学(pharmacokinetics/pharmacodynamics, PK/PD)相关的信息数据库(admetSAR和MetaADEDB)、与临床相关的信息数据库(FDA Orange Book, NIH/NCATSdata set, 美国政府临床实验登记网站)等。目前为止,多种相关数据库的构建为组合药物的发展提供有力的支持。
在目前版本的药物组合数据库(drug combination database,DCDB)中,共收集了1363种药物组合。DCDB提供了每个组合药物的测试活性、适应症、可能的作用机制、组分间的药物相互作用以及当前的发展状态等信息。在1813种组合药物用法中,1445种的临床有效性和安全性显著提高[36]。解剖治疗化学(anatomical therapeutic chemical,ATC)分类系统对治疗药物进行分类,获得治疗相似性,提高药物使用安全性[37]。
此外,台湾中医药数据库(TCM Database@Taiwan),TCMID,Herb Ingredients'Targets(HIT),中药系统药理学数据库和分析平台(TCMSP)和CHEM-TCM,收集中药的化学成分信息[38-42];Biogrid (Biological General Repository for Interaction Datasets)收集蛋白质和基因的相互作用数据[43];Dream 3(Dialogue for Reverse Engineering Assessments and Method),药物基因组学知识库(pharmgkb)和 String收集基因数据[44-46];TTD,PubMed 文献和 DrugBank 收集所有单药的药物信息以及单药对应的靶标信息[47]。
2.2 网络药理学常用的算法和软件 网络药理学基于上述生物学数据库及临床和实验研究结果,通过有效的算法和应用软件,在分析“疾病表型-基因-靶点-药物”相互作用网络的基础上,从系统生物学角度出发观察药物对病理网络的干预和影响,使新的组合药物更趋近于疾病的实际情况,实现复杂疾病的安全有效治疗,成为组合药物设计的重要手段。
在很多学者的努力下,网络药理学发展演绎了用于疾病基因预测(CIPHER和CIPHER-HIT)、药物靶标预测(drug CIPHER)、疾病分子机制挖掘(CSPN,sGSCA,ClustEx和 LMMA)及药物作用机制挖掘(comCIPHER,CMAP,DMIM,NADA 和DGPsubNet)和药物组合设计(NIMS,SAF和GIFT)等方面的算法。例如,模块化因子图(Modularized Factor Graph,http://ctsb.is.wfubmc.edu/publications/modularizedfactor-graph.php)是一种参数估计方法,提供模块化因子图的 RPPA实验数据和 Matlab源代码[32];CMAP (The Connectivity Map,https://portals.broadinstitute.org/cmap/) 是一种药物-基因-疾病的网络共模块分析方法,采用生物活性小分子和简单的模式匹配算法,通过常见基因表达变化的暂时性特征寻找药物、基因和疾病之间的功能连接[48]。 Batagelj等[49]开发了应用软件Pajek,用于疾病表型候选网络的发现。
这些网络药理学相关数据库的构建,相关算法和软件的开发和应用为组合药物的开发和药物相互作用的预测提供蛋白、基因、靶点、化学成分及相互作用等基本信息,同时为组合药物的设计提供方法和手段。
3 现有的组合药物研究方法
药物相互作用(drug-drug interactions,DDIs)是指两种或两种以上的药物同时应用时所发生的药效变化,即产生协同(增效)、相加(增加)、拮抗(减效)作用[50]。合理的药物相互作用可以增强疗效或降低药物不良反应,反之可导致疗效降低或毒性增加,还可能发生一些异常反应,干扰治疗,加重病情。随着人口老龄化的加重和一些慢性疾病的出现,药物相互作用的效果也受到越来越多的关注。尽管过去的几十年为开发新的组合药物做出很多努力,但临床使用的有效组合药物大多数是通过临床试验和经验从已批准的单药中筛选出可能的药物组合[51]。随着单药数量的增加,可能的组合药物数也会增加,但确定最佳和最有效的药物组合是一项费资耗时的工作。随着网络药理学、多向药理学、生物信息学、系统生物学等多学科的出现,多种可用于组合药物预测和发现的网络建模方法也不断涌现,为协同药物组合的预测提供重要指导方法。本文将从药物-药物相互作用、药物靶标间相互作用以及“药物-靶标-疾病-基因”多级相互作用三个层面介绍现有的组合药物研究方法(表2)。

表2 现有的组合药物研究方法

表2(续)

表2(续)

表2(续)

表2(续)
3.1 药物-药物相互作用 这类分析方法依据化合物结构相似性、功能相似性、综合(结构和功能)相似性、药效学和药代动力学(PK/PD)等来预测药物间的相互作用。如,Wang等[52]基于药物的治疗效果及药物相互作用提出了一种网络生物学方法——“药物鸡尾酒”网络(drug cocktail network)统计模型,研究药物组合及其靶蛋白在遗传相互作用网络和相关通路中的作用,以便了解有效药物组合的作用机制,从而发现潜在的协同作用药物。作者对比分子空间中的药物组合,尤其对遗传相互作用网络和靶蛋白通路中的药物相互作用进行探究,将有效药物组合和随机组合进行对比,发现组合药物的靶蛋白更趋向于具有遗传相互作用。
赵鹏姚等[53]通过对拓扑特性和分子机理的分析,发现了药物相互作用的网络药理学特征,同时还提出一种基于药物功能相似性的组合药物预测方法。该方法基于相似药物具有相似作用的假设,从药物靶点模块性和ATC结构的角度,通过相似性的测量和标准相互作用数据集的整合,预测新的相互作用药物对。采用试者工作特征曲线(receiver operating characteristic curve,ROC)评价该预测模型,得到基于药物ATC结构相似性和药物模块相似性方法的AUC值分别为0.95和0.92,表明该方法具有较高的准确性。但作者仅单独分析了药物相互作用与蛋白质相互作用网络、药物靶点、ATC结构和化学结构之间的关系,并没有将变量整合分析,也未将新得到的相互作用药物对与标准的药物对的药理学分类和临床效应相结合。
Cheng等[54]通过机器学习结合全面药品不良反应网络表型相似性,ATC治疗相似性,来自SMILES数据的化学结构相似性以及使用DrugBank和治疗靶标数据库建立的药物-靶标相互作用网络的基因组相似性,提出异构网络辅助推理(heterogeneous networkassisted inference,HNAI)框架对药物相互作用进行预测。这种利用药物综合相似性的预测方法可弥补利用单一特征变量进行预测的不足,且预测性能较好。然而,该方法缺少明确药物间无相互作用的标准;当药物结构或表型特征除去或无法获得时,模型的预测性能较差;模型使用的药物不良反应(adverse drug reactions,ADRs)关联网络和药物靶标间相互作用(drug-target interaction,DTI)网络都不完整;可能存在与每个相似性相关的附加限制,即2D和3D结构并不是单一对应的关系。上述局限性在Vilar等[55]的方法中得到改善,他们使用综合相似度测量,包括2D和3D分子结构、相互作用谱、靶标和副作用相似性,预测新型药物相互作用。但该方法的预测性能依赖于标准数据集的全面性,当分析初始数据库中未记录的药物或最新上市药物的相互作用时,模型预测性能将受到限制。同样,组合药物设计(combinatorial drug design,CDD)也存在对药物不良反应考虑不足的缺陷[56]。
Zhao等[50]基于已获得FDA批准的组合药物信息:药物靶蛋白、相关下游通路、医疗指示区以及来自ATC分类系统的治疗作用和副作用建立了一种模型;Li等[57]基于药物机构特征、ATC代码和靶标副作用药物相似性特征,以及靶标序列、PPI中靶标相互作用和GO语义(gene ontology,GO)靶标相似性特征建立 probability ensemble approach(PEA),使用Bayesian网络计算相似率(likelihood ratio,LR),将每个药物的以上六个特征组合,用于评估已知药物的相互作用的相似性;Chandrasekaran等[58]采用化学基因组学数据预测抗生素药物组合,作者首先实验测量了15个药物的105种相互作用;然后将测量所得的产生协同或拮抗作用的药物相互作用数据和化学基因组学数据结合,作为INDIGO(computational modelentitled INferring Drug Interactions using chemo-Genomicsand Orthology)的训练数据集;该方法仅需要个别药物的化学基因组学数据就可输出新的药物组合。这四种模型往往利用药物靶点、ATC代码和化学结构等药物性质,但药物协同作用的依赖性较强,病型和药物剂量往往可以调节药物的疗效,同时模型应该综合考虑药物性质和药物治疗相关信息,提高预测性能。
Takeda等[59]通过PD和PK相关网络,探究药物2D结构的相似性对其相互作用的影响,并以两种药物华法林和辛伐他汀为例,进行药物相互作用的预测。验证表明,这种逻辑回归模型可以有效预测药物相互作用,与靶标和酶相关的组合可以最大化地增加药物间相互作用的预测能力。然而,这种预测方法强烈依赖检查药物(a drug to be examined,De)或查询药物(a query drug,Dq)的酶或转运蛋白信息。另外,该方法仅适用于小分子药物(即不适用于肽或核酸),因此,整合其他方法可能是提高预测药物间相互作用性能的有效方式。
3.2 药物靶标间相互作用 这类方法依据蛋白质结构相似性(拓扑结构相似性)及基因相似性等来预测药物间相互作用或推断药物在体内的作用模式。程飞雄[60]提出药物相似性推理、靶标相似性推理、网络推理和加权网络推理方法,用于药物-靶标间相互作用预测和药物重定位研究,有效地发现药物的潜在靶标。另外,通过加权网络推理方法模拟研究,首次提出药物-靶标网络中的“弱相互作用假设”。这种方法具有一定的创新性,然而目前方法只考虑了静态药物-靶标网络性质,即主要考察药物-靶标网络的亲合力、选择性或杂泛性,忽略了药物时效性,即药物在靶部位的停留时间(target residence time)和药物-靶标结合动力学过程。
在组合靶标优化方面,Penrod等[61]采用基于差异共表达和帕累托优化(pareto optimization)的高通量转录组学数据预测药物诱导肿瘤抵抗的组合靶点。以这种方法,几种肿瘤必需基因被确定为抑制乳腺癌的可用药物靶标,同时将大量药效学数据和药物靶标结合,预测特定癌细胞的选择性靶点,最大限度地发挥抑癌作用。在网络建模方面,已有很多学者使用蛋白质-蛋白质相互作用网络来预测药物相互作用[63-64]。 如邹俊等[65]构建药物组合网络(drug combination network, DCN),采用“neighbor community”通过在DCN的背景下指定药物靶标与其邻近蛋白之间的相互作用来表征协同药物之间的关系。“neighbor communities”更适合表征组合药物之间的关系并进行药物组合预测。DCN可以达到88%的预测准确率和0.95 AUC,且这种基于网络的方法只需要试验药物的靶标信息,不需要难以获得的组学或药理学数据,也不需要生化反应的详细动力学参数值。Luo等[66]通过绘制化合物-靶蛋白相互作用网络图,建立药物相互作用-药物-化学相互作用工具(DDI-CPI,https://cpi.bio-x.cn/ddi/),用于预测药物间相互作用,并寻找与药物相互作用相关的PK/PD蛋白。虽然,该方法的预测准确度高达82%,但仍然无法保证化合物对所有蛋白质的对接精度,同时忽略药物代谢物可能会引起药物相互作用的这一事实。Salvatore等[67]改进了Cheng等[54]的方法,通过综合表型、结构和基因组相似性,在药物相互作用网络的基础上,使用机器学习方法进行药物相互作用预测,并建立DT-Web(https://alpha.dmi.unict.it/dtweb/)。 用户可查询每种药物的特征信息,预测和验证靶标,相关酶和转运蛋白,还可以根据多药物、多靶点、多途径的特点分析,确定可能对其产生间接影响的药物基因。
近年来,有很多学者采用基于模式的统计/学习模型(Pattern-based statistical/learning model)预测药物相互作用或组合协同作用,主要应用于无动力学参数或明确定义的网络靶标的复杂疾病研究。Li等[72]提出了基于靶标的多组分协同识别网络(NIMS:network target-based identification of multicomponent synergy),通过整合靶标网络拓扑模式和靶向相关疾病/表型相似性来筛选中药的协同作用组合,从网络靶标的角度来描述多组分协同作用的潜在机制;Xu等[73]利用“药物鸡尾酒网络”(The drug cocktail network)拓扑特征预测可能的药物组合。
Lee等[48]开发了一种药物组装的方法(combinatorial drug assembler, CDA, http://cda.i-pharm.org/)基于基因组谱数据的富集分析来预测药物组合并提出功能性见解。然而,该方法的富集分析是基于差异表达的基因,对疾病驱动因子无明确定义,因此,可能忽略靶向疾病因果功能障碍信号传导模块的药物。为了克服上述问题,Huang等[23]开发了一种新型的计算工具DrugComboRanker(drug combination discovery based on target network analysis),通过对药物和癌症的基因组谱进行综合分析来预测癌症特异性网络多个信号模块的药物组合;Jansen等[74]开发了一种生物信息学驱动方法,有效预测组合药物的协同作用;Sun等[17]建立了一个半监督学习模型(rankingsystem of anti-cancer synergy, RACS),预测癌症治疗的药物协同作用,通过实验和文献发现对乳腺癌预测准确率高达63.6%;基于此方法,余蔚明[83]从化学和基因空间出发,在已知的四类药物-靶标相互作用网络中最大限度地利用标签数据去推测未标签数据,提出了一种半监督学习方法来预测潜在的药物-靶标对;闫龑[84]结合机器学习的方法,对药物疾病关系网络以及与其相关的受体配体关系网络和蛋白质相互作用网络中的数据进行了挖掘。
3.3 “药物-靶标-疾病-基因”多级相互作用 这类方法依据 CombiGEM(combinatorial genetics en masse)方法、全基因组关联分析(genome-wide association studies,GWAS)和网络建模等方法推测药物对疾病的相互作用。Manolio等[76]检查潜在的遗传来源,并提出基因组关联研究策略,阐明复杂性疾病的遗传学特征,有效地进行疾病预防或治疗;CombiGEM通过分析多因素遗传学特征,快速预测药物组合,揭示产生强表型途径之间的相互作用,利用系统生物学方法发现网络交互作用。Cheng等[77]将该方法应用于癌症研究中,并根据不同的基因敲除模型评估癌细胞对药物的敏感性。网络建模是基于细胞通路和相互作用的详细拓扑图来预测细胞行为的方法。网络建模方法已实现结合自上而下的方法应用,可用于预测模型常数或根据实验数据训练网络特征参数。通过考察特征参数对整个网络的影响,判断药物之间的协同或拮抗作用[78]。
基于网络的药物协同作用评估方法通路相互作用 网络 (“pathway-pathway interaction” network,WWI)比以往基于靶标的方法具有更好的性能,且可更好地阐明某些组合药物的潜在协同机制[85]。该方法以 WNS-score(WWI network-based synergy score)来评估和优化药对,是非常简单的且无模型训练的评估方法,可以实现很好的性能。与Luo等[66]的方法一样,该方法仅考虑了药物靶标干扰的药理学通路的相互作用关系,忽略了组合药物的代谢过程中的协同机制。同时,由于通路相互作用和通路组成的不完整性,WNS-score只能作用于已知通路的药物靶点;由于通路间增强和衰减效应等的界定不完整,只能确定两种途径间是否具有相互作用,并不能确定其强度变化。
此外,有很多基于无监督学习模型的建立,用于协同药物组合的预测[80-82,85]。该预测模型的数据主要来源于药物治疗转录表达数据(drug-treated transcriptional expression data),由于信息的性质可以充分反映生物途径中药物作用机制。但由于目前尚无处理转录表达谱数据的标准规则;差异表达基因的选择在很大程度上取决于个体研究者;且依据不同的算法,重要的基因列表将存在较大的差异性;基于转录表达数据的模型,不同的研究人员将根据不同的微阵列数据的处理方法构建不同的网络,而没有统一标准界定,使最终解释变得困难等,使得模型有一定的局限性。
以上网络药理学的网络建模方法从药物、靶标、疾病、基因方面出发,有效地预测药物相互作用,推测药物对疾病的影响,计算药物的临床有效率等,为组合药物间药物相互作用的预测、组合药物的设计以及协同药物组合的筛选提供新思路和新方法。
4 展望
本文聚焦几年来组合药物在中西药研究领域中的应用,网络药理学可用的数据资源、算法和软件以及现有的组合药物研究方法等,系统论述了网络药理学研究策略在组合药物研究中的应用,为组合药物研究提供理论支撑和方法依据。但目前也存在一些弊端:第一,中医理论将人体视为一个有机的整体,与自然界有着密切的联系,而中药在中医理论指导下,具有多靶点、多途径、整体协同的特点。中药配伍是指有目的地按病情需要和药性特点,有选择地将两味以上药物配合同用,达到增效减毒的作用。这一理论与组合药物的组合原则相一致,更有利于中药在组合药物领域中的应用和发展。因此,应致力于推广中药组合药物的发展和应用。第二,经调研发现,现有的组合药物研究方法在预测组合药物间相互作用时尚未对方法的准确率、召回率、灵敏度和特效性等进行考察与评估,无法对预测方法的性能进行准确评价,具有一定的局限性;采用单一标准的方法预测组合药物时存在或多或少的缺陷,不能达到精准预测的目的。针对这些问题,需对预测方法的精度评价设定统一标准,进行方法的筛选和评估,选择最为优质的方法进行组合药物药效相关性评价。同时发现,采用基于PK/PD相关网络的药物结构相似性的方法来预测组合药物间相互作用及有效性更为优越。因此,联合使用多种方法可提高预测药物间相互作用的准确度和灵敏度,有利于有效组合药物的设计和开发。第三,一些网络建模,如PPI网络、基因-蛋白相互作用网络以及药物靶标相互作用网络等在预测模型中均未考虑代谢网络。药物的吸收和分布在疾病治疗中发挥重要作用,但几乎没有预测模型将药物代谢考虑在内。协同药物组合模型应充分利用疾病信号通路、药物靶标通路以及药物吸收、分布、代谢、排泄等药物代谢过程等信息,综合预测协同药物组合,提高预测性能。第四,随着网络药理学研究的出现和不断深入,网络建模方法的建立、常用数据库资源的开发、算法以及软件的应用等受到国内外学者的广泛关注。网络药理学多成分、多途径和多靶点复杂关系网络的建立与组合药物机制研究相符。同时,有效的网络分析平台的建立为新型药物组合靶点与疾病治疗的网络靶标的筛选,组合药物的设计和优化,以及新的组合药物的创制等提供了关键技术支撑,为组合药物的研发和临床应用提供了一条新途径。
【参考文献】
[1]陈 畅,马 兴,满国峰,等.组合药物-现代药物研发的新趋势[J].中国实验方剂学杂志,2012,18(14):310-312.
[2]Listed N.A once-daily combination tablet(Atripla) for HIV[J].Med Lett Drugs Ther, 2006,48(1244):78-79.
[3]James JS.FDA approves two combination pills, Epzicom and Truvada; comment on commercial race to once-a-day nucleosides[J].AIDS Treat News,2004(403):6.
[4]Goltsov A, Faratian D, Langdon SP, et al.Features of the reversible sensitivity-resistance transition in PI3K/PTEN/AKT signalling network after HER2 inhibition[J].Cell Signal,2012,24(2):493-504.
[5]Dickson MA, Tap WD, Keohan ML, et al.Phase II trial of the CDK4 inhibitor PD0332991 in patients with advanced CDK4-amplified well-differentiated or dedifferentiated liposarcoma[J].J Clin Oncol,2013,31(16):2024-2028.
[6]Robert C, Karaszewska B, Schachter J, et al.Improved overall survival in melanoma with combined dabrafenib and trametinib[J].New Engl J Med,2015,372(1):30-39.
[7]Nair PM,Javad H,Al-Mandhiry ZA.Organophosphate poisoning in children-atropine, pralidoxime or both[J].Saudi Med J,2001,22(9):814-815.
[8]Doyle KP, Simon RP, Stenzel-Poore MP.Neuropharmacology-special issue on cerebral ischemia mechanisms of ischemic brain damage-review article[J].Neuropharmacology,2008,55(3):310-318.
[9]彭世陆,刘丽芳,朱华旭,等.组合药物及黄连解毒汤对小檗碱在大鼠脑组织中药动学的影响[J].中草药,2016,47(16):2877-2882.
[10]Zhang YY, Li HX,Chen YY, et al.Convergent and divergent pathways decoding hierarchical additive mechanisms in treating cerebral ischemia-reperfusion injury[J].Cns Neurosci Ther,2014,20(3):253-263.
[11]Li F, Zhang Y, Zeng D, et al.The combination of three components derived from sheng maisan protects myocardial ischemic diseases and inhibits oxidative stress via modulating MAPKs and JAK2-STAT3 signaling pathways based on bioinformatics approach[J].Front Pharmacol,2017,8:21.
[12]梁 晨.三氧化二砷联合粉防己碱对乳腺癌血管新生及肺转移的影响[D].北京:北京中医药大学,2016.
[13]Zhang Y,Huang F,Lai G.Combination drug approved to treat HIV[J].FDA Consum,2001,22(6):631-638.
[14]Cirstea D, Hideshima T, Rodig S, et al.Dual inhibition of akt/mammalian target of rapamycin pathway by nanoparticle albuminbound-rapamycin and perifosine induces antitumor activity in multiple myeloma[J].Mol Cancer Ther,2010,9(4):963-975.
[15]Mayer LD,Harasym TO,Tardi PG,et al.Ratiometric dosing of anticancer drug combinations:controlling drug ratios after systemic administration regulates therapeutic activity in tumor-bearing mice[J].Mol Cancer Ther,2006,5(7):1854-1863.
[16]Soldi R, Cohen AL, Cheng L, et al.A genomic approach to predict synergistic combinations for breast cancer treatment[J].Pharmacogenomics J,2013,13(1):94-104.
[17]Sun Y, Sheng Z, Ma C, et al.Combining genomic and network characteristics for extended capability in predicting synergistic drugs for cancer[J].Nat Commun,2015,6:8481.
[18]Park HK,Lee JE,Lim J,et al.Combination treatment with doxorubicin and gamitrinib synergistically augments anticancer activity through enhanced activation of bim[J].BMC Cancer,2014,14:431.
[19]Sun X, Bao J, Nelson KC, et al.Systems modeling of anti-apoptotic pathways in prostate cancer:psychological stress triggers a synergism pattern switch in drug combination therapy[J].PLoS Comput Biol,2013,9(12):e1003358.
[20]Goltsov A, Faratian D, Langdon SP, et al.Compensatory effects in the PI3K/PTEN/AKT signaling network following receptor tyrosine kinase inhibition[J].Cell Signal,2011,23(2):407-416.
[21]邱永丰.药物组合对膀胱癌T24细胞抑制作用的初步实验研究[D].长春:吉林大学,2011.
[22]刘 磊,赵羲和,李 凯.帕妥珠单抗与厄洛替尼对荷瘤小鼠的疗效研究[J].解剖科学进展,2008,14(3):277-280.
[23]Huang L, Li F, Sheng J, et al.DrugComboRanker:drug combination discovery based on target network analysis[J].Bioinformatics,2014,30(12):i228-i236.
[24]Miller ML, Molinelli EJ, Nair JS, et al.Drug synergy screen and network modeling in dedifferentiated liposarcoma identifies CDK4 and IGF1R as synergistic drug targets[J].Sci Signd,2013,6(294):ra85.
[25]Flaherty KT.Dividing and conquering:controlling advanced melanoma by targeting oncogene-defined subsets[J].Clin Exp Metastasis,2012,29(7):841-846.
[26]Tyner JW, Yang WF, Bankhead A 3rd, et al.Kinase pathway dependence in primary human leukemias determined by rapid inhibitor screening[J].Cancer Res,2013,73(1):285-296.
[27]Robert C, Thomas L, Bondarenko I, et al.Ipilimumab plus dacarbazine for previously untreated metastatic melanoma[J].N Engl J Med,2011,364(26):2517-2526.
[28]张治洲.从系统生物学到配伍西药:中药现代化的一个核心战略[J].中国生物工程杂志,2007,27(4):153-156.
[29]朴香梅,姜俊哲,朴林虎,等.赖诺普利氢氯噻嗪在中老年性高血压治疗中的临床疗效[J].中国继续医学教育,2016,8(2):157-158.
[30]Chan R, Park KC, Lee MH, et al.A randomized controlled trial of the efficacy and safety of a fixed triple combination(fluocinolone acetonide 0.01%, hydroquinone 4%, tretinoin 0.05%) compared with hydroquinone 4%cream in Asian patients with moderate to severe melasma[J].Br J Dermatol,2008,159(3):697-703.
[31]Peng H, Peng T, Wen J, et al.Characterization of p38 MAPK isoforms for drug resistance study using systems biology approach[J].Bioinformatics,2014,30(13):1899-1907.
[32]Wang Z, Jing ZW, Zhou CX, et al.Fusion of core pathways reveals a horizontal synergistic mechanism underlying combination therapy[J].Eur J Pharmacol,2011,667(1-3):278-286.
[33]Pawson T, Linding R.Network medicine[J].FEBS Lett,2008,582(8):1266-1270.
[34]张彦琼,李 梢.网络药理学与中医药现代研究的若干进展[J].中国药理学与毒理学杂志,2015,29(6):883-892.
[35]Xiong J, Rayner S, Liang F, et al.The Principle of Rational Design of Drug Combination and Personalized Therapy Based on Network Pharmacology[J].Chin J Pharmacol Toxicol,2013,27(1):1-5.
[36]Liu Y, Wei Q, Yu G, et al.DCDB 2.0:a major update of the drug combination database[J].Database(Oxford),2014,2014:bau124.
[37]Library WP.Anatomical therapeutic chemical classification system[J].2015.
[38]Chen CY.TCM Database@ Taiwan:the world's largest traditional Chinese medicine database for drug screening in silico[J].Plos One,2011,6(1):e15939.
[39]Xue R,Fang Z,Zhang M,et al.TCMID:traditional Chinese medicine integrative database for herb molecular mechanism analysis[J].Nucleic Acids Res,2013,41(Database issue):D1089-D1095.
[40]Ye H, Ye L, Kang H, et al.HIT: linking herbal active ingredients to targets[J].Nucleic Acids Res,2011,39(Database issue):D1055-D1059.
[41]Ru J, Li P, Wang J, et al.TCMSP:a database of systems pharmacology for drug discovery from herbal medicines[J].J Cheminformatics,2014,6:13.
[42]Yao X, Hao H, Li Y, et al.Modularity-based credible prediction of disease genes and detection of disease subtypes on the phenotype-gene heterogeneous network[J].Bmc Syst Biol,2011,5(1):1-11.
[43]Chatr-Aryamontri, Oughtred R, Boucher L, et al.The BioGRID interaction database:2017 update[J].Nucleic Acids Res,2017,45(D1):D369-D379.
[44]Stolovitzky G,Monroe D,Califano A.Dialogue on reverse-engineering assessment and Methods:the DREAM of high-throughput pathway inference[J].Ann N Y Acad Sci,2007,1115:1-22.
[45]Thorn CF,Klein TE,Altman RB.PharmGKB:the Pharmacogenomics knowledge base[J].Methods Mol Biol,2013,1015:311-320.
[46]Szklarczyk D, Franceschini A, Wyder S, et al.STRING v10:protein-protein interaction networks, integrated over the tree of life[J].Nucleic Acids Res,2015,43(Database issue):D447-D452.
[47]刘彦斌.组合药物数据库构建及药物协同作用机理分析[D].杭州:浙江大学,2011.
[48]Lee JH, Kim DG, Bae TJ, et al.CDA:combinatorial drug discovery using transcriptional response modules[J].PLoS One,2012,7(8):e42573.
[49]Batagelj V, Mrvar A.Pajek-analysis and visualization of large networks[M]//Graph Drawing Software.Springer Berlin Heidelberg,2004.
[50]Zhao XM,Iskar M,Zeller G,et al.Prediction of drug combinations by integrating molecular and pharmacological data[J].PLoS Comput Biol,2011,7(12):e1002323.
[51]Wu M, Sirota M, Butte AJ, et al.Characteristics of drug combination therapy in oncology by analyzing clinical trial data on Clinical-Trials.gov[J].Pac Symp Biocomput,2015:68-79.
[52]Wang YY, Xu KJ, Song J, et al.Exploring drug combinations in genetic interaction network[J].BMC Bioinformatics,2012,13(Suppl 7):S7.
[53]赵鹏姚.药物相互作用的网络药理学分析与预测[D].北京:北京交通大学,2016.
[54]Cheng F, Zhao Z.Machine learning-based prediction of drug-drug interactions by integrating drug phenotypic, therapeutic, chemical,and genomic properties[J].J Am Med Inform Assoc,2014,21(e2):e278-e286.
[55]Vilar S, Uriarte E, Santana L, et al.Similarity-based modeling in large-scale prediction of drug-drug interactions[J].Nat Protoc,2014,9(9):2147-2163.
[56]Sun X, Vilar S, Tatonetti NP.High-throughput methods for combinatorial drug discovery[J].Sci Transl Med,2013,5(205):205rv1.
[57]Li P, Huang C, Fu Y, et al.Large-scale exploration and analysis of drug combinations[J].Bioinformatics,2015,31(12):2007-2016.
[58]Chandrasekaran S,Cokol-Cakmak M,Sahin N,et al.Chemogenomics and orthology-based design of antibiotic combination therapies[J].Mol Syst Biol,2016,12(5):872.
[59]Takeda T,Hao M,Cheng T,et al.Predicting drug-drug interactions through drug structural similarities and interaction networks incorporating pharmacokinetics and pharmacodynamics knowledge[J].J Cheminform,2017,9:16.
[60]程飞雄.系统药物设计方法发展及应用研究[D].上海:华东理工大学,2013.
[61]Penrod NM,Greene CS,Moore JH.Predicting targeted drug combinations based on pareto optimal patterns of coexpression network connectivity[J].Genome Med,2014,6(4):33.
[62]Tang J, Karhinen L, Xu T, et al.Target inhibition networks:predicting selective combinations of druggable targets to block cancer survival pathways[J].PLoS Comput Biol,2013,9(9):e1003226.
[63]Herrero-Zazo M, Segura-Bedmar I, Hastings J, et al.DINTO:using OWL ontologies and SWRL rules to infer drug-drug interactions and their mechanisms[J].J Chem Inf Model,2015,55(8):1698-1707.
[64]Huang H, Zhang P, Qu XA, et al.Systematic prediction of drug combinations based on clinical side-effects[J].Sci Rep,2014,4:7160.
[65]邹 俊,杨胜勇,魏于全,等.基于药物组合网络的药物联合作用预测方法[P].2015.
[66]Luo H, Zhang P, Huang H, et al.DDI-CPI, a server that predicts drug-drug interactions through implementing the chemical-protein interactome[J].Nucleic Acids Res,2014,42(Web Server issue):W46-W52.
[67]Alaimo S, Bonnici V, Cancemi D, et al.DT-Web:a web-based application for drug-target interaction and drug combination prediction through domain-tuned network-based inference[J].BMC Syst Biol,2015,9(Suppl 3):S4-S4.
[68]Iadevaia S, Lu Y, Morales FC, et al.Identification of optimal drug combinations targeting cellular networks:integrating phospho-proteomics and computational network analysis[J].Cancer Res,2010,70(17):6704-6714.
[69]Araujo RP, Petricoin EF,Liotta LA.A mathematical model of combination therapy using the EGFR signaling network[j].Biosystems,2005,80 (1):57-69.
[70]Aldridge BB, Saez-Rodriguez J, Muhlich JL, et al.Fuzzy logic analysis of kinase pathway crosstalk in TNF/EGF/insulin-induced signaling[J].PLoS Comput Biol,2009,5(4):e1000340.
[71]Molinelli EJ, Korkut A, Wang W, et al.Perturbation biology:inferring signaling networks in cellular systems[J].PLoS Comput Biol,2013,9(12):e1003290.
[72]Li S, Zhang B, Zhang N.Network target for screening synergistic drug combinations with application to traditional Chinese medicine[J].BMC Syst Biol,2011,5(Suppl 1):S10.
[73]Xu KJ, Song J, Zhao XM.The drug cocktail network[J].BMC Syst Biol,2012;6(Suppl 1):S5.
[74]Jansen G, Lee AY, Epp E, et al.Chemogenomic profiling predicts antifungal synergies[J].Mol Syst Biol,2009,5:338.
[75]Chen X,Ren B,Chen M,et al.NLLSS:predicting synergistic drug combinations based on semi-supervised learning[J].PLoS Comput Biol,2016,12(7):e1004975.
[76]Manolio TA, Collins FS, Cox NJ, et al.Finding the missing heritability of complex diseases[J].Nature,2009,461(7265):747-753.
[77]Cheng AA,Ding H,Lu TK.Enhanced killing of antibiotic-resistant bacteria enabled by massively parallel combinatorial genetics[J].Proc Natl Acad Sci USA,2014,111(34):12462-12467.
[78]Zhang Y, Smolen P, Baxter DA,et al.Computational Analyses of Synergism in Small Molecular Network Motifs[J].PLoS Comput Biol,2014,10(3):e1003524.
[79]Wu Z, Zhao XM, Chen L.A systems biology approach to identify effective cocktail drugs[J].BMC Syst Biol,2010,4(Suppl 2):S7.
[80]Tan VY, Févotte C.Automatic relevance determination in nonnegative matrix factorization with the β-divergence[J].IEEE Trans Pattern Anal Mach Intell,2013,35(7):1592-1605.
[81]Jin G, Zhao H, Zhou X, et al.An enhanced Petri-net model to predict synergistic effects of pairwise drug combinations from gene microarray data[J].Bioinformatics,2011,27(13):i310-i316.
[82]Yang J,Tang H,Li Y,et al.DIGRE:drug-induced genomic residual effect model for successful prediction of multidrug effects[J].CPT Pharmacometrics Syst Pharmacol,2015,4(2):91-97.
[83]余蔚明.药物-靶标相互作用网络预测方法研究[D].上海:华东师范大学,2012.
[84]闫 龑.重要药物-疾病互作网络的数据挖掘方法研究[D].上海:华东师范大学,2014.
[85]Chen D, Zhang H, Lu P, et al.Synergy evaluation by a pathway-pathway interaction network:a new way to predict drug combination[J].Mol Biosyst,2016,12(2):614-623.
