俄罗斯卡尔梅克共和国传统奶酪中细菌多样性研究
2018-04-17侯强川孙志宏孙天松刘文俊
任 敏, 李 晶, 侯强川, 孙志宏, 孙天松, 刘文俊
(内蒙古农业大学 乳品生物技术与工程教育部重点实验室/农业部奶制品加工重点实验室,内蒙古 呼和浩特 010018)
俄罗斯卡尔梅克共和国是一个以草原畜牧业为主的国家,拥有丰富的羊奶和牛奶资源,当地传统奶酪是最古老的发酵食品之一[1]。传统奶酪是以天然乳清作为发酵剂添加到牛奶中进行发酵,排出乳清后将凝乳放入模具中成型制成[2]。众所周知,微生物间的相互作用对传统奶酪形成尤为重要,而作为发酵剂的天然乳清中包含着丰富且复杂的微生物群落。Masoud等[3]对丹麦原奶酪的研究表明,发酵剂中的细菌会成为奶酪中的优势菌群。然而,Almeida等[4]研究不同传统奶酪时发现静止嗜冷杆菌(Psychrobacterimmobilis)和假单胞菌(Pseudoalteromonashaloplanktis)等虽然最初并没有作为发酵剂接种,但却是发酵过程中的优势菌种。因此,了解传统奶酪中的细菌群落组成,获得优势菌群[5],了解其发酵机理,同时挖掘新的微生物资源,对传统奶酪产品的工业化、标准化生产和益生菌开发利用具有重要的意义,但目前针对俄罗斯卡尔梅克共和国传统奶酪样品中细菌多样性的研究却少之又少。
454焦磷酸测序技术被广泛应用于许多发酵产品中微生物多样性的分析,例如发酵蔬菜、发酵乳以及奶酪等[6-9]。本研究应用6份俄罗斯卡尔梅克共和国传统奶酪样品的454焦磷酸测序数据,通过生物信息学分析,以地域和奶酪中细菌群落的关系研究为侧重点,对样品中细菌多样性进行详细分析,希望能够充实俄罗斯传统奶酪理论研究数据,为其中微生物的开发应用提供理论依据。
1 材料与方法
1.1 实验材料
6份传统奶酪样品采自俄罗斯卡尔梅克共和国,具体信息见表1。
1.2 实验方法
以本研究团队采用454焦磷酸测序技术获得的6份奶酪样品序列数据为分析对象,序列已上传至MG-RAST数据库,编号为No.4682839.3—4682844.3。
1.2.1高质量序列的筛选
测序得到的原始数据确认引物所在位置,认为引物之间的区域为可变区,从原始序列中筛选可变区长度大于300 bp的序列,tag按照样本-tag编号筛选,必须为首7位且不允许错配。控制整条序列上质量大于20的碱基所占比例高于93%。经过筛选,可以认为提取出长度、质量、引物、tag各方面均有效的高质量序列[10-11]。
1.2.2高质量序列的生物信息学分析
应用QIIME(quantitative insights into microbial ecology)软件对得到的高质量序列进行生物信息学分析。用PyNAST[12]进行校准、排齐,在序列100%相似性下进行UCLUST[13]归并,建立无重复的16S rRNA序列,在给定相似度97%的水平上划分分类操作单元(operational taxonomic units,OTU)。将去除含有嵌合体序列的OTU序列使用RDP(ribosomal database project)[14]和Greengenes(Version_3.8)[15]数据库进行序列同源性比对,并在分类学的门、纲、目、科、属和种水平上进行鉴定。使用FastTree[16]软件构建基于OTU代表性序列的系统发育进化树,随后计算香农指数(Shannon-Wiener index)、发现物种数(observed species)、超1(chao1)指数和辛普森(Simpson)指数共4个Alpha多样性指标,分别对样品菌群构成的丰度和多样性进行评价。同时,采用香农多样性指数(Shannon,s diversity index)和稀疏曲线(rarefaction curve)评估每个样本测序的多样性,并判断当前测序量是否能够满足后续分析的需要。使用UniFrac距离进行加权(weighted)和非加权(unweighted)的主坐标分析,揭示各个样本之间菌群结构的差异[17]。
2 结果与分析
2.1 细菌序列丰度和多样性
6份奶酪样品的454焦磷酸测序序列信息和α多样性指数如表2。
由表2可知,6份奶酪样品共得到89 393条高质量细菌基因序列,每个样品平均序列为14 899条(序列数范围:13 485~17 261)。根据97%的序列相似度划分分类操作单元(OTU),共得到369条具有代表性的细菌OTU序列。结果显示,R6号样品的α多样性指数均高于其余5份样品,表明R6号样品微生物多样性最高。根据表2信息绘制样品稀疏曲线图(a)和香农指数图(b)分别用于分析样品中微生物的丰度和多样性,结果如图1。
由图1(a)可知,6份奶酪样品的稀疏曲线均未进入平台期,这表明随着测序量的增加,新的细菌种系将被发现。而图1(b)显示的Shannon曲线已经饱和,这表明随着测序量的增加细菌多样性将不再改变,因此当前测序量可以满足后续分析的需要。

表2 样品的测序序列信息和α多样性指数

图1 稀疏曲线和香农指数图Fig.1 Rarefaction curves and Shannon diversity index curves
2.2 样品中细菌相对含量和构成
进一步使用BLAST同源性比对,采用RDP和Greengenes数据库,将所有序列鉴定到7个门、50个属。在门水平上,6份样品主要由硬壁菌门(Firmicutes,80.15%)和变形菌门(Proteobacteria,19.80%)组成,如图2。值得注意的是,R1-R4号样品中Firmicutes都占有绝对优势(相对含量均在95%以上),R6号样品Firmicutes含量为74.6%,而R5号样品中相对含量最高的菌门为Proteobacteria(85.1%)而并非Firmicutes(14.8%)。

除Proteobacteria和Frimicutes两个菌门外,其余菌门相对含量较少,图中未明显显示。图2 门水平上奶酪样品的细菌组成Fig.2 Relative abundance of major bacteria in cheeses at phylum level
在属水平上,6份样品中相对含量大于1%的优势菌属包括乳球菌属(Lactococcus,42.81%)、链球菌属(Streptococcus,29.44%)、柠檬酸杆菌属(Citrobacter,8.88%)、不动杆菌属(Acinetobacter,5.04%)、醋酸杆菌属(Acetobacter,2.00%)、肠球菌(Enterococcus,1.96%)、巨型球菌(Macrococcus,1.89%)、明串珠菌属(Leuconostoc,1.47%)和乳杆菌属(Lactobacillus,1.23%),如图3。除此之外还鉴定到了一些相对含量较低的罕见菌属。这些菌属的鉴定丰富了本研究团队前期对该地区传统奶酪样品中优势菌属的描述,更加全面地揭示了样品的细菌多样性。值得一提的是,6份样品的优势菌属出现了与门水平中大致相同的情况,即R1-R4号以及R6号样品中的优势菌属为Lactococcus或Streptococcus,而R5号样品中的优势菌属为Citrobacter和Acinetobacter。
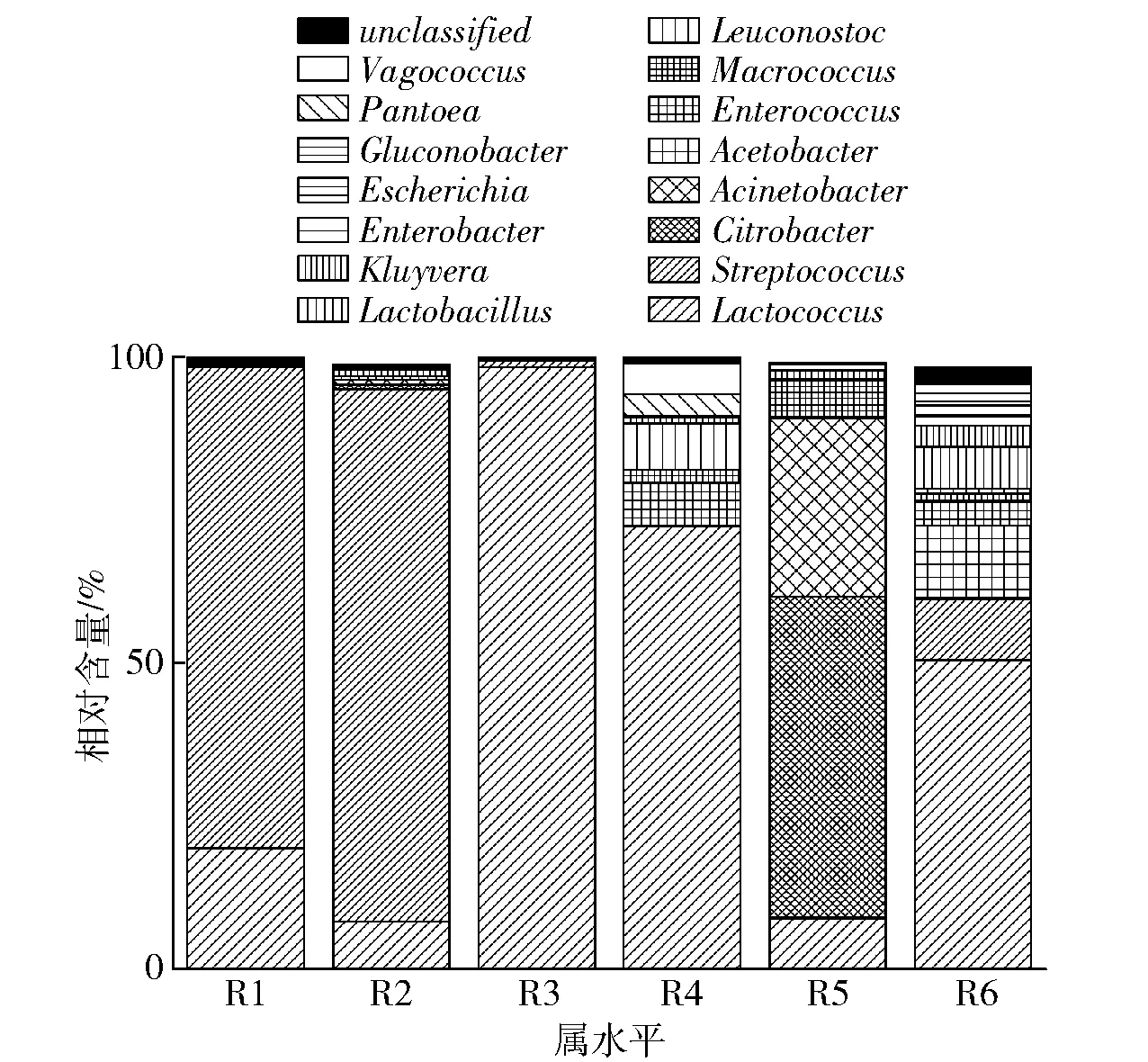
图3 属水平上奶酪样品的细菌组成Fig.3 Relative abundance of major bacteria in cheeses at genus level
2.3 基于OTU水平上传统奶酪样品中细菌组成
由于α多样性分析是基于OTU水平的,因此基于OTU水平对样品中细菌菌群做进一步分析,见图4。由图4可知,6份样品共产生369个OTU,6份样品共享的OTU仅有11个,占OTU总数的2.98%,但其包含的序列数为40 671,占质控合格总序列数的45.5%。每份样品独有的OTU有239个,占OTU总数的64.8%。由此可知,6份样品中共同存在的OTU数目很少,但是包含的序列信息却很多,那么这部分OTU很有可能反映出了样品中的核心菌群,但还有很大一部分OTU是每份样品独有的。因此,即使6份样品的核心菌群在很大程度上相似,但其特有的微生物使样品间存在差异。
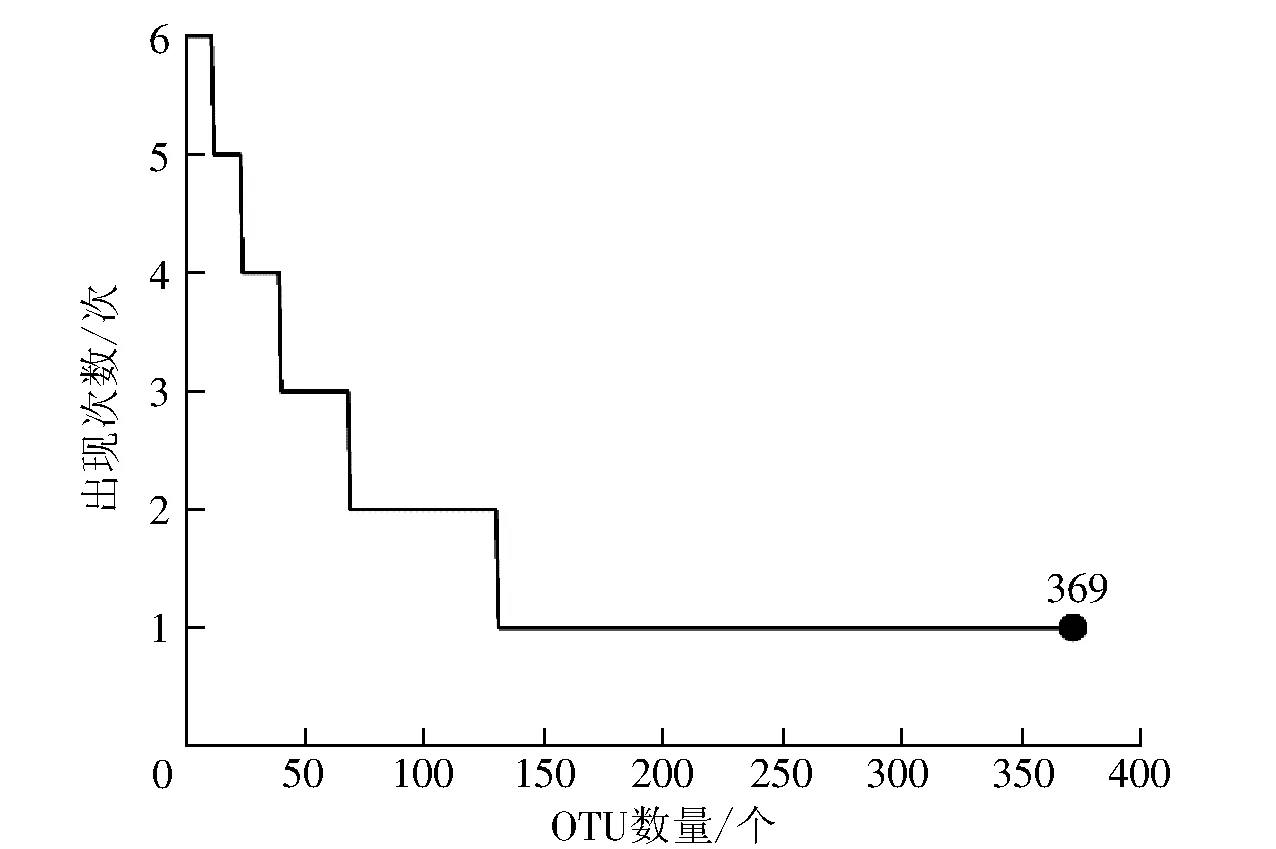
图4 奶酪样品中OTU出现次数Fig.4 Frequency of occurences of OTU in cheeses
图5进一步反映了奶酪样品中核心OTU的相对含量,结果显示6份样品主要的菌群为Lactococcus和Streptococcus,是发酵乳制品中常用的发酵剂菌种。

图5 奶酪样品间核心OTU的相对含量 Fig.5 Relative abundance of core OTU in cheeses
为了探究6份传统奶酪样品中核心菌群间的相关关系,根据核心菌属的相对含量计算其相关性,结果如图6。由图6可知,克雷伯氏菌属(Klebsiella)与肠杆菌属(Enterobacter)和Lactobacillus之间有极显著的正相关关系(p<0.01)。

** p<0.01,上方和左侧的非加权组平均法聚类树均依照各核心菌属相对含量进行计算。图6 奶酪样品间核心菌属的相关性Fig.6 Correlation of core bacterial community among cheeses
2.4 基于多元统计方法的传统奶酪样品细菌群落结构比较
基于UniFrac距离的加权和非加权主坐标(principal coordinate analysis, PCoA)法对6份传统奶酪样品细菌群落结构进行分析。之前的研究仅仅将这些样品数据看作一个整体与其他国家和地区发酵产品进行比较,并未详细分析6份样品本身存在的菌群结构差异。本研究基于加权和非加权UniFrac距离进行主坐标分析,结果如图7 。由图7可见,第一主成分和第二主成分的贡献率分别为88.33%、6.91%和29.86%、23.63%,从图7(a)可知,采自卡尔梅克共和国亚什库尔镇地区的大部分样品有明显聚类趋势。

图7 基于非加权与加权UniFrac距离的主坐标分析得分图Fig.7 PCoA scores plot based on unweighted and weighted UniFrac analysis
3 讨 论
目前,采用454焦磷酸测序技术对发酵乳制品细菌多样性的研究越来越多,本研究团队也采用该测序技术对俄罗斯卡尔梅克共和国地区的发酵酸牛奶[18]以及传统奶酪样品中的细菌微生物进行了一些研究。但对该地区的传统奶酪样品只进行了优势菌属的简要描述,并将其作为对照与其他国家和地区的发酵乳[19]和奶酪[20]进行了比较,并没有对奶酪样品本身细菌群落的构成进行详细分析。
本研究基于454焦磷酸测序数据对俄罗斯卡尔梅克共和国的6份传统奶酪样品进行细菌多样性的分析。在门水平上,Firmicutes和Proteobacteria为优势菌门;在属水平上,6份传统奶酪样品共鉴定到50个细菌属。杨彦荣等[21]使用纯培养方法对该地区的传统奶酪样品进行乳酸菌分离鉴定,共得到37株乳酸菌,分别归属于Enterococcus、Lactobacillus和Leuconostoc3个菌属。侯强川等[6]利用纯培养和454焦磷酸测序技术结合的方法对俄罗斯卡尔梅克共和国的2份发酵蔬菜进行细菌多样性研究,结果显示,与传统方法相比,采用高通量测序技术的确能够鉴定到更多的细菌种类,从而全面了解样品中的细菌多样性。
依据这6份传统奶酪样品中细菌相对含量的不同,可以将这些细菌分为三大类。第一类是由Lactococcus和Streptococcus组成的优势菌属(相对含量大于10%),这个结果与许多地区奶酪中的优势菌属一致,例如Quigley 等[22]采用454焦磷酸测序技术发现爱尔兰奶酪中的优势菌属为Lactococcus。Dalmasso等[23]对意大利传统奶酪的研究发现其优势菌属也是由Lactococcus、Streptococcus和Lactobacillus组成。有研究表明,这两个菌属的某些菌株有助于奶酪的酸化、凝乳以及酪蛋白水解,而这些功能对奶酪的形成至关重要;此外,奶酪的风味也会受到菌株代谢产物的影响,例如氨基酸和脂肪代谢等[24]。第二类是传统奶酪样品中的次优势菌属(相对含量在1%~10%),包括Citrobacter、Acinetobacter、Acetobacter、Enterococcus、Macrococcus、Leuconosto和Lactobacillus。有研究显示Leuconosto与Enterococcus能够对奶酪的味道和质地产生影响[25],Lactobacillus中瑞士乳杆菌(Lactobacillushelveticus)凭借其蛋白水解能力可以减少奶酪苦味[26]。值得一提的是,在个别样品中发现了Macrococcus,该菌属中最典型的菌种为溶酪大球菌(Macrococcuscaseolyticus),吴燕涛等[27]对Macrococcuscaseolyticus的发酵特性进行分析,发现该菌种具有脂肪与蛋白分解活性,由此推断在奶酪中也可能起到相同作用。第三类是在个别样品中检出含量很低的菌属(相对含量在0.1%~1%),包括有克吕沃菌属(Kluyvera)、漫游球菌属(Vagococcus)、泛生菌属(Pantoea)、金黄杆菌属(Chryseobacterium)、葡萄球菌属(Staphylococcus)、葡糖杆菌属(Gluconobacter)、Enterobacter、Klebsiella、假单胞菌(Pseudomonas)、水细菌属(Enhydrobacter)和劳特菌属(Raoultella)等罕见的菌属。Dalmasso等[23]对意大利奶酪进行454焦磷酸测序分析时也鉴定到了相对含量较低的Vagococcus和Klebsiella等菌属。这些菌属的存在大多意味着产品加工过程中卫生条件较差,但这些菌属所占比例较少,并且也没有出现主要的食源性病原体。基于本研究结果,俄罗斯卡尔梅克共和国地区可能需要加快对传统奶酪制品的现代化和工业化,使其品质得到保证。
与其他采自同一地区的5份样品不同,R5号样品的优势菌属是Citrobacter和Acinetobacter。此外,基于多元统计方法对6份样品中的细菌群落结构进行比较分析,结果显示采自同一区域的4份样品(R1-R4)有明显的聚类趋势,与R5号样品存在明显的分离趋势。这可能是由于不同地区之间的气候因素、制作工艺以及地理环境不同等因素导致的。目前也有越来越多的研究表明,环境因素也是影响乳制品微生物多样性的原因。Sun等[28]采用高通量测序技术对采自中国内蒙古、四川、甘肃以及蒙古国的17个传统酸奶样品进行分析时发现,传统酸奶中菌群多样性与其所在地理位置有密切的联系。Liu等[18]和Xu等[29]的研究表明,来自不同地理位置的自然发酵乳中微生物多样性存在明显差异。
4 结 论
本研究应用454焦磷酸测序数据对6份采自俄罗斯卡尔梅克共和国的传统奶酪样品进行细菌多样性的分析。研究发现,其中5份样品的优势菌属与其他国家和地区的奶酪样品基本一致,证实了传统奶酪中的优势菌属大多为Lactococcus和Streptococcus,但仅有R5号样品其优势菌属为Citrobacter和Acinetobacter。此外,本研究在奶酪样品中鉴定到的Vagococcus和Klebsiella等菌属,意味着该地区奶酪制作过程中卫生条件较差,提示当地传统奶酪产品急需现代化和工业化。6份传统奶酪样品之间存在的菌群结构差异,这可能意味着地理环境不同对奶酷菌群产生了影响,进一步的原因需增加样品量进行深入分析。
参考文献:
[1] 陈鹏. 欧洲的蒙古民族——卡尔梅克人[J]. 中国民族, 1986(11):46-47.
CHEN P. The Mongolian nation of Europe-Kalmyk[J]. Chinese Nation,1986(11):46-47.
[2] FUKA M M, WALLISCH S, ENGEL M, et al. Dynamics of bacterial communities during the ripening process of different Croatian cheese types derived from raw ewe’s milk cheeses[J]. Plos One, 2013, 8(11):e80734.
[3] MASOUD W, VOGENSEN F K, LILLEVANG S, et al. The fate of indigenous microbiota, starter cultures,Escherichiacoli,ListeriainnocuaandStaphylococcusaureusin Danish raw milk and cheeses determined by pyrosequencing and quantitative real time (qRT)-PCR[J]. International Journal of Food Microbiology, 2012, 153(1/2):192-202.
[4] ALMEIDA M, HÉBERT A, ABRAHAM A L, et al. Construction of a dairy microbial genome catalog opens new perspectives for the metagenomic analysis of dairy fermented products[J]. Bmc Genomics, 2014, 15(1):1101.
[5] 吴进菊, 胡佳琪, 于博,等. 传统发酵食品中微生物多样性和群落结构动态变化研究进展[J]. 中国酿造, 2016, 35(9):20-23.
WU J J, HU J Q, YU B,et al.Research progress on microbial diversity and dynamic changes of community structure in the traditional fermented food[J],China Brewing,2016, 35(9):20-23.
[6] 侯强川, 郭壮, 张家超,等. 俄罗斯卡尔梅克共和国发酵蔬菜中细菌多样性研究[J]. 食品与发酵工业, 2014, 40(7):16-22.
HOU Q C,GUO Z,ZHANG J C, et al. Study on bacteria diversity in fermented vegetable of Russian Republic of Kalmykia Elista[J].Food And Fermentation Industries,2014, 40(7):16-22.
[7] SUN Z, LIU W, BAO Q, et al. Investigation of bacterial and fungal diversity in tarag using high-throughput sequencing[J] .J Dairy Sci, 2014, 97(10): 6085-6096.
[8] LIU W J, XI X X, SUDU Q, et al. High-throughput sequencing reveals microbial community diversity of Tibetan naturally fermented yak milk[J] .Ann Microbiol, 2015,65(3): 1741-1751.
[9] ALDRETETAPIA A, ESCOBARRAMREZ M C, TAMPLIN M L, et al. High-throughput sequencing of microbial communities in Poro cheese, an artisanal Mexican cheese.[J]. Food Microbiology, 2014, 44(6):136-141.
[10] 张冬蕾. 应用焦磷酸测序技术对新疆地区传统发酵乳制品中微生物多样性的研究[D]. 呼和浩特:内蒙古农业大学, 2016.
[11] 郭壮. 应用焦磷酸测序技术对不同人群肠道微生物群落结构的研究[D]. 无锡: 江南大学, 2013.
[12] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al. PyNAST: a flexible tool for aligning sequences to a template alignment[J]. Bioinformatics, 2010, 26(2):266-267.
[13] EDGAR R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19):2460-2461.
[14] COLE J R, CHAI B, FARRIS R J, et al. The ribosomal database project (RDP-Ⅱ): introducing myRDP space and quality controlled public data[J]. Nucleic Acids Research, 2007, 35:D169-D172.
[15] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied & Environmental Microbiology, 2006, 72(7):5069-5072.
[16] PRICE M N, DEHAL P S, ARKIN A P. Fast tree: computing large minimum evolution trees with profiles instead of a distance matrix[J]. Molecular Biology & Evolution, 2009, 26(7):1641-1650.
[17] LOZUPONE C, KNIGHT R. UniFrac: a new phylogenetic method for comparing microbial communities[J]. Applied & Environmental Microbiology, 2005, 71(12):8228.
[18] LIU W, ZHENG Y, KWOK L Y, et al. High-throughput sequencing for the detection of the bacterial and fungal diversity in Mongolian naturally fermented cow's milk in Russia[J]. Bmc Microbiology, 2015, 15(1):15-45.
[19] ZHONG Z, HOU Q, KWOK L, et al. Bacterial microbiota compositions of naturally fermented milk are shaped by both geographic origin and sample type[J]. J of Dairy Sci, 2016, 99(10):7832-7841.
[20] LI J, ZHENG Y, XU H, et al. Bacterial microbiota of Kazakhstan cheese revealed by single molecule real time (SMRT) sequencing and its comparison with Belgian, Kalmykian and Italian artisanal cheeses[J]. Bmc Microbiology, 2017, 17(1):13.
[21] 杨彦荣, 任艳, 德亮亮,等. 传统奶酪样品中乳酸菌的分离鉴定[J]. 中国乳品工业, 2015, 43(9):19-22.
YANG Y R,REN Y,DE L L,et al. Isolation and identification of lactic acid bacteria from traditional cheese in the Republic of Kalmykia in Russian[J].China Dairy Industry,2015, 43(9):19-22.
[22] QUIGLEY L, O’SULLIVAN O, BERESFORD T P, et al. High-throughput sequencing for detection of subpopulations of bacteria not previously associated with artisanal cheeses[J]. Applied & Environmental Microbiology, 2012, 78(16):5717-5723.
[23] DALMASSO A, MARIA D L, CIVERA T, et al. Chara-cterization of microbiota in plaisentif cheese by high-throughput sequencing[J]. LWT-Food Science and Technology, 2016, 69:490-496.
[24] MCSWEENEY P L H. Biochemistry of cheese ripening: introduction and overview[J]. Cheese Chemistry Physics & Microbiology, 2004, 1(1):347-360.
[25] MONTEL M C, BUCHIN S, MALLET A, et al. Traditional cheeses: rich and diverse microbiota with associated benefits[J]. International Journal of Food Microbiology, 2014, 177(54):136-154.
[26] GRIFFITHS M W, TELLEZ A M.Lactobacillushelveticus: the proteolytic system[J]. Frontiers in Microbiology, 2013, 4(1):30.
[27] 吴燕涛, 赵谋明, 孙为正,等. 内源性发酵剂Macrococcuscaseolyticus发酵广式腊肠的风味物质成分分析[J]. 食品工业科技, 2011, 32(7):207-209.
WU Y T,ZHAO M M,SUN W Z, et al.Analysis of volatile compounds of cantonese sausage fermented byMacrococcuscaseolyticusseparated from cantonese sausage[J].Science and Technology of Food Industry,2011, 32(7):207-209.
[28] SUN Z, LIU W, BAO Q, et al. Investigation of bacterial and fungal diversity in tarag using high-throughput sequencing[J]. Journal of Dairy Science, 2014, 97(10):6085-6096.
[29] XU H, LIU W, GESUDU Q, et al. Assessment of the bacterial and fungal diversity in home-made yogurts of Xinjiang, China by pyrosequencing[J]. Journal of the Science of Food & Agriculture, 2015, 95(10):2007-2015.
