QX200微滴式数字PCR方法检测转基因大豆GTS-40-3-2
2018-03-07吕贝贝王金斌武国干胡为一唐雪明
吴 潇,吕贝贝*,蒋 玮,王金斌,4,白 蓝,武国干,胡为一,唐雪明**
(1上海市农业科学院生物技术研究所,上海201106;2农业部转基因植物环境安全监督检验测试中心(上海),上海201106;3上海市农业遗传育种重点实验室,上海201106;4上海瑞丰农业科技有限公司,上海201106;5上海安景科技有限公司,上海200051)
微滴式数字PCR(Droplet digital PCR,ddPCR)是近年来迅速发展起来的一种突破性地检测和定量核酸的技术,其基本原理是通过将模板极度稀释,将所有样品在相同条件下进行PCR扩增,实现理论上的单分子扩增,有PCR扩增荧光信号记为1,无荧光信号记为0,反应结果采用泊松概率分布公式,便可计算出样本的原始浓度[1-5]。
外源基因的检测是转基因生物安全评价的重要评价指标之一[6]。目前,外源基因拷贝数和含量分析多采用实时荧光定量PCR技术(Real-time Quantitative PCR,qPCR)。qPCR技术在分析外源基因拷贝数时必须依赖于已知拷贝数的内参基因和标准曲线的建立,是一种相对定量方法,且标准曲线的建立易受到模板纯度、引物和探针的浓度等诸多因素影响[7]。与qPCR方法相比,微滴式数字PCR技术不依赖于标准曲线,实现了DNA检测从相对定量到绝对定量的飞跃[8-14]。QX200微滴式数字PCR系统(QX200TMDroplet DigitalTMPCR)获得了欧盟体外诊断(in vitro diagnostic,IVD)产品 CE认证(CE IVD marking),这是该领域首个获得CE认证的数字PCR系统。QX200 ddPCR系统可以直接从分析器同时读取外源基因和内标基因的拷贝数,快速计算出外源基因的百分含量,测定过程更为方便和快捷。
目前,尚无QX200 ddPCR技术应用于转基因生物外源基因检测分析的相关报道。本试验研究转基因大豆GTS-40-3-2的微滴式数字QX200 PCR检测方法,以期为转基因生物中外源基因拷贝数和外源基因含量的精确测定提供一种新方法。
1 材料与方法
1.1 材料
转基因大豆GTS-40-3-2阳性品(100%)和转基因大豆GTS-40-3-2标准品(转基因大豆GTS40-3-2含量为10%)购于European Reference Materials公司,Quant-iTTMPicoGreen©dsDNA Kit购于美国Invitrogen公司,ddPCR Master Mix、Droplet Generation Oil、Droplet Reader Oil购于美国 Bio-Rad公司,DNeasy Plant Mini Kit购于天根生化科技有限公司。
1.2 仪器
Nanodrop ND-1000核酸蛋白定量仪(Thermo Scientific,美国),QX200TMDroplet DigitalTMPCR系统(Bio-Rad,美国,包括微滴生成仪和微滴分析仪),ABI StepOne Plus定量 PCR仪(Applied Biosystem,美国)。
1.3 方法
1.3.1 DNA提取和纯化
转基因大豆标准品使用DNeasy Plant Mini Kit纯化,具体步骤见试剂盒说明书。提取的基因组DNA使用Nanodrop ND-1000核酸蛋白定量仪测定纯度和浓度(步骤略)。
1.3.2 引物和探针
涉及的PCR扩增引物和探针序列参照GB/T 19495.5—2004《转基因产品检测核酸定量PCR检测方法》,由生工生物工程(上海)股份有限公司合成,具体序列见表1。

表1 引物和探针序列Table 1 Sequences of primers and probes
1.3.3 qPCR反应体系和条件
转基因大豆GTS40-3-2阳性品提取的DNA含量为1.762×105copies/μL,按照10倍梯度稀释为:1.762×104copies/μL、1.762×103copies/μL、1.762×102copies/μL、1.762×101copies/μL,用于构建实时荧光定量PCR标准曲线。实时荧光定量PCR反应体系和反应条件参见GB/T 19495.5—2004《转基因产品检测核酸定量PCR检测方法》。每个模板重复3个平行扩增。
1.3.4 ddPCR反应体系和条件
ddPCR反应分为4个步骤:PCR体系配制、微滴生成、PCR扩增和微滴分析。ddPCR扩增体系为20μL,包含10μL 2×ddPCR Master Mix,10μmol/L正向引物和反向引物各1.8μL(2对)、探针0.5μL(2条),DNA模板1μL,H2O 0.8μL。将20μL PCR Mix和70μL微滴生成油加入微滴生成卡中生成微滴,将生成的微滴混合液加入96孔板中,用锡箔纸密封后置入ABI-PCR仪中运行。PCR扩增程序:95℃预变性10 min;94℃变性30 s,59℃退火1 min,40个循环;98℃固化10 min。每个模板重复3个平行检测。扩增结束后,将96孔板置入微滴读取仪中读取信号,有荧光信号的微滴为阳性,无荧光信号的为阴性,软件统计每个样品里阳性和阴性微滴数,并使用软件QuantaSoft Version 1.7分析数据。
1.3.5 实时荧光定量PCR计算转基因成分的含量
内标基因拷贝数已知,因此首先根据内标基因的荧光数据构建标准曲线,按照公式:转基因成分的含量=外源基因拷贝数/内标基因拷贝数,计算出转基因成分的含量[15]。用10%的标准品作为待测样品进行测定。
1.3.6 ddPCR计算外源基因拷贝数和含量
ddPCR采用直接计数的方法进行拷贝数分析。PCR扩增结束后,认为有荧光信号反应单元中至少包含1个拷贝的目标分子,整体符合泊松分布[2,16-17]。根据数字PCR反应总微滴数和有荧光信号的微滴数以及样品的稀释倍数,就可以得到样本最初的浓度和拷贝数。用10%的标准品作为待测样品进行测定。
2 结果与分析
2.1 qPCR拷贝数分析结果
根据不同浓度模板测得的荧光值构建标准曲线,如图1所示。内标基因标准曲线的回归方程为:C t=-3.2318x+36.631,线性相关系数R2为0.999。外源基因标准曲线的回归方程为:C t=-3.3251x+37.082,线性相关系数R2为0.998。把待测标准品测得的C t值代入两个标准曲线中,再通过1.3.5的公式计算出转基因大豆的含量为10%,与标准品实际转基因成分的含量一致。

图1 内标基因和结构特异性基因的标准曲线Fig.1 Standard curves of the endogenous reference gene and structure-specific gene
2.2 ddPCR分析
2.2.1 ddPCR信号识别
有效的微滴生成数目是ddPCR的关键,只有生成的微滴数大于8 000时,目标分子的分布才能适用于泊松分布的统计学原理,才能准确计算出目标分子的拷贝数[18-20]。ddPCR通过扩增形成的阳性微滴和阴性微滴如图2和表2所示。由表2可见,反应中形成的微滴数为10 863—13 859,均大于10 000,满足ddPCR微滴的分析要求。由图2可见,阴性微滴和阳性微滴明显分为两个区域,ddPCR系统可准确检测出阳性微滴和阴性微滴的数目。根据每个反应生成的微滴数来看,本研究建立的ddPCR体系微滴生成稳定,具有良好的重复性,能够用于转基因大豆GTS40-3-2外源基因拷贝数的分析。
2.2.2 转基因含量分析
根据软件读取的外源基因和内标基因的微滴数,便可计算出外源基因和内标基因的浓度,通过1.3.5的公式,计算出外源基因的拷贝数,分析最低检测限;同时可计算出待测样品转基因成分的含量(表2)。
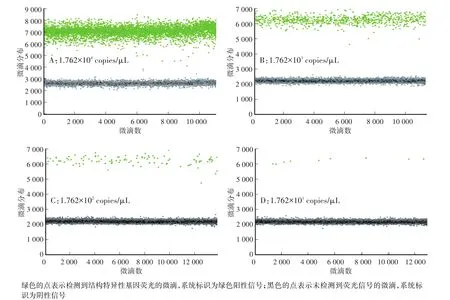
图2 微滴式数字PCR测定结构特异性基因Fig.2 Detection of structure-specific gene by ddPCR

图3 微滴式数字PCR测定Lectin基因Fig.3 Detection of Lectin gene by ddPCR
从表2可见,生成的微滴数最少为10 863,最多为13 859,完全满足ddPCR数据分析的需要,并且可计算出ddPCR方法的最低检出限约为2个拷贝/(20μL)。在4个稀释梯度下,均能测出转基因成分的含量,前3个稀释梯度的转基因成分的含量非常接近10%,最后一个稀释梯度由于模板含量太低,转基因成分含量的计算值出现一定的偏差。如果外源基因和内标基因的拷贝数关系为1∶1,那么可以从微滴读取器的界面通过微滴数直接算出转基因成分的含量。如本研究中,测定外源结构特异性基因的微滴数为78,测定内标基因Lectin的微滴数为774,待测样品的含量为78/774=10.07%.

表2 ddPCR基因拷贝数分析结果Table 2 Genetic copy number analysis by ddPCR
3 讨论
随着转基因技术的广泛应用,如何精准测定转基因植物、动物的外源基因拷贝数以及计算转基因成分的含量,已经成为现今转基因产品检测技术研究的一个关键点[19-20]。传统的Southern Blot方法分析基因拷贝数所需的周期长、工作量大、准确性较差。qPCR是目前最常用的转基因成分检测技术[21],使用qPCR进行外源基因的拷贝数分析必须依赖标准品或者构建标准质粒分子,PCR体系也需要摸索和优化,对未知品浓度的计算依赖于标准曲线的构建,因此是一种相对定量的分析方法,结果并非十分精确[20]。
本研究同时使用qPCR和ddPCR对10%的标准品进行了转基因成分含量的测定。相比ddPCR方法,qPCR需要更多的模板量和引物,加样的数量也比较多,容易发生错误。采用QX200TMDroplet DigitalTMPCR系统,将两套引物和探针加入到同一PCR管中,减少了加样数量,也节省了模板量。在数据分析方面,qPCR方法需要根据荧光数值构建标准曲线,然后通过计算获得基因的拷贝数和转基因成分的含量;而ddPCR方法可以在电脑的分析软件上直接读取数据,相比之下,ddPCR方法所需时间更短,获取数据也简单便捷。此外,ddPCR方法检测限可以达到2个拷贝/(20μL),ddPCR在线性范围、检测极限和定量极限等方面都更胜qPCR。ddPCR方法的优点获得越来越多的认可[22-24],在临床方面为癌症、肿瘤等疾病检测提供了新的诊断工具,被用于基因拷贝数变化分析、基因突变检测、基因的分型等,并取得了突破性的进展[25-30]。
在转基因检测方面,Corbisier等[11]利用数字PCR分析了玉米种子DNA中外源基因的拷贝数,显示与利用qPCR方法检测的结果相同。姜羽等[20]利用ddPCR技术分析了转基因水稻T1c-19和转人乳铁蛋白基因山羊134外源基因的拷贝数,并与qPCR和Southern Blot方法的检测结果进行比较,结果表明:T1c-19杀虫晶体蛋白基因的qPCR和ddPCR测定结果比较一致,约为2个拷贝;转人乳铁蛋白基因山羊134的qPCR和ddPCR的分析结果比较一致,HLF基因的拷贝数约为1[20]。
本研究通过建立的QX200 ddPCR体系,分析了转基因大豆GTS-40-3-2外源构建特异性基因。ddPCR不依赖标准曲线,能够通过不同荧光的微滴数目直接获得靶基因的拷贝数,并精确计算出转基因成分的含量;能够有效节省试剂和样品、具有更高的灵敏度,是一种精确、灵敏的转基因生物外源基因拷贝数分析和含量测定的新方法。
[1]HINDSON B,NESSK,MASQUELIER D,et al.High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J].Analytical Chemisty,2011,83(22):8604-8610.
[2]林彩琴,姚波.数字 PCR技术进展[J].化学进展,2012,24(12):2415-2423.
[3]SANDERSR,HUGGETT JF,BUSHELLCA,et al.Evaluation of digital PCR for absolute DNA quantification[J].Analytical Chemistry,2011,83(17):6474-6484.
[4]ZIMMERMANN B G,GRILL S,HOLZGREVE W,et al.Digital PCR:a powerful new tool for noninvasive prenatal diagnosis[J].Prenatal Diagnosis,2008,28(12):1087-1093.
[5]HINDSON CM,CHEVILLET JR,BRIGGSH A,et al.Absolute quantification by droplet digital PCR versus analog real-time PCR[J].Nature Methods,2013,10(10):1003-1005.
[6]VAUCHERETH,BECLIN C,ELMAYAN T,et al.Transgene-induced gene silencing in plants[J].The Plant Journal,1998,16(6):651-659.
[7]CANKAR K,ŠTEBIH D,DREO T,et al.Critical points of DNA quantification by Real-time PCR-effects of DNA extractionmethod and sample matrix on quantification of geneticallymodified organisms[J].BMC Biotechnology,2006,6(4):37-52.
[8]柳方方,张玲,王晶,等.数字PCR测定DNA含量及测量结果不确定度评定[J].化学分析计量,2013,22(1):18-22.
[9]BURNSM J,BURRELL A M,FOY C A.The applicability of digital PCR for the assessment of detectionlimits in GMO analysis[J].Eur Food Res Technol,2010,231(3):353-362.
[10]HUA Z S,JEREMY L R,ALLEN E,et al.Multiplexed real-Time polymerase chain reaction on a digital microfluidic platform[J].Analysis Chemical,2011,82(6):2310-2316.
[11]PHILIPPEC,SOMANATH B,LINA P,etal.Absolute quantification of geneticallymodified MON810maize(Zeamays L.)by digital polymerase chain reaction[J].Anal Bioanal Chem,2010,396(6):2143-2150.
[12]PINHEIRO L B,COLEMAN V A,HINDSON CM,et al.Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification[J].Anal Chem,2012,84(2):1003-1011.
[13]李亮,隋志伟,王晶,等.基于数字PCR的单分子DNA定量技术研究进展[J].生物化学与生物物理进展,2012,39(10):1017-1023.
[14]胡伟,陈荣华,张晨,等.微滴式数字PCR技术用于生物样品种属鉴定和绝对定量[J].法医学杂志,2014,30(5):342-345.
[15]WENG H,PAN A,YANG L,et al.Estimating number of transgene copies in transgenic rapeseed by Real-time PCR assay with HMG I/Y as an endogenous reference gene[J].Plant Molecular Biology Reporter,2004,22(3):289-300.
[16]洪来法.基于核酸精准检测的微滴式数字PCR检测技术[C]//新发传染病研究热点研讨会论文集.2012.
[17]HINDSON B J,NESSK D,MASQUELIER D A,etal.High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J].Anal Chem,2011,83(22):8604-8610.
[18]MORISSETD,STEBIH D,MILAVECM,et al.Quantitative analysis of food and feed sampleswith droplet digital PCR[J].PLoSOne,2013,8(5):e62583.
[19]ZHANG D,GUO J.The development and standardization of testingmethods for geneticallymodified organisms and their derived products[J].Journal of Integrative Plant Biology,2011,53(7):539-551.
[20]姜羽,胡佳莹,杨立桃.利用微滴数字PCR分析转基因生物外源基因拷贝数[J].农业生物技术学报,2014,22(10):1298-1305.
[21]仇有文,高学军,张明辉,等.抗除草剂转基因大豆插入拷贝数及其旁侧序列分析[J].生物技术,2011,21(6):31-35.
[22]万艳,李丽玲,陈小佳.应用实时荧光定量PCR检测外源基因拷贝数的新方法[J].暨南大学学报(自然科学与医学版),2009,30(3):310-312.
[23]HAYDEN R,GU Z,INGERSOLL J,etal.Comparison of droplet digital PCR to Real-time PCR for quantitative detection of cytomegalovirus[J].Journal of Clinical Microbiology,2013,51(2):540-546.
[24]SEDLAK R,JEROME K.Viral diagnostics in the era of digital polymerase chain reaction[J].Diagnostic Microbiology and Infectious Disease,2013,75(1):1-4.
[25]WHALE A,HUGGETT J,COWEN S,et al.Comparison of microfluidic digital PCR and conventional quantitative PCR for measuring copy number variation[J].Nucleic Acids Research,2012,40(11):e82.
[26]董莲华,张玲,姜君,等.大肠杆菌 O157:H7微滴数字 PCR定量方法的建立[J].分析化学研究报告,2015,43(3):319-324.
[27]杨冬燕,杨永存,李浩,等.双重 PCR鉴别牛、羊肉掺假[J].中国卫生检验杂志,2014,24(23):3379-3382.
[28]王珊,李志娟,苗丽.微滴式数字PCR与实时荧光PCR检测羊肉制品中羊源和猪源性成分方法的比较[J].肉类工业,2015(7):38-41.
[29]王青,刘宝瑞.液滴数字PCR在肿瘤个体化治疗方面的应用[J].现代肿瘤医学,2015,23(12):1771-1774.
[30]吴斌,申翠翠,张晨曦,等.数字RT-PCR检测鲤春病毒血症的方法建立与应用[J].中国动物检疫,2015,32(7):72-76.
