特香型白酒酿造过程中真核微生物菌群演替
2017-11-13李凯敏付桂明吴酬飞刘成梅郑福平
李凯敏,付桂明,*,吴酬飞,刘成梅,万 茵,潘 菲,郑福平*
(1.南昌大学 食品科学与技术国家重点实验室,食品学院,江西 南昌 330047;2.湖州师范学院生命科学学院,浙江 湖州 313000;3.北京工商大学食品学院,北京 100048)
特香型白酒酿造过程中真核微生物菌群演替
李凯敏1,付桂明1,*,吴酬飞2,刘成梅1,万 茵1,潘 菲1,郑福平3,*
(1.南昌大学 食品科学与技术国家重点实验室,食品学院,江西 南昌 330047;2.湖州师范学院生命科学学院,浙江 湖州 313000;3.北京工商大学食品学院,北京 100048)
采用高通量测序技术对特香型白酒酿造过程中的真核微生物菌群演替动态变化进行分析。方法:分别于发酵窖池内采集底糟样品(0、30 d)和表层与下层酒醅样品(0、3、6、10、15、20、25、30 d),洗脱表面微生物,提取基因组DNA,进行聚合酶链式反应扩增和高通量测序分析。结果表明:在整个发酵周期内,酒醅中的真核微生物菌群多样性与丰度呈现明显下降趋势,优势菌目为Saccharomycetales(酵母目),优势菌属依次为Saccharomyces(酵母属)、Pichia(毕赤酵母)、Galactomyces(耐碱酵母属);相比酒醅而言,底糟真核微生物菌群构成更为复杂,主要优势菌群包括Saccharomycetales(酵母目)、Eurotiales(散囊菌目)、Capnodiales(煤炱目)和Tremellales(银耳目)等;对整个发酵过程中的菌群结构进行主坐标分析,结果显示表层酒醅与下层酒醅在真核微生物菌群结构上没有明显的差异。
特香型白酒;发酵过程;真核微生物;多样性
白酒作为我国传统优势蒸馏酒,以谷物为原料,采用大曲、小曲或麸曲为发酵剂,用双边或固态糖化发酵,蒸馏取酒,经过贮存、勾兑生产出香气浓郁的蒸馏酒,是世界著名的六大蒸馏酒之一,因酿酒原料、地理位置与酿造工艺的差异,所酿白酒呈现出不同的风味特点[1];按香型可分为浓香型、清香型、酱香型、特香型[2]等。
特香型白酒是一种“浓头酱尾清中间”、“三香具备尤不靠”的特殊香型,以四特、樟树贡、临川贡酒为主要代表。其酿造工艺不同于其他白酒,是以红锗条石建造窖池,“三进四出”的续渣发酵,混蒸混烧“四甑”生产工艺,以优质大米为原料进行酿酒[3-4]。特香型白酒窖池底部铺有一层酒醅称为底糟,底糟要在窖池底部发酵1 a以上才会启封蒸酒,且底糟所蒸白酒香气浓郁,常作为特香型白酒产品的调味酒。研究表明,微生物在白酒的酿造过程中发挥着重要作用[5],分析酿酒过程中微生物菌群演替可以了解白酒的发酵机理,解析酿酒过程中的功能微生物[6]。传统菌群分析采用涂布分离纯菌株,再进行分子鉴定,生理生化鉴定,操作复杂,且最大的弊端在于绝大多数的微生物是不能够被培养或者很难被培养,大量菌群的遗漏导致检测结果不准确[7]。近年来,16S rRNA克隆建库[8]、变性梯度凝胶电泳[9]、温度梯度凝胶电泳[10]、高通量测序(high throughput sequencing,HTS)[11]等分子生物学技术的发展为环境微生物的检测提供了条件。HTS是一种全新的测序技术,它能一次对多个样品进行分析,具备一次对几十万到几百万条DNA分子同时进行测序的能力,节省了诸多繁琐的实验步骤[12]。该方法具有通量高、快速、低成本等优点,广泛应用于不同的环境中,例如土壤[13]、湖泊淤泥[14]等生态环境。近年来,HTS也逐步应用于传统发酵食品中菌群多样性研究,如香肠[15]、米酒[16]、醋[17]等发酵食品。
白酒酿造过程中存在大量微生物,其中霉菌、酵母菌等真核微生物在发酵过程中扮演重要角色。丝状真菌产生大量淀粉酶、糖化酶、蛋白酶等,可以大量水解淀粉与蛋白质,提高消化率与生理活性,甚至一些真菌对白酒风味物质的合成有很大贡献[18-19];酿酒酵母发酵可产生大量乙醇,而一些非酿酒酵母则可产生多种香气成分[20]。目前有关特香型白酒微生物的研究较少,尚鲜见对特香型白酒酿造过程中真核微生物菌群的相关研究报道。本研究采用宏基因组学对特香型白酒酿造过程中酒醅与底糟中的真核微生物群落结构进行分析,为解析特香型白酒酿造过程提供理论依据。
1 材料与方法
1.1 材料与试剂
Soil DNA kit试剂盒 北京天根生化科技有限公司;DNA Marker 宝生物工程(大连)有限公司。
1.2 仪器与设备
聚合酶链式反应(polymerase chain reaction,PCR)仪赛默飞世尔科技有限公司;DYY-8C电泳仪 北京六一仪器公司;HTS仪 美国Illum ina公司。
1.3 方法
1.3.1 样品采集
采样点为江西樟树贡酒厂的生产窖池,取样时间为2016年4—5月,样品发酵时间分别为0 d(酒醅入窖当天)、3、6、10、15、20、25 d和30 d;窖池深度2 m;酒醅按纵向分表层(0~20 cm)与下层(80~180 cm)、窖池底糟(180~200 cm),按横向分中心区域(中心区75 cm以内)和周边区域(距池边5~20 cm环带);发酵期间内窖池密封,取样时使用取样器通过取样口直接插入酒醅进行取样,不破坏窖池整体的密封环境,同区域取6 点样均匀混合;混匀后,置无菌容器中于-80 ℃冰箱保藏。
1.3.2 样品基因组DNA提取
分别称取酒醅样品2 g,用无菌水对微生物进行洗脱,并用玻璃珠破碎法,对样品进行破碎处理,采用Soil DNA kit试剂盒进行提取宏基因组DNA。
1.3.3 目的区段扩增及高通量测序
根据实验设计对18S rRNA基因进行扩增,采用引物SSU 1196R:TCTGGACCTGGTGAGTTTCC和SSU0817F:TTAGCATGGAATAATRRAATAGGA[21]。为了对所有样品进行区分,上游引物前加一段barcode序列。PCR扩增程序:95 ℃预变性3 m in,95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,30 个循环;最后72 ℃延伸10 m in。PCR体系参见Liu Wenjun等[22]报道,PCR扩增结束后,产物经2%琼脂糖凝胶电泳纯化,并对样本浓度定量,送至上海美吉生物医药科技有限公司,利用M iSeq PE300平台进行高通量测序分析。
1.4 数据分析
用M othur软件以97%为划定阈值对序列划分操作分类单元(operational taxonom ic units,OUT),根据各样品物种丰富度情况,利用得出的OTU结果计算样品中生物多样性指数、丰富度指数(Chao、ACE指数)、多样性指数(Shannon、Sim pson指数)、覆盖率指数(Coverage)等。此外,在测序前需要将各序列的引物和barcode去掉,并对序列长度进行筛选,以减少错误的测序,确保测序质量[12]。经过处理后的序列与SILVA数据库进行比对,然后基于分类学信息,在各个分类水平上进行群落结构的统计分析。使用加权UniFrac距离算法进行主坐标分析(principal coordinates analysis,PCoA)[23]。
2 结果与分析
2.1 18S rDNA的扩增结果

图1 基因组DNA的PCR扩增产物电泳图Fig. 1 Electrophoretogram of PCR amplification products of genomic DNA
图1是以提取的样品宏基因组DNA为模板,对18S rDNA进行PCR扩增,由琼脂糖凝胶电泳后的成像图可以看出,17 个样品长度均保持在550 bp左右,与目标片段相一致。
2.2 样本序列及微生物多样性分析
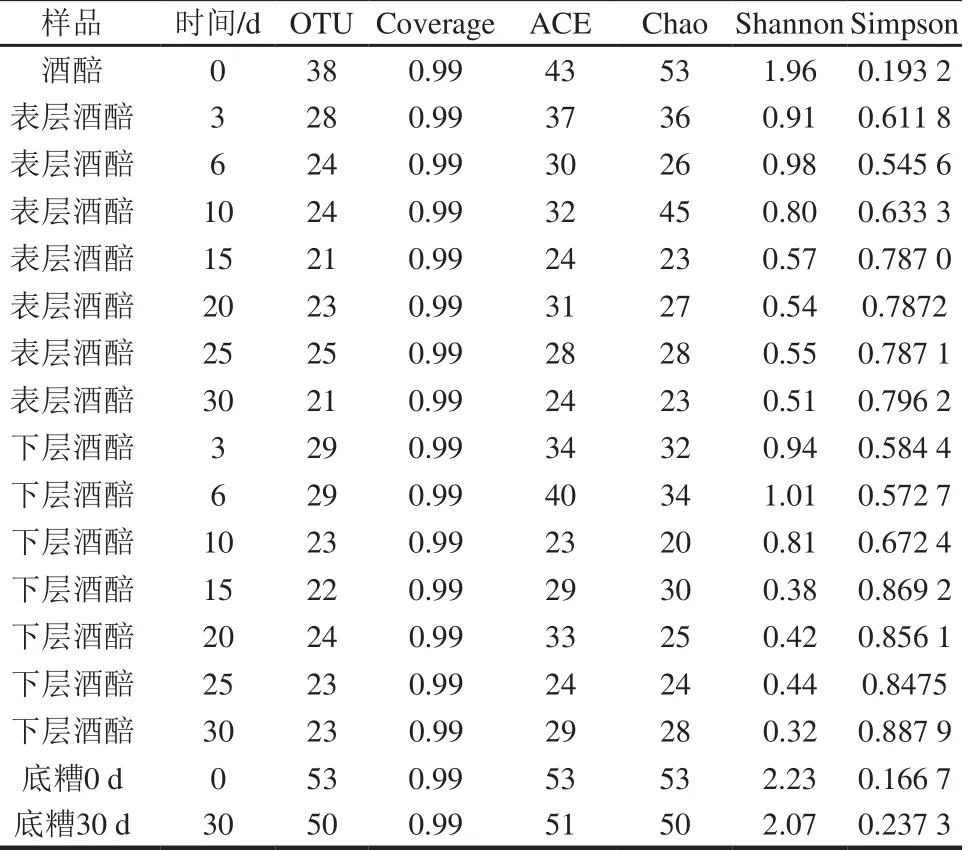
表1 样品多样性指数Table 1 Alpha-diversity indexes of m icrobial communities from fermented grain samp les
如表1所示,所有样本的Coverage指数均达到0.99,证明本次测序覆盖了样本中绝大部分真核微生物,数据结果准确可靠。结果表明:底糟的Shannon指数最高,达到2.23(Shannon指数越高,多样性越高),说明底糟样品中的微生物多样性要远高于发酵期酒醅,可能是由于底糟不参与规律性的蒸馏出酒,经过多轮、长时间发酵,其微生物种类丰富、数量庞大[24]。从真核微生物多样性上来看,发酵周期的前10 d,窖池内下层酒醅与表层酒醅样品的微生物多样性没有明显差异,从发酵10 d到发酵周期结束,窖池表层酒醅的微生物多样性要明显高于下层样品。这可能是由于发酵前期窖池内整体变化保持一致,使得表层与下层酒醅真核微生物没有较大的差异;到发酵中后期窖池处于低pH值、低氧含量、高酒精度等“不利”环境中,且随窖池深度增加,酸和酒精含量逐渐增高、氧含量逐渐降低[25],对微生物的抑制作用增强,以至于真核微生物多样性越来越低。

图2 不同发酵时期真核微生物在属水平上的数目变化情况Fig. 2 Changes in eukaryotic m icroorganism s during different fermentation periods at genus level
图2表明,刚入池发酵的酒醅样品(0 d)中真核微生物多样性最为复杂,OTU数达到28 种,随着发酵的进行,酒醅中真核微生物种类呈逐渐递减趋势。这是由于刚入窖的酒醅中不仅含有大曲,还包含大米,稻壳,酒糟等酿酒原料,这些原料中富含大量的微生物,因此发酵0 d的酒醅中的微生物种类最高[18,26]。
发酵启动阶段(0、3 d),真核微生物多样性出现大幅度下降,可能原因是微生物开始发酵后,由于采用隔绝空气的厌氧发酵,氧气含量均大幅度减少,大量好氧菌生长受到抑制,真核微生物多样性显著降低。随后进入酿酒开始阶段(3、6、10 d),真核微生物趋于稳定,没有产生明显的下降。这是由于能够进行厌氧发酵而存活的酵母菌等真核微生物经过数天适应,开始大量生长,并开始发酵产生酒精等各种代谢产物,但积累量不大,不足以对酒醅理化性质进行大幅度改变,对微生物的抑制作用不强,所以该阶段真核微生物菌群多样性相对复杂且保持稳定[26]。
随着发酵的进行,进入发酵中后期(15、20、25、30 d),菌群多样性开始逐步降低,直到发酵周期结束后,真核微生物只有10 种存在。这是由于发酵进入了中后期,酒醅中的营养物质与氧气消耗殆尽,同时积累了大量代谢产物,例如酿酒酵母生成大量的酒精、丝状霉菌产生柠檬酸,醋酸菌和乳酸菌分别分泌产生醋酸、乳酸[26-27]等。这些代谢产物造成酒醅中微生物处于低pH值和高酒精环境中,生长受到抑制,使酒醅中真核微生物菌群变得更加单一。
2.3 发酵过程中酒醅优势真核微生物变化情况分析

图3 门水平(a)、目水平(b)、属水平(c)上各样品真核微生物菌群结构Fig. 3 Fungal communities of fermented grain samp les at phylum level (a),order level (b) and genus level (c)
对整个发酵周期的酒醅进行鉴定分析,门水平真核微生物菌群结构如图3a所示,在酒醅入窖(0 d),主要真核微生物是由Ascomycota(子囊菌门,72.6%)、Zygomycota(接合菌门,24.6%)和Basidiomycota(担子菌门,2.3%)构成;随着发酵时间的延长,从发酵3 d起,Ascomycota成为酒醅中的唯一的优势真核菌门。结果表明整个发酵周期内酒醅真核微生物菌群结构在门水平上较稳定。
如图3b所示,在整个发酵过程中,Saccharomycetales(酵母目)与Eurotiales(散囊菌目)构成了主要的酒醅真核微生物菌群。Saccharomycetales从发酵0 d的52.9%,3 d内迅速升高至97.1%且维持到发酵结束;与此同时Eurotiales由发酵开始的19.2%迅速下降到2.1%,并随着发酵时间的延伸继续下降。由此可见,Saccharomycetales是整个酿酒过程中的绝对优势菌,可能是由于酒醅发酵过程处于密封环境,进行厌氧固态发酵,且发酵后期酒醅环境恶劣,氧气含量严重不足,酵母菌相比其他真核微生物具有更高的耐受性[20]。
如图3c所示,在属水平上Saccharomyces(酵母属)、Galactomyces(耐碱酵母)和Pichia(毕赤酵母)是酿造过程中的优势菌群;Saccharomyces具有发酵能力强,高产酒精能力,是酿酒过程中最主要的功能菌株;Pichia是酿酒过程中重要的产酯微生物[27];Galactomyces(耐碱酵母)、Aspergillus(曲霉属)和Thermoascus(热子囊菌)随发酵时间的延长呈现明显下降趋势,到发酵结束后丰度仅存2.9%、0.3%和0.1%左右;部分真核微生物在发酵中后期才被检测出,例如Brettanomyces(生香酵母),发酵前期(0~10 d)未检出,于发酵增香阶段(15~25 d)才被检测到;生香酵母是一类产酯酵母,是中国白酒风味中酯香的主要产生菌之一;有研究表明于特香型白酒酒醅中加入一定量的生香酵母可以有效提高所酿白酒香味成分的含量,且不会对原白酒风味产生任何不利影响,更有助于香味物质的积累[28]。
2.4 底糟真核微生物分析
对窖池底糟进行高通量测序分析,发现两个底糟样品微生物菌群结构、多样性上没有明显的差异,仅在某些微生物的丰度上略有变化。结果表明Ascomycota(子囊菌门)、Zygom ycota(接合菌门)和Basidiom ycota(担子菌门)是主要的优势菌门(图3a);Ascomycota序列数高达90%以上,其次是Basidiomycota。共有25 个目的真核微生物被检测到(图3b),其中Saccharomycetales(酵母目)、Eurotiales(散囊菌目)、Capnodiales(煤炱目)和Tremellales(银耳目)是优势菌目。经过一轮酿酒发酵,Capnodiales丰度上稍有提高,而Saccharomycetales略有下降,其余菌群均未发生明显改变。
图3a表明,无论是酒醅还是底糟,Ascomycota均是其中真核微生物的主导门类,但是相较酒醅而言,底糟中含有更高丰度的Basidiomycota,主要原因是底糟长期处于窖池底层,进行厌氧发酵,水分含量高,更利于担子菌的生长[29]。如图3b所示,Saccharom ycetales虽在底糟样品中也有最高的丰度,但要远低于其在酒醅样品中所占比例;Eurotiales和Capnodiales在底糟样品中分别达到21.2%和6.2%,远超其在酒醅中所占比例;且诸多真核微生物在仅在底糟中被检测,例如Capnodiales、Cystofilobasidiales、Leucosporidiales、Liliopsida、Malasseziales。
在属水平上(图3c),Aspergillus(曲霉属)、Brettanomyces(生香酵母属)、Cladosporium(芽枝霉属)、Galactomyces(耐碱酵母属)、Thermoascus(热子囊菌属)等菌群在底糟中占到较高的比例。由于底糟微生物菌群构成丰富,富含大量风味成分,其蒸馏物作为特香型白酒产品的调味酒体,可以有效的增强产品酒的风味,使其后味爽净,诸香协调,更具特色。
2.5 PCoA结果
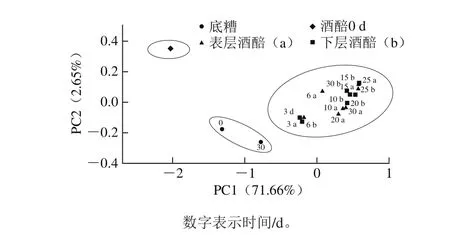
图4 所有样品真核微生物菌群的PCoAFig. 4 PCoA of fungal communities in all sam p les obtained by using high throughput sequencing
PCoA可通过降维找出影响样本群落组成差异的潜在主成分,用来研究样本群落组成的相似性或差异性[23]。本研究通过PCoA对整个特香型白酒发酵过程中表层酒醅、下层酒醅和底糟的真核微生物菌群结构的相似性进行评估,如图4所示,所有样品在PCoA图上分为3 个集群;酒醅(0 d)、酒醅(3~30 d)与底糟三者各集中于一簇,表现出明显的差异;在整个固态发酵过程中,3~30 d表层与下层酒醅的真核微生物菌群结构没有明显的差异,表明特香型白酒酒醅真核微生物菌群构成不随窖池深度的改变而发生明显变化,具有整体性。
3 结 论
本研究采用高通量测序方法系统分析了特香型白酒酿造过程中表层酒醅、下层酒醅和底糟中的真核微生物菌群多样性变化及其优势菌群演替过程。结果发现在整个发酵过程中,Ascomycota是酒醅真核微生物的优势菌门,由Saccharomycetales与Eurotiales构成,其中前者作为酒醅微生物的优势菌目,主要包括Pichia、Galactomyces和Saccharomyces;且整个发酵过程中,表层酒醅与下层酒醅在真核微生物菌群结构上不存在显著差异。
对底糟样品进行测序分析,结果表明:相较酒醅而言,底糟中真核微生物菌群更为丰富、复杂,主要包括Ascomycota、Basidiom ycota两个优势菌门和Saccharomycetales、Eurotiales、Capnodiales、Tremellales 4 个优势菌目。底糟所酿白酒作为特香型白酒产品酒的调味剂,更具特香型白酒特色,风味物质含量非常丰富,对其进行进一步分析来提高特香型白酒的风味非常有意义。对特香型白酒酿造过程中真核微生物的菌群结构和优势菌的动态变化进行研究,对解释特香型白酒产香机理、指导白酒生产、稳定产品质量具有重要的理论指导和实践价值。在后续实验研究中,可对发酵周期内酒醅的理化性质和风味成分做进一步分析,结合酒醅中微生物菌群的演替,为解释特香型白酒产香机理提供理论支持。
[1] ZHENG X W, HAN B Z. Baijiu (白酒), Chinese liquor: history,classifi cation and manufacture[J]. Journal of Ethnic Foods, 2016, 3(1):19-25. DOI:10.1016/j.jef.2016.03.001.
[2] 赵爽, 杨春霞, 窦岫. 白酒生产中酿酒微生物研究进展[J]. 中国酿造,2012, 31(4): 5-10.
[3] 严伟, 吴生文, 刘建文, 等. 特香型年份酒香味物质初探[J]. 酿酒科技, 2013(8): 54-57.
[4] 章肇敏, 吴生文. 特香型白酒酿造工艺分析[J]. 中国酿造, 2012,31(5): 164-167.
[5] LI X R, MA E B, YAN L Z, et al. Bacterial and fungal diversity in the traditional Chinese liquor fermentation process[J]. International Journal of Food M icrobiology, 2011, 146(1): 31-37. DOI:10.1016/j.ijfoodm icro.2011.01.030.
[6] WANG L, WANG Y Y, WANG D Q, et al. Dynam ic changes in the bacterial community in Moutai liquor fermentation process characterized by deep sequencing[J]. Journal of the Institute of Brew ing, 2015, 121(4): 603-608. DOI:10.1002/jib.259.
[7] SUN W, XIAO H, PENG Q, et al. Analysis of bacterial diversity of Chinese Luzhou-fl avor liquor brewed in different seasons by Illum ina M iseq sequencing[J]. Annals of M icrobiology, 2016, 66(3): 1-9.DOI:10.1007/s13213-016-1223-5.
[8] LI K, ZHANG Q, ZHONG X T, et al. M icrobial diversity and succession in the Chinese Luzhou-flavor liquor fermenting cover lees as evaluated by SSU rRNA profi les[J]. Indian Journal of M icrobiology,2013, 53(4): 425-431. DOI:10.1007/s12088-013-0382-3.
[9] X IONG X, HU Y, YAN N, et al. PCR-DGGE analysis of the m icrobial communities in three different Chinese “Baiyunbian” liquor fermentation starters[J]. Journal of M icrobiology & Biotechnology,2014, 24(8): 1088. DOI:10.4014/jmb.1401.01043.
[10] HUR J M, PARK D H. Making soy sauce from defatted soybean meal without the mejus process by submerged cultivation using thermophilic bacteria[J]. Journal of Food Science and Technology, 2015, 52(8):5030-5038. DOI:10.1007/s13197-014-1536-y.
[11] LI P, LIN W, LIU X, et al. Effect of bioaugmented inoculation on m icrobiota dynam ics during solid-state fermentation of Daqu starter using autochthonous of Bacillus, Pediococcus, Wickerhamomyces and Saccharomycopsis[J]. Food M icrobiology, 2017, 61: 83-92.DOI:10.1016/j.fm.2016.09.004.
[12] METZGER J, TONDA R, BELTRAN S, et al. Next generation sequencing gives an insight into the characteristics of highly selected breeds versus non-breed horses in the course of domestication[J].BMC Genom ics, 2014, 15(1): 1-13. DOI:10.1186/1471-2164-15-562.
[13] ZHOU X, LI S, LI W, et al. M yxobacterial community is a predom inant and highly diverse bacterial group in soil niches[J].Environm en tal M ic robio logy Repo rts, 2014, 6(1): 45-56.DOI:10.1111/1758-2229.12107.
[14] LI S, ZHOU X, LI P, et al. The existence and diversity o f m yxobacteria in lake mud-a previously unexp lored m yxobacteria habitat[J]. Environmental M icrobiology Reports, 2012, 4(6): 587-595.DOI:10.1111/j.1758-2229.2012.00373.x.
[15] POLKA J, REBECCHI A, PISACANE V, et al. Bacterial diversity in typical Italian salam i at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J]. Food M icrobiology, 2015, 46: 342-356. DOI:10.1016/j.fm.2014.08.023.
[16] WANG P, MAO J, MENG X, et al. Changes in flavour characteristics and bacterial diversity during the traditional fermentation of Chinese rice w ines from Shaoxing region[J]. Food Control, 2014, 44: 58-63.DOI:10.1016/j.foodcont.2014.03.018.
[17] NIE Z, ZHENG Y, WANG M, et al. Exploring m icrobial succession and diversity during solid-state fermentation o f Tian jin duliu mature vinegar[J]. Bioresource Technology, 2013, 148(8): 325-333.DOI:10.1016/j.biortech.2013.08.152.
[18] LV X C, HUANG Z Q, ZHANG W, et al. Identification and characterization of filamentous fungi isolated from fermentation starters for Hong Qu glutinous rice w ine brew ing[J]. The Journal of General and Applied M icrobiology, 2012, 58(1): 33-42. DOI:10.2323/jgam.58.33.
[19] LV X C, CA I Q Q, KE X X, et al. Characterization of fungal community and dynam ics during the traditional brew ing of W uyi Hong Qu glutinous rice w ine by means of multiple culture-independent m ethods[J]. Food Control, 2015, 54: 231-239. DOI:10.1016/j.foodcont.2015.01.046.
[20] WU Q, CHEN L, XU Y. Yeast community associated w ith the solid state fermentation of traditional Chinese Maotai-flavor liquor[J].International Journal of Food M icrobiology, 2013, 166(2): 323-330.DOI:10.1016/j.ijfoodmicro.2013.07.003.
[21] ROUSK J, BAATH E, BROOKES P C, et al. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. Isme Journal M ultidisciplinary Journal of M icrobial Ecology, 2010, 4(10): 1340-1351. DOI:10.1038/ismej.2010.58.
[22] LIU W J, ZHENG Y, KWOK L Y, et al. High-throughput sequencing for the detection of the bacterial and fungal diversity in Mongolian naturally fermented cow’s m ilk in Russia[J]. BMC M icrobiology,2015, 15(1): 1-12. DOI:10.1186/s12866-015-0385-9.
[23] LI P, LIN W, LIU X, et al. Environmental factors affecting m icrobiota dynam ics during traditional solid-state fermentation of Chinese Daqu starter[J]. Frontiers in M icrobiology, 2016, 7: 1-12. DOI:10.3389/fm icb.2016.01237.
[24] HU X, DU H, REN C, et al. Illum inating anaerobic m icrobial community and co-occurrence patterns across a quality gradient in Chinese liquor fermentation Pit muds[J]. Applied & Environmental M icrobiology, 2016, 82(8): 2506-2515. DOI:10.1128/AEM.03409-15.
[25] DING X, WU C, HUANG J, et al. Characterization of interphase volatile compounds in Chinese Luzhou-flavor liquor fermentation cellar analyzed by head space-solid phase m icro extraction coupled w ith gas chromatography mass spectrometry (HS-SPME/GC/MS)[J].LWT-Food Science and Technology, 2016, 66: 124-133. DOI:10.1016/j.lw t.2015.10.024.
[26] CHEN B, WU Q, XU Y. Filamentous fungal diversity and community structure associated w ith the solid state fermentation of Chinese Maotai-fl avor liquor[J]. International Journal of Food M icrobiology,2014, 179(2): 80-84. DOI:10.1016/j.ijfoodm icro.2014.03.011.
[27] WU Q, ZHU W, WANG W, et al. Effect of yeast species on the terpenoids profile of Chinese light-style liquor[J]. Food Chem istry,2015, 168(1): 390-395. DOI:10.1016/j.foodchem.2014.07.069.
[28] 余伟民, 徐小明, 林培, 等. 生香酵母对特香型白酒风味风格的影响研究[J]. 酿酒科技, 2012(1): 24-27.
[29] ZHOU L W, VLASAK J, DECOCK C, et al. Global diversity and taxonom y of the Inonotus linteus com p lex (Hymenochaetales,Basidiom yco ta): Sanghuangporus gen. nov. Tropicoporus excentrodendri and T. guanacastensis gen. et spp. nov. and 17 new combinations[J]. Fungal Diversity, 2016, 77(1): 335-347.DOI:10.1007/s13225-015-0335-8.
Dynamics of Eukaryotic M icrobial Community Succession during the Traditional Fermentation of Special-Flavor Liquor
LI Kaimin1, FU Guiming1,*, WU Choufei2, LIU Chengmei1, WAN Yin1, PAN Fei1, ZHENG Fuping3,*
(1. State Key Laboratory of Food Science and Technology, College of Food Sciense and Technology, Nanchang University,Nanchang 330047, China; 2. College of Life Science, Huzhou University, Huzhou 313000, China;3. School of Food and Chemical Engineering, Beijing Technology and Business University, Beijing 100048, China)
High throughput sequencing (HTS) was used to investigate the dynam ics of eukaryotic m icrobial community succession during the fermentation process of special-flavor liquor. M icrobial samples were collected by washing the surface of fermented grains from the bottom of fermentation cellar (0 and 30 d) and from the top and m iddle (0, 3, 6, 10, 15, 20, 25,and 30 d), and their genetic DNA was then extracted. The 18S rRNA regions of fungal rRNA genes were amplified by PCR.The dom inant m icrobial populations and their succession dynam ics were determ ined by pyrosequencing. The diversity and abundance index of fungal microbial communities showed a decreasing tendency during the fermentation process. Moreover,Saccharomycetales was the predom inant order and Saccharomyces, Pichia and Galactomyces were the predom inant genra on top and middle fermented grains during the whole fermentation process. The eukaryotic m icrobial communities identified from bottom fermented grains were more complicated, w ith the dom inant species being Saccharomycetales, Eurotiales,Capnodiales and Tremellales. Furthermore, principal coordinate analysis (PCoA) results revealed that no obvious difference in fungal communities between the top and m iddle fermented grains was observed during the fermentation process of special-flavor liquor.
special-flavor liquor; fermentation process; eukaryotic m icrobial; diversity
10.7506/spkx1002-6630-201722020
TS26
A
1002-6630(2017)22-0131-06
李凯敏, 付桂明, 吴酬飞, 等. 特香型白酒酿造过程中真核微生物菌群演替[J]. 食品科学, 2017, 38(22): 131-136.
DOI:10.7506/spkx1002-6630-201722020. http://www.spkx.net.cn
LI Kaim in, FU Guim ing, WU Choufei, et al. Dynam ics of eukaryotic m icrobial community succession during the traditional fermentation of special-flavor liquor[J]. Food Science, 2017, 38(22): 131-136. (in Chinese w ith English abstract)
DOI:10.7506/spkx1002-6630-201722020. http://www.spkx.net.cn
2016-12-14
食品科学与技术国家重点实验室开放课题(SKLF-KF-201612);江西省教育厅科技重点项目(GJJ150018)
李凯敏(1994—),男,硕士,研究方向为发酵工程。E-mail:13767010782@163.com
*通信作者:付桂明(1972—),男,教授,博士,研究方向为微生物。E-mail:fuguim ing@ncu.edu.cn郑福平(1969—),男,教授,博士,研究方向为风味化学。E-mail:zhengfp@th.bubu.edu.cn
