Genbank中陆地棉表达序列标签(EST)与基因组序列(GSS)的SNP特征
2016-03-01胡根海
胡根海, 董 娜
(河南科技学院, 现代生物育种河南省协同创新中心, 河南 新乡 453003)
Genbank中陆地棉表达序列标签(EST)与基因组序列(GSS)的SNP特征
胡根海, 董 娜
(河南科技学院, 现代生物育种河南省协同创新中心, 河南 新乡 453003)
陆地棉基因组精细定位需要更加丰富的分子标记,为了阐明单核苷酸多态性(SNP)标记资源开发利用的前景,对GenBank数据库中陆地棉表达序列标签(EST)和基因组序列(GSS)进行分析。下载GenBank数据库中公布的陆地棉EST序列及GSS序列,利用DNAStar软件进行叠连群构建及其候选SNP位点分析。结果表明:陆地棉EST序列307 414条及GSS序列242 015条,EST序列构建3 737个叠连群,序列累计10 477 241 bp。由4条及以上序列组成的叠连群累计序列长度为3 761 800 bp,发现候选SNP位点1 007 258个,叠连群平均出现1个SNP位点最低频率为2.32%。GSS序列共构建1 517个叠连群,序列累计1 625 700 bp,发现SNP位点574 296个,叠连群平均出现1个SNP位点最低频率为9.18%。陆地棉的EST和GSS均有频率较高SNP位点,GSS出现SNP频率高于EST序列,开发SNP引物3 254对。
陆地棉; 叠连群; SNP频率; GenBank
我国现有的陆地棉品种来自47个基础种质,品种间亲缘关系近,分子标记多态性较低。据最新报道[1],陆地棉高密度连锁图谱共整合2 292个标记位点,覆盖基因组5 115.16 cM,平均2.23 cM有1个标记,该标记数量和密度尚不能满足陆地棉基因组精细定位的需要,高密度精细定位是进一步解析基因功能的基础,棉花重要农艺性状的基因组精细定位需要加大现有图谱密度。新型分子标记SNP(Single Nucleotide Polymorphism)的出现,为进一步加密陆地棉图谱提供可能。SNP是广泛存在于植物基因组中的一类DNA高频序列变异,是标记密度最高的一种遗传多样性标记,SNP比SSR(simple sequence repeats)标记具有更高的稳定性[2],在群体中符合孟德尔遗传定律,可用于遗传关联分析[3],还可用于材料的基因分型[4-5]。SNP通常用DNA芯片自动化检测[6-7],也可用简化的EcoTILLING 技术[8]和普通PCR方法实现检测[4,9]。目前,SNP已成为分子标记辅助育种的一种新型工具。但诸多作物SNP开发刚起步仅在荔枝[10]、花生[11]、菊花[12]、葡萄[13]、栽培番茄[3]和大白菜[14]等作物中进行开发。在陆地棉上利用数据库开发EST(expressed sequence tag)-SSR标记已有报道[15],但开发SNP标记尚未见报道。已有记载显示陆地棉SNP具有良好分型效果[16],笔者拟利用NCBI的GenBank数据库中公布的陆地棉GSS(genomic survey sequence)和 EST序列数据,使用生物信息学软件分析陆地棉SNP标记的潜在存在数量,对陆地棉SNP进行开发,以期为陆地棉基因组精细定位提供理论基础。
1 材料与方法
1.1 数据材料
陆地棉EST和GSS序列下载于NCBI(美国国家生物信息中心)的GenBank数据库,下载序列以FASTA格式保存,下载时间为2015年5月5号。
1.2 序列拼接形成叠连群
下载序列解压后,采用EST-trimmer和cross-match程序清理下载序列中的短序列和载体序列,屏蔽低复杂度序列,处理后序列用DNAStar软件包的子程序SeqMan进行叠连群分析。
1.3 SNP位点分析及引物开发
使用SeqMan软件找出候选SNP,SNP出现频率=候选SNP的数目/叠连群长度×100%;根据候选SNP位点序列,使用 Primer Premier 5.0设计陆地棉SNP引物,基本检索条件:叠连群长度≥500 bp,组成序列条数≥10;筛选引物标准:长度18~24 bp,GC含量40%~60%,退火温度(Tm) 40~60℃,不产生引物二聚体、发夹结构和引物错配,引物中无 6个连续碱基出现,PCR扩增产物长度为200~500 bp。
2 结果与分析
2.1 陆地棉EST与GSS序列的叠连群构成及SNP出现频率
在GenBank数据库中总计下载307 414条陆地棉EST序列,通过剔除短序列和载体,屏蔽低复杂度序列,获得有用序列255 002条,叠连群构建后,有9 002条序列未组装成叠连群,序列组装共使用246 000条序列,构建3 737个叠连群。叠连群最长达3 912 bp,其SNP候选位点 2 972个,出现SNP频率75.97%,最短1个叠连群长度仅 142 bp,无候选SNP位点存在。叠连群累计长度3 761 800 bp,鉴定候选SNP位点1 007 258个,SNP位点出现的平均频率为26.77%。从叠连群的长度与数量组成看,较长(≥2000 bp)53条,较短(≤499 bp)90条,叠连群长度主要集中在500~1 500 bp,约占86.76%,其SNP出现频率随叠连群长度增加也在增加,但陆地棉的SNP位点分布在EST中不均匀。
最长叠连群长度为10 498 bp,有7 345个SNP候选位点,叠连群SNP出现频率69.97%;最短1个叠连群长度仅220 bp,无候选SNP位点存在。由表1可知,不同长度叠连群的数目表现很大差异,叠连群越长其数目越少,统计叠连群累计长度3 761 800 bp,鉴定出候选SNP位点574 296个,SNP平均出现频率为44.57%。从叠连群的长度与数量看,叠连群长度主要集中在500~2 000 bp,约占91.67%,其SNP出现频率随叠连群长度增加而增加,但分析陆地棉的SNP位点在GSS叠连群中分布并不均匀。
2.2 陆地棉 EST 与 GSS序列叠连群组成条数及SNP 出现频率
对3 737条叠连群进行构成叠连群序列条数分析显示,仅有2条序列构成的叠连群高达1 801个,由3条序列构成叠连群有752条,这些叠连群因组成序列低于4条,故未检测到SNP。由表2可见,4~6条序列构成的叠连群,SNP出现频率为2.32%,随着组成叠连群的序列条数增加叠连群中出现SNP的频率也在增加,最高叠连群是2个由112条、119条序列构成叠连群,其出现SNP频率均值为46.36%。
表1 陆地棉EST与GSS序列的叠连群构成与SNP出现频率
Table 1 Contigs constitute and SNP frequency of EST and GSS sequences fromG.hirsutum
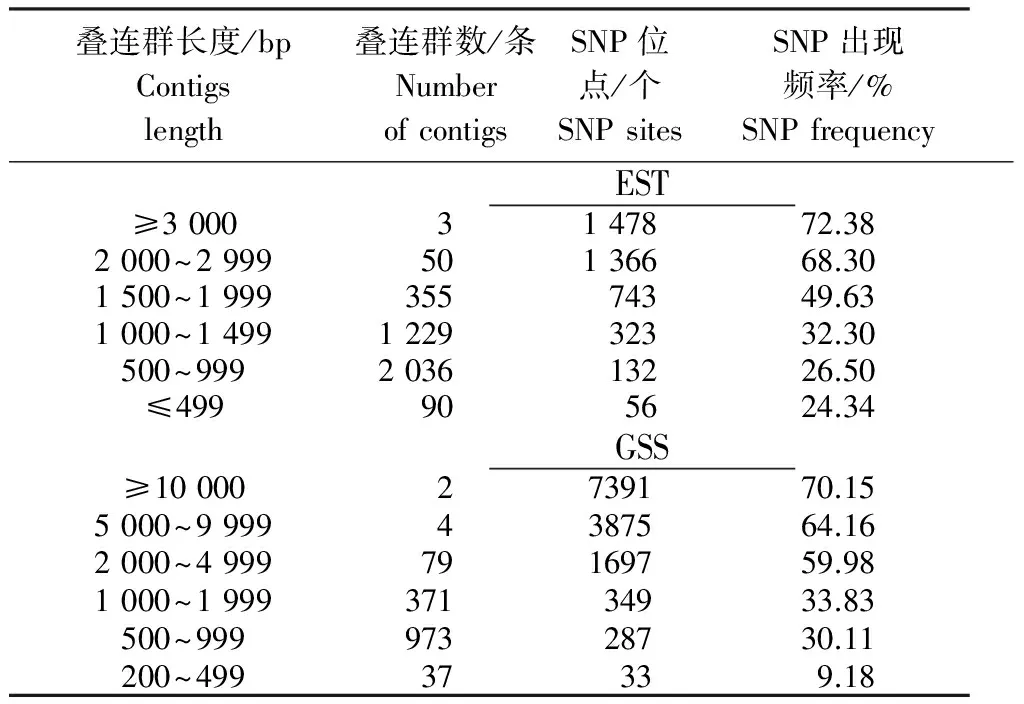
叠连群长度/bpContigslength叠连群数/条NumberofcontigsSNP位点/个SNPsitesSNP出现频率/%SNPfrequencyEST≥30003147872.382000~299950136668.301500~199935574349.631000~1499122932332.30500~999203613226.50≤499905624.34GSS≥100002739170.155000~99994387564.162000~499979169759.981000~199937134933.83500~99997328730.11200~49937339.18
表2 陆地棉 EST序列组成叠连群的序列条数及候选SNP 出现频率
Table 2 Number of sequences of contigs and candidate SNP frequency of EST sequences fromG.hirsutum
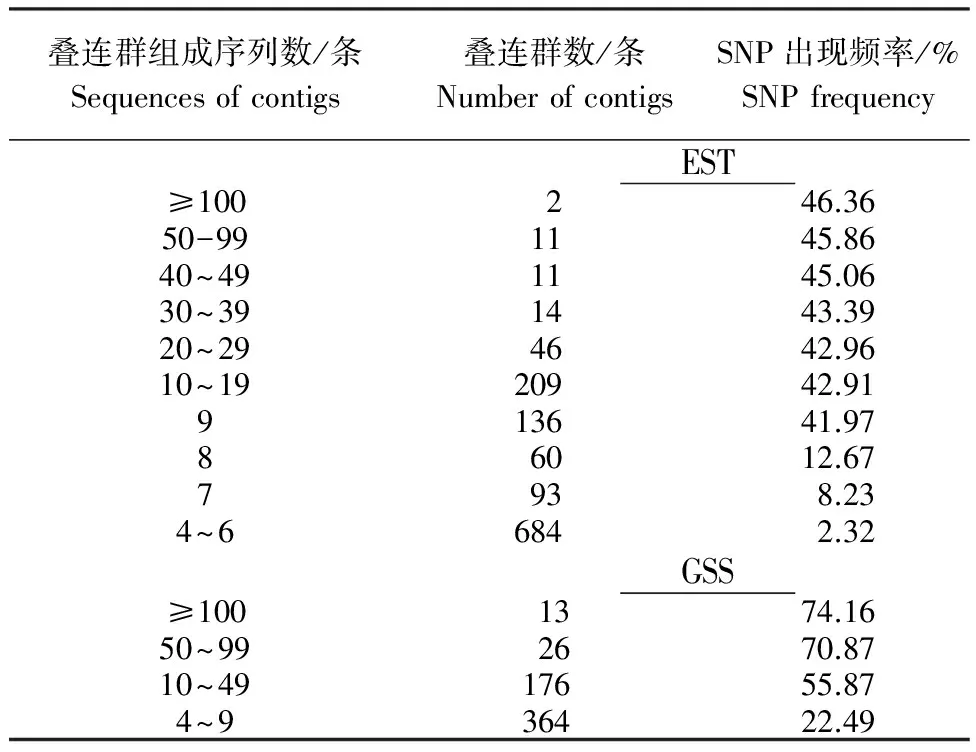
叠连群组成序列数/条Sequencesofcontigs叠连群数/条NumberofcontigsSNP出现频率/%SNPfrequencyEST≥100246.3650-991145.8640~491145.0630~391443.3920~294642.9610~1920942.91913641.9786012.677938.234~66842.32GSS≥1001374.1650~992670.8710~4917655.874~936422.49
在GenBank数据库中总计下载242 015条陆地棉GSS序列,通过剔除短序列和载体,屏蔽低复杂度序列,最后获得有用序列24 144条,其中11 393条序列未组装成叠连群,序列组装共使用11 234条序列,构建1 517个叠连群。组成叠连群序列最多的有656条序列。由表2可知,在4条以上序列组成叠连群中,主要有4~49条序列组成,随着组成叠连群的序列数目增加,其SNP位点数和SNP出现频率也在增加。但在计算统计分析时发现,随着组成叠连群序列的减少,出现SNP位点数极少或无位点的频率也在增加。
2.3 陆地棉SNP 引物的开发
叠连群组成序列≥10条的共有508个叠连群,依据SNP位点两侧序列和引物筛选的基本原则,保守设计即可得到3 254对引物,表明陆地棉的SNP可设计的引物数量非常多,该标记在育种利用中有较大挖掘潜力。
3 结论与讨论
单核苷酸多态性在植物基因组中广泛存在,较高的多态性使材料的遗传多样性变得容易区分,也让生产上种子纯度鉴定变得简单[14],SNP是单核苷酸变异,可能发生在基因的内含子区、表达调控区和基因功能区,因此部分SNP位点可能和基因的功能直接相关。EST是测序获得的短cDNA 序列,是完整基因的一部分。因此,基于EST的SNP更有可能与功能基因及农艺性状相关联。
本研究中分析307 414条陆地棉EST序列,平均每43.1 bp EST序列中出现1个SNP位点,SNP位点出现频率约为2.32%,比菊花[12]EST的SNP平均出现频率(0.15%)略高,但远远低于对吴茱萸SOD基因分析得到的SNP 位点出现频率(20%)[17]。这种结果的差异可能因为目前菊花EST数据偏少,很多SNP位点未被发现;陆地棉EST发现SNP位点少于吴茱萸的SOD基因,可能因EST一般为短cDNA 序列,仅完整基因的一小部分,而吴茱萸SOD基因分析是完整基因。从陆地棉GSS数据中获得574 296个候选SNP位点,SNP的出现频率最低为9.18%,平均每10.89 bp出现1个候选SNP位点。已有结果显示,在水稻基因组序列中,平均每89 bp出现1个SNP位点[18];在花生栽培种基因组DNA的测序数据中,平均每27.86 bp出现1个SNP位点[11];玉米基因组DNA中平均每61 bp序列有1个SNP[19];试验数据显示陆地棉GSS序列的SNP出现频率高于水稻、花生和玉米,比较4个物种可见物种间差别较大,这可能是物种不同引起。比较陆地棉EST和GSS构建的各个叠连群中含有的SNP数量,可见构成叠连群的序列条数越多,出现的SNP位点数量也越多,与Duran C等[20]在大麦EST序列的研究结果基本一致。EST序列和GSS序列出现SNP的频率不均匀,与前人在菊花[12]上研究结果一致。
比较由EST和GSS获得结果可知,利用GSS序列检测到其有更多的SNP位点和更高出现频率,这可能是因为GSS序列是基因组序列,含有非编码区,这些区域的突变不会直接导致植物体变异,具有更低选择压和更高突变频率。陆地棉基因组序列候选SNP出现频率较高还可能与其为异源四倍体起源有关,这与唐月异[11]研究作物染色体倍数越大SNP频率越高一致。本研究首次对陆地棉的GSS和EST序列进行了SNP标记出现频率及可能数量进行分析并开发了3 254对SNP检测引物, SNP变异比SSR丰富,是更有前景的新一代标记。
[1] Yuzhen Shi,Wentan Li,Aiguo Li,et al.Constructing a high density linkage map for Gossypium hirsutum×Gossypium barbadense and identifying QTLs for lint percentage[J].Journal of Integrative Plant Biology,2015,57(5):450-467.
[2] 李 雪,田红丽,王凤格,等.SSR和SNP两种标记技术在玉米品种真实性鉴定中的比较分析[J].分子植物育种,2014,12(5):1000-1004.
[3] 郝转芳,苏治军,李 亮,等.基于SNP标记的关联分析在玉米耐旱研究中的应用[J].作物杂志,2009(6):1-7.
[4] 李亚玲,李景富,康立功,等.番茄Mi-1基因的SNP分型[J].东北农业大学学报,2010,41(10):36-42.
[5] 姚远颋,谢青轩,杜培粉,等.利用SNP分型技术鉴别油菜FLC1与FLC3基因的AC组基因型[J].分子植物育种,2009,7(2):330-334.
[6] Olivier M.SNP genotyping using invader technology[J].Mutation Res,2005,573(1/2):103-110.
[7] 曹廷杰,谢菁忠,吴秋红,等.河南省近年审定小麦品种基于系谱和SNP标记的遗传多样性分析[J].作物学报,2015,41(2):197-206.
[8] 滕 斌,李泽福,罗志祥,等.利用EcoTILLING简化技术进行水稻基因型鉴定及单核苷酸多态性(SNP)检测[J].农业生物技术学报,2012,20(3):254-260.
[9] Michaels S D,Amasino R M.A robust method for detecting single-nucleotide changes as polymorphic markers by PCR[J].Plant J.,1998,14(3):381-385.
[10] 孙清明,李永忠,向 旭,等.利用SNP和EST-SSR分子标记鉴定荔枝新种质御金球[J].分子植物育种,2013,11(3):403-414.
[11] 唐月异,张建成,王秀贞,等.GenBank中花生栽培种基因组DNA及EST序列的SNP分析[J].花生学报,2010,39(2):21-23.
[12] 万志兵,王言言,陈 黎.菊花EST-SNP的发掘与特征分析[J].东北林业大学学报,2013,41(5):84-86,90.
[13] 李 猛,郭大龙,刘崇怀,等.葡萄EST-SNP位点的信息与特征[J].浙江大学学报:农业与生命科学版,2012,38(3):263-270.
[14] 赵 新,王 永,兰青阔,等.基于复合EST-SSR标记的大白菜种子纯度鉴定及SNP位点获取[J].中国蔬菜,2013(14):31-38.
[15] 李华盛,范术丽,沈法富.从棉花ESTs数据库中筛选微卫星标记的初步研究[J].棉花学报,2005,17(4):211-216.
[16] 郑炜佳,曲延英,谢元元,等.ARMS-PCR对棉花SNP分型的研究[J].新疆农业科学,2013,50(12):2182-2188.
[17] 吴 波,高 丹,潘超美,等.吴茱萸SOD基因片段克隆和SNP分析[J].江西农业大学学报,2011,33(6):1206-1211.
[18] Nasu S,Suzuki J, Ohta R,et a1.Search for and analysis of single nucleotide polymorphisms(SNPs) in rice(Oryza sativa,Oryza rufipogon)and establishment of SNP markers[J].DNA Res.,2002,9:163-171.
[19] Ching A,Caldwell K S,Jung M,et a1.SNP frequency,haplotype structure and linkage disequilibrium in elite maize inbred lines[J].BMC Genet,2002,3:19.[20] Duran C,Appleby N,Vardy M,et a1.Single nucleotide polymorphism discovery in badey using auto SNPdb[J].Plant Biotechnology Journal,2009,7:326-333.
(责任编辑: 刘忠丽)
SNP Discovery in GSS and ESTs ofGossypiumhirsutumfrom GenBank
HU Genhai, DONG Na
(HenanCollaborativeInnovationCenterofModernBiologicalBreeding,HenanInstituteofScienceandTechnology,Xinxiang,Henan453003,China)
In order to explore the SNP marker resources development prospects, which can provide abundant number of molecular markers for cotton genome fine positioning. 242 015 GSS sequences and 307 414 ESTs ofG.hirsutumdownloaded from GenBank in NCB1 were analyzed. 3 737 contigs were assembled from the EST sequences by DNAStar, and the length of the total contigs was 1 047 7241 bp. The length of the tota1 contigs with≥4 ESTs was 3 761 800 bp, from which 1 007 258 candidate SNPs were identified directly, and the average frequency of SNPs was 2.32%. 1 517 contigs were assembled from the GSS, and the length of the total contigs was 1 625 700 bp. From which 574 296 candidate SNPs were identified directly, and the average frequency of SNPs was 9.18%. Conclusion: The upland cotton EST and GSS all had higher frequency of SNP loci, SNP frequency of GSS was higher than that of ESTs, it was more valuable that SNP loci of GSS was utilized. 3 254 pairs of SNP primers were designed by Primer Premier 5.0 following the basic criteria.
Gossypiumhirsutum; contigs; SNP frequency; GenBank
2015-08-28; 2016-03-24修回
国家自然科学基金项目“棉花早熟不早衰基因型的根系形态和生理机制研究”(31271648);河南省科技攻关计划项目“转基因耐盐碱陆地棉新材料创制”(142102110126)
胡根海(1966-),男,副教授,博士,从事棉花分子育种研究。E-mail: hgh1013@126.com
1001-3601(2016)04-0150-0021-03
S562; Q946.2
A
