塔里木河上游不同生境细菌群落结构及多样性分析
2024-04-28姚宇翔王国强王可伊莎杨欣雅罗晓霞
姚宇翔 王国强 王可 伊莎 杨欣雅 罗晓霞
doi:10.6048/j.issn.1001-4330.2024.03.021
摘 要:【目的】研究新疆塔里木河上游不同生境细菌群落结构及多样性。
【方法】采用免培养和可培养方法,研究塔里木河上游流域原始生境与人工开发后生境细菌群落结构及多样性差异,同时对分离的1株潜在新种进行多相分类鉴定。
【结果】原始生境与人工开发后生境细菌群落结构组成差异明显,人工开发后生境细菌α多样性低于原始生境,不同生境细菌β多样性差异较大。变形菌门和放线菌门为优势菌群,并获1株链霉菌属潜在新物种。
【结论】塔里木河上游不同生境样品中含有丰富的微生物资源,其细菌群落组成受人为活动的影响较大。
关键词:不同生境;细菌群落组成;多样性;多相分类鉴定
中图分类号:S188 文献标志码:A 文章编号:1001-4330(2024)03-0708-11
收稿日期(Received):
2023-07-15
基金项目:
新疆生产建设兵团中青年科技创新领军人才计划(1119119);塔里木放线菌种质资源库建设(TDZKKY202201);
塔里木大学自治区研究生科研创新项目(TDGRI202204)
作者简介:
姚宇翔(1999-),女,河南周口人,硕士研究生,研究方向为微生物资源及其利用,(E-mail)1366307897@qq.com
通讯作者:
罗晓霞(1982-),女,新疆阿拉尔人,教授,博士,硕士生导师,研究方向为微生物资源及其利用,(E-mail)xxluo415@163.com
0 引 言
【研究意义】塔里木河自然植被、生物多样性、土壤次生盐渍化等细菌群落变化已有研究[1-4]。以往微生物群落分析仅使用传统培养的方法,无法检测环境中大多数微生物群落[5-6],特别是不可培养的微生物群落[7-8]。宏基因组学又叫微生物环境基因组学、元基因组学[4]。宏基因组学通过与生物信息学的有机结合,在揭示微生物之间、微生物与环境之间相互作用的规律中发挥了作用,拓展了环境微生物的研究思路与方法,为从群落水平上全面认识微生物的生态特征和功能开辟了新的途径[9]。对塔里木河上游原始生境与开发后的生境微生物的多样性进行探究,对于科学管理绿洲生态系统具有重要意义。【前人研究进展】目前研究较多集中于湖泊、河流、森林湿地、热泉冷泉、海洋等细菌多样性的分布组成[10-11],而对于塔里木河上游微生物群落结构的研究多集中于植物根际,例如柽柳[12]、胡杨[13-16]等。【本研究切入点】原始生境与人工开发后生境细菌群落结构组成差异明显,需研究塔里木河上游不同生境细菌群落结构及多样性。【拟解决的关键问题】以塔里木河上游流域不同生境的水体和土壤样品为研究材料,比较分析原始生境和人为开发后的生境的细菌群落结构,为指导细菌群落多样性、群落结构差异及群落潜在功能提供参考。
1 材料与方法
1.1 材 料
1.1.1 土样采集
采集地点位于塔里木河上游(40°32′2″N,81°17′58″E)不同生境的土壤样品,包括农田排碱渠底泥(S1)、阿拉尔人工湖底泥(S3)、东湖底泥(S-Y)、台兰河底泥(S-m)、塔里木河底泥(S4)、塔里木河底泥(S-T)、农田灌溉水底泥(S2)、农田耕作土样(S-M)。采用等距采样法,用直径5 cm的不锈钢土钻取0~20 cm的土壤样品,取5钻,每钻点取样量一致,并均匀混合,保存于灭菌密封。
1.1.2 水样采集
采集地点位于塔里木河上游(40°32′2″N,81°17′58″E)不同生境的水体样品,包括:农田灌溉水样(EG),阿拉尔人工湖水样(ZF),东湖(BM),塔里木河水样(TA)。采用等距采样法:取水深20 cm处,每个样品25 L放于灭菌密封桶中。
1.2 方 法
1.2.1 基因组DNA提取和测序
土样使用SDS高盐法手提土壤基因组后再使用Ezup柱式土壤DNA抽提试剂盒进行纯化。水样使用0.22 μm的滤膜对10 L水样样品进行过滤富集。样品委托南京派森诺生物科技有限公司基于Illumina MisSeq技术测序平台构建文库,进行高通量双末端测序。根据相似度,将序列聚类为操作分类单元(OTU),相似度阈值为97%。使用RDP分类器对序列进行系统分类,并在各分类级别上对物種进行统计。
1.2.2 生物信息学分析
使用QIIME2软件对获得的高质量序列,按97%的序列相似度进行归并和OTU(operational taxonomic units,OTU)划分,细菌基于Silva参考数据库进行比对,得到每个OTU对应的物种分类信息[12];根据OTU划分和分类地位鉴定结果,使用QIIME2软件,获得每个样本在各分类水平的群落组成[13];使用QIIME2软件分别对每个样本计算Ace、Chao1、Shannon,Simpso指数等4种α多样性指数;使用R软件,基于UniFrac距离算法PcoA(Principal coordinate analysis),进行β多样性分析。
1.2.3 菌株筛选
采用IChip技术分离不同区域样品的可培养菌种,选取11种培养基进行菌种分离。菌株分离纯化后保存于20%的甘油管中于-20℃低温保存[18-19]。
1.2.4 细菌多相分类鉴定
采用《放线菌快速鉴定与系统分类》对菌株基因特征、形态特征、生理生化特征、化学特征、全基因组测序、注释及分析等进行鉴定[17]。
1.3 数据处理
采用QIIME2软件计算样品的Chaol指数、Shannon指数和Simpson指数用于表征其丰富度和多样性。
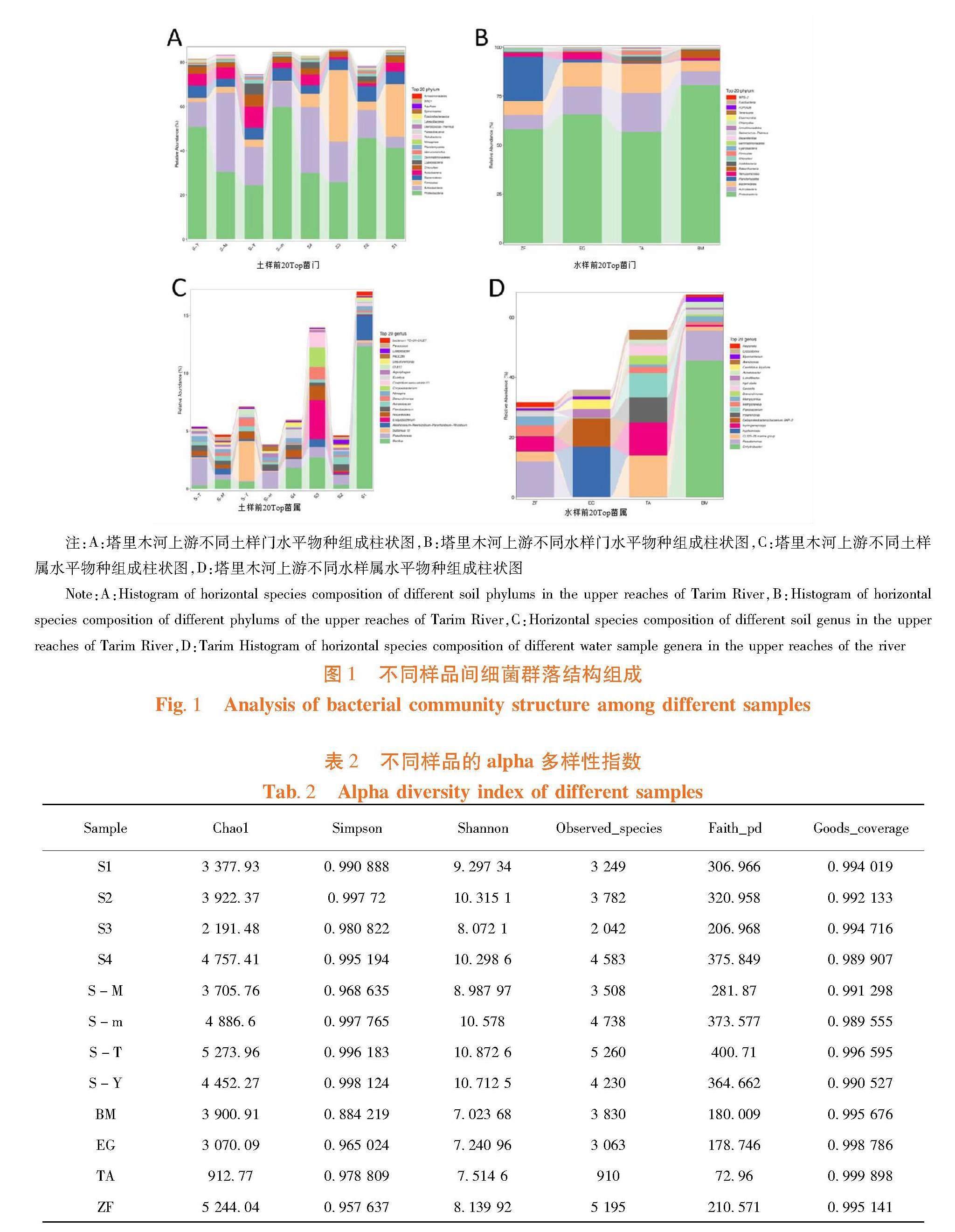
Rank-abundance曲线可用来解释多样性的两个方面,即物种丰度和物种均匀度。Rank-abundance在水平方向,物种的丰度由曲线的宽度来反映,物种的丰度越高,曲线在横轴上的范圍越大;曲线的形状(平滑程度)反映了样品中物种的均度,曲线越平缓,物种分布越均匀。以各个样品的OTU序列数由大到小等级排序,再以OTU 等级为横坐标,以每个OTU 中所含的序列数为纵坐标做图。
Beta多样性是指沿着环境梯度变化的不同群落之间,物种组成的相异性或物种沿环境梯度的更替速率,也被称为生境间多样性(between-habitat diversity)。根据不同样本的OTU丰度计算Bray curtis,加权Unifrac和未加权Unifrac距离,评估不同样本间的微生物群落结构差异。Beta多样性分析通过PcoA(主坐标分析)解析各个样本之间微生物群落结构的差异和不同分类对样本差异的影响。研究采用QIIME2软件分析塔里木河上游不同生境样品微生物Beta分析,包括PCoA分析及样本间物种组成的遗传距离。
使用层次聚类方法,使用R语言stat包的uclust函数,对Bray-Curtis距离矩阵采用UPGMA算法进行聚类分析,计算土样和水样各样品间距离。
借助ASVs(Amplification Sequence Variants)丰度表制作韦恩图进行分析。
使用热图进行反映各样品间微生物群落中物种组成相似性、差异性、物种聚类关系物种组成分析。
2 结果与分析
2.1 塔河流域上游不同生境16SrRNA基因序列多样性
研究表明,水样样品共得到336 046条高质量序列,其中TA、ZF、EG、BM样品分别为130 988、106 818、102 301、113 829条,土壤样品共得到696 234条高质量序列,其中S-T、S4、S-M、S-Y、S-m、S3、S2、S1样品分别为58 271、77 641、90 095、94 229、71 627、113 845、78 668、111 858条。按97%的序列相似度进行归并和OUT聚类,水样获得了2 468个可分类的操作单元,土样获得了3 721个可分类的操作单元。水样可划分为88门、294纲、436目、601科、861属、287种,土样可划分为212门、504纲、836目、1 020科、830属、220种。原始生境样品可划分为133门、327纲、552目、694科、630属、47种,人工改造的生境样品可划分为167门、372纲、720目、927科、1061属、341种。表1
2.2 塔河流域上游不同生境微生物群落组成
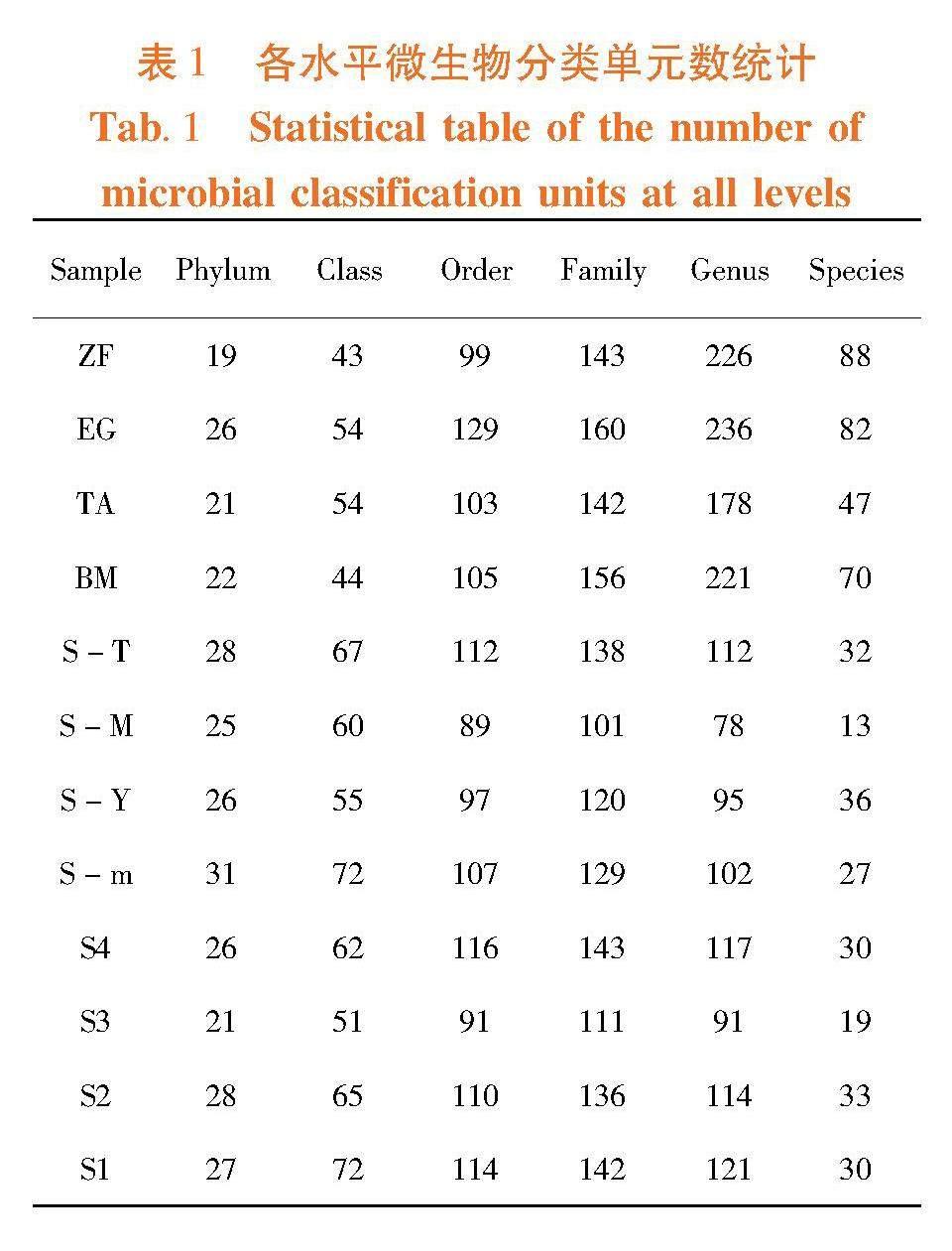
研究表明,土样经鉴定得到212门、504纲、836目、1 020科、830属的细菌物种信息,水样经鉴定得到88门、294纲、436目、601科、861属的细菌物种信息。在门水平上,土样均以变形菌门(Proteobacteria)(38.45%)、未分类细菌门(unclassified_Bacteria)(17.81%)、放线菌门(Actinobacteria)(17.12%)为主,其中农田排碱渠底泥以变形菌门(Proteobacteria)(41.06%)、厚壁菌门(Firmicutes)(23.81%)、未分类细菌门(unclassified_Bacteria)(14.37%)为主(图1-A)。水样细菌群落菌门主要属于4个类群,分别是变形菌门(Proteobacteria)(65.25%)、放线菌门(Actinobacteria)(12.15%)、拟杆菌门为主(Bacteroidetes)(9.91%)和浮霉菌门(Planctomycetes)(6.20%)。农田灌溉水样和塔里木河水样以变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门为主(Bacteroidetes)(图1-B)。人工湖水样以变形菌门和浮霉菌门为主,东湖水样以变形菌门为主。在属水平上,土样中共存在830个属,包括芽孢杆菌(Bacillus)(20.93%)、γ变形菌属(γ-proteobacteria)(8.12%)、伯克氏菌属(Burkholderiaceae),(8.05%)、根瘤菌属(Rhizobiales)(5.64%)、动球菌属(Planococcaceae)(9.02%)、红杆菌属(Rhodobacteraceae)(6.32%)、伯克霍尔德氏菌属(Burkholderiaceae)(5.41%)等8个主要的细菌群落菌属类群,其中未分类细菌属占36.12%(1-C)。农田灌溉水底泥样品共注释到114个属,阿拉尔人工湖底泥样品共注释到91个属,塔里木河底泥样品共注释到117个属,农田耕作土样品共注释到78个属,台兰河底泥样品共注释到102个属,塔里木河底泥样品共注释到112个属,东湖底泥样品共注释到95个属,结果表明原始生境的土壤样品细菌群落组成更为丰富。水样细菌群落菌属主要有8个类群,包括生丝单胞菌属(Hyphomonas)(16.56%)、微杆菌属(Microbacteriaceae)(7.24%)、出芽菌属(Gemmataceae)(21.42%)、假单胞菌属(Pseudomonas)(12.00%)、氢噬胞菌属(Hydrogenophaga) (10.95%)、极胞菌属(Polaromonas)(8.40%)、黄杆菌属(Flavobacterium)(8.07%)、水栖菌属(Enhydrobacter)(45.59%)等 。农田灌溉水样共注释到236个属,人工湖水样共注释到226个属,东湖水样共注释到221个属,塔里木河水样共注释到178个属。图1
2.3 微生物群落结构多样性
2.3.1 微生物组成alpha多样性
研究表明,
所有样品共获得的细菌有效序列总数为1 032 580。其中水样中获得的细菌平均有效序列总数高于土样,且塔里木河水样获得的细菌有效序列总数最多。阿拉尔人工湖底泥样品中的Chaol指数和Shannon指數最低,土样中台兰河底泥和塔里木河底泥中的Chaol指数、Shannon指数最高,水样中农田灌溉水、东湖样本中的Chaol指数、Shannon指数最高。水样中东湖、农田灌溉水样和阿拉尔人工湖水样Chaol指数和Shannon指数较高,而塔里木河水样Chaol指数和Shannon指数较低,结果表明开发后生境水样样品比原始生境水样样本群落的丰富度高,物种亲缘关系较远。表2
在土样中的原始生境与人工改造后生境土样相比,原始生境群落丰富度要高、物种亲缘关系更远、均匀度高,在水样中人工改造后生境丰富度要高、物种亲缘关系更远、均匀度高。塔里木河上游的水样与人工改造后的生境群落结构α多样性表现则相反。图2
2.3.2 Beta多样性分析
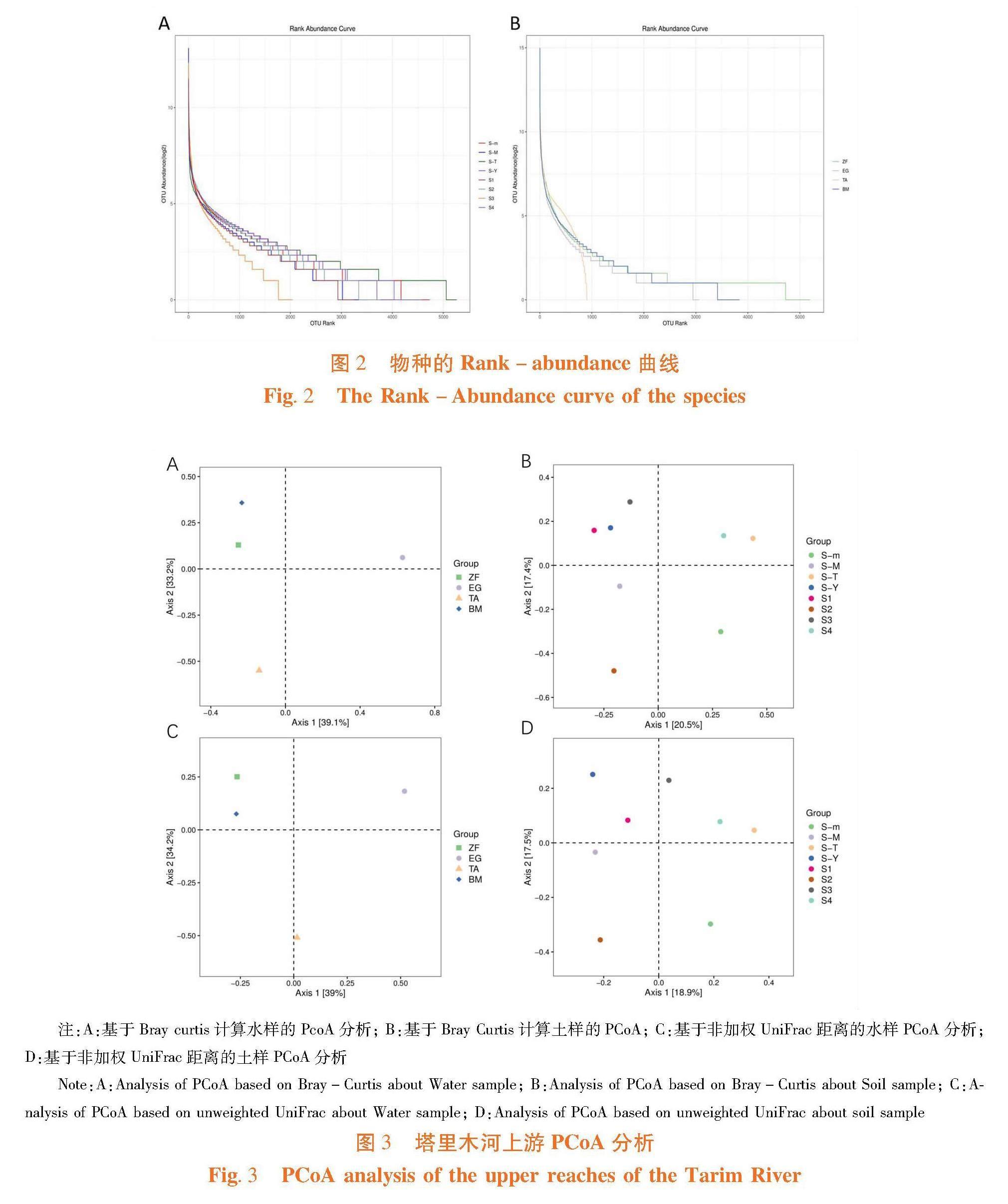
研究表明,水样中塔里木河水样与其他水样相比,微生物群落结构差异最大,土样中塔里木河底泥和台兰河底泥微生物群落结构相比较其他土样有明显差异。塔里木河底泥、台兰河底泥及塔里木河水样细菌群落结构差异较大,进化关系较远。图3
土样样本在距离为 0.035 处聚成3 组,水样样本在距离为 0.032 处聚成2组。在土样中8个土样样本共聚为3大分支,塔里木河底泥细菌群落结构相似度最高,与农田耕作土样关系较近,农田灌溉水底泥、农田耕作土样、农田排碱渠底泥与阿拉尔人工湖底泥细菌群落相似度较高,东湖底泥细菌群落单独成一个分支。在水样中4个水样样本共聚为2大分支,东湖水样与阿拉尔人工湖水样细菌群落结构相似度最高,农田灌溉水单独成一个分支。图4,图5
2.3.3 不同样品间物种差异
研究表明,在土样中,塔里木河底泥的ASVs数目最多,阿拉尔人工湖底泥的ASVs数目最少;8个土样样品中共有12个相同的ASVs,占ASVs总数的0.053%。由于塔里木河床土样长期处于裸露状态,因此塔里木河底泥(S4)与塔里木河床土样(S-T)相同ASVs非常少,而塔里木河河床土样特有ASVs非常丰富。在水样中,阿拉尔人工湖水样样品细菌丰富度最高,含4 808个ASVs。丰富度最低的是塔里木河水样样品,ASVs数为912。4个水样样品中有11个相同的ASVs,占总样本数ASVs数的0.09%,4个样本中细菌组成基本不相似。东湖水样特有的ASVs为3 467,农田灌溉水样特有的ASVs为3 006,塔里木河水样特有的ASVs为729,阿拉尔人工湖特有的ASVs为4 808。图5,图6
在土样中,原始生境在物种组成上较为相似,均含有一些黄杆菌属(Flavobacterium)、粘细菌属(Phaselicystis)、地上杆菌属(Pedobacter)等。塔里木河底泥与台兰河底泥都含有拉氏噬冷菌属(Algoriphagus)、假单胞菌属(Pseudomonas)等。塔里木河河床土样与塔里木河底泥均含有Wstewater metagenome、CL500-29 marine group、ADurb.BinO63-1等,其余的人工改造生境土样均各自含有特殊的菌属,差异性较大。在水样中,阿拉尔人工湖水样与东湖水样在物种组成上较为相似,都含有ngcl 群、假单胞菌属(Pseudomonas)、噬甲基菌属(Methylophilus)、厌氧绳菌属(Reyranella)等。塔里木河水样与农田灌溉水样均具有独特菌属。原始生境细菌群落较为相似,有较多共同的菌属,而原始生境与人工改造生境细菌群落差异明显。图6
2.4 可培养法揭示塔里木河上游细菌的多样性
研究表明,采用甘油精氨酸培养基、高氏培养基和P4培养基对8个土样和1个水样进行细菌分离培养。共分离到270株菌。从土样和水样中共分离到58个物种,252株放线菌,18株变形菌,分为3个属:链霉菌属(Streptomyeces)、北里孢菌属(Kitasatosporia)、诺卡氏菌属(Nocardia)。从塔里木河底泥样品分离到的放线菌,在种属和数量水平上以链霉菌属为优势属,占比97.76%。分离的252株放线菌中,有17株菌株相似性小于98.65%,为潜在新物种。图7
2.5 TRM49032菌株的多相分类鉴定
研究表明,从塔里木河底泥中分离得到的菌株TRM 49032为1株链霉菌,该菌株基内菌丝为棕色,气生菌丝为白色,且菌丝体伴有杆状分生孢子(直径1.5~2.0 μm);菌株TRM 49032与其最相似菌株相似性为97.64%,经系统进化分析成独立的分支,为1株潜在新物种。该菌株基因组DNA的G+C含量为70.4%。菌株在ISP1、ISP2、ISP4、ISP6及高氏Ⅰ号培养基上生长良好,且在上述除ISP1培养基外的四种培养基上均会产生棕色可溶性色素;菌株生长温度耐受温度为16~51℃,最适生长温度为31℃,pH耐受范围为5.0~12.0,最适pH为8.0,耐受NaCl浓度为0%~8%,最适NaCl浓度为2%~3%;菌株能够以葡萄糖、果糖、甘露醇、木糖、阿拉伯糖、棉子糖、鼠李糖作为唯一碳源利用,其中最适碳源为葡萄糖;菌株明胶液化反应和硝酸盐还原反应、氧化酶和过氧化氢酶反应、脂肪酶产生与纤维素分解为阳性,牛奶胨化、硫化氢产生、黑色素产生为阴性;菌株TRM 49032全细胞壁糖组分由核糖、甘露糖和葡萄糖组成,主要醌类型为MK-9(H2),MK-9(H4),MK-9(H6)和MK-9(H8) ,极性脂类型为双磷脂酰甘油(DPG),磷脂酰甲基乙醇胺(PME),磷脂酰乙醇胺(PE),磷脂酰胆碱(PC),磷脂酰肌醇(PI);主要脂肪酸类型为iso-C16∶0,anteiso-C17∶0,anteiso-C15∶0和iso-C16∶1。基于菌株TRM 49032基因型特征、形态学特征、生理生化特征、化学分类特征,确定TRM 49032为链霉菌属的一株新物种。图8
3 讨 论
3.1
农田排碱渠底泥、农田灌溉水底泥、农田耕作土和台兰河底泥均含有大量的γ-变形菌,在农田灌溉水样、塔里木河水样和东湖水样中含有大量的α-变形菌。γ-变形细菌在沿海沉积物中的暗碳固着中占主导地位。α-变形菌在分解和同化有机沉积物中有显著作用,γ-变形菌、α-变形菌和Deltaproteobacteria是大多数烃类污染海洋沉淀物的主要优势菌,且通常具有降解石油、石油烃、芳烃等碳氢化合物的功能[18-22]。农田灌溉水样中的主要菌属为生丝单胞菌属(Halobacillus)(16.56%)、变形菌属(Deltaproteobacteria)(9.39%)、未培养生丝单胞菌属(8.62%)、未分类微杆菌属(7.24%);Deltaproteobacteria在天然有机卤化物循环中起重要作用,生丝单胞菌属是一种发芽的假肢细菌,主要存在于海洋环境中,大多数是从石油降解微生物群落中分离出来的[23-24]。
3.2
塔里木河上游水样中氢噬胞菌属(10.95%)、极胞菌属(8.40%)和黄杆菌属(8.07%)为优势菌属。黄杆菌生活在温带和极地地区的各种环境中,包括陆地、湖泊、海洋、冰川、植物、动物等[25],目前共发现7种耐冷黄杆菌属物种,均从冰川中收集到[26],极胞菌属是冰川微生物群系的重要组成部分,目前共发现9种极孢菌属物种[27-28],塔里木河上游水温较冷,主要水源来自冰川,塔里木河上游水样中丰度较高的极胞菌属和黄杆菌属可能来自该冰川融水。黄杆菌可除氮和磷及有机物的降解[27],氢噬胞菌属能够脱氮并降解多种有机物[29]。后期可设计特异性培养基针对黄杆菌属和氢噬孢菌属特殊功能菌株进行分离,丰富和完善塔里木河上游极端环境微生物菌种库。
4 结 论
在人为因素影响下的生境与原始生境细菌群落结构差异明显,细菌α多样性低于原始生境,细菌β差异性明显减弱。人工开发生境细菌结构相互之间差异性减小。8个土样1个水样分离并测序得到3屬,58种,270株菌株。并得到了1株链霉菌潜在新物种,并对其进行了新种多相分类鉴定。
参考文献(References)
[1]
刘海燕,王敬敬,赵维,等.塔里木河中下游流域棉田及胡杨林土壤细菌群落结构及多样性研究[J].微生物学通报,2019,46(9):2214-2230.
LIU Haiyan,WANG Jingjing,ZHAO Wei,et al.Structure of soil bacteria community and diversity in cotton field and Euphrates poplar forest in the middle and lower reaches of Tarim river basin [J].Microbiology China,2019,46(9):2214-2230.
[2]孙晨,贡璐,马勇刚,等.塔里木河上游典型绿洲土地利用时空格局分析[J].西北农林科技大学学报(自然科学版),2020,48(7):113-122.
SUN Chen,GONG Lu,MA Yonggang,et al.Temporal and spatial pattern analysis of land use in typical oasis in the upper reaches of the Tarim River[J].Journal of Northwest A & F University(Natural Science Edition),2020,48(7):113-122.
[3]罗建桦,陶晔,邢鹏等.湖泊微生物宏基因组学研究进展[J].湖泊科学,2020,32(1):271-280.
LUO Jianhua,TAO Ye,XING Peng,et al.Mini-review:Advances of metagenomics research for lake microbiomes [J].Journal of Lake Sciences,2020,32(1):271-280.
[4]吴庆龙,邢鹏,李化炳,等.草藻型稳态转换对湖泊微生物结构及其碳循环功能的影响[J].微生物学通报,2013,40(1):87-97.
WU Qinglong,XING Peng,LI Huabing,et al.Impacts of regime shift between phytoplankton and macrophyte on the microbial community structure and its carbon cycling in lakes [J].Microbiology China,2013,40(1):87-97.
[5]Han D,Ha H K,Hwang C Y,et al.Bacterial communities along stratified water columns at the Chukchi Borderland in the western Arctic Ocean [J]. Deep-Sea Research Part II:Topical Studies in Oceanography,2015,120:52-60.
[6]吴庆龙,江和龙.中国湖泊微生物组研究[J].中国科学院院刊,2017,32(3):273-279.
WU Qinglong,JIANG Helong.China lake microbiome project[J].Bulletin of Chinese Academy of Sciences,2017,32(3):273-279.
[7] Ininbergs K,Bergman B,Larsson J,et al.Microbial metagenomics in the Baltic Sea:Recent advancements and prospects for environmental Monitoring[J].Ambio,2015,44(Suppl 3):439-450.
[8]张庆芳,杨超,于爽,等.黄海海域海洋沉积物细菌多样性分析[J].微生物学通报,2020,47(2):370-378.
ZHANG Qingfang,YANG Chao,YU Shuang,et al.Bacterial diversity of marine sediments in the Yellow Sea[J].Microbiology China,2020,47(2):370-378.
[9]Vavourakis C D,Andrei A S,Mehrshad M,et al.A metagenomics roadmap to the uncultured genome diversity in hypersaline soda lake sediments [J].Microbiome,2018,6(1):168.
[10]吴悦妮,冯凯,厉舒祯等.16S/18S/ITS扩增子高通量测序引物的生物信息学评估和改进[J].微生物学通报.2020,47(9):2897-2912.
WU Yueni,FENG Kai,LI Shuzhen,et al.In-silico evaluation and improvement on 16S/18S/ITS primers for amplicon high-throughput sequencing[J].Microbiology China,2020,47(9):2897-2912.
[11]Fung C,Rusling M,Lampeter T,et al.Automation of QIIME2 Metagenomic Analysis Platform[J].Current Protocols.2021.1(9):e254.
[12]Ling L L,Schneider T,Peoples A J,et al.A new antibiotic kills pathogens without detectable resistance [J].Nature, 2015,517(7535):455-459.
[13]Sherpa R T,Reese C J,Montazeri A H.Application of iChip to Grow "Uncultivable" Microorganisms and its Impact on Antibiotic Discovery [J] .J Pharm Pharm Sci, 2015,18(3):303.
[14]Lodhi A F,Zhang Y,Adil M,et al.Antibiotic discovery:combining isolation chip(iChip) technology and co-culture technique.[J] .Appl Microbiol Biotechnol,2018,102(17):7333-7341.
[15]阮继生,黄英.放线菌快速鉴定与系统分类[M].北京:科学出版社,2011.
RUAN Jisheng,HUANG Ying. Rapid identification and systematic classification of Actinomyces sp [M].Beijing:Science Press,2011.
[16]雷艳娟.骆驼蓬内生放线菌多样性及其次级代谢产物初探[D].阿拉尔:塔里木大学,2020.
LEI Yanjuan.The Diversity of Endophytic Actinobacteria from Peganum harmala and Primary Investigation of Their Secondary Metabolites [D].Aral:Tarim University,2020.
[17]王扬.一株糖霉菌新物种的多相分类及糖霉菌属CRISPR/Cas系统多样性分析[D].阿拉尔:塔里木大学,2018.
WANG Yang.The Polyphasic Taxonomy of a New Species of Glycomyces and Analyses of CRISPR/Cas System Diversity in Glycomyces [D].Aral:Tarim University,2018.
[18]劉峰,冯民权,王毅博.汾河入黄口夏季微生物群落结构分析[J].微生物学通报,2019,46(1):54-64.
LIU Feng,FENG Minquan,WANG Yibo.Microbial community structure of estuary of the Fenhe River into the Yellow River in summer[J].Microbiology China,2019,46(1):54-64.
[19]Kimura Z I,Okabe S.Acetate oxidation by syntrophic association between Geobacter sulfurreducens and a hydrogen-utilizing exoelectrogen [J] .ISME J,2013,7(8):1472-1482.
[20]Eyiuche N J,Asakawa S,Yamashita T,et al.Community analysis of biofilms on flame-oxidized stainless steel anodes in microbial fuel cells fed with different substrates [J].BMC Microbiol,2017,17(1):145.
[21]Ciok A,Budzik K,Zdanowski M K,et al.Plasmids of Psychrotolerant Polaromonas spp.Isolated From Arctic and Antarctic Glaciers - Diversity and Role in Adaptation to Polar Environments [J].Front Microbiol,2018,9:1285.
[22]Gan H M,Lee Y P,Austin C M.Nanopore Long-Read Guided Complete Genome Assembly of Hydrogenophaga intermedia,and Genomic Insights into 4-Aminobenzenesulfonate,p-Aminobenzoic Acid and Hydrogen Metabolism in the Genus Hydrogenophaga[J].Front Microbiol,2017,8:1880.
[23]Mu J,Cui X,Shao M J,et al.Microbial origin of bioflocculation components within a promising natural bioflocculant resource of Ruditapes philippinarum conglutination mud from an aquaculture farm in Zhoushan,China [J].PLoS One,2019,14(6):e0217679.
[24]Vester J K,Glaring M A,Stougaard P.Improved cultivation and metagenomics as new tools for bioprospecting in cold environments [J].Extremophiles,2015,19(1):17-29.
[25]Yang Q,Cai S,Dong S W,et al.Biodegradation of 3-methyldiphenylether(MDE) by Hydrogenophaga atypical strain QY7-2 and cloning of the methy-oxidation gene mdeABCD [J].Sci Rep.,2016,6:39270.
[26]Zhao T,Hu K,Li J,et al.Current insights into the microbial degradation for pyrethroids:strain safety,biochemical pathway,and genetic engineering[J].Chemosphere,2021,279:130542.
[27]Zhou S L,Sun Y,Yu M H,et al.Linking Shifts in Bacterial Community Composition and Function with Changes in the Dissolved Organic Matter Pool in Ice-Covered Baiyangdian Lake,Northern China[J].Microorganisms.,2020,8(6):883.
[28]肖方南,姜夢,李媛媛,等.塔里木河下游柽柳灌丛土壤真菌群落结构及多样性分析[J].干旱区地理,2021,44(3):759-768.
XIAO Fangnan,JIANG Meng,LI Yuanyuan,et al.Community structure and diversity of soil fungi in Tamarix chinensis shrubs in the lower reaches of Tarim River[J].Arid Land Geography,2021,44(3):759-768.
[29]李媛媛.塔里木河下游胡杨根际微生物群落结构与土壤理化性质的相关性分析[D].石河子:石河子大学,2022.
LI Yuanyuan.Structure of Soil Microbial Community in the Rhizosphere of Populus euphratica in the Lower Reaches of Tarim River and Its Correlation with Soil Physicochemical Properties [D].Shihezi:Shihezi University,2022.
Analysis of the bacterial communities structure and diversty of the Tarim River in different habitats
YAO Yuxiang1,2,WANG Guoqiang2,WANG Ke2,YI Sha2,YANG Xinya2,LUO Xioaxia1,2
(1.Key Laboratory Biological Resources Conservation and Utilization in Tarim Basin,Xinjiang Production and Construction Corps,Tarim University,Aral Xinjiang 843300,China; 2 School of Life Science and Technology,Tarim University,Aral Xinjiang 843300,China)
Abstract:【Objective】 Analysis of the bacterial communities structure and diversty of the Tarim River in different habitats.
【Methods】 The culture-free and cultivable methods were used in this study to explore the bacterial community structure and diversity of Tarim River,and also identified a potential new species by using the polyphasic taxonomy method.
【Results】 There were significantly difference of bacterial community between the original habitat and the artificially developed habitat.There was lower alpha diversity of the artificially developed habitat than the original habitat,and it had multiple difference of the beta diversity among the different habits.Based on the cultivable methods,the Proteobacteria and Actinomycota were dominant bacterial groups in these habitats.According to the polyphasic taxonomy method,a potential new species was identified.
【Conclusion】 There are multiple microbial resources in different habitats of the Tarim River.The bacterial community structure has been affected by human activities.
Key words:different habitats; bacterial community structure; diversity;the polyphasic taxonomy
Fund projects: Young and middle-aged scientific and technological innovation leader program of XPCC(1119119); Construction of the Germplasm Resource Bank of Actinomyces tarimus(TDZKKY202201); Tarim University Autonomous Region Postgraduate Research Innovation Project(TDGRI202204)
Correspondence author: LUO Xiaoxia(1982-),female,from Aral,Xinjiang,professor,research interests:microbial resources and their utilization,(E-mail) xxluo415@163.com
