PCR-HRM方法快速鉴定布鲁氏菌的初步应用
2015-06-24梁雪妮崔步云毛玲玲于静波薛文成孟冬娅
梁雪妮,崔步云,毛玲玲,任 微,于静波,薛文成,孟冬娅
PCR-HRM方法快速鉴定布鲁氏菌的初步应用
梁雪妮1,崔步云2,毛玲玲3,任 微4,于静波4,薛文成4,孟冬娅4
目的 本研究旨在建立准确、快速地鉴定布鲁氏菌菌种及生物分型的PCR-HRM(高分辨率熔解曲线)方法。方法 根据目的基因序列,参考文献合成6对基因扩增引物(1对布鲁氏菌属特异引物,5对种间特异引物),应用PCR-HRM方法鉴定布鲁氏菌属6个种19个生物型的标准菌株,并初步应用到临床分离的35株布鲁氏菌中。结果 采用布鲁氏菌属特异引物(Bspp),6个种19个生物型的标准菌株均扩增出同样形状的溶解曲线,与其它对照菌株的曲线不同;采用5对种特异引物,6个种的标准菌株均有特征性PCR-HRM曲线;临床分离的35株布鲁氏菌的PCR-HRM曲线与羊种布鲁氏菌标准菌株的一致。结论 该研究采用的PCR-HRM分析方法,获得了布鲁氏菌属及6个种标准菌株的不同曲线图,可准确鉴定临床分离的羊种布鲁氏菌,可用于临床微生物实验室疑似布鲁氏菌感染的初步鉴定。
布鲁氏菌;分子鉴定;高分辨溶解分析(PCR-HRM)
Supported by the grant from the Key Technologies R&D Program of Liaoning Province (No. 2011225021), and the Major National Science and Technology Special Project (No. 2009ZX10004-209) Corresponding author: Meng Dong-ya, Email: mengdongya@hotmail.com
布鲁氏菌属人畜共患病病原体,是潜在的致残性生物恐怖战剂,其引起的疾病为乙类传染病或二类动物疫病。布鲁氏菌属分为6个种19个生物型,即羊种(生物型1~3),牛种(生物型1~7和9),猪种(生物型1~5),绵羊副睾种,沙林鼠种及犬种(各1个生物型)[1]。根据中国疾病预防控制中心传染病预防控制所已收集到的核酸鉴定结果显示,我国已分离到除猪种4型、5型,羊种2型和沙林鼠种外的15个生物型,但在人群中主要致病菌株为羊种布鲁氏菌。布鲁氏菌生长缓慢,准确的生化鉴定及特异性凝集试验需要一些特殊试剂,费时、费力也不适合在普通实验室开展。临床急需一种能够准确、快速鉴定布鲁氏菌菌种的方法。本文旨在探讨PCR-HRM技术对布鲁氏菌种进行准确快速鉴定的可行性及初步应用。
1 材料与方法
1.1 菌株来源 由中国疾病预防控制中心提供的6个种19个生物型的标准菌株,即羊种(1~3型),牛种(1~7,9型),猪种(1~5型),绵羊副睾种、沙林鼠种和犬种DNA样品。2010—2013年9月间沈阳军区总医院及周边医院细菌培养分离的35株羊种布鲁氏菌,由中国疾病预防控制中心鉴定分型。阴性对照(苍白杆菌、流感嗜血杆菌、大肠埃希菌、铜绿假单胞菌、 鲍曼不动杆菌、黏质沙雷菌)均分离自沈阳军区总医院微生物实验室,经16s rRNA基因测序验证。
1.2 主要仪器与试剂 罗氏LightCycler®480 PCR扩增仪,SYBR®Premix Ex TaqTMII (Perfect Real Time)(宝生物,大连),High Resolution Melting Master(Roche)。所用引物参照文献[1]设计,引物序列、靶基因及用途等见表1。

表1 研究所需引物序列及用途
1.3 反应体系 通过预实验对反应体系进行优化,总体系20 μL,Master Mix为10 μL,每次试验固定其它加入量,分别做MgCl2(25 mmol/L)、Primer(10 μmol/L)、模板DNA梯度实验,剩余用H2O补足,根据各自梯度所对应的最佳溶解曲线选择出最适合此实验的反应体系。最终优化好的反应体系为Master Mix 10 μL,MgCl22.4 μL(终浓度3 mmol/L),Primer F、R各 0.4 μL(终浓度0.2 μmol/L),H2O 5.8 μL,模板DNA 1 μL(50 pg)。
1.4 PCR-HRM分析 通过预实验对PCR反应条件进行优化,优化后的PCR-HRM反应条件:预变性95 ℃ 10 min,变性95 ℃ 10 s、退火65 ℃~53 ℃(touchdown PCR:每个循环降低0.5 ℃)15 s、延伸72 ℃ 20 s(收集荧光信号)、扩增45个循环。PCR扩增结束后直接运行HRM程序:95 ℃变性1 min,40 ℃降温1 min,75 ℃~95 ℃连续升温(每上升1 ℃收集25次荧光信号),冷却40 ℃ 10 s。使用LightCycler®480 Gene Scanning Software分析软件,分析各种HRM曲线。
1.5 结果比对 将PCR-HRM鉴定结果与中国疾病预防控制中心的鉴定结果进行比较,评估HRM分析应用于布鲁氏菌分型的准确性及效果。
2 结 果
2.1 布鲁氏菌属PCR-HRM分析鉴定方法的建立 以布鲁氏菌属特异性引物Bspp进行PCR扩增,对其扩增产物进行HRM分析,可得到荧光强度变化率-温度熔解曲线,软件根据曲线的峰形、主峰的起落点和Tm值等对曲线进行归类。实验使用该方法对已知DNA序列的布鲁氏菌6个种19个生物型标准菌株进行鉴定,所有菌种的标准菌株均扩增出了同样形状的HRM曲线,计算机分析软件成功将其划分为同一类,与其它阴性对照(苍白杆菌、流感嗜血杆和其它阴性杆菌)菌株很好地鉴别开来,结果如图1所示。
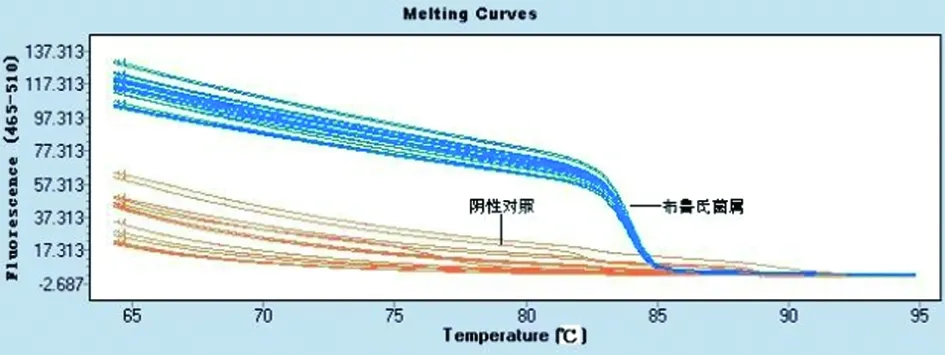
Primer Bspp; abscissa stands for temperature; ordinate stands for fluorescence.
2.2 布鲁氏菌属内不同种型菌株鉴别的PCR-HRM分析方法建立 分别以Bmel、Boa、Bcan、Bsui、Bneo为引物,对布鲁氏菌6个种19个生物型标准菌株进行PCR-HRM分析鉴定,得到了5套鉴定布鲁氏菌种的HRM标准曲线,详见图2-6。这5套标准曲线可以将6种布鲁氏菌相互鉴别。
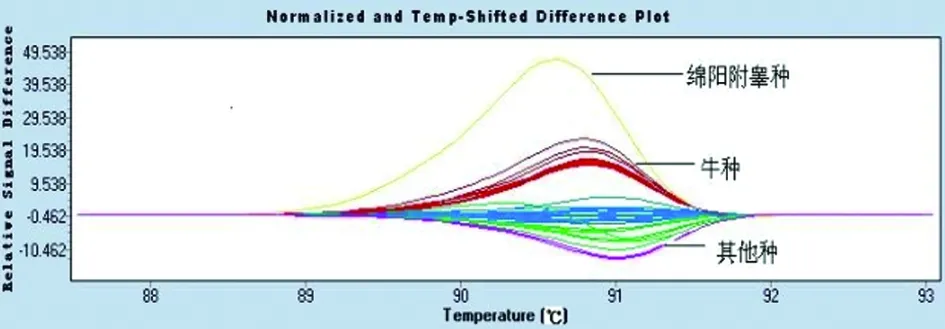
Primer Boa; abscissa stands for temperature; ordinate stands for relative fluorescence.
2.3 羊种布鲁氏菌临床分离株的PCR-HRM鉴定的初步应用 2010—2013年9月间,收集沈阳及周边地区临床分离的可疑布鲁氏菌35株。经中国疾病预防控制中心用传统培养鉴定方法及血清学凝集实验证实35株为羊种布鲁菌;布鲁氏菌属特异的PCR-HRM分析证实35株为布鲁氏菌属,再用Bmel标准PCR-HRM分析其均为羊种布鲁氏菌,PCR-HRM方法与传统培养血清学凝集试验鉴定方法的鉴定结果完全一致。微生物自动鉴定仪将35株临床分离株中的5株误鉴定为人苍白杆菌、流感嗜血杆菌、支气管鲍特菌、解脲寡源杆菌等,这5株经16S rRNA基因测序鉴定为羊种布鲁菌。PCR-HRM方法与微生物自动鉴定仪结果比较见表2。统计分析采用Fisher确切概率法,计算得出P=0.107 2,P≥0.05,差异无统计学意义。
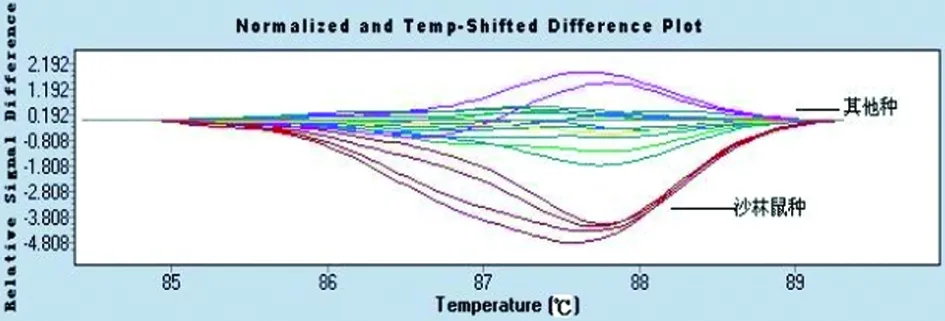
Primer Bneo; abscissa stands for temperature; ordinate stands for relative fluorescence.
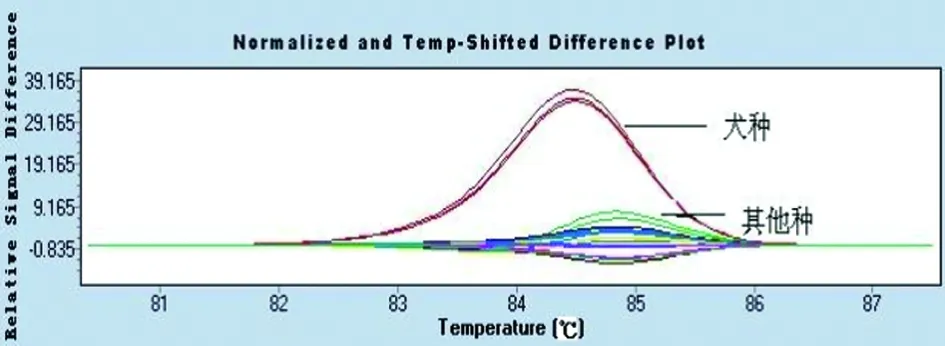
Primer Bcan; abscissa stands for temperature; ordinate stands for relative fluorescence.
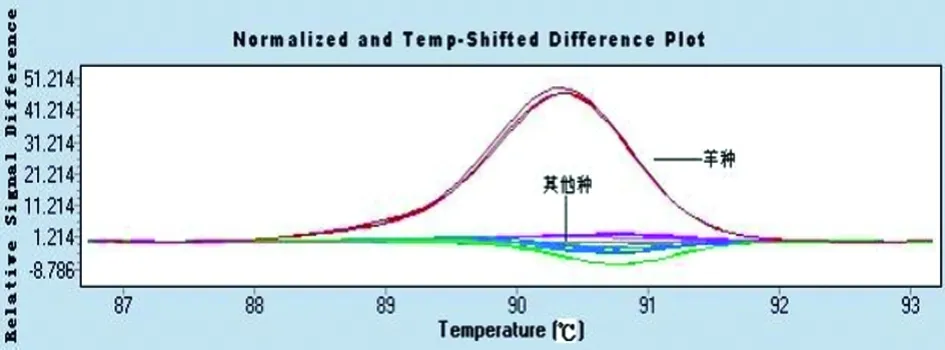
Primer Bmel; abscissa stands for temperature; ordinate stands for relative fluorescence.
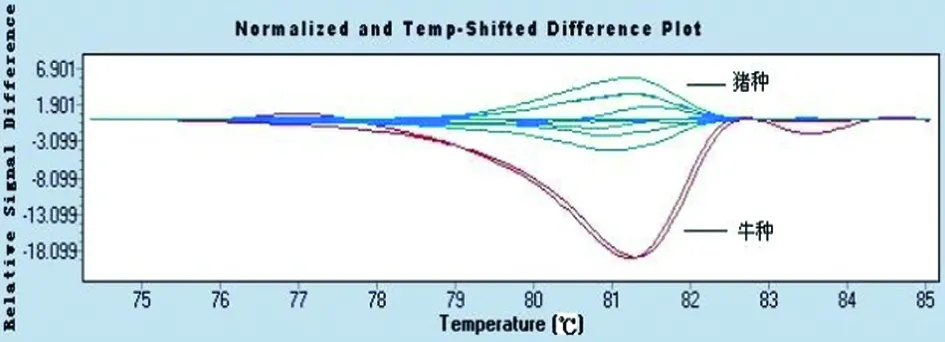
Primer Bsui; abscissa stands for temperature; ordinate stands for relative fluorescence.

表2 PCR-HRM方法和微生物自动鉴定仪对35株羊种布鲁氏菌的鉴定结果
3 讨 论
布鲁氏菌病为乙类传染病,布鲁氏菌可以通过实验室直接接触和气溶胶传播,国内外常有实验室防护不当导致操作人员感染报道,甚至是爆发性的人群感染[2-3]。实际工作中,在缺乏相关临床资料的情况下,实验人员也不会针对培养物采取特殊的防护,极易造成实验室感染[4]。由于该菌生长较慢,细菌鉴定仪器经常将布鲁氏菌鉴定为其它非传染性疾病病原菌[5-6]。
PCR-HRM方法具有快速、准确、不需特殊设备、重复性好、灵敏度高等优点。其分析原理[7-8]是在标准PCR试剂的基础上加入可与双链DNA结合的饱和性荧光染料, PCR扩增结束后即开始运行HRM程序, 将PCR产物自低到高(60 ℃~95 ℃)逐步升温,每次升温0.02 ℃~0.1 ℃,此过程双链DNA解链,荧光染料脱落,不再产生荧光信号。以荧光强度变化率为纵坐标,以温度为横坐标作图,即可得出对应于特定双链DNA的特征曲线。因DNA 序列的长度,GC 含量以及碱基互补性差异,每一段DNA的主峰高度、溶解温度Tm(主峰最高处所对应的温度)不同,因而每一段DNA均具有独特的PCR-HRM曲线。
本研究首先采用以布鲁氏菌属特异引物Bssp为基础的PCR-HRM分析方法,得到布鲁氏菌属特有的曲线图,并通过了该实验验证,即布鲁氏菌属中的6个种、19个生物型标准菌株及临床分离的35株布鲁氏菌均扩增出形态完全一致的HRM分析曲线,该溶解曲线形状与Jonas M.Winchell等报道[1]基本一致。而其它与布鲁氏菌亲缘关系较近或形态相似的革兰阴性小杆菌,如苍白杆菌和流感嗜血杆菌等均未得到特征性扩增曲线。证明该研究所应用的引物、优化后的PCR-HRM反应体系及反应条件能够将布鲁氏菌属与其它菌鉴别,适合常规微生物实验室对疑似布鲁氏菌属感染的初步鉴别诊断。
在此基础上,又合成了针对不同种布鲁氏菌株的5对引物,得到了不同种间特异的HRM曲线图。绵羊附睾种的glk基因有两个G-A突变,牛种有一个G-A突变,其它种不存在突变,因此在图2中引物Boa能够将绵羊附睾种、牛种与其它种分开;沙林鼠种的Glk基因有A-G突变,与其它种相比,沙林鼠种出现较晚的溶解曲线,图3中引物Bneo将沙林鼠种与其它种区分开;图4犬种的int-hyp基因有G-A突变,其溶解曲线与其它种不同;图5中,引物Bmel扩增的125bpDNA片段存在G-T突变,溶解曲线能将羊种与其它种区分开;图6中,引物Bsui将猪种、牛种与其它种分开;因此这些引物联合应用可以将不同种的布鲁菌相互鉴别开。
引起人类感染的布鲁氏菌主要为羊种(B.melitensis)、猪种(B.suis)、牛种(B.abortus)及犬种(B.canis),这些种在不同地区分布差异明显,本实验收集的菌株主要为羊种布鲁氏菌。实验利用Bmel引物为基础的PCR-HRM分析方法,成功地将35株沈阳军区总医院和周边医院临床分离的羊种布鲁氏菌与其它几种布鲁氏菌相鉴别,有助于对地区布鲁氏菌流行病学调查研究。
综合分析实验结果,布鲁氏菌PCR-HRM快速鉴定方法初步建立,其临床应用价值可观,但初步应用于本地区临床分离株后,经统计分析得出差异无统计学意义,尚不能认为微生物自动鉴定仪和PCR-HRM方法鉴定结果的准确率有明显差别,可能受样本数量不足影响,尚需大量样本进一步验证。从实验过程得出PCR-HRM方法具有快速、准确、灵敏度高等优点,推荐实验室在遇到可疑病例时,先用种属特异引物扩增分析,确定是否为布鲁氏菌,再根据本地流行病学资料,首先选择适合本地主要流行株鉴别的引物进行PCR-HRM分析,应能满足大部分临床诊断需求。接下来我将尝试应用PCR-HRM方法区分羊种布鲁氏菌的3个生物型。
[1]Winchell JM, Wolff BJ, Tiller R, et al. Rapid identification and discrimination ofBrucellaisolates by use of real-time PCR and High-Resolution Melt analysis[J]. J Clin Microbiol, 2010, 48(3): 697-702. DOI: 10.1128/JCM.02021-09
[2]Maley MW, Kociuba K, Chan RC. Prevention of laboratory-acquired brucellosis: significant side effects of prophylaxis[J]. Clin Infect Dis, 2006, 42(3): 433-434.
[3]Staszkiewicz J, Lewis CM, Colville J, et al. Outbreak ofBrucellamelitensisamong microbiology laboratory workers in a community hospital[J]. J Clin Microbiol, 1991, 29(2): 287-290.
[4]Noviello S, Gallo R, Kelly M, et al. Laboratory-acquired brucellosis[J]. Emerg Infect Dis, 2004, 10(10): 1848-1850.
[5]Xue WC, Ren XQ, Meng DY, et al. Analysis on the reason and clinical influence of mistaken identify ofBrucellainfection by automated microbial identification system[J]. Chin J Clin (Electron Ed), 2012, 6(21): 6943-6944. (in Chinese) 薛文成,任晓庆,孟冬娅,等.自动细菌鉴定仪对布鲁菌误鉴定原因及临床影响分析[J].中华临床医师杂志(电子版),2012,6(21):6943-6944.
[6]Yang J, Ren XQ, Meng DY, et al. Three misidentification cases ofBrucellaspecies by automated microbial identification system and literature review[J]. Int J Lab Med, 2012, 33(20): 2491-2494. (in Chinese) 杨婧,任晓庆,孟冬娅,等.细菌自动鉴定仪3例布鲁氏菌误鉴定及文献复习[J].国际检验医学杂志,2012,33(20):2491-2494.
[7]Writtwer CT, Read GH, Gundry CN, et al. High-resolution genotyping by amplicon melting analysis using LCGreen[J]. Clin Chem, 2003, 49(6 Pt 1): 853-860.
[8]Xue WC, Rao XC, Meng DY. Universal PCR coupled with high-resolution melting analysis for rapid detection and identification of microorganism: strategies and perspective[J]. Rev Med Microbiol, 2012, 23(1): 5-8. DOI:10.1097/MRM.0b013e32834f9e62
Clinical application of PCR and high resolution melting analysis for rapid identification ofBrucellaisolates
LIANG Xue-ni1,CUI Bu-yun2,MAO Ling-ling3, REN Wei4,YU Jing-bo4,XUE Wen-cheng4,MENG Dong-ya4
(1.PostgraduateTrainingBase,GeneralHospitalofShenyangMilitaryCommand,LiaoningMedicalUniversity,Jinzhou121001,China; 2.StateKeyLaboratoryforInfectiousDiseasePreventionandControl,CollaborativeInnovationCenterforDiagnosisandTreatmentofInfectiousDisease,NationalInstituteforCommunicableDiseaseControlandPrevention,Beijing102206,China; 3.LiaoningProvincialCenterforDiseaseControlandPrevention,Shenyang110005,China; 4.ClinicalLaboratory,GeneralHospitalofShenyangMilitaryCommand,Shenyang110016,China)
The aim of this study is to develop a rapid and accurately species typing method forBrucellaisolates by using High Resolution Melting (HRM) analysis. Six pairs of primers were used according to the reference for the sequence of purpose gene. Nineteen biotypes of six speciesBrucellastandard strains were identified by PCR-HRM analysis and this analysis was used to detect the 35 clinical isolates. Results showedBrucellaamplified specific melting curves were different from contrasted strains with primer Bspp. The six speciesBrucellastandard strains have own characteristic curve shape from each others by PCR-HRM analysis with five pairs of primers. Thirty-five clinical isolates ofBrucellahave entirely consistent with PCR-HRM curve shape withBrucellamelitensisstandard strains. So, PCR-HRM analysis methods can accurately identifyBrucellastrains, especially clinical isolatedBrucellamelitensis, and may be used in clinical microbiology laboratories.
Brucella; molecular diagnosis; PCR-HRM analysis
10.3969/cjz.j.issn.1002-2694.2015.03.015
辽宁省科技攻关计划资助项目(2011225021)和国家科技重大专项课题(2009ZX10004-209)联合资助(梁雪妮和崔步云同等贡献)
孟冬娅,Email:mengdongya@hotmail.com
1.辽宁医学院沈阳军区总医院研究生培养基地,锦州 121001; 2.传染病预防控制国家重点实验室,感染性疾病诊治协同创新中心,中国疾病预防控制中心传染病预防控制所,北京 102206; 3.辽宁省疾病预防控制中心,沈阳 110005; 4.沈阳军区总医院检验科,沈阳 110016
R378.5
A
1002-2694(2015)03-0255-05
2013-12-04;
2014-10-23
