C2株蓝氏贾第鞭毛虫SUMO特异性蛋白酶基因的克隆、生物信息学分析及其催化活性区的原核表达
2015-06-24李少东周英斌刘晓莉李思瑾田喜凤
李少东,周英斌,刘晓莉,禇 晗,李思瑾,田喜凤,王 洋
C2株蓝氏贾第鞭毛虫SUMO特异性蛋白酶基因的克隆、生物信息学分析及其催化活性区的原核表达
李少东,周英斌,刘晓莉,禇 晗,李思瑾,田喜凤,王 洋
目的 克隆C2株蓝氏贾第鞭毛虫(Giardialamblia,简称贾第虫)的SUMO-Specific Protease(SENP)基因,并对其序列进行生物信息学分析,原核表达贾第虫SENP的催化活性区。方法 提取C2株贾第虫基因组DNA,以基因组DNA为模板获得SENP编码区全长片段,连入克隆载体pGM-T,测序后进行生物信息学分析;根据分析结果克隆SENP的催化活性区,构建其原核表达载体pET-28a(+)-SENPc,在E.coliRosetta(DE3)中诱导表达,SDS-PAGE及Western blot观察表达结果。结果 成功克隆了C2株贾第虫SENP编码区全长序列,生物信息学分析显示C2株贾第虫SENP蛋白序列与WB株相同,二级结构以无规则卷曲为主,其催化活性区位于126-497aa,被一段插入序列分割成两个部分;构建了SENP催化活性区原核表达载体并在大肠杆菌中高效表达,在相对分子量约43 kD的位置出现目的蛋白条带,与理论值相符。结论 成功克隆了贾第虫SENP基因并原核表达了其催化活性区,为贾第虫SENP蛋白功能的研究提供了基础。
蓝氏贾第鞭毛虫;SUMO特异性蛋白酶;原核表达;生物信息学
小泛素相关修饰物(small ubiquitin-related modifier, SUMO)是一种与泛素结构和作用方式类似的小分子蛋白,由大约100个氨基酸残基组成,在真核生物中高度保守,广泛存在于真菌、原生动物、后生动物和植物中。SUMO能够通过酶促反应共价结合到靶蛋白上,修饰调节靶蛋白的功能,这个过程被称作SUMO化,是一种重要的翻译后修饰机制,广泛存在于所有的真核生物,其反应过程和作用方式类似于泛素化。与泛素化导致蛋白降解的功能不同,蛋白的SUMO化修饰会产生多种不同的生物学效应,参与包括胞质/核物质运输、转录调控、凋亡、调节蛋白稳定性、细胞周期、DNA修复、应激反应在内的许多重要的生物学过程[1-4]。SUMO化是一个动态可逆的过程,SUMO化的蛋白可以被SUMO特异性蛋白酶(SUMO-specific protease,SENP)催化而脱去结合的SUMO蛋白,这个过程称为去SUMO化。SENP属于C48半胱氨酸蛋白酶家族,具有两种功能,一方面能够特异性剪切SUMO蛋白C末端的甘氨酸和底物赖氨酸之间的异肽键,使SUMO蛋白从底物上脱落下来,即去SUMO化作用;另一方面,SENP还参与SUMO前体蛋白的成熟过程继而间接影响SUMO的结合。SENP存在于几乎所有的真核生物中,提示SENP对于真核生物的生存具有非常关键的作用。
蓝氏贾第鞭毛虫(Giardialamblia,简称贾第虫)是一种常见的肠道寄生性原虫,可以寄生在人和多种哺乳动物的小肠,引起以腹泻、腹痛、吸收不良为主要表现的贾第虫病。贾第虫是最早分化出来的真核生物之一,被称为“生物活化石”,在生物进化研究中处于重要地位[5]。贾第虫SUMO化研究目前未见相关报道,在之前的研究中,我们证实了贾第虫有单一种类的SUMO蛋白存在,且与其他物种SUMO蛋白在进化过程中距离较远[6]。通过对GenBank检索,我们发现WB株贾第虫基因组中存在编码SENP基因的可能序列,但未经过实验验证。本研究在C2株贾第虫中分离克隆SENP基因的同源基因,根据测序结果对SENP蛋白进行了生物信息学分析,并原核表达了其催化活性区,为贾第虫SENP功能的研究提供了前提。
1 材料与方法
1.1 材料 C2株贾第虫,大肠杆菌菌株EcoliDH5α、Rosetta(DE3),原核表达质粒pET-28a (+)均由本实验室保存;引物合成及测序工作由上海生工生物技术有限公司完成;2×Pfu Master Mix购自近岸蛋白质科技有限公司;限制性内切酶NcoⅠ和XhoⅠ购自NEB公司;T4 DNA连接酶购自大连宝生物公司;Taq DNA聚合酶、pGM-T连接试剂盒、血液基因组提取试剂盒、琼脂糖凝胶回收试剂盒、通用型DNA纯化回收试剂盒购自北京TIANGEN生化科技有限公司;鼠源抗His单克隆抗体、HRP标记的羊抗鼠IgG抗体购自北京康为世纪生物科技有限公司。
1.2 方法
1.2.1 虫体的培养 将液氮内冻存的C2株贾第虫复苏。置含改良TYI-S-33培养基的硼酸硅培养管内,于37 ℃培养。48~72 h后,虫体即呈对数生长期。选取虫体生长旺盛的培养管,置4 ℃冰浴,15 min后取出,在双手掌间多次滚搓培养管,使贴壁生长的虫体完全自管壁脱落。用血球计数板计数虫数,再用培养基将虫液浓度调为6×106~10×106个滋养体/mL。
1.2.2 C2株贾第虫SENP编码区的克隆 离心收集虫体团块,采用血液基因组提取试剂盒提取虫体基因组DNA。根据GenBank中WB株贾第虫SENP(XM_001705896.1)核酸序列设计扩增SENP全长编码区的引物SENP-SN(5′-ATGACTGCTGAACTGTTGCAGC-3′)及SENP-ASN(5′- CTAGCAGAGCTCGTCCAGATC -3′)。以贾第虫基因组DNA为模板,采用2×Pfu Master Mix扩增SENP全长编码区,反应条件:94 ℃预变性5 min;94 ℃ 30,52 ℃ 30 s,72 ℃ 2 min,共30个循环;72 ℃延伸10 min。反应完成后向反应体系中加入0.5 μL Taq DNA聚合酶,72 ℃延伸15 min加A尾。PCR产物中的目的带经琼脂糖凝胶回收试剂盒回收。回收产物估浓度后与pGM-T连接,连接产物转化EcoliDH5α,挑取阳性克隆提取质粒,经EcoRⅠ单酶切鉴定正确后送测序。
1.2.3 生物信息学分析 将C2株贾第虫SENP编码区测序结果通过软件翻译成蛋白质,应用PSIPRED、PredictProtein、APSSP2、SOPMA等多个蛋白序列分析平台在线分析该蛋白的二级结构特征。采用PredictProtein、 Psort服务器预测贾第虫SENP蛋白的亚细胞定位,PROSITE和InterPro服务器分析贾第虫SENP蛋白的催化活性区域。采用Phyre2服务器同源建模预测贾第虫SENP蛋白三级结构。
1.2.4 C2株贾第虫SENP催化区的克隆及原核表达载体的构建 根据PROSITE和InterPro预测的SENP催化区设计引物SENPc-SN(5′-CATGCCATGGGGACTCTCAGTGGCGCC-3′)和SENPc-ASN(5′-CCGCTCGAGACGTGCAATCATCTTCATA-3′),上下游引物分别含有NcoⅠ和XhoⅠ酶切位点(下划线部分)。以包含SENP全长序列的pGM-T-SENP为模板,扩增催化区编码序列, PCR产物经DNA纯化回收试剂盒回收。回收产物和pET-28a(+)载体同时用NcoⅠ和XhoⅠ双酶切,回收目的片段和线性载体并估浓度,以适当比例混合进行连接反应,连接产物转化EcoliDH5α。通过卡那霉素(50 μg/mL)抗性平板筛选阳性克隆,挑菌扩大培养,提取质粒后双酶切鉴定。
1.2.5 SENP催化活性区的诱导表达和鉴定 将双酶切鉴定正确的质粒转化原核表达菌株Rosetta(DE3),涂卡那霉素(50 μg/mL)和氯霉素(34 μg/mL)双抗平板,挑取单菌落于含双抗的LB培养基中,37 ℃震荡培养过夜,按1%的接种量转接于新鲜双抗LB培养基中。当OD600 达到0.4~0.6时加入终浓度1 mmol/L的异丙基-β-D-硫代半乳糖苷(IPTG)于30 ℃诱导表达5 h。煮沸裂解获得全菌体蛋白,分别经SDS-PAGE和Western blot检测融合蛋白表达情况。Western blot以抗His-Tag鼠源单克隆抗体(1∶1 000)为一抗,HRP标记的羊抗鼠IgG(1∶2 000)为二抗, DAB显色观察结果。
2 结 果
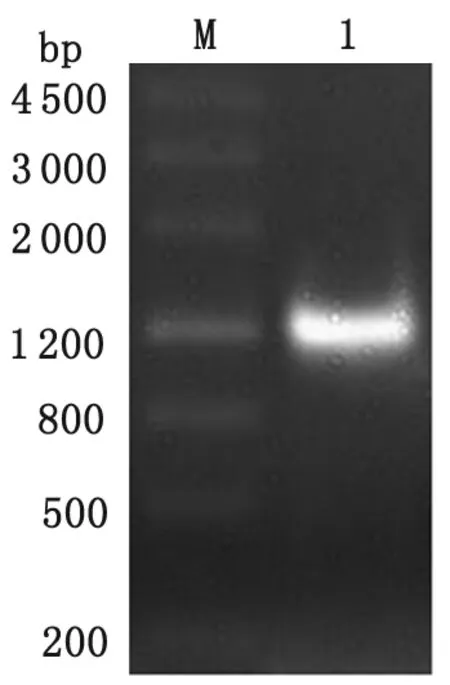
2.1 C2株贾第虫SENP全长编码区的克隆 PCR产物电泳显示,在大约1 620bp处出现特异性片段,与预期大小相符(图1);将目的片段连接到pGM-T载体后提取质粒经EcoRI单酶切,电泳可见1 620 bp左右的SENP目的片段 及3 015 bp的pGM-T载体两条电泳条带(图2),与预期结果一致。测序结果显示pGM-T-SENP克隆载体构建成功。
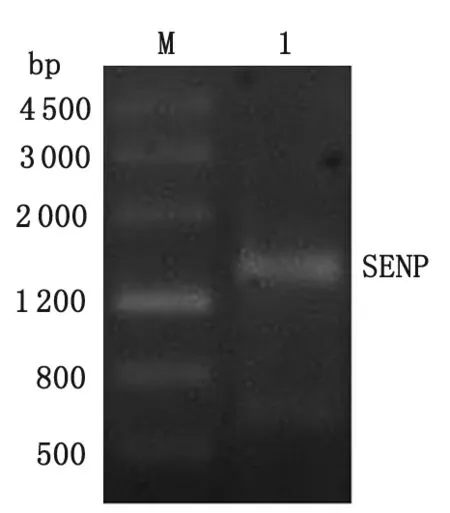
M:DNA分子量标准;1:PCR产物
2.2 C2株贾第虫SENP蛋白的生物信息学分析 BLAST序列比对显示C2株贾第虫SENP编码区序列与WB株完全相同。将贾第虫SENP核苷酸序列通过Gene Runner软件翻译成蛋白,结果显示贾第虫SENP蛋白由539个氨基酸组成,分子量约59.7 kD。PSIPRED服务器预测SENP蛋白的二级结构以无规则卷曲为主,占蛋白全长的66.98%;α螺旋结构占蛋白全长的27.46%;β折叠链结构占蛋白全长5.57%。这一结果与APSSP2、PredictProtein、SSpro4等服务器预测的结果大致相同。PredictProtein和Psort服务器预测该蛋白定位于核浆中。PROSITE和InterPro服务器预测该蛋白属于C48半胱氨酸蛋白酶家族,其ULP蛋白酶催化活性区域位于126-497aa。该催化活性区域又被一段插入序列分为两个关键的活性区段,即126-247aa和476-497aa。
M:DNA分子量标准;1:pGM-T-SENP载体EcoRI单酶切产物
Phyre2服务器选取可信度(100%)和一致性(33%)最高,且具有对应PDB 数据的d2iy1a1(腺病毒半胱氨酸蛋白酶Adenain)作为模版进行贾第虫SENP蛋白三级结构预测,其模建残基范围在101-528aa,涵盖了贾第虫SENP的关键催化区域,基本能够反映该蛋白的三级结构(图3)。
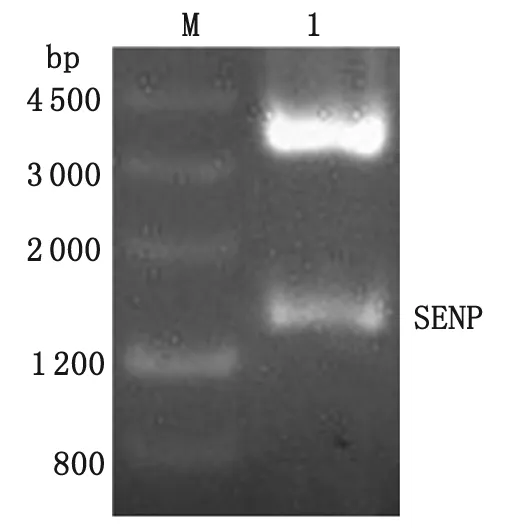
2.3 贾第虫SENP催化区域原核表达载体的构建 PCR产物电泳显示,在大约1 157 bp处出现特异性片段,与预期大小相符(图4);将目的片段连接到pET-28a(+)载体后提取质粒经NcoI和XhoI双酶切,电泳可见1 150 bp左右的SENPc目的片段及5 369 bp的载体pET-28a(+)两条电泳条带(图5),与预期结果一致,提示pET-28a(+)-SENPc表达载体构建成功。
2.4 C2株贾第虫SENPc融合蛋白的诱导表达和鉴定 菌液加入终浓度1.0 mmol/L IPTG,30 ℃诱导5 h后取少量菌液进行SDS-PAGE电泳,与未加IPTG诱导的pET28a(+)-SENPc转化菌比较,在43 kD处出现目的条带,与预期结果一致(图6)。
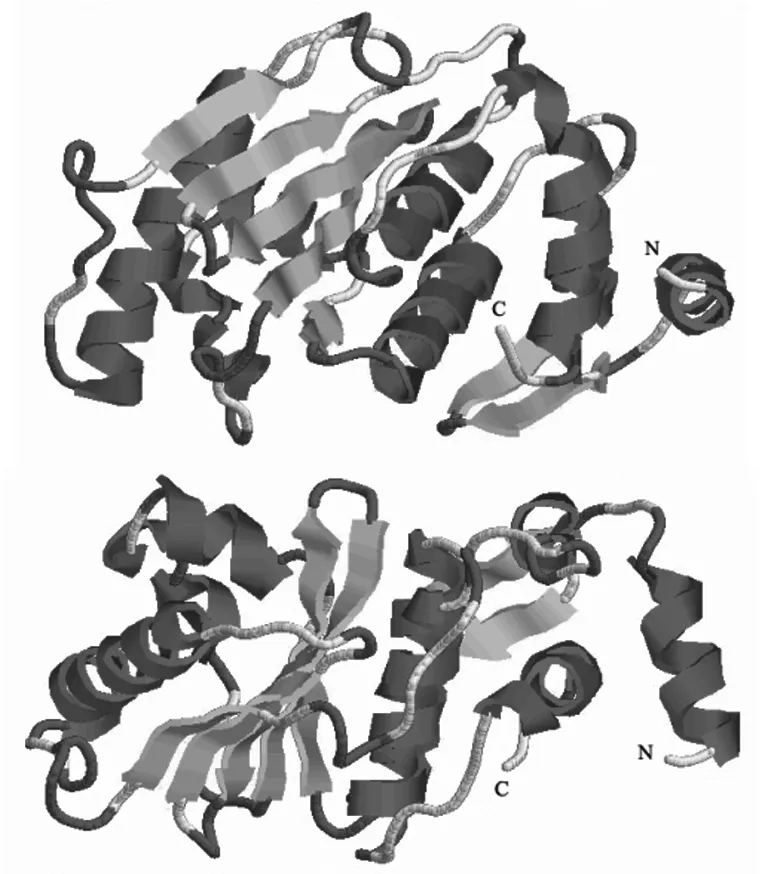
图3 Phyre2服务器由Adenain建模的贾第虫SENP蛋白三维模型
M:DNA分子量标准;1:PCR产物
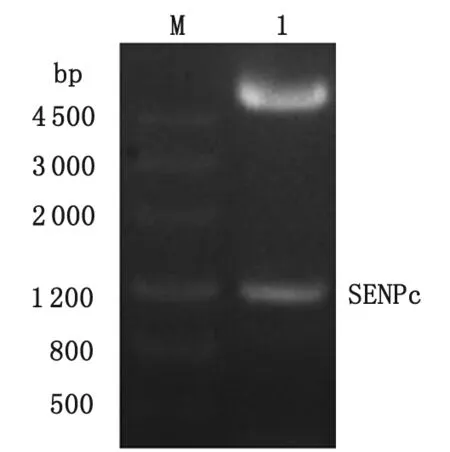
M:DNA分子量标准;1:pET-28a(+)-SENPc载体NcoI和XhoI双酶切产物
Western blot结果显示,在43 kD大小处出现一条阳性条带(图7)。
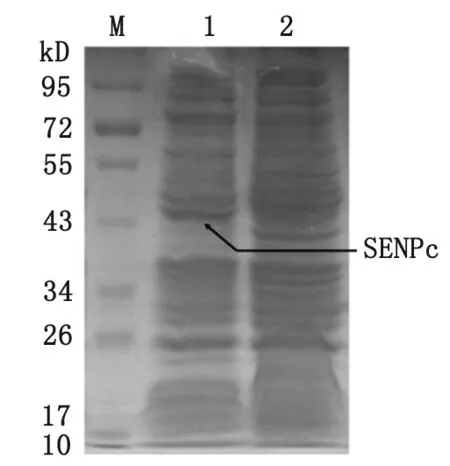
M:蛋白分子量标准;1:IPTG诱导后的pET28a(+)-SENPc/Rosetta(DE3)全菌体裂解物;2:未经诱导的pET28a(+)-SENPc/Rosetta(DE3)全菌体裂解物
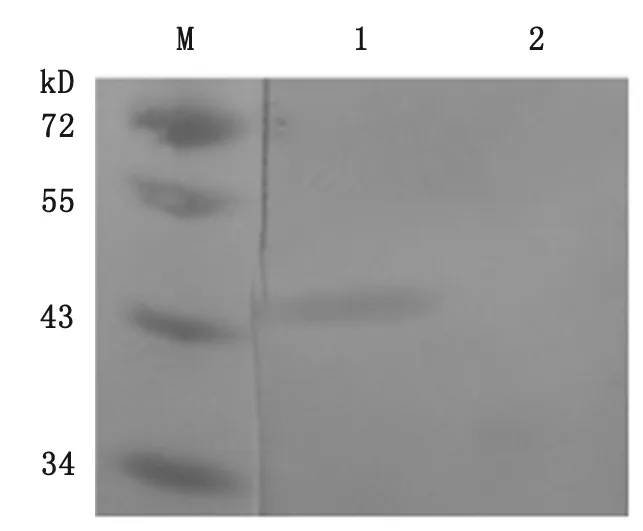
M:蛋白分子量标准;1:IPTG诱导后的pET28a(+)-SENPc/Rosetta(DE3)全菌体裂解物;2:未经诱导的pET28a(+)-SENPc/Rosetta(DE3)全菌体裂解物
3 讨 论
SENP是SUMO化修饰中的关键酶,在SUMO化和去SUMO化过程中均发挥着重要的作用。在细胞内,新合成的SUMO蛋白通常以无活性的前体形式存在,其C末端带有一段约2~11氨基酸长短不等的短肽。SENP将其C末端短肽切除掉,暴露出保守的C端Gly-Gly模序,此时SUMO转变为成熟的功能蛋白,成熟的SUMO蛋白在E1活化酶、E2结合酶以及E3连接酶的催化下通过其C端最后一个Gly的羧基末端与靶蛋白Lys残基上的ε氨基以异肽键结合。在去SUMO化过程中,SENP发挥异肽酶的作用,破坏Gly-Gly与靶蛋白Lys之间的异肽键,将靶蛋白释放。SENP对于真核生物的生存是必需的,将酵母的SENP——U1p1(Ubiquitin-like specific protease 1)敲除后,酵母不能生存[7-8]。
酿酒酵母的Ulp1是第一个鉴定出来的SENP,通过序列比对分析,陆续鉴定出第二个酵母SENP——Ulp2以及其它各种生物的SENP。所有SENP都存在一个约200aa保守的ULP 结构域,即催化结构域,绝大部分位于C端,只占了SENP全蛋白的一小部分。而占蛋白全长大部分的N端非催化结构域保守性很差,决定了该种SENP的亚细胞定位及其对底物的识别特异性。有研究显示,SENP对SUMO分子和底物的识别主要依赖底物的空间结构,而不像其他蛋白酶只识别特定的氨基酸序列[9]。多数真核生物都存在一种以上的SENP,例如酵母有2种SENP,在哺乳动物中目前已发现6 种SENP,而某些植物,例如拟南芥,存在多达7种SENP,其中一些SENP还存在剪接造成的异构体,进一步加大了SENP家族的多样性。但通过序列比对,我们发现贾第虫仅存在此一段带有ULP结构域的同源蛋白,且同源性较低,这一结果进一步支持了贾第虫在进化中的原始性。
虽然SUMO分子与泛素分子在空间结构上非常相似,且SUMO化和泛素化是两个程序和机制非常类似的修饰过程,SENP与去泛素蛋白酶的相似性却很低,而与病毒蛋白酶具有很高的相似性[10]。因此,在贾第虫SENP三级结构模型构建上,选择了相似性最高的腺病毒半胱氨酸蛋白酶Adenain作为模版。对贾第虫SENP的催化活性区的分析显示,贾第虫SENP与哺乳动物的SENP6/7最为类似,其ULP催化活性区被一段插入序列分隔成了两个部分[11]。其预测核浆内定位的特点也类似于SENP6/7,但哺乳动物的SENP6/7主要作用SUMO2/3且没有SUMO前体加工的能力[12]。贾第虫SENP的亚细胞定位和酶活性仍需进一步实验证实。
由于贾第虫基因组具有缺乏内含子的原始特性,在贾第虫SENP编码基因的克隆中,我们直接用C2株贾第虫的基因组DNA取代cDNA作为模板,简化了实验操作的步骤。此外,鉴于贾第虫SENP编码序列中含有大量大肠杆菌的稀有密码子,我们采用了携带稀有密码子tRNA编码质粒的Rosetta(DE3)菌株,从而避免因大肠杆菌密码子使用频率差别导致的表达限制。C2株贾第虫SENP蛋白的克隆、生物信息学分析以及活性区的原核表达,证实了贾第虫中该蛋白的存在,为贾第虫SENP功能的研究提供了基础。
[1]Hannoun Z, Greenhough S, Jaffray E, et al. Post-translational modification by SUMO[J]. Toxicology, 2010, 278(3): 288-293. DOI: 10.1016/j.tox.2010.07.013
[2]Lee SW, Lee MH, Park JH, et al. SUMOylation of hnRNP-K is required for p53-mediated cell-cycle arrest in response to DNA damage[J]. EMBO J, 2012, 31(23): 4441-4452. DOI: 10.1038/emboj.2012.293
[3]Almedawar S, Colomina N, Bermudez-Lopez M, et al. A SUMO-dependent step during establishment of sister chromatid cohesion[J]. Curr Biol, 2012, 22(17): 1576-1581. DOI: 10.1016/j.cub.2012.06.046
[4]Psakhye I, Jentsch S. Protein group modification and synergy in the SUMO pathway as exemplified in DNA repair[J]. Cell, 2012, 151(4): 807-820. DOI: 10.1016/j.cell.2012.10.021
[5]Adam RD. Biology ofGiardialamblia[J]. Clin Microbiol Rev, 2001, 14(3): 447-475. DOI: 10.1128/CMR.14.3.447-475.2001
[6]Wang Y, Wang Y, Li WW, et al. Cloning, prokaryotic expression and bioinformatics analysis ofGiardialambliaC2 strain SUMO gene[J]. Chin J Zoonoses, 2013,29(8): 785-790. DOI: 10.3969/cjz.j.issn.1002-2694.2013.08.011 (in Chinese) 王洋, 王沂, 李巍伟,等. C2株蓝氏贾第鞭毛虫SUMO基因的克隆、原核表达和生物信息学分析[J]. 中国人兽共患病学报, 2013,29(8): 785-790.
[7]Li SJ, Hochstrasser M. A new protease required for cell-cycle progression in yeast[J]. Nature, 1999, 398(6724): 246-251. DOI: 10.1038/18457
[8]Li SJ, Hochstrasser M. The Ulp1 SUMO isopeptidase: distinct domains required for viability, nuclear envelope localization, and substrate specificity[J]. J Cell Biol, 2003, 160(7): 1069-1081. DOI: 10.1083/jcb.200212052
[9]Mukhopadhyay D, Dasso M. Modification in reverse: the SUMO proteases[J]. Trends Biochem Sci, 2007, 32(6): 286-295. DOI: 10.1016/j.tibs.2007.05.002
[10]Iyer LM, Koonin EV, Aravind L. Novel predicted peptidases with a potential role in the ubiquitin signaling pathway[J]. Cell Cycle, 2004, 3(11): 1440-1450. DOI: 10.4161/cc.3.11.1206
[11]Lima CD, Reverter D. Structure of the human SENP7 catalytic domain and poly-SUMO deconjugation activities for SENP6 and SENP7[J]. J Biol Chem, 2008, 283(46): 32045-32055. DOI: 10.1074/jbc.M805655200
[12]Alegre KO, Reverter D. Swapping small ubiquitin-like modifier (SUMO) isoform specificity of SUMO proteases SENP6 and SENP7[J]. J Biol Chem, 2011, 286(41): 36142-36151. DOI: 10.1074/jbc.M111.268847
Cloning and bioinformatics analysis ofGiardialambliaC2 strain SENP gene and prokaryotic expression of SENP catalytic domain
LI Shao-dong,ZHOU Ying-bin,LIU Xiao-li,ZHU Han,LI Si-jin,TIAN Xi-feng,WANG Yang
(CollegeofLifeSciences,HebeiUnionUniversity,Tangshan063000,China)
SUMOylation is a post-translational modification involved in various cellular processes. SUMO-specific protease (SENP) regulates SUMOylation by removing SUMO from conjugated substrates (deSUMOylation) and promoting maturation of SUMO precursor. In order to expressGiardialambia(C2 strain) SENP catalytic domain inE.coli, the full-length open reading frame of SENP was amplified by PCR fromGiardialambliagenome DNA. The PCR product about 1 620 bp was cloned into cloning vector pGM-T. Sequencing result showed the sequence of SENP in C2 strain was identical with that inGiardiaWB strain. Bioinformatics analysis showed that SENP protein possessed a 372 aa discontinuous ULP catalytic domain at C-terminal. The catalytic domain of SENP was cloned into prokaryotic expression vector pET-28a(+). The recombinant vector pET-28a(+)-SENPc was transformed intoE.coliRosetta(DE3), then the recombinant SENPc protein was expressed by IPTG induction. SDS-PAGE and Western blot using anti-His Tag antibody showed that the expression product of SENPc was a fusion protein with a molecular weight of 43 kD. The successful prokaryotic expression and bioinformatics analysis ofGiardialambliaSENP protein provide basis for further functional study of SENP.
Giardialamblia; SUMO-specific protease; prokaryotic expression; bioinformatics
Wang Yang, Email: konig718@163.com
10.3969/cjz.j.issn.1002-2694.2015.03.003
国家自然科学基金(No. 31471954)和河北省青年科学基金(No.C2012401039)联合资助
王洋,Email:konig718@163.com
河北联合大学生命科学学院,唐山 063000
R382.21
A
1002-2694(2015)03-0203-05
Supported by the National Natural Science Foundation of China (No. 31471954) and the Hebei Province Science Foundation for Youths (No. C2012401039)
2014-08-19;
2014-10-23
