宁波野生鼠类携带的沙粒病毒检测和种系进化分析
2015-06-24马思杰邹春颖童淑梅
胡 群,马思杰,邹春颖,童淑梅,梅 勇
宁波野生鼠类携带的沙粒病毒检测和种系进化分析
胡 群,马思杰,邹春颖,童淑梅,梅 勇
目的 对宁波地区野生鼠类携带的沙粒病毒进行检测并分析病毒基因序列特征。方法 设计沙粒病毒L、S基因的简并引物,采用RT-PCR方法对宁波口岸捕获的鼠类样品进行沙粒病毒检测,分离病毒,对PCR产物进行测序及系统发生分析。结果 共检测73份鼠类样品,其中12份褐家鼠经检测为阳性,分离获得沙粒病毒株DX1401,该病毒株S片段扩增片段大小为413 bp,L片段扩增大小1 204 bp。S片段的系统发生分析显示DX1401与Mobala 病毒株ACAR3080位于同一分支,与Mobala 病毒株ACAR3080遗传距离最为接近,值为0.467;L片段的系统发生分析显示DX1401与Lassa病毒株Josiah、NL、Z148、Bamba-R114、Soromba-R、Nig08-A37、Nig08-A47, Mobala病毒株ACAR3080,Morogoro病毒株13017/2004,Mopeia病毒株Mozambique、AN 21366-BNI位于同一组,与Mobala 病毒株ACAR 3080遗传距离最为接近,值为6.953。结论 本研究证实了宁波地区野生鼠类中存在沙粒病毒流行。
鼠类;沙粒病毒;检测;系统发生分析
Funded by the Science Project of Natural Science Foundation of Ningbo (No. 2013A610240)
沙粒病毒(arenavirus)属于沙粒病毒科(arenaviridae)沙粒病毒属(arenavirus),为有包膜的单股负链RNA病毒,基因组由L和S两个节段组成,已知至少由23种病毒组成,一些新的沙粒病毒还在不断的发现中[1]。主要引起的人类疾病有淋巴细胞性脉络丛脑膜炎、拉沙热、阿根廷出血热、和玻利维亚出血热等,其中鼠类携带和感染淋巴细胞性脉络丛脑膜炎病毒(Lymphocytic choriomeningitis virus,LCMV),主要分布在欧美和亚洲,国内曾有少数病例。拉沙热病毒(Lassa virus,LASV)主要发生在西非,能引起发热、头疼、肌痛等症状,严重有出血和休克,病死率为15%~25%。鸠宁病毒(Junin virus,JUNV)、马秋波病毒(Machupo virus,MACV)、瓜纳瑞托病毒(Guanarito virus)能引起人类出血性疾病,而且感染性极高[2-3]。虽然,我国目前尚未有沙粒病毒感染病例的报道,但是为了了解宁波地区鼠样品中是否存在沙粒病毒的自然感染情况,我们依据沙粒病毒特有蛋白质氨基酸序列,设计了两对分别扩增沙粒病毒L、S片段特定基因的简并引物,以宁波口岸地区捕获的73只鼠类样品为模板,进行沙粒病毒筛查,并对分离的病毒株进行了遗传学分析,现将结果报告如下。
1 材料与方法
1.1 样品来源 2013年6月至12月期间从宁波北仑港区监测点捕获73只鼠类样品,鼠种构成为黑线姬鼠(Apodemus agrarius)26只、褐家鼠(Rattus norvegicus)25只、臭鼩(Suncus murinus)13只、小家鼠(Mus musculus)6只、社鼠(Rattus niviventer)3只。
1.2 试剂 Ribounclease inhibition、100bp ladder marker、琼脂糖均购自宝生物工程(大连)有限公司,病毒RNA 提取试剂盒、一步法 RT-PCR 试剂盒购自德国Qiagen公司。
1.3 方法
1.3.1 病毒RNA提取 取约50 mg鼠肺样品,研磨匀浆后参照Qiagen公司RNA提取试剂盒使用说明书提取病毒RNA,用40 μL无RNA酶的超纯水溶解,于-80 ℃保存备用。
1.3.2 引物合成 根据基因库中不同沙粒病毒属的病毒聚合酶蛋白氨基酸序列和核蛋白N氨基酸序列,设计扩增沙粒病毒L基因片段和S基因片段两对简并引物,委托上海英骏生物工程公司合成,引物序列见表1。

表1 沙粒病毒L和S基因片段引物
1.3.3 RT-PCR反应体系 采用一步法RT-PCR试剂盒进行病毒模板目标基因片段扩增,反应体系(50 μL):其中 RNase Free Water 19 μL ,5×RT-PCR Buffer 10 μL,10 mmol/L dNTP Mixture 2 μL,Enzyme Mix 2 μL,Ribonuclease Inhibitor 1 μL,20 μmol/L 上游引物 4 μL,下游引物 2 μL,RNA模板10 μL。反应程序为60 ℃ 1 min,42 ℃ 10 min,50 ℃ 30 min,95 ℃ 15 min反转录结束后,94 ℃ 30 s,52 ℃ 30 s,72 ℃ 1 min,35个循环,72 ℃ 7 min,4 ℃保存。
1.3.4 扩增产物序列分析 取5 μL PCR扩增产物用1.5%的琼脂糖凝胶电泳,凝胶成像系统观察结果。剩余扩增产物送上海英俊生物工程公司进行测序,测序后得到的片段序列登录NCBI进行同源性比对,选择同源性较为接近的沙粒病毒代表株进行种系进化分析,利用Mega5.2软件构建系统发生树和计算遗传距离。
2 结 果
2.1 RT-PCR检测和病毒分离 12份从褐家鼠中分离的鼠肺样品经RT-PCR检测阳性,褐家鼠病毒阳性率达到48%,S基因片段与L基因片段检测结果一致。选择阳性较强的3份鼠肺,制成20%悬液接种于Vero-E6细胞培养后,成功分离到1株病毒株,命名为DX1401,该病毒株再次经沙粒病毒L基因和S基因简并引物进行PCR扩增,扩增产物电泳后可见明显的阳性扩增条带,条带大小约为413 bp和1 204 bp,与预期大小一致。电泳条带位置,见图1。
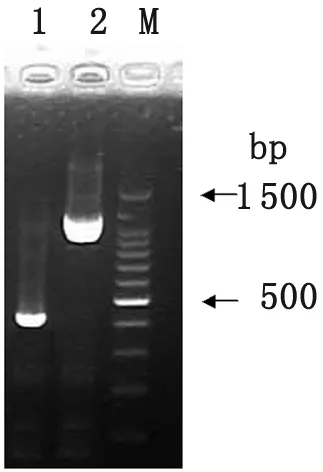
1:S基因RT-PCR扩增产物,大小约413 bp;2:L基因RT-PCR扩增产物,大小约1 204 bp;M:分子量对照
2.2 测序及同源性比对 将病毒株DX1401的 S、L片段测序序列,上传至GenBank,获得登录号分别为KJ144198.1和KJ144197.1。通过Blast检索与GenBank收录的病毒株亲源性比较,检索所获得的100个相似序列均为沙粒病毒属病毒相应基因片段,S片段与其他病毒基因片段相似度范围为72%~83%,L片段与与其他病毒基因片段相似度范围为66%~93%。
2.3 种系进化分析 根据Blast同源性比对结果,选择与病毒株DX1401亲缘关系较近的不同沙粒病毒代表株,分别构建S基因和L基因种系进化树。S基因种系进化分析结果见图2可知,DX1401与Mobala病毒ACAR3080位于同一分支。L基因种系进化分析结果见图3可知,DX1401与Lassa病毒株Josiah、NL、Z148、Bamba-R114、Soromba-R、Nig08-A37、Nig08-A47, Mobala病毒株ACAR3080,Morogoro病毒株13017/2004,Mopeia病毒株Mozambique、AN 21366-BNI位于同一组。
2.4 遗传距离分析 从表3可知, DX1401与沙粒病毒属Mobala 病毒株ACAR 3080遗传距离最为接近,S基因和L基因所对应的遗传距离分别为0.467和6.953。

表2 DX1401与不同沙粒病毒间S基因和L基因基因距离
注:“-”表示GenBank里未收录该病毒株所对应的基因序列
Note:“-” means the corresponding gene sequences of the virus strain were not recorded in the GenBank.

图2 不同沙粒病毒 S基因PCR扩增产物构建的系统发生树
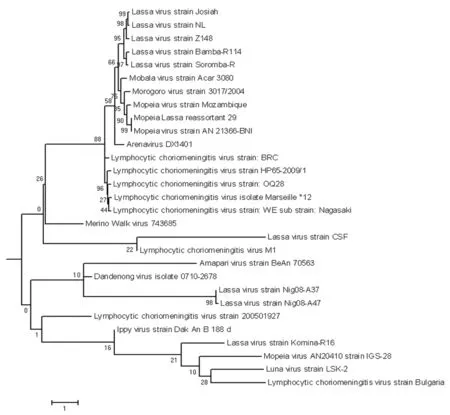
图3 不同沙粒病毒 L基因PCR扩增产物构建的系统发生树
3 讨 论
沙粒病毒是一类由啮齿动物为宿主、能够以多种方式感染并引起人类不同程度病死率疾病的 RNA 病毒, 近来陆续有各种沙粒病毒被检测分离的报道[4-7]。但是目前国内的沙粒病毒的检测研究依然非常少,在鼠类自然疫原性感染方面的研究更处于空白。
本研究采用了CODEHOP方法设计沙粒病毒科的简并引物,所设计引物由3′端简并的核心区和5′端为非简并夹板区2个部分组成,与常规方法设计的简并引物相比具有简并度低、退火温度高、特异性更强等优势[8-11]。沙粒病毒基因组RNA分大小两个片段:大片段L长7.2~7.5 kb,包含3′的L基因和5′端的Z基因,分别编码病毒的RNA聚合酶蛋白L和一种可能具有转录复制调节功能的Z蛋白。小片段S长3.4~3.5 kb,包含3′端的N基因和5′端的G基因,分别编码病毒的核蛋白N和病毒前体糖蛋白G。根据CODEHOP方法设计原理,首先从基因库里获取不同沙粒病毒株RNA聚合酶蛋白L和核蛋白N的氨基酸序列,然后分别寻找蛋白质上的保守氨基酸区域,进一步挑选两个保守区域作为上下游引物的设计位点,所获得的两对简并引物能够分别扩增沙粒病毒L片段和S片段的基因片段进行扩增,从而能够检测沙粒病毒科内不同基因型别的病毒[12],从宁波口岸监测捕获的鼠类样品检测结果来看,证实了宁波口岸鼠类样品中存在沙粒病毒感染的情况,褐家鼠为该病毒的自然宿主,阳性率高达48%。
将分离的沙粒病毒株DX1401扩增产物基因序列上传至GenBank进行Blast同源性检索,所获得的100个同源性较为接近的序列来源均为沙粒病毒,随后从中选择典型的沙粒病毒株进行进一步的系统进化分析,从构建的两个系统进化树和遗传距离分析来看,DX1401与Mobala 病毒株ACAR 3080的亲源关系最为接近,该病毒株最早是在1983年从非洲的柔毛鼠(African soft-furred rat)中分离[13],已有的资料显示,Mobala病毒并不引起人类的疾病[14-15]。
[1]Xiong CH, Jiang QW. Review: Arenaviruses[J]. Chin J Dis Ctrl Prev, 2009, 13(3): 362-365. (in Chinese) 熊成龙, 姜庆五. 沙粒病毒的研究进展[J]. 中国疾病控制杂志, 2009, 13(3): 362-365.
[2]Ishii A, Thomas Y, Moonga L, et al. Molecular surveillance and phylogenetic analysis of Old World arenaviruses in Zambia[J]. J Gen Virol, 2012, 93(10): 2247-2251. DOI: 10.1099/vir.0.044099-0
[3]Emonet SE, Urata S, de la Torre JC. Arenavirus reverse genetics: New approaches for the investigation of arenavirus biology and development of antiviral strategies[J]. Virology, 2011, 411(2): 416-425. DOI: 10.1016/j.virol.2011.01.013
[4]Gonzalez JP, McCormick JB, Saluzzo JF, et al. An arenavirus isolated from wild-caught rodents (Pramys species) in the Central African Republic[J]. Intervirology, 1983, 19(2): 105-112.
[5]Yama IN, Cazaux B, Britton-Davidian J, et al. Isolation and characterization of a new strain of lymphocytic choriomeningitis virus from rodents in southwestern France[J]. Vector Borne Zoonotic Dis, 2012, 12(10): 893-903. DOI: 10.1089/vbz.2011.0892
[6]Kessell A, Hyatt A, Lehmann D, et al. Cygnet River virus, a novel orthomyxovirus from ducks, Australia[J]. Emerg Infect Dis, 2012, 18(12): 2044-2046. DOI: 10.3201/eid1812.120500
[7]Palacios G, Savji N, Hui J, et al. Genomic and phylogenetic characterization of Merino Walk virus, a novel arenavirus isolated in South Africa[J]. J Gen Virol, 2010, 91(5): 1315-1324. DOI: 10.1099/vir.0.017798-0
[8]Rose TM, Henikoff JG, Henikoff S. CODEHOP (COnsensus-DEgenerate Hybrid Oligonucleotide Primer) PCR primer design[J]. Nucleic Acids Res, 2003, 31(13): 3763-3766. DOI: 10.1093/nar/gkg524
[9]Rose TM, Schultz ER, Henikoff JG, et al. Consensus-degenerate hybrid oligonucleotide primers for amplification of distantly related sequences[J]. Nucleic Acids Res, 1998, 26(7): 1628-1635.
[10]Zlateva KT, Crusio KM, Leontovich AM, et al. Design and validation of consensus-degenerate hybrid oligonucleotide primers for broad and sensitive detection of corona-and toroviruses[J]. J Virol Methods, 2011, 177(2): 174-183. DOI: 10.1016/j.jviromet.2011.08.005
[11]Baines JE, McGovern RM, Persing D, et al. Consensus-degenerate hybrid oligonucleotide primers (CODEHOP) for the detection of novel papillomaviruses and their application to esophageal and tonsillar carcinomas[J]. J Virol Methods, 2005, 123(1): 81-87.
[12]Hu Q, Ma SJ, Qiu JL, et al. Development of CODEHOP RT-PCR to detect Arenaviruses carried by rodent[J]. J Pan Biol, 2013, 8(12): 1074-1077. (in Chinese) 胡群, 马思杰,裘炯良,等.鼠类感染沙粒病毒CODEHOP RT-PCR检测方法的建立[J].中国病原生物学杂志,2013,8(12):1074-1077.
[13]Gonzalez JP, Mccormick JB, Georges AJ, et al. Mobala virus: Biological and physicochemical properties of a new arenavirus isolated in the Central African Republic[J]. Virologie, 1984, 135(2): 145-158. DOI: 10.1016/S0769-2617(84)80051-9
[14]Vieth S, Drosten C, Lenz O, et al. RT-PCR assay for detection of Lassa virus and related Old World arenaviruses targeting the L gene[J]. Trans R Soc Trop Med Hyg, 2007, 101(12): 1253-1264.
[15]Gunther S, Hoofd G, Charrel R, et al. Mopeia virus-related arenavirus in natal multimammate mice, Morogoro, Tanzania[J]. Emerg Infect Dis, 2009, 15(12): 2008-2012. DOI: 10.3201/eid1512.090864
Detection and phylogenetic analysis of arenavirus carried by wild rodents in Ningbo, China
HU Qun,MA Si-jie,ZHOU Chun-yin,TONG Shu-mei,MEI Yong
(DaxieExit-EntryInspectionandQuarantineBureau,Ningbo315812,China)
To detect and phylogeneticaly analyze arenavirus carried by wild rodents in Ningbo, China, two pairs of degenerate-primers were designed to amplify the S and L gene of arenavirus, and then RT-PCR was applied to detect arenavirus carried by rodents which captured from Ningbo port area. All 73 rodents samples were detected, of which 12Rattusnorvegicuswere positive, an arenavirus virus strain named DX1401 were separated. The S gene amplified products of DX1401 was about 413 bp, and the L gene was 1 204 bp. The phylogenetic analysis of S segments showed that DX1401 strain was in one branch of phylogenetic tree with Mobala virus strain ACAR3080. The genetic distance to Mobala virus strain ACAR3080 was the closest, with the value of 0.467; the phylogenetic analysis of L segments showed that DX1401 strain were in one group of phylogenetic tree with Lassa virus strain Josiah, NL, Z148, Bamba-R114, Soromba-R, Nig08-A37, Nig08-A47, Mobala virus strain ACAR3080, Morogoro virus strain 13017/2004, Mopeia virus strain Mozambique, and AN 21366-BNI. The genetic distance to Mobala virus strain ACAR3080 was the closest, with the value of 6.953. In conclusion, the study confirmed the existence of arenavirus popular in wild rodents in Ningbo, China.
rodents; arenavirus; detection; phylogenetic analysis
10.3969/cjz.j.issn.1002-2694.2015.03.010
宁波市自然科学基金项目(2013A610240)
大榭出入境检验检疫局,宁波 315812; Email: nbhuqun@sina.com
R373.9
A
1002-2694(2015)03-0235-05
2014-06-09;
2014-10-22
