香蕉枯萎病菌内源报告基因比卡菌素聚酮合酶编码基因Bik1的鉴定及应用
2022-03-25王艳玮曾凡云漆艳香丁兆建谢艺贤张欣彭军
王艳玮 曾凡云 漆艳香 丁兆建 谢艺贤 张欣 彭军




摘 要:香蕉枯萎病是由尖孢鐮刀菌古巴专化型( f. sp. , Foc)引起的香蕉毁灭性土传病害,其中4号生理小种(Foc4)能感染几乎所有的香蕉品系,危害最严重。本研究克隆鉴定Foc4比卡菌素聚酮合酶编码基因( gene, ),编码一个由2036个氨基酸组成的多功能酶,具有I型PKS聚酮合酶的保守结构域,包含酰基转移酶功能域[acyl-carrier protein (ACP) transacylase, SAT],β-酮脂酰合成酶功能域(β-ketoacyl synthase, KS),丙二酰酰基转移酶功能域(acyltransferase, AT),脱氢酶(dehydratase, DH)以及硫酯酶(thioesterase, TE)等多个保守蛋白结构域。采用Split-marker同源重组技术获得基因敲除突变体,通过比较突变体和野生菌株生长速度、产孢量、致病力等差异,证明突变体仅影响次生代谢产物比卡菌素的生物合成,不影响病原菌的其他生理表型及致病力;其次,突变体摇培后菌液呈白色,与野生型Foc4的暗红色形成鲜明对比,说明基因符合作为内源报告基因的条件。本研究以为内源靶标基因,利用水稻恶苗病菌()的基因编辑载体pUC-fFuCas9-HTB-hph,尝试以质粒体内表达Cas9和sgRNA的方式探索Foc4中CRISPR/Cas9编辑的可行性。gRNA序列由在线网站设计,构建的sgRNA162导入质粒构建靶向的pUC-fFuCas9-HTB-hph-Foc4Bik1基因编辑载体,与供体质粒pUC19-Foc4Bik1-HDR一起导入原生质体通过同源重组修复(homology directed repair, HDR)的方式进行基因编辑。经过潮霉素抗性筛选获得的(HDR)基因编辑后的敲除转化子进行PCR检测和摇培,白色菌液表型与PCR检测阳性的敲除转化子相互对应,证实了Foc4基因编辑的可行性。此外,可作为香蕉枯萎菌Foc4内源报告基因并评估探索新的分子生物学技术在香蕉枯萎菌上应用的可能性。
关键词:香蕉枯萎菌4号生理小种(Foc4);比卡菌素聚酮合酶基因;基因敲除;表型分析;CRISPR/Cas9基因编辑中图分类号:S668.4 文献标识码:A
Identification and Feasibility Analysis the Endogenous Reporter Gene of gene () in f. sp. race 4
WANG Yanwei, ZENG Fanyun, QI Yanxiang, DING ZhaoJian, XIE Yixian, ZHANG Xin, PENG Jun
1. Environment and Plant Protection Institute, Chinese Academy of Tropical Agricultural Sciences / Key Laboratory of Integrated Pest Management on Tropical Crops, Ministry of Agriculture and Rural Affairs / Hainan Key Laboratory for Monitoring and Control of Tropical Agricultural Pests / Haikou, Hainan 571101, China; 2. Department of Biological Science, Qiongtai Normal University, Haikou, Hainan 571127, China
f. sp. (Foc) causes wilt (Panama disease), one of the most devastating diseases of banana ( spp.). Foc race 4 (Foc4) is currently known as a major concern in global banana production. Previous studies reported that gene encoded a polyketide synthase responsible for the biosynthesis of a red pigment bikaverin in , and the mutant totally lost the ability to produce pigmented mycelia. In this study, the orthologous gene (FOIG_14908) in Foc4 was cloned and identified. The objective of this study was to identify the gene (FOIG_14908) in Foc4 which had potential as an endogenous reporter gene of Foc4, which could serve as a promising target gene for assessing the feasibility of newly developed CRISPR/Cas9 gene editing technique in Foc4. Foc4Bik1 is a typical type I fungal multifunctional polyketide synthase (PKS) composed of 2036 amino acids that contains conserved protein domain. SMART revealed that Foc4Bik1 contained conserved protein structure such as acyl-carrier protein (ACP) transacylase (SAT), β-ketoacyl synthase (KS), acyltransferase (AT), dehydratase (DH) and thioesterase (TE). The gene-knockout mutants were obtained by the split-marker homologous recombination technique. After two rounds of PCR amplification, the upstream and downstream recombination fragments required for protoplast transformation were successfully amplified by the split-marker approach. The knockout mutant was obtained via PEG-mediated protoplast transformation, the positive transformants were verified with the PCR method. The gene-knockout mutants showed no significant effects on fungal growth, conidial production, and pathogenicity to banana plantlet (Cavendish, AAA), albeit the gene-knockout mutants totally lost the ability to produce reddish pigmented mycelia, and thus the mycelia turned white when cultured in liquid PDB media. Therefore, could be selected as an endogenous reporter gene to verify whether the feasibility of CRISPR/Cas9 gene editing technology available for the filamentous fungus Foc4 via the plasmid CRISPR/Cas9 system. TheCRISPR/Cas9 vector pUC-fFuCas9-HTB-hph (Addgene, #121092) was used in this study. gRNA sequence was designed via the online software, and the sgRNA162 sequence was introduced into gene editing plasmid resulted the Foc4 CRISPR/Cas9 gene editing vector, pUC-fFuCas9-HTB-hph-Foc4Bik1, associated with donor plasmid pUC19-Foc4Bik1-HDR served as homology directed repair (HDR) template co-transformation into Foc4 protoplasts. Specifically, mutants totally lost the ability to produce pigmented mycelia appeared white mycelia. Using the white mycelia as the judging phenotype, the candidate (HDR) gene replacement mutants with white mycelia when grown in PDB liquid medium were selected for subsequent PCR detection. However, the white phenotypes were coupled with the positive PCR amplification bands. It is important that the positive (HDR) gene replacement mutants could be picked up according to the white phenotype by eyes without PCR procedures. The results proved that CRISPR/Cas9 system is stable and can efficiently disrupt the genes of interest, and Foc4Bik1 gene can serve as the endogenous reporter gene for further Foc4 molecular biology research.
f. sp.; - gene (); gene knockout; phenotype analysis; CRISPR/Cas9 gene editing
10.3969/j.issn.1000-2561.2022.03.005
镰刀菌属包括禾谷镰刀菌()、串珠镰刀菌()、尖孢镰刀菌()、拟枝孢镰刀菌()等主要的植物病原真菌,能够产生多种次生代谢产物。其中,聚酮类化合物的种类最为丰富,它以乙酰辅酶A、丙二酰辅酶A或其衍生物为底物,经聚酮合酶(PKS)催化而成。比卡菌素(bikaverin)是一种红色的四环聚酮化合物,具有抗菌、抗肿瘤的活性,拥有巨大的应用潜力。比卡菌素的生物合成的基因簇以及生物合成途径已经解析清楚,基因编码催化抗生性聚酮化合物合成的关键酶。在植物模式病原真菌水稻恶苗病菌()中证明了I型聚酮合酶pks4参与了红色次生代谢产物比卡菌素的生物合成,(现更名为)突变体不能产生比卡菌素,bikaverin生物合成基因簇中包含有6个基因都参与比卡菌素的合成,依次命名为。其中全长6270 nt,含有3个内含子,编码2009个氨基酸。编码I型聚酮合酶,将1个乙酰辅酶A和8个丙二酰辅酶A聚合形成比卡菌素前体物,负责红色色素比卡菌素的生物合成。编码FAD依赖性单加氧酶(Bik2),编码o-甲基转移酶(Bik3),编码A样家族结构域蛋白,编码一个真菌特异性的Zn(II)2Cys6转录因子,编码一个MFS转运蛋白。基因簇中的每一个基因的缺失都会导致比卡菌素合成量的全部丢失或显著减少。敲除突变体中无法合成比卡菌素;和影响比卡菌素的合成量,敲除后僅产生微量的比卡菌素。具有成为内源报告基因的特征和潜力。是目前丝状真菌中鉴定到的最长的编码基因,此外,参与催化合成的红色色素比卡菌素的生物合成,敲除突变体不能合成比卡菌素从而丧失了红色色素合成能力,摇培后的菌液呈白色。在构建棉花枯萎菌( f. sp. , Fov)的高效CRISPR/Cas9基因编辑体系中,FovBik1作为内源报告基因评估编辑的效率,原核表达Cas9蛋白后,与体外转录的sgRNA体外组装成核糖体核蛋白复合体RNP,连同供体DNA一并原生质体转化敲除靶标基因,敲除成功的突变体摇培后的菌液呈白色,与野生型的红色菌液对比明显,直接通过颜色差异即可判断基因编辑是否成功。
香蕉枯萎病是破坏香蕉维管束导致植株死亡的毁灭性土传病害,其病原菌为尖孢镰刀菌古巴转化型( f. sp., Foc),其中4号生理小种(Foc4)是致病力最强的小种,其致病机制和防控措施是香蕉产业的重要问题,也是热带地区植物病理学研究的热点和难题之一。利用反向遗传学基因功能日趋成熟,通过基因敲除(gene knockout)、RNAi干扰(RNA gene silencing, RNAi)等技术创造突变体并研究突变体的表型从而阐述基因的生物学功能。对Foc4而言,这些反向遗传学操作都需要一系列的操作,成功率受到很多因素制约。基因敲除可选择的筛选标记有限,而有些特殊的致死基因敲除后无法获得敲除突变体。利用RNAi基因沉默不能完全消除背景基因的影响,并且RNAi发夹中同源序列的dsRNA可能干扰其他非靶标基因出现脱靶现象。
成簇的规律间隔的短回文重复序列(clustered regulatory interspaced short palindromic repeats/ CRISPR-associated protein 9, CRISPR/Cas9)是近年开展的一种方便灵活的基因组编辑技术,可直接在基因组上进行DNA序列的插入、敲除、定点突变以及组合编辑等,尤其是在水稻恶苗病菌()以及尖孢镰刀菌棉花专化型(f. sp. , Fov)上的成功编辑极大地促进了镰刀菌分子生物学和遗传学的研究。但是Foc4目前尚无CRISPR/Cas9基因编辑的相关报道,因此,建立可靠的遗传转化技术并高效地删除目的基因是进行基因功能分析的重要前提。
尖孢镰刀菌()中也有比卡菌素的生物合成途径。通过生物信息学功能域分析软件分析,Foc4菌株含有I型PKS聚酮合酶同源基因(FOIG_14908),开展基因敲除及表型分析,观察基因敲除缺失突变体是否出现菌液颜色变化并评估作为内源报告基因的可行性。其次,香蕉枯萎病目前尚无CRISPR/Cas9基因编辑技术的报道,借鉴水稻恶苗病菌()的基因编辑载体pUC-fFuCas9-HTB-hph(Addgene, #121092),尝试构建了以为靶标基因的基因编辑体系,评估Foc4中构建基因编辑的可行性,为后续的香蕉枯萎菌的分子生物学研究提供有力的技术支撑。
材料与方法
材料
1.1.1 试验菌株及香蕉品种 香蕉枯萎病菌4号生理小种( f. sp. race 4, Foc4)由本实验室鉴定保存。试验接种的香蕉品种为‘巴西蕉’(Cavendish cv. Brazil, AAA)。
1.1.2 试剂 2× Master Mix DNA聚合酶(#P112)、DNase I(#EN401)、Realtime PCR试剂盒ChamQ Universal SYBR qPCR Master Mix(#Q711)、Phata Max Super-fidelity DNA高保真酶(#P505)、ClonExpress II One Step Cloning Kit同源重组试剂盒(#C112)等购自南京诺唯赞(Vazyme)生物科技股份有限公司;Trizol RNA提取试剂(Invitrogen, Carlsbad, CA, USA)、酵母轉化试剂盒Alkali-Cation Yeast Transformation Kit(#112200200)以及其他分析纯及试剂购自生工生物工程(上海)股份有限公司;引物由北京华大基因合成(PAGE纯化)。
方法
1.2.1 真菌DNA的制备 采用CTAB方法提取真菌的基因组DNA。将实验室保存的Foc4野生型菌株接种到PDA平板上,28℃培养3~5 d后,刮取新鲜的菌丝,按照CTAB方法提取菌丝DNA。其他突变体的DNA提取按照Foc4的操作方法和步骤进行。
1.2.2 Bik1蛋白结构域分析及基因敲除片段扩增利用在线蛋白功能域预测分析软件SMART(simple modular architecture research tool, http:// smart.embl.de)分析基因的保守功能域。参考的基因序列(FOIG_14908)和已发布的Foc4野生株基因组序列(JH658279.1),设计引物LBCK和LB-R,RB-F和RBCK(表1)扩增基因的上下游片段;设计HYG-F和HYG-R引物(表1)扩增验证潮霉素片段;重组上下游片段则分别通过引物LB-F、HYG-R1和HYG-F1、LB-R(表1)来扩增。Outside引物检测靶标基因是否被重组片段替换(敲除),而Inside引物检测内源基因。
1.2.3 原生质体制备及转化 香蕉枯萎病菌原生质体制备参照王飞燕等的方法进行。PEG介导的原生质体转化参照徐齐君等的方法。以培养3 d的Foc4野生株为原始菌株制备浓度为1×10个/mL的原生质体,分别取3~5 µg上下游片段回收产物与1 mL原生质体混匀,冰上放置30 min;取2 mL 60% PTC分3次缓慢加入到混合液,冰上放置15~20 min;将预冷的STC溶液加入到混合液至25 mL,4℃,5000 r/min离心15 min弃上清液;加入无抗性的SR液体培养基3 mL,再加入含有100 µg/mL潮霉素和50 µg/mL链霉素的SR固体培养基混匀后均匀倒在培养皿上,28℃培养箱中培养3~5 d后得到转化子,并连续培养3代得到稳定转化子。
1.2.4 敲除突变体PCR鉴定 敲除重组DNA片段需要包含靶标基因5上游约1 kb的同源序列、抗性基因序列、3下游约1 kb的同源序列。Split-marker重组方法参考WANG等的方法构建Split-marker基因敲除重组片段,包含2个重组DNA片段,具体分2步进行。第1轮PCR扩增3个片段:HYG-F/HYG-R扩增HYG抗性基因1.4 kb;Foc4基因组DNA为模板,引物Bik1-HYG-LBCK/Bik1-HYG-LB-R扩增左端LB1 1830 bp;Foc4基因组DNA为模板,引物Bik1- HYG-RB-F/Bik1-HYG-RBCK扩增右端RB 2133 bp。第2轮PCR扩增2个重组DNA片段:左端(5上游)重组DNA片段扩增(LB2+HY):Bik1-HYG- LB-F/HYG-R1为引物,Bik1-LB1+HYG-1.4kb为模板,扩增产物大小2713 bp;右端(3下游)重组DNA片段扩增(YG+RB2):HYG-F1/Bik1- HYG-RB-R为引物,HYG-1.4kb+Bik1-RB1为模板,扩增产物2386 bp。将5上游重组DNA和3下游重组DNA等体积混合后按照上述的原生质体转化方法进行转化。采用CTAB方法提取真菌的基因组DNA。配制20 µL PCR反应体系,反应程序:94℃,5 min;95℃,40 s;58℃,45 s;72℃,2 min;72℃,5 min;16℃,5 min,35个循环扩增验证敲除体中的目标片段,其中引物Outside-F/Outside-R检测靶标基因是否敲除;而引物Inside-F/Inside-R对敲除体内源基因进行检测(表1)。
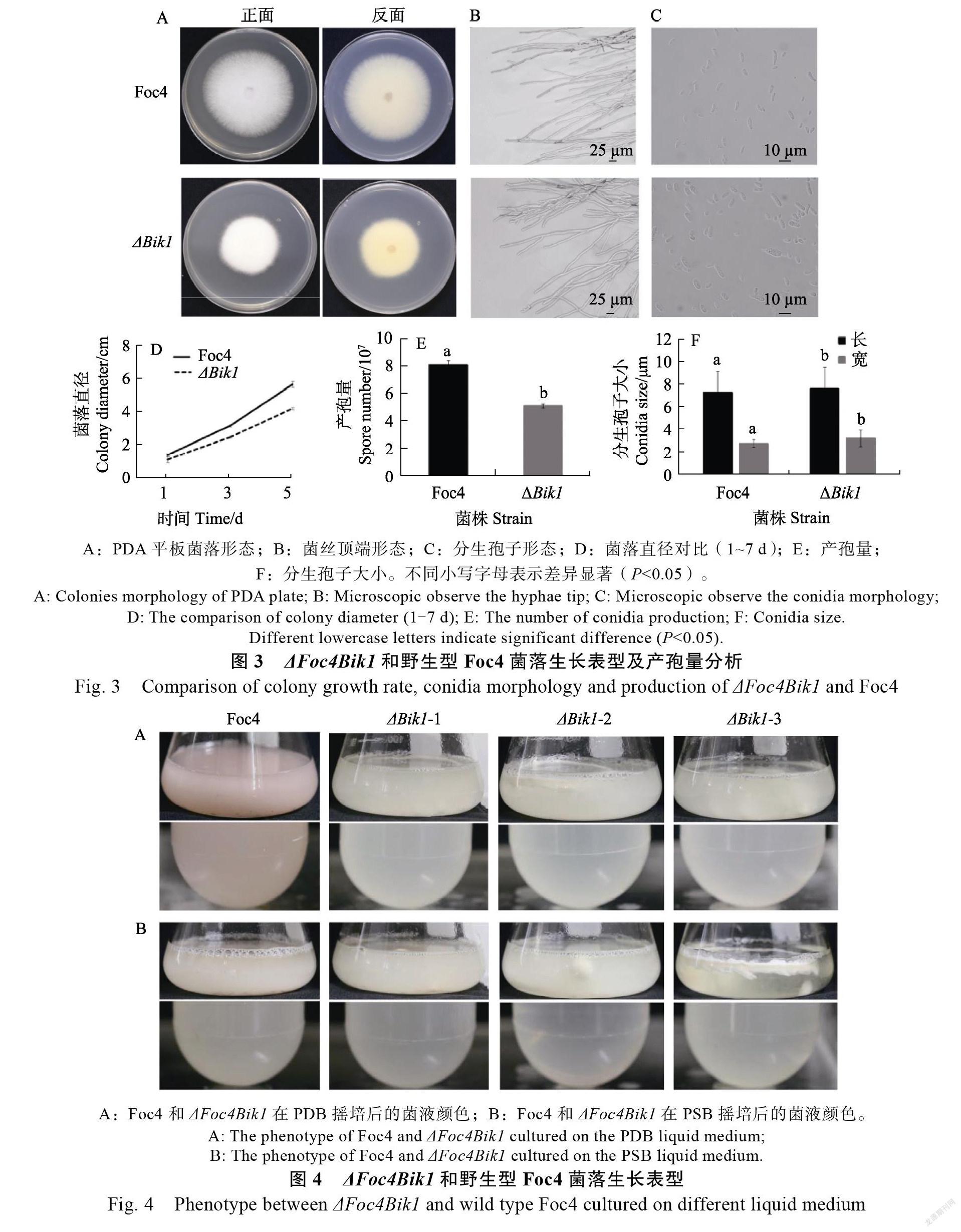
1.2.5 突变体生长特性分析 (1)生长形态和生长速率测定。挑选3个培养3~5 d的突变体和Foc4野生株,分别取5 mm菌饼接种到无抗性的PDA平板上,28℃培养,并分别在培养1、3、5、7 d测量菌落直径并拍表型图,每次设置3个重复。第7天观察结束后从PDA平板上取少量菌丝,制成载玻片进行显微观察形态。(2)孢子形态和产孢量分析。取突变体和Foc4野生株的5 mm新鲜菌饼分别加入到无抗性PDB液体培养基中,28℃摇培7 d,观察菌液颜色;同时,三层滤纸过滤后借助血球计数板在显微镜观察并统计其孢子数量。
1.2.6 突变体致病力分析 取突变体和Foc4野生株的5 mm新鲜菌饼分别接种到100 mL PDB液体培养基中,28℃摇床、150 r/min摇培5 d,用三层滤纸将菌丝过滤,收集分生孢子液,用无菌水将分生孢子液浓度调节为2×10个/mL。采用盆栽伤根灌淋法测定菌株致病性。其中突变体和野生株分别处理15株香蕉苗,每株苗浇灌15 mL孢子液,以无菌水作为空白对照,3次重复,60 d后纵向切开香蕉苗球茎,观察球茎褐变程度,参考MOHAMED等的病情调查分级标准,记录每株苗的发病等级并对病情指数进行统计。
1.2.7 CRISPR/Cas9基因编辑技术体系 借鉴水稻恶苗病菌()的基因编辑载体pUC-fFuCas9-HTB-hph(Addgene, #121092),探索采用5SrRNA启动子和HTB核定位信号的质粒编辑载体,尝试构建以为靶标基因的基因编辑技术。
sgRNA序列的设计以及编辑载体构建。人工合成263 nt的Ff5SrRNA-Bik1 sgRNA全基因序列,并以此为模板扩增后续的sgRNA序列后同源重组进入基因编辑质粒。guide RNA由Eukaryotic Pathogen CRISPR guide RNA/DNA Design Tool网站设计,综合分析后确定gRNA的序列,Foc4Bik1-gRNA162:AGAGAGAAAATCGATTCC CGagg。以人工合成的序列为模板,分别用引物对5SrRNA-U-F-EcoRI/BIK1-sgRNA162-R、BIK1- sgRNA162-F/5SrRNA-U-R-EcoRⅠ扩增5SrRNA- Bik1 sgRNA的上、下游两段序列;pUC-f FuCas9- HTB-hph载体RⅠ酶切后,利用ClonExpress II One Step Cloning Kit重组克隆试剂盒,将5SrRNA-Bik1 sgRNA的两端扩增序列同源重组导入质粒构建成靶向Foc4Bik1的pUC-fFuCas9- HTB-hph-Foc4Bik1基因编辑载体,该载体采用体内表达的Cas9和sgRNA。
同源重组修复供体质粒pUC19-Foc4Bik1- HDR的构建。pUC19-BIK1-LB-F/pUC19-BIK1- LB-R、pUC19-BIK1-RB-F/pUC19-BIK1-RB-R分别扩增基因的上游1419 bp同源序列、下游1315 bp同源序列,同源重组导入pUC19构成供体质粒pUC19-Foc4Bik1-HDR作为同源重组修复(homology directed repair, HDR)的HDR模板。
基因编辑操作方法。分别取5 µg构建的pUC- fFuCas9-HTB-hph-Bik1、pUC1-Bik1-HDR质粒原生质体共转化后筛选阳性转化子,筛选方法与基因敲除阳性转化子筛选方法一致。
结果与分析
进化树分析
基因(FOIG_14908)CDS全长6111 bp,编码一个由2036个氨基酸组成的氨基酸多功能酶,包含多个保守结构功能域:初始单位酰基转移酶功能域[starter unit: acyl-carrier protein (ACP) transacylase, SAT],β-酮脂酰合成酶功能域(β-ketoacyl synthase, KS),丙二酰酰基轉移酶功能域(acyltransferase, AT),脱氢酶(dehydratase, DH)以及硫酯酶(thioesterase, TE)(图1A)。
经BLAST分析、Clustal X比对,Foc4Bik1与水稻恶苗病菌()的BIK1蛋白(AJ278141)、尖孢镰刀菌棉花专化型Fov的BIK1(FOTG_08225)蛋白序列高度同源,具有高度保守的功能域结构,同源性达到99%以上(图1B)。
2.2 基因敲除及检测
采用Split-marker重组技术原理,进行二轮PCR扩增重组DNA片段,左端(5上游)重组DNA片段(LB2+HY)扩增(LB2+HY)2713 bp;右端(3下游)重组DNA片段(YG+RB2)扩增2386 bp(图2A)。将5上游重组DNA和3下游重组DNA等体积混合后按照上述原生质体转化方法进行转化。经过潮霉素抗性筛选和再生培养,获得敲除转化子。分别提取Foc4野生型菌株基因组DNA和敲除转化子基因组DNA,并以此为模版,分别用Outside、Inside和HYG引物进行PCR扩增,鉴定阳性转化子。结果显示,Outside引物对从Foc4中扩增到6832 bp,而敲除突变体中扩增到1800 bp;Inside引物对仅从Foc4中扩增到746 bp,而敲除突变体中无扩增条带;HYG检测显示仅敲除突变体中扩增到1376 bp条带,Foc4中无扩增条带,这些结果表明,这3个转化子中的基因已成功敲除(图2B)。
2.3 生长特性和产孢量分析
将Foc4和分别接种于PDA培养基上,每天测量菌落直径大小,生长速度略慢于野生株Foc4,其菌落直径为4.18 cm,野生株Foc4菌落直径5.65 cm,但是二者差异不显著(图3A,图3D)。显微观察结果显示,与野生株对比,菌丝生长和菌丝顶端形态无明显差异,菌丝生长正常(图3A,图3B)。产孢量统计显示,产孢量为(5.1± 0.16)×10个/mL,野生株Foc4产孢量为(8.1± 0.3)×10个/mL,产孢量明显降低但是差异不明显(图3C,图3E)。分生孢子形态和大小发生改变,野生菌株Foc4小型分生孢子卵圆形或肾型,无色,单胞或双胞,而分生孢子长度不变,稍微变宽,卵圆形或近球形(图3C)。分生孢子平均大小为(7.88± 1.96)μm × (3.32 ± 0.78)μm,Foc4分生孢子平均大小为(7.56±1.93)μm × (2.84±0.36)μm(图3F)。
2.4 摇培表型分析
取突变体和Foc4野生株的5 mm新鲜菌饼分别加入到无抗性PDB、PSB液体培养基中,28℃摇床摇培7 d,观察菌液颜色;同时,观察三层滤纸过滤后的滤液分生孢子颜色。结果显示,在PDB(pH 4.0)液体培养基中,Foc4野生株摇培3 d后的菌液颜色为红色,而突变体摇培3 d后的菌液颜色为白色,差异明显(图4A)。结果说明,在Foc4中基因负责红色次生代谢产物比卡菌素的生物合成,与水稻恶苗病菌()的研究结果一致。
RODRÍGUEZ-ORTIZ等的研究表明,在高氮培养基(ICI medium)中加入蔗糖能显著提高水稻恶苗病菌()中比卡菌素的生物产量。因此,将PDB培养基中的葡萄糖用蔗糖替代的PSB培养基摇培,观察蔗糖是否能够显著提高Foc4中比卡菌素的生物产量。PSB(pH 4.0)液体培养基中,突变体摇培颜色偏淡粉色,与Foc4野生株摇培3 d后的颜色差异不明显(图4B)。研究结果证实PDB培养基中的红色表型比PSB的更加明显,推测产生差异的原因可能是由于菌株差异以及培养基组分差异。
2.5 致病力分析
将Foc4野生型菌株和突变体菌株的菌丝分别接种‘巴西蕉’幼苗。30 d后观察香蕉苗外部症状和内部症状。以HO作为空白对照的‘巴西蕉’苗叶片嫩绿,叶片未出现症状,球茎无褐化现象。接种野生株Foc4的‘巴西蕉’苗下部叶片出现黄化枯萎现象,植株下部叶片褪绿变黄,并且纵向切开球茎后球茎褐化明显,出现典型的坏死斑;而接种突变株的‘巴西蕉’苗均与接种野生株的症状相似,叶片黄化现象,球茎褐化明显(图5A)。病情指数统计显示,接种突变株平均病情指数(54.17)与Foc4接种的平均病情指数(57.08)并无显著差异(图5B),结果证实基因敲除后对致病力并无明显影响,基因并不参与调控病原菌的致病力。
Foc4Bik1為靶标评估基因编辑可行性
采用Pol III型启动子5S rRNA启动sgRNA体内表达,选用HTB作为核定位信号,以水稻恶苗病菌()的基因编辑载体pUC- fFuCas9-HTB-hph(Addgene, #121092)构建以为靶标基因的CRISPR/Cas9基因编辑体系,编辑载体见图6A。在线软件Eukaryotic Pathogen CRISPR guide RNA/DNA Design Tool(http://grna.ctegd.uga.edu/)设计gRNA序列- gRNA162: AGAGAGAAAATCGATTCCCG agg,gRNA在基因上的作用位点以及供体质粒pUC19-Foc4Bik1-HDR见图6B,构建好的编辑载体pUC-fFuCas9-HTB-hph-Foc4Bik1与供体质粒pUC19-Foc4Bik1-HDR共转化原生质体。
CRISPR/Cas9基因编辑载体pUC-fFuCas9- HTB-hph-Foc4Bik1与供体质粒pUC19-Foc4Bik1- HDR共转化原生质体后,获得基因编辑后的(HDR)敲除转化子,在PDB(pH 4.0)液体培养基摇培3 d后,基因编辑转化子菌液颜色为白色,差异明显(图6C)。分别提取Foc4野生型菌株基因组DNA和(HDR)基因编辑敲除转化子基因组DNA,分别进行PCR检测以鉴定阳性基因编辑敲除转化子。结果显示,
Outside引物对从Foc4中扩增到6832 bp,而(HDR)基因编辑敲除转化子中扩增到450 bp;Inside引物对仅从Foc4中扩增到746 bp,而(HDR)基因編辑敲除转化子中无扩增条带;此外,以及基因也仅从(HDR)基因编辑敲除转化子中扩增获得。这些结果表明,基因编辑后成功敲除了转化子(HDR)中的内源报告基因(图6D)。
讨论
CRISPR/Cas9基因组编辑技术借助特异性DNA双链断裂(DNA double-strand breaks, DSBs)激活细胞天然的修复机制,包括同源重组修复(homology directed repair, HDR)和非同源末端连接(non-homologous DNA end joining, NHEJ)2条途径。同源重组是将外源基因靶向导入受体细胞染色体上的方法,借助在该位点有与导入基因同源的序列,通过单一或双交换,新基因片段可替换原基因片段。这是一种具有相对高保真度的修复过程。CRISPR/Cas9基因组编辑技术在代谢工程以及分子遗传育种等方面具有广阔的应用前景。镰刀菌中水稻恶苗病菌()以及尖孢镰刀菌棉花专化型( f. sp. )都有基因编辑的相关报道,目前,Foc4尚无基因编辑技术的报道,因此,鉴定并筛选表型容易分辨的内源报告基因评估基因编辑技术在枯萎菌上应用的可行性具有重要意义。
报告基因(reporter gene)是一种编码可被检测的蛋白质或酶的基因,非常容易鉴定其表达产物。报告基因的种类很多,根据报告基因的来源及分析方法的不同,可将其分为外源报告基因和内源报告基因。植物中最常用的外源报告基因如β-葡糖醛酸酶(GUS)报告基因,绿色荧光蛋白(GFP)基因、萤火虫荧光素酶(luciferase)基因等;内源报告基因是自身编码的基因,植物中应用最广泛的是八氢番茄红素脱氢酶(phytoene desaturase, PDS),基因沉默会出现典型的白化表型。目前,香蕉枯萎菌应用最广泛的是GFP外源报告基因。荧光蛋白GFP标记枯萎菌后有利于观察枯萎菌侵染、定殖过程、评估不同生理小种之间的毒力差异以及不同香蕉品种中的抗性差异,目前尚无香蕉枯萎病菌内源报告基因的相关报道。
本文鉴定的基因可以作为内源报告基因探索和评估基因编辑在Foc4上的可行性。基因符合作为内源报告基因的几大特征条件:(1)表型容易分辨。突变株摇培后的菌液颜色呈白色,与野生株的红色菌液颜色差异明显。(2)突变体不影响病原菌的其他生理表型和致病力,仅影响代谢产物中比卡菌素的生物合成。(3)其表达产物能够定量测定。负责比卡菌素(bikaverin)的生物合成,含量可以量化测定。(4)表达可调控。的表达可以受培养基和外界因素的调控表达,比卡菌素的合成主要受4个方面的影响:AreA的正调节、PacC的抑制、时间的调节和内部基因的相互调节。基因簇中任何一个相关基因突变,其他基因的表达均有明显的下降。此外,比卡菌素产量还受培养条件的调控。研究表明,培养基中的N源缺乏以及高C/N比有利于比卡菌素的生物合成。蔗糖显著提高比卡菌素的生物产量。培养基中添加钙提高其产量,而添加磷酸盐和硫酸盐负调控产量,培养过程中的通气量以及水分活度(water activity, a)也影响比卡菌素生物产量。
丝状真菌的基因编辑有2种主要的操作方式,一种方式是基于质粒的体内表达系统,质粒上携带Cas9和sgRNA表达系统。sgRNA由Pol II型启动子,比如U6等启动表达;而Cas9由真菌组成型启动子表达。另外一种是体外表达Cas9,与体外转录的sgRNA结合形成RNP核糖核蛋白复合体后转化。Cas9单独转化到Foc4后并不影响其致病力(结果未显示),与稻瘟菌的结果不一致,为后续采用质粒型编辑载体提供了前期保证。此外,同源重组修复是将外源基因靶向导入受体细胞染色体上的方法,本文构建的CRISPR/ Cas9基因组编辑技术依赖同源重组修复完成靶标基因敲除,需要外源供体DNA模板导入,借助在该位点有与导入基因同源的序列,通过单一或双交换,供体DNA序列可替换原基因片段。这是一种具有相对高保真度的修复过程。因此,本文采用质粒体内表达的方式探索Foc4基因编辑的可行性。以为靶标基因,在线网站设计gRNA构建pUC-fFuCas9-HTB-hph-Foc4Bik1后,与供体质粒pUC19-Bik1-HDR一起导入到原生质体,潮霉素筛选的转化子经过表型分析和PCR鉴定。阳性转化子PDB摇培后的菌液呈白色,与野生型Foc4差异明显;DNA测序结果也证实通过基因编辑的方式敲除了基因,结果说明了Foc4中基因编辑的可行性,可以通过颜色差异直接筛选阳性转化子。
綜上,基因编码聚酮合酶在代谢产物比卡菌素的生物形成中发挥重要作用,敲除突变体菌液颜色发生改变外,不影响病原菌的生长发育、产孢及致病力,是Foc4良好的内源报告基因。以为靶标基因,初步构建了Foc4的基因编辑技术,后续将继续优化Cas9序列,NLS核定位信号,完善香蕉枯萎病的基因编辑技术,为病原菌的功能基因组学提供有力的技术支撑。
参考文献
- Chelkowski J, Zajkowski P, Visconti A. Bikaverin production by species[J] . Mycotoxin Research, 1992, 8(2): 73-76.
- 张晓伟, 张 栋. 镰孢菌属真菌次生代谢产物的研究进展[J]. 植物生理学报, 2013, 49(3): 201-216.ZHANG X W, ZHANG D. Recent advances of secondary metabolites in genus [J]. Plant Physiology Journal, 2013, 49(3): 201-216. (in Chinese)
- Linnemannstöns P, Schulte J, del Mar Prado M, Proctor R H, Avalos J, Tudzynski B. The polyketide synthase gene 4 from encodes a key enzyme in the biosynthesis of the red pigment bikaverin[J]. Fungal Genetics and Biology, 2002, 37(2): 134-48.
- Proctor R H, Desjardins A E, Plattner R D, Hohn T M. A polyketide synthase gene required for biosynthesis of fumonisin mycotoxins in mating population A[J]. Fungal Genetics and Biology. 1999, 27(1): 100-112.
- Wang Q, Cobine P A, Coleman J J. Efficient genome editing in based on CRISPR/Cas9 ribonucleoprotein complexes[J]. Fungal Genetics and Biology, 2018, 117: 21-29.
- Lin B R, Shen H F. f. sp.[M]. Springer Singapore: Biological Invasions and Its Management in China, 2017: 225-236.
- Shi T Q, Gao J, Wang W J, Wang K F, Xu G Q, Huang H, Ji X J. CRISPR/Cas9-based genome-editing in the filamentous fungus and its application in strain engineering for gibberellic acid production[J]. ACS Synthetic Biology, 2019, 8(2): 445-454.
- Lebeau J, Petit T, Dufossé L, Caro Y. Putative metabolic pathway for the bioproduction of bikaverin and intermediates thereof in the wild LCP531 strain[J]. AMB Express, 2019, 9(1): 186.
- Lebeau J, Petit T, Clerc P, Dufossé L, Caro Y. Isolation of two novel purple naphthoquinone pigments concomitant with the bioactive red bikaverin and derivates thereof produced by [J]. Biotechnology Progress, 2019, 35(1): e2738.
- 王飞燕, 郭立佳, 杨腊英, 汪 军, 王国芬, 黄俊生. 尖孢镰刀菌古巴专化型4号生理小种基因敲除与表型分析[J]. 热带作物学报, 2015, 36(8): 1462-1468.WANG F Y, GUO L J, YANG L Y, WANG J, WANG G F, HUANG J S. Construction and phenotype analysis of the deletion mutantsof the fungal pathogenf. sp.[J]Chinese Journal of Tropical Crops, 2015, 36(8): 1462-1468. (in Chinese)
- 徐齐君, 胡小平, 陈 婷, 赵俊兴. PEG介导的棉花枯萎病菌原生质体转化体系的建立[J]. 棉花学报, 2012, 24(3): 222-228.XU Q J, HU X P, CHEN T, ZHAO J X. Protoplast transformation of f. sp. mediated by polyethylene Glyol[J]. Cotton Science, 2012, 24(3): 222-228. (in Chinese)
- Wang W Q, Tang W H. Generation of knockout mutants by the split-marker recombination approach[J] . Bio-101, 2018: e2976.
- Mohamed A A, Mak C, Liew K W, Ho YW. Early evaluation of banana plants at nursery stage for wilt tolerance[M] . In: Molina A B, Nik Masdek N H, Liew K W (eds). Seminar on banana wilt management towards sustainable cultivation, Pahang, Malaysia. International Network for the Improvement of Banana and Plantain, Montpellier, 1999: 174-185.
- Rodríguez-Ortiz R, Mehta B J, Avalos J, Limón M C. Stimulation of bikaverin production by sucrose and by salt starvation in [J]. Applied Microbiology Biotechnology, 2010, 85(6): 1991-2000.
- Geissman T A, Verbiscar A J, Phinney B O, CRAGG G. Studies on the biosynthesis of gibberellins from (-)-kaurenoic acid in cultures of [J]. Phytochemistry, 1966, 5: 933-947.
- Li C Y, Chen S, Zuo C W, Sun Q G, Ye Q, Yi G J, Huang B Z. The use of GFP-transformed isolates to study infection of banana with f. sp. race 4[J]. European Journal of Plant Pathology, 2011, 131(2): 327-340.
- Guo L J, Yang L Y, Liang C C, WANG G F, DAI Q D, HUANG J S. Differential colonization patterns of bananas ( spp.) by physiological race 1 and race 4 isolates of f. sp. [J]. Journal of Phytopathology, 2015, 163(10): 807-817.
- Li C, Yang J, Li W, Sun J, Peng M. Direct root penetration and rhizome vascular colonization by f. sp. are the key steps in the successful infection of Brazil Cavendish[J]. Plant disease, 2017, 101(12): 2073-2078.
- Studt L, Wiemann P, Kleigrewe K, Humpf H U, Tudzynski B. Biosynthesis of fusarubins accounts for pigmentation of perithecia[J]. Applied and Environmental Microbiology, 2012, 78(12): 4468- 4480.
- Giordano W, Avalos J, Cerdá-Olmedo E, DOMENECH C E. Nitrogen availability and production of bikaverin and gibberellins in [J]. FEMS Microbiology Letters, 1999, 173: 389-393.
- Lazzaro I, Busman M, Battilani P, BUTCHKO R A E. and gene expression contribute to describe fumonisin and bikaverin synthesis in [J]. International Journal of Food Microbiology, 2012, 160(2): 94-98.
