辣椒属叶绿体基因组特征及进化
2022-03-25刘潮韩利红代小波刘宸语
刘潮 韩利红 代小波 刘宸语


摘 要:辣椒( L.)屬于重要的蔬菜和香料作物,在世界范围广泛栽培。种质资源是育种和生产的基础,开展辣椒属叶绿体基因组特征研究对阐明辣椒种质资源遗传多样性、培育和改良栽培品种具有重要的理论和实践价值。本研究从NCBI数据库下载13个辣椒属物种叶绿体基因组序列,利用REPuter、MISA和mVISTA等软件,对辣椒属叶绿体基因组特征、序列重复、结构变异和系统发育进行分析。结果显示:辣椒属叶绿体基因组均具有保守的四分体结构,一对反向重复区(IRs)将大单拷贝区(LSC)和小单拷贝区(SSC)分开,基因组全长介于156 583~158 077 bp之间,包含113个unique基因,其中蛋白编码基因、tRNA基因和rRNA基因数分别为79、30和4;含有60~127对长重复序列,其中正向重复序列和回文重复序列比例较高,30~39 bp的重复序列含量较高;发现150~164个SSR位点,以单核苷酸和二核苷酸重复为主,多为A或T碱基重复;基因组序列同源性较高,未发现基因重排现象,非编码区序列变异高于基因编码区,LSC和SSC的多样性位点多于IR;发现-、、--和等4个核苷酸多态性热点,LSC和SSC的序列多样性高于IR;主要栽培种辣椒()与多花辣椒()亲缘关系较近,中华辣椒()与灌木状辣椒()和紫绿花辣椒()亲缘关系较近。研究中筛选的SSR和核苷酸多态性热点可作为辣椒属物种鉴定和遗传多样性分析的分子标记,研究明确了辣椒属物种的系统发育关系,为辣椒属物种遗传多样性和生物育种奠定基础。
关键词:辣椒属;叶绿体基因组;核苷酸多态性热点;系统发育中图分类号:S718.46 文献标识码:A
Characteristics and Phylogenetics of the Complete Chloroplast Genomes of Species
LIU Chao, HAN Lihong, DAI Xiaobo, LIU Chenyu
Yunnan Engineering Research Center of Fruit Wine, College of Biological Resource and Food Engineering, Qujing Normal University, Qujing, Yunnan 655011, China
Pepper ( spp.) is widely used as food, spice, decoration, and medicine. Multiple cultivated species belong to important vegetable and spice crops. The analysis of the chloroplast genome of has important theoretical and practical value for clarifying the genetic diversity of germplasm resources and cultivating and improving cultivated varieties. The availability of complete chloroplast genome sequences has offered enormous opportunity to researchers to study the details of gene number, characterization, phylogenetic relationships and diversification of species. The complete chloroplast genome sequences of 13 species were downloaded from the NCBI database, and analyzed by online softwares, such as REPuter, MISA, and mVISTA. The chloroplast genome of had a conserved quadripartite structure, a pair of inverted repeats (IRs) separated a large single copy (LSC) region and a small single copy (SSC) region. The size of chloroplast genome sequences ranged from 156 583 bp ()-158 077 bp (). A total of 113 unique genes were predicted, including 79 protein-coding genes, 30 tRNA genes, and four rRNA genes. About 60-127 pairs of repeats were identified in chloroplast genomes, and the proportion of forward repeats and palindromic repeats and 30-39 bp repeats were all high in the chloroplast genomes. About 150-164 potential SSRs were reported in chloroplast genomes, and SSRs mainly consisted of mononucleotide and dinucleotide repeats, and most of the repeats were composed of A or T base. Comparative genomics of the 13 species revealed a high sequence similarity in genome-wide organization and a lower sequence divergence in the IRs than LSC and SSC. The fragments of r-,,-- and were identified as variable regions in species, and the nucleotide variability of LSC and SSC was higher than that of IR. The phylogenetic analysis showed that the main cultivated species was closely related to , and was closely related to and . The SSR and variable hotspots identified could be used as molecular markers for species identification and genetic diversity analysis of species. Overall, our study would highlight the taxonomic utility of the chloroplast genomes in species, and lay a foundation for the evolution, genetic diversity, conservation and biological breeding of species.
; chloroplast genome; variable hotspots; phylogeny
10.3969/j.issn.1000-2561.2022.03.002
辣椒属( L.)属于茄科一年或有限多年生植物,由约30余个物种构成。辣椒起源于中美洲和安第斯山脉区域,目前在热带和温带地区广泛栽培。辣椒属物种包含多个野生驯化种,其果实外形和风味高度不同。辣椒属的5个栽培种为重要的蔬菜和香料作物,包括辣椒()、灯笼辣椒()、中华辣椒()、灌木状辣椒()和绒毛辣椒(),其果实富含多种生物活性物质,其中类胡萝卜素可作为天然色素和抗氧化剂,广泛应用于多种疾病的防控和治疗。
叶绿体是绿色植物进行光合作用的场所,具有独立的遗传系统,其遗传结构相对稳定,较少发生基因重组,因此叶绿体基因组序列被广泛用于植物系统发育和物种鉴定。被子植物的叶绿体基因组高度保守,由一个大单拷贝区(large single copy region,LSC)、一个小单拷贝区(small single copy region,SSC)和一對反向重复区(inverted repeat regions,IRs)构成。细胞核核糖体DNA内部转录间隔区(rDNA-ITS)作为DNA条形码,被广泛用于物种鉴定和遗传多样性评估。研究发现辣椒的rDNA-ITS发生了不完全协同进化,辣椒的遗传多样性是所有辣椒属物种中最低的,中华辣椒可能是灌木状辣椒的栽培变种。CAROLINA等使用叶绿体基因、和核基因对34个辣椒属物种的种间关系、地理起源和扩散进行系统研究。TANG等对辣椒的线粒体基因家族进行了鉴定和表征。ZHONG等利用SSR标记对147份辣椒属种质资源的遗传多样性进行研究。MAGDY等通过
泛质体基因组对辣椒属分类和鉴定进行研究,发现、、位点多态性可用于辣椒属物种系统进化分析。BARBOZA等在南美安第斯热带森林中发现了4个辣椒属新种,并利用、rpl32-trnL、、和对该属物种进行系统发育分析。D’AGOSTINO等测序了8个辣椒属叶绿体基因组,发现、、、和位于高变区,可用于辣椒属物种的鉴定。
种质资源是辣椒育种和生产的基础。对基因组数据库1618份分子材料的分析表明,分类学上的模糊性和错误通常给辣椒属物种的形态学分类带来困难。辣椒由多个商业栽培品种组成,经过长期的人工驯化和连续栽培,品种遗传背景变窄,遗传多样性降低,人类对辛辣等性状的偏好为驯化辣椒遗传变异产生了重要影响。因此,了解辣椒属种质资源间的遗传多样性,对于物种资源的收集、保护和利用至关重要。目前,辣椒属的系统发育关系已被广泛研究,多个辣椒属物种叶绿体基因组被公布,但由于分子数据的限制,辣椒属的种间关系仍存在较大争议。本研究基于叶绿体基因组序列,针对辣椒属物种资源亲缘关系和遗传多样性展开分析,将为我国现有辣椒栽培品种的改良选育提供理论依据。
材料与方法
序列获取
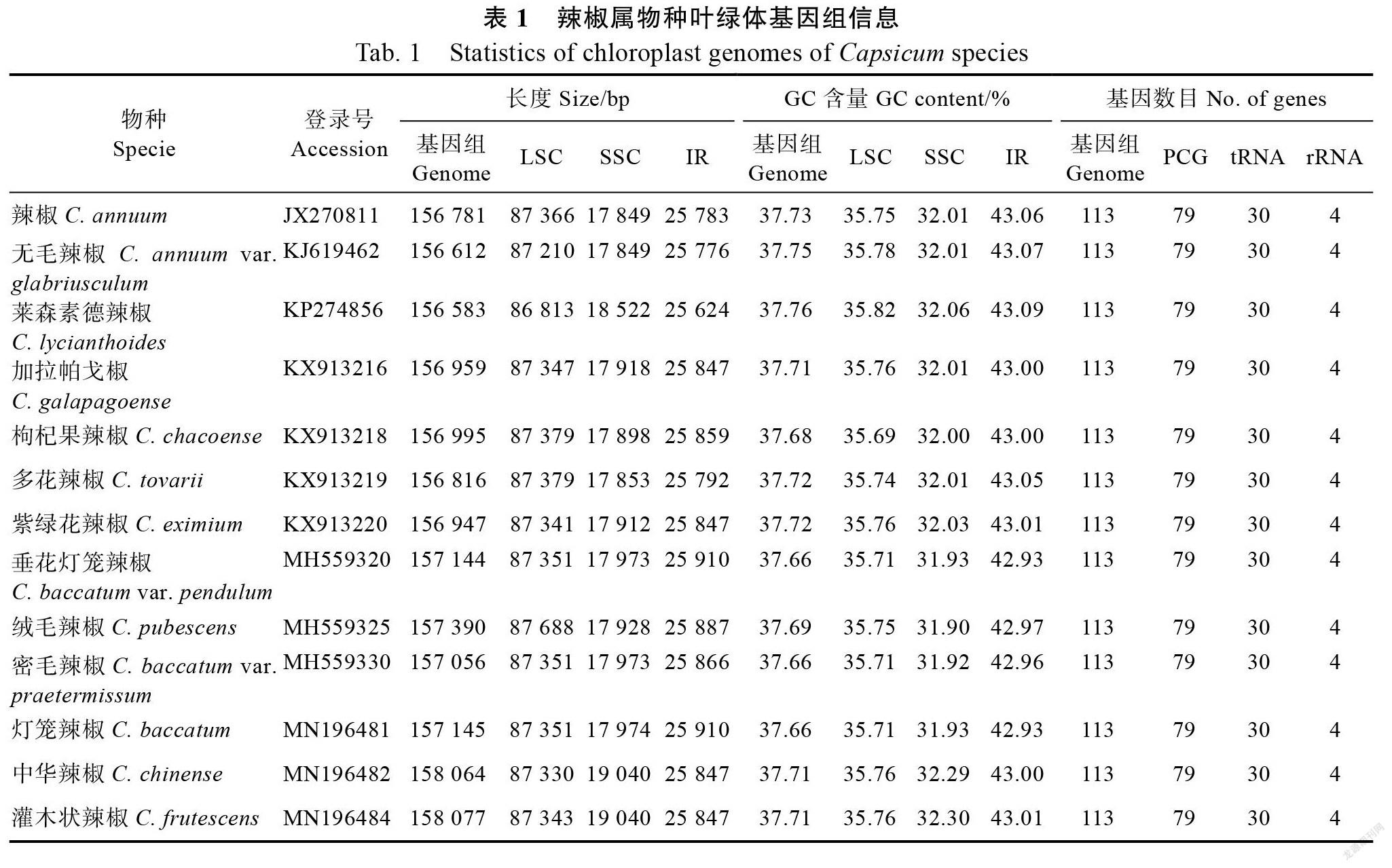
从NCBI数据库(https://www.ncbi.nlm.nih. gov/)下载13个辣椒属物种的叶绿体基因组信息,见表1。
方法
1.2.1 重复序列分析 使用REPuter软件分析叶绿体基因组序列重复,包括互补(complementary,C)、正向(forward,F)、反向(reverse,R)和回文(palindromic,P)重复,最小重复长度30 bp,序列同源性90%,Hamming距离为3。使用MISA在线工具检测SSR数量及位置分布,设置最小阈值为单核苷酸重复次数为8,二核苷酸和三核苷酸重复次数均为4,四核苷酸、五核苷酸和六核苷酸重复次数均为3。
1.2.2 序列多样性分析 使用mVISTA在线软件,以辣椒叶绿体基因组(JX270811)序列为参照,对辣椒属物种叶绿体基因组序列变异进行可视化比对。使用MAFFT v. 7.450软件对辣椒属物种叶绿体基因组序列进行多序列比对,使用DnaSP v. 6.10软件分析叶绿体基因组序列核苷酸多样性(),以确定辣椒属的高度可变区,设置搜索窗口长度为600 bp,步长为200 bp,使用R程序绘图。
1.2.3 系统发育分析 利用13个辣椒属物种的叶绿体基因组序列,以番茄(,AM087200)和马铃薯(,DQ386163)为外类群,使用MAFFT v. 7.450软件进行多序列比对,通过IQ-TREE v. 2.1.1软件基于
最大似然法(maximum likelihood,ML)构建系统进化树,建树模型为KPu+F+R,步长值为1000。
结果与分析
叶绿体基因组特征
所有辣椒属叶绿体基因组均具有完整的LSC、SSC、IRa和IRb四部分(表1)。叶绿体基因组全长介于156 583()~158 077 bp(),平均GC含量为37.70%。LSC长度介于86 813()~87 688 bp(),SSC长度介于17 849(var. )~19 040 bp(),IR长度介于25 624()~25 910 bp(),各部分平均GC含量依次為35.75%、32.03%和43.01%。所有辣椒属叶绿体基因组均包含113个unique基因,其中蛋白编码基因79个、tRNA基因30个、rRNA基因4个(表1)。蛋白编码基因、、、、、和均含有2个拷贝,、、、、、、、、和均含有1个内含子,和含有2个内含子。
叶绿体基因组重复序列
由图1可看出,辣椒属物种含有60~127对长重复序列,明显多于番茄和马铃薯的40对和33对。与茄科代表物种不同,辣椒属物种正向重复序列普遍占比均超过50%(除莱森素德辣椒外)(图1A)。与茄科代表物种相比,辣椒属长重复序列长度分布更加多样化,30~39 bp长度的序列最多,占42%~60%,其次为40~49 bp序列(图1B)。
辣椒属物种叶绿体基因组中含有150~164个SSR位点(图2)。主要以单核苷酸和二核苷酸重复为主,其中单核苷酸均大于100个,二核苷酸平均43个,四核苷酸重复略多于三核苷酸,仅有辣椒、多花辣椒和莱森素德辣椒含有六核苷酸重复(图2A)。辣椒属二核苷酸SSR明显多于番茄和马铃薯。单核苷酸主要为A/T重复,占单碱基重复的98%,C/G重复仅占2%;二核苷酸重复主要为AT/AT、AG/CT和AC/GT 3种类型,未检测到CG/GC重复,其中AT/AT重复占60%,AG/CT占38%;三、四、五和六核苷酸重复序列相对较少,且均含有较高的A或T碱基(图2B)。
2.3 叶绿体基因组结构变异
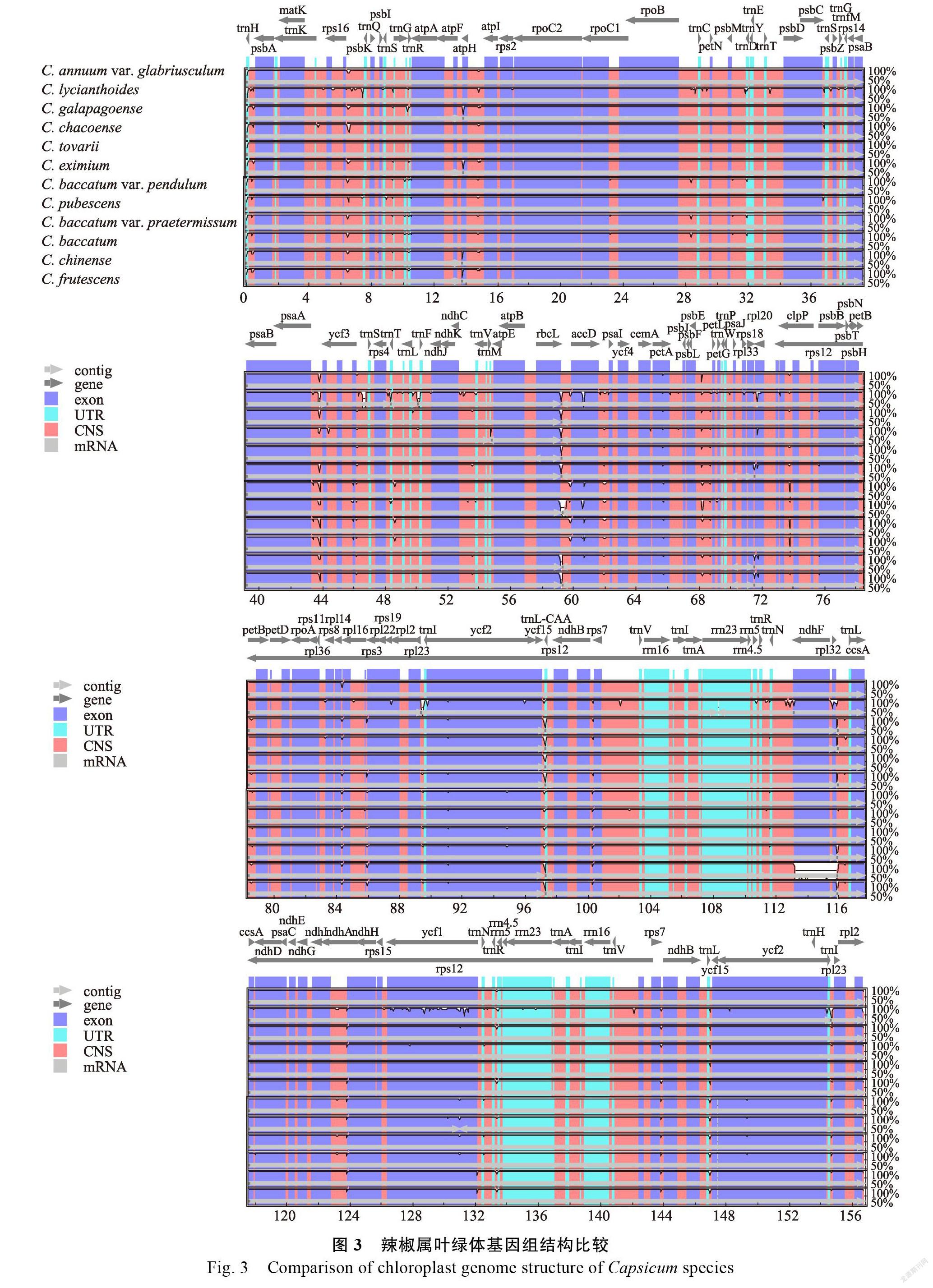
以辣椒叶绿体基因组JX270811为参考,利用mVISTA软件比较辣椒属叶绿体基因组序列的同源性(图3)。结果显示,辣椒属叶绿体基因组未发生基因重排现象,非编码区序列变异高于基因编码区,LSC和SSC的多样性位点多于IR。与其他物种相比,莱森素德辣椒具有较高的序列变异。
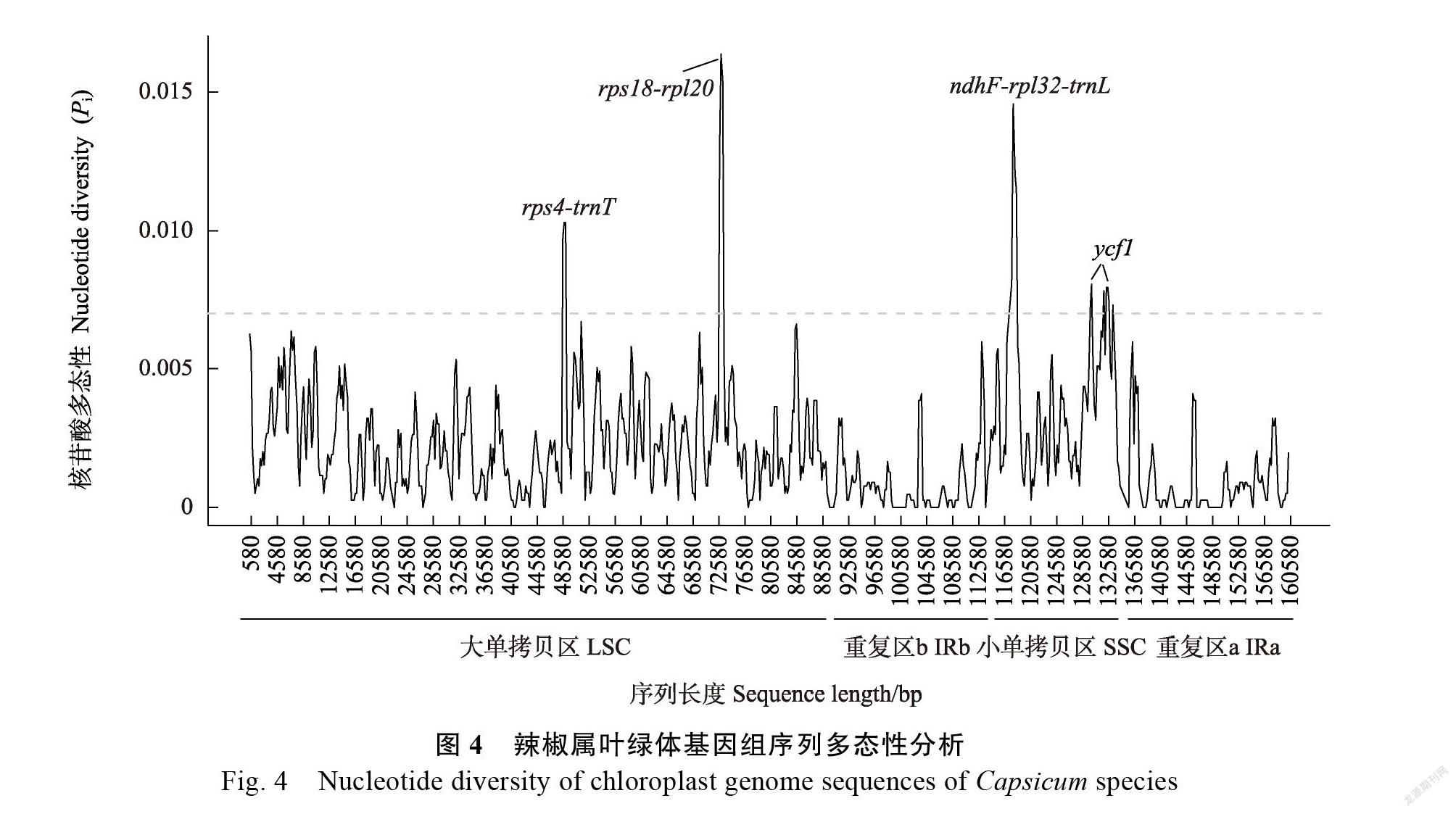
使用DnaSP软件对辣椒属13个物种叶绿体基因组核苷酸多态性()进行分析(图4)。共检测到1514个多态性位点,LSC、SSC和IR区平均值分别为0.002 39、0.003 83和0.000 86,发现3个非编码区核苷酸多态性热点(hotspots)r-、、--和1个编码区热点,其中r-(=0.016 37)和(=0.010 30)位于LSC,-- (=0.014 57)和(=0.008 06)位于SSC。基因组中LSC和SSC区多态性位点数较多,IR区变异较少,表现为相对保守。
辣椒属系统发育
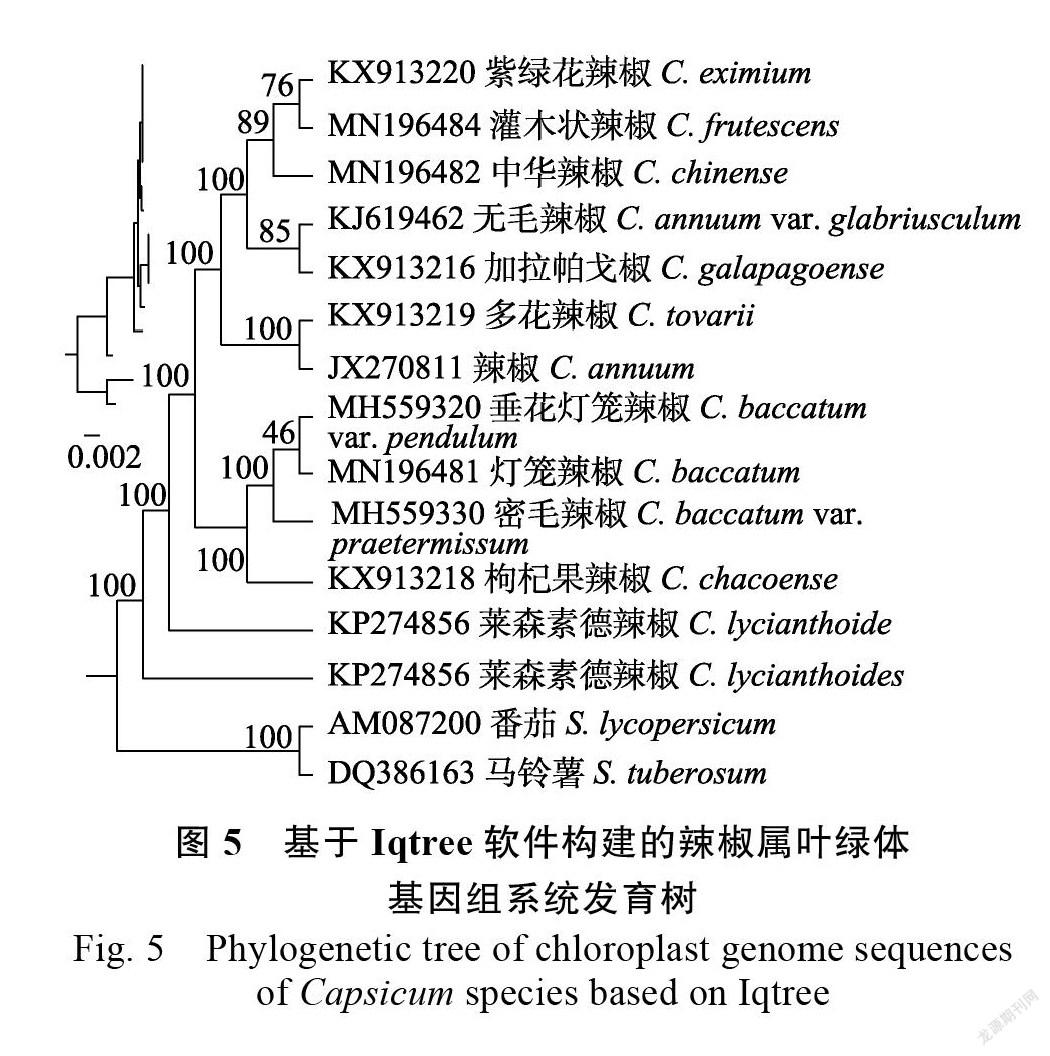
基于叶绿素基因组数据,构建了辣椒属物种系统发育关系(图5)。莱森素德辣椒位于辣椒属系统发育树的基部,随后是绒毛辣椒,节点支持率均为100%。辣椒和多花辣椒以100%的支持率聚成一支,中华辣椒以89%的支持率与灌木状辣椒和紫绿花辣椒聚成一支,密毛辣椒以100%的支持率与灯笼辣椒和垂花灯笼辣椒聚成一支。
討论
随着高通量测序技术的发展,植物叶绿体基因组序列为分类学、物种鉴定和系统发育提供了有价值的遗传信息,并被广泛用于评估物种遗传多样性和物种亲缘关系。目前,多个辣椒属叶绿体基因组已被公布,本研究对辣椒属物种叶绿体基因组序列特征、结构变异和系统发育进行分析,发现13个辣椒属物种叶绿体基因组均具有典型的四分体结构,2个IR将LSC和SSC分开,物种间叶绿体基因组大小差异1.5 kb以内,编码的基因种类和数目一致,显示基因组结构高度保守。
遗传多样性评价对于种质资源的收集和高效利用具有重要意义。重复序列在叶绿体基因组重排和序列变异中发挥重要作用,基因组SSR标记可用作物种群体遗传多样性、遗传演化和基因组图谱分析,在植物群体遗传学和分子育种中具有重要的应用价值。辣椒属叶绿体基因组中正向重复比例最高,其次是回文重复,反向重复和互补重复比例较低,这与其他茄科物种一致,而与杨属、芸苔属等结果不同,说明物种的亲缘关系与重复序列的种类及数量存在一定的相关性。辣椒属叶绿体基因组中以单核苷酸和二核苷酸SSR为主,且含有较高的AT含量,这支持了植物叶绿体基因组中的SSR通常由多聚A或多聚T重复组成,较少包含串联G或C重复,这些结果与其他物种类似。不同科或属的植物叶绿体基因组核苷酸多态性位点有较大差异,但一般情况下,非编码区的变异位点比例高于编码区,2个IR区相对LSC或SSC含有较少的序列变异。辣椒属叶绿体基因组LSC和SSC序列变异高于IR,识别的4个核苷酸多态性热点位于LSC或SSC。热点和与D’AGOSTINO等对11个辣椒物种叶绿体基因组变异的研究结果一致。蒿属()、茄科枸杞属()和木兰科(Magnoliaceae)叶绿体基因组中也分别鉴定到了、和属于核苷酸多态性热点。本研究中鉴定的重复序列和核苷酸多态性位点可作为遗传多样性标记,用于辣椒属种间物种鉴定和谱系分析。
目前,基于DNA条形码、质体基因和SSR标记等对辣椒属物种多样性和种间关系开展了多项研究,然而辣椒属种间关系仍存在争议,基于叶绿体基因组的遗传多样性和序列多态性分析对辣椒品种改良具有重要意义。本研究基于已公布的辣椒属物种叶绿体基因组序列构建系统发育树,中华辣椒与灌木状辣椒聚成一个高支持率的系统发育支,二者叶绿体基因组SSC和IR区大小一致,仅在LSC区相差7 bp,结果支持了中华辣椒可能是灌木状辣椒的栽培变种的结论。辣椒与多花辣椒亲缘关系较近,二者叶绿体基因组仅相差35 bp。本研究结果可为辣椒属植物系统发育关系和遗传育种提供理论参考。
本研究结果表明,辣椒属植物叶绿体基因组全长介于156 583 bp~158 077 bp之间,平均GC含量为37.70%,基因组结构保守。长重复序列以正向重复和回文重复为主,30~39 bp长度的序列比较最高。SSR位点主要为单核苷酸和二核苷酸重复,通常由多聚A或多聚T组成。基因组非编码区序列变异高于基因编码区,4个核苷酸多态性热点中,r-和位于LSC,--和位于SSC。辣椒与多花辣椒、中华辣椒与灌木状辣椒和紫绿花辣椒、密毛辣椒与灯笼辣椒和垂花灯笼辣椒分别具有较近的亲缘关系。
参考文献
- AGUILAR-MELENDEZ A, MORRELL P L, ROOSE M L, KIM S C. Genetic diversity and structure in semiwild and domesticated chiles (; Solanaceae) from Mexico[J]. American Journal of Botany, 2009, 96(6): 1190- 1202.
- CAROLINA G C, BARFUSS M H, SEHR E M, BARBOZA G E, SAMUEL R, MOSCONE E A, EHRENDORFER F. Phylogenetic relationships, diversification and expansion of chili peppers (, Solanaceae)[J]. Annals of Botany, 2016, 118(1): 35-51.
- TRIPODI P, RABANUS-WALLACE M T, BARCHI L, KALE S, ESPOSITO S, ACQUADRO A, SCHAFLEITNER R, VAN ZONNEVELD M, PROHENS J, DIEZ M J, BORNER A, SALINIER J, CAROMEL B, BOVY A, BOYACI F, PASEV G, BRANDT R, HIMMELBACH A, PORTIS E, FINKERS R, LANTERI S, PARAN I, LEFEBVRE V, GIULIANO G, STEIN N. Global range expansion history of pepper ( spp.) revealed by over 10,000 genebank accessions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(34): e2104315118.
- AKHTAR A, ASGHAR W, KHALID N. Phytochemical constituents and biological properties of domesticated species: a review[J]. Bioactive Compounds in Health and Disease, 2021, 4(9): 201-225.
- ZHU B, QIAN F, HOU Y, YANG W, CAI M, WU X. Complete chloroplast genome features and phylogenetic analysis of (Brassicaceae)[J]. PLoS One, 2021, 16(3): e0248556.
- AMIRYOUSEFI A, HYVÖNEN J, POCZAI P. The chloroplast genome sequence of bittersweet (): Plastid genome structure evolution in Solanaceae[J]. PLoS One, 2018, 13(4): e0196069.
- 黄琼林. 高良姜叶绿体基因组测序与特征分析[J]. 热带作物学报, 2021, 42(1): 1-6.HUANG Q L. Complete sequencing and analysis of chloroplast genome from Hance[J]. Chinese Journal of Tropical Crops, 2021, 42(1): 1-6. (in Chinese)
- 杜致辉, 杨 澜, 张朝君, 许红娟, 陈之林. 黑喉石斛叶绿体基因组特征及比较分析[J]. 热带作物学报, 2021, 42(11): 3111-3119.DU Z H, YANG L, ZHANG C J, XU H J, CHEN Z L. Characteristics of the complete chloroplast genome of and its comparative analysis[J]. Chinese Journal of Tropical Crops, 2021, 42(11): 3111-3119. (in Chinese)
- SHIRAGAKI K, YOKOI S, TEZUKA T. Phylogenetic analysis and molecular diversity of based on rDNA-ITS region[J]. Horticulturae, 2020, 6(4): 87.
- TANG B, XIE L, YI T, LV J, YANG H, CHENG X, LIU F, ZOU X. Genome-wide identification and characterization of the mitochondrial transcription termination factors (mterfs) in L[J]. International Journal of Molecular Sciences, 2019, 21(1): 269.
- ZHONG Y, CHENG Y, RUAN M, YE Q, WANG R, YAO Z, ZHOU G, LIU J, YU J, WAN H. High-throughput SSR marker development and the analysis of genetic diversity in [J]. Horticulturae, 2021, 7(7): 187.
- MAGDY M, OU L, YU H, CHEN R, ZHOU Y, HASSAN H, FENG B, TAITANO N, VAN DER KNAAP E, ZOU X, LI F, OUYANG B. Pan-plastome approach empowers the assessment of genetic variation in cultivated species[J]. Hortic Research, 2019, 6(1): 108.
- BARBOZA G E, CARRIZO GARCIA C, LEIVA GONZALEZ S, SCALDAFERRO M, REYES X. Four new species of (Solanaceae) from the tropical Andes and an update on the phylogeny of the genus[J]. PLoS One, 2019, 14(1): e0209792.
- D’AGOSTINO N, TAMBURINO R, CANTARELLA C, DE CARLUCCIO V, SANNINO L, COZZOLINO S, CARDI T, SCOTTI N. The complete plastome sequences of eleven genotypes: insights into DNA variation and molecular evolution[J]. Genes (Basel), 2018, 9(10): 503.
- RAVEENDAR S, LEE K J, SHIN M J, CHO G T, LEE J R, MA K H, LEE G A, CHUNG J W. Complete chloroplast genome sequencing and genetic relationship analysis of Jacq[J]. Plant Breeding and Biotechnology, 2017, 5(4): 261-268.
- SEBASTIN R, LEE K J, CHO G T, SHIN M J, KIM S H, HYUN D Y, LEE J R. The complete chloroplast genome sequence of a Bolivian wild chili pepper, Hunz. (Solanaceae)[J]. Mitochondrial DNA Part B, 2019, 4(1): 1634-1635.
- JO Y D, PARK J, KIM J, SONG W, HUR C G, LEE Y H, KANG B C. Complete sequencing and comparative analyses of the pepper ( L.) plastome revealed high frequency of tandem repeats and large insertion/deletions on pepper plastome[J]. Plant Cell Reports, 2011, 30(2): 217- 229.
- WEN F, WU X, LI T, JIA M, LIU X, LIAO L. The complete chloroplast genome of and compared analysis revealed adaptive evolution of subfamily Lardizabaloideae species in China[J]. BMC Genomics, 2021, 22(1): 161.
- PARK H S, LEE J, LEE S C, YANG T J, YOON J B. The complete chloroplast genome sequence of Jacq. (Solanaceae)[J]. Mitochondrial DNA Part B, 2016, 1(1): 164-165.
- CUI Y, ZHOU J, CHEN X, XU Z, WANG Y, SUN W, SONG J, YAO H. Complete chloroplast genome and comparative analysis of three (Solanaceae) species with medicinal and edible properties[J]. Gene Reports, 2019, 17: 100464.
- ZHOU J, ZHANG S, WANG J, SHEN H, AI B, GAO W, ZHANG C, FEI Q, YUAN D, WU Z, TEMBROCK L R, LI S, GU C, LIAO X. Chloroplast genomes in (Salicaceae): comparisons from an intensively sampled genus reveal dynamic patterns of evolution[J]. Scientific Reports, 2021, 11(1): 9471.
- SHAHZADI I, ABDULLAH, MEHMOOD F, ALI Z, AHMED I, MIRZA B. Chloroplast genome sequences of and : Comparative analyses, mutational hotspots in genus and phylogeny in family Asteraceae[J]. Genomics, 2020, 112(2): 1454-1463.
- IRAM S, HAYAT M Q, TAHIR M, GUL A, ABDULLAH, AHMED I. Chloroplast genome sequence of : Comparative analyses and screening of mutational hotspots[J]. Plants (Basel), 2019, 8(11): 476.
- DENG Y, LUO Y, HE Y, QIN X, LI C, DENG X. Complete chloroplast genome of and a comparative analysis with four Magnoliaceae species[J]. Forests, 2020, 11(3): 267.
