基于RAD-seq简化基因组测序的西藏黄牛遗传多样性研究
2021-09-11邓兵晋美旺杰任增帮次旦欧珠达瓦罗布杨锐陈仕毅邝良德
邓兵 晋美旺杰 任增帮 次旦欧珠 达瓦罗布 杨锐 陈仕毅 邝良德


摘要:为在基因组水平研究西藏黄牛的遗传多样性,并通过选择信号分析发掘重要种质特性基因,利用RAD-seq简化基因组测序鉴定拉萨黄牛、阿沛甲咂牛、日喀则驼峰牛和樟木黄牛的SNP标记,计算群体遗传结构和遗传进化,鉴定基因组受选择区域和受选择基因。结果表明,共鉴定出1 355 274个SNP标记。阿沛甲咂牛和樟木黄牛遗传多样性最为丰富,观察杂合度分别为0.185 6、0.164 4,核苷酸多样性分别为0.202 2、0.202 6,拉萨黄牛和日喀则驼峰牛的遗传多样性相对低些。近交系数最高的为阿沛甲咂牛,而日喀则驼峰牛的近交系数最低。拉萨黄牛与日喀则驼峰牛之间的遗传分化系数最高(0.092 7),而樟木黄牛和阿沛甲咂牛之间的遗传分化系数最低(-0.001 1)。聚类分析结果表明,日喀则驼峰牛和阿沛甲咂牛之间的亲缘关系最近,而与樟木黄牛的亲缘关系最远。通过选择信号分析,在西藏黄牛群体中检测出22个基因组区域受到选择,包含96个受选择基因。GO富集分析表明,这些受选择基因显著富集在心血管系统发育、炎症反应、细胞间连接、染色质结合等生物学通路。本研究从基因组水平揭示西藏黄牛的种质特性,为进一步开展西藏黄牛种质资源保护及利用提供了重要理论依据。
关键词:西藏黄牛;遗传多样性;简化基因组测序
中图分类号:S823.8+12 文献标志码: A 文章编号:1002-1302(2021)16-0153-04
西藏黄牛是西藏的主要地方家畜品种之一,集中分布于西藏农区、半农半牧区和林区,主要分布在海拔 3 000~4 200 m,且分布数量随海拔高度的增加而减少。雅鲁藏布江中下游、喜马拉雅山东段和三江流域下游地区分布较集中,占全区黄牛总数的50%以上。西藏黄牛体型外貌以及生产性能由于分布区域的不同存在一定差异,经过长期自然选择形成了拉萨黄牛、阿沛甲咂牛、日喀则驼峰牛、樟木黄牛等4个主要地方类群。西藏黄牛以产乳为主,乳、肉、役兼用,具有体型小、成熟晚、耐寒、耐粗饲、适应高海拔、抗逆性强等特点,是我国宝贵的地方畜禽遗传资源[1-3]。
通过利用微卫星标记和线粒体DNA标记来进行遗传多样性和遗传进化分析已经在不同品种牛上广泛开展[4-7],然而这些DNA标记在整个牛基因组中的覆盖范围是极其微小的,所代表的基因组遗传变异信息量也是非常有限的。而新一代测序技术的出现实现了SNP分子标记的高通量检测,为从整个基因组水平上研究牛的遗传进化提供了准确高效的研究方法[8]。RAD-seq简化基因组测序是通过限制性内切酶对基因组进行打断,然后进行高通量测序从而得到大量遗传多态性标签序列,该技术流程简单、费用较低,能有效降低基因组测序复杂度,目前,已广泛应用在动物分子遗传标记开发、群体遗传及进化学、高密度遗传图谱构建等研究中[9-10]。因此,本研究利用RAD-seq简化基因组测序对西藏黄牛4个类群的遗传多样性和系统进化进行研究,能为西藏黄牛遗传保种提供科学依据,为进一步进行品种改良和优良新品种培育奠定基础。
1 材料与方法
1.1 试验动物
以西藏黄牛的4个地方类群,即拉萨黄牛(L)、阿沛甲咂牛(J)、日喀则驼峰牛(T)和樟木黄牛(Z)为研究对象,于2020年7月在西藏拉萨、林芝、日喀则随机采集每个类群30头成年牛(公母各半)血液样本,尽量避免个体间存在亲缘关系,合计采集120头成年西藏黄牛血液样本,-20 ℃保存备用。
1.2 试验方法
1.2.1 RAD-seq简化基因组测序 利用天根血液基因组DNA提取试剂盒(DP318-02)提取样品基因组DNA,经质检合格后,利用Super GBS技术[1]构建pair-end测序文库(300~500 bp),在Illumina PE150平台上对构建好的文库进行RAD-seq簡化基因组测序。测序由上海欧易生物医学科技有限公司完成。
1.2.2 SNP质控 测序结果用GATK 和Samtools程序进行SNP鉴定,质控条件为:Q20>95%,ddRAD depth>60%,SNP Call rate>90%,MAF>0.05。
1.2.3 统计方法 利用Pop Gen 软件进行观察杂合度(Ho)、近交系数(Fis)和群体遗传分化系数(Fst)计算,利用Treemix软件进行基因流(Nm)计算,利用PLINK 软件进行连锁不平衡(LD)分析和选择信号检测,利用MEGA 软件UPGMA聚类法进行群体聚类分析。
2 结果与分析
2.1 基因组SNP标记鉴定
经过两步法数据质控,在西藏黄牛4个类群中共鉴定出SNP 1 355 274个,其中,常染色体上鉴定出SNP 1 326 666个,性染色体上鉴定出SNP 28 588个,总体上,SNP数量与染色体长度呈正相关。
2.2 西藏黄牛不同类群遗传多样性分析
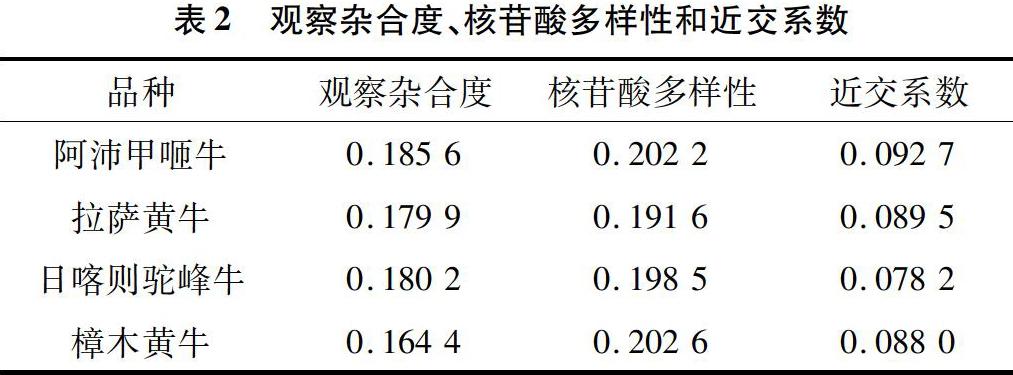
由表2可知,阿沛甲咂牛和樟木黄牛遗传多样性最为丰富,观察杂合度分别为0.185 6、0.164 4,核苷酸多样性分别为0.202 2、0.202 6,拉萨黄牛和日喀则驼峰牛的遗传多样性相对低些,核苷酸多样性分别为0.191 6、0.198 5,与LD连锁不平衡分析结果(图1)相一致。近交系数最高的是阿沛甲咂牛,为0.092 7,日喀则驼峰牛的近交系数最低,为0.078 2。
2.3 西藏黄牛不同类群遗传进化分析
西藏黄牛不同类群遗传分化系数(Fst)和基因流(Nm),由表3可知,拉萨黄牛与日喀则驼峰牛之间的遗传分化系数最高(0.092 7),而樟木黄牛和阿沛甲咂牛之间的遗传分化系数最低(-0.001 1),相对应的,拉萨黄牛与日喀则驼峰牛之间的基因流最高(0.021 0),而樟木黄牛和阿沛甲咂牛之间的基因流最低(-0.000 3)。UPGMA 聚类结果,由图2可知,日喀则驼峰牛和阿沛甲咂牛之间的亲缘关系最近,而与樟木黄牛的亲缘关系最远。
2.4 西藏黃牛群体选择信号分析
结合核苷酸多样性比率(Pi)和遗传分化系数(Fst)的选择信号分析发现,22个基因组区域受到选择,共有96个受选择基因。由图3可知,功能分析结果受选择基因主要富集在心血管系统发育(VSIG8、TEK、ADM、HOPX)、炎症反应(CCL24、VSIG8、CCL26)、细胞间连接(TEK、SLAMF8、KDR)、染色质结合(KLF8、MAK、MCMBP、CCNT2)等生物学通路。
3 讨论
3.1 西藏黄牛群体基因组SNP鉴定和遗传多样性
本研究通过简化基因组测序在西藏黄牛4个地方类群中共鉴定出1 355 274个SNP位点,主要分布在1~19号常染色体上,且数量均在40 000个以上 说明西藏黄牛群体具有丰富的遗传多样性。本试验克服了微卫星标记和线粒体DNA技术在基因组上覆盖度较低的缺点,为从基因组水平进行西藏黄牛遗传进化和关联分析奠定了基础,也为构建西藏黄牛SNP芯片提供了必要条件。
在西藏黄牛不同类群中,阿沛甲咂牛遗传多样性最丰富,观察杂合度为0.185 6,核苷酸多样性为0.202 2。同时,其近交系数最高为0.092 7,提示在阿沛甲咂牛的保种和选种过程中应严格控制近交系数的过快增加,维持群体遗传多样性的相对稳定。而拉萨黄牛的遗传多样性最低,观察杂合度为0.179 9,核苷酸多样性为0.191 6。总体来看这与LD连锁不平衡分析结果相一致。拉萨黄牛与日喀则驼峰牛之间的遗传分化系数最高(0.092 7),说明两者之间存在中等程度的遗传分化,而拉萨黄牛与樟木黄牛以及阿沛甲咂牛之间的遗传分化系数分别为0.008 5、0.010 1,遗传分化系数较小。UPGMA聚类分析表明,日喀则驼峰牛和阿沛甲咂牛之间的亲缘关系最近,而与樟木黄牛的亲缘关系最远。总体上,遗传聚类分析结果与西藏黄牛4个地方类群间遗传分化、基因流以及地理分布相一致。
3.2 西藏黄牛群体选择信号
大量研究表明,在动物进化过程中,无论是先天自然选择,还是后天人工选择都会在动物基因组上留下选择信号,受到选择区域会产生高度遗传分化。因此,进行选择信号分析有助于进一步了解基因组遗传分化对动物表型的影响,发掘筛选出影响动物性状的重要的功能基因。目前,国内外学者利用基因组测序或基因芯片技术已经在猪、羊、鸡等畜禽上开展了选择信号研究,发掘出大量种质特性基因和经济性状相关基因[11-14]。针对牛而言,Zinovieva等利用高密度SNP基因分型芯片对2个俄罗斯本地牛品种进行选择信号研究,检测出了新的基因组受选择区域和受选择候选基因[15]。Ben-Jemaa 等利用SNP芯片对北非牛进行基因组选择信号研究,确定了36个受选择的基因组区域,筛选到了GH1、ACE、ASIC3、HSPH1等与炎热相关的受选择基因[16]。Mariadassou等利用全基因组测序对利木赞牛进行选择信号研究,找到了57个受选择基因组区域以及68个受选择候选基因,其中包括MSTN、NCKAP5、RUNX2等可能与利木赞牛重要表型性状相关的基因[17]。Xia等利用全基因组测序评估了中国郏县红牛基因组多样性和选择信号特征,采用FST方法找到了17个受选择基因,这些基因可能与郏县红牛的饲料转化率(CCSER1)、肉品质性状(ROK2、PPP1R12A、CYB5R4、EYA3、PHACTR1)、生育能力(RFX4、SRD5A2)和免疫应答(SLAFF1、CD84和SLAFF6)密切相关[18]。吕世杰等通过简化基因组测序对中国南阳牛和安格斯牛进行选择信号分析,筛选得到33个受选择基因组区域,其中,16个基因组区域与生长性状相关QTLs重合,找到了4个基因(FXR1、ADAR、IGF1和MNF1)与骨生长、肌肉发育和生长调控有关[19]。
在青藏高原上,缺氧是影响动物机体生长发育的主要因素,生活在青藏高原上的西藏黄牛具有耐寒、耐粗饲、适应高海拔、抗逆性强的特点。本试验中,通过选择信号分析在西藏黄牛群体中发现22个基因组区域受到选择,找到了VSIG8、TEK、ADM、HOPX等4个显著富集在心血管系统发育的受选择基因,而心血管系统发育与动物对高海拔缺氧环境的适应性密切相关。因此,这4个受选择基因极有可能是西藏黄牛对高海拔适应性的候选基因。其中HOPX基因已被证实在心脏发育和心肌细胞增殖中发挥着重要作用[20-21],而ADM基因参与血管系统调节[22],VSIG8和TEK在心血管系统中的作用尚未证实,需要进一步研究。同时,与炎症反应相关的基因CCL24、VSIG8、CCL26也受到了选择,CCL24和CCL26是趋化性细胞因子家族成员,能够促进动物体内炎症部位的各种白细胞的补充、激活及黏附[23]。推测CCL24、VSIG8、CCL26这3个基因与西藏黄牛的抗逆性强相关,因而在进化上受到选择。
4 结论
综上所述,本研究利用简化基因组测序准确揭示了西藏黄牛4个类群基因组水平的遗传多样性和系统进化,选择信号分析表明,选择作用主要集中在高海拔适应性、抗逆性等方面的塑造。该研究结果进一步为西藏黄牛种质资源的保护及利用提供了重要参考依据。
参考文献:
[1]唐建华,陈晓英,宋天增,等. 西藏黄牛种质资源保护与利用研究[J]. 中国牛业科学,2016,42(3):48-51.
[2]刘锡武,刘鹏,高玉君. 西藏日喀则市黄牛改良现状与对策[J]. 中国牛业科学,2009(4):63-65.
[3]石 达. 西藏畜禽品种遗传资源[M]. 北京:中国农业大学出版社,2010.
[4]黄 兴,柴志欣,信金伟,等. 西藏牛亚科部分群体线粒体DNA遗传多样性研究[J]. 西南民族大学学报(自然科学版),2019,45(2):117-124.
[5]張桂香,郑友民,王志刚,等. 我国部分黄牛品种线粒体D-loop区遗传多样性与起源分化[J]. 遗传,2009,31(2):160-168.
[6]姬秋梅. 西藏牦牛mtDNA cytb基因的序列多态性及其系统进化分析[J]. 中国畜牧兽医文摘,2013(12):77.
[7]汪 琦,钟金城,柴志欣,等. 三江黄牛mtDNA Cytb基因序列多态性及其系统进化分析[J]. 中国畜牧杂志,2016,52(15):20-27.
[8]Davey J W,Hohenlohe P A,Etter P D,et al. Genome-wide genetic marker discovery and genotyping using next-generation sequencing[J]. Nature Reviews Genetics,2011,12(7):499-510.
[9]韩 威,朱云芬,殷建玫,等. 基于RAD-seq简化基因组测序的19个地方鸡种遗传进化研究[J]. 畜牧兽医学报,2020,51(4):670-678.
[10]Hohenlohe P A,Amish S J,Catchen J M,et al. Next-generation RAD sequencing identifies thousands of SNPs for assessing hybridization between rainbow and westslope cutthroat trout[J]. Molecular Ecology Resources,2011,11 (S1):117-122.
[11]Chen Q,Huang Y,Wang Z,et al. Whole-genome resequencing reveals diversity and selective signals in Longlin goat[J]. Gene,2021,771:145371.
[12]Wang M S,Zhang R W,Su L Y,et al. Positive selection rather than relaxation of functional constraint drives the evolution of vision during chicken domestication[J]. Cell Research,2016,26(5):556-573.
[13]Wu F,Sun H,Lu S,et al. Genetic diversity and selection signatures within diannan Small-Ear Pigs revealed by Next-Generation sequencing[J]. Frontiers in Genetics,2020,11:733.
[14]Wang X,Zhang H,Huang M,et al. Whole-genome SNP markers reveal conservation status,signatures of selection,and introgression in Chinese Laiwu pigs[J]. Evolutionary Applications,2021,14(2):383-398.
[15]Zinovieva N A,Dotsev A V,Sermyagin A A,et al. Selection signatures in two oldest Russian native cattle breeds revealed using high-density single nucleotide polymorphism analysis[J]. PLoS One,2020,15(11):e0242200.
[16]Ben-Jemaa S,Mastrangelo S,Lee S H,et al. Genome-wide scan for selection signatures reveals novel insights into the adaptive capacity in local North African cattle[J]. Scientific Reports,2020,10(1):19466.
[17]Mariadassou M,Ramayo-Caldas Y,Charles M,et al. Detection of selection signatures in Limousin cattle using whole-genome resequencing[J]. Animal Genetics,2020,51(5):815-819.
[18]Xia X,Zhang S,Zhang H,et al. Assessing genomic diversity and signatures of selection in Jiaxian Red cattle using whole-genome sequencing data[J]. BMC Genomics,2021,22(1):43.
[19]吕世杰,陈付英,金 磊,等. 利用简化基因组测序筛选安格斯牛生长相关的受选择基因[J]. 畜牧兽医学报,2020,51(4):713-721.
[20]Chen F,Kook H,Milewski R,et al. Hop is an unusual homeobox gene that modulates cardiac development[J]. Cell,2002,110(6):713-723.
[21]Trivedi C M,Zhu W,Wang Q,et al. Hopx and Hdac2 interact to modulate Gata4 acetylation and embryonic cardiac myocyte proliferation[J]. Developmental Cell,2010,19(3):450-459.
[22]史洪岩,贺 綦,程 敏,等. HOPX基因过表达对鸡前脂肪细胞增殖的影响[J]. 中国农业科学,2015,48(8):1624-1631.
[23]高云飞,余艳红,李志琴 . ADM基因在正常孕妇与子痫前期孕妇妊娠晚期胎盘组织表达的研究[J]. 南方医科大学学报,2006,26(12):1828-1830.颜世卿,马 轲,陈 巍. 鸡肠源益生口乳杆菌的分离鉴定与生物学特性[J]. 江苏农业科学,2021,49(16):157-161.
