MIMLRBF预测谷物蛋白质功能方法的改进
2021-06-21崔双龙曹洪伟
刘 静,崔双龙,曹洪伟,管 骁*
(1.上海海事大学 信息工程学院,上海201306;2.上海理工大学 医疗器械与食品学院,上海200093)
谷物食品是人类营养摄入的主要来源,在我们的膳食中有举足轻重的地位[1]。在我国,谷物品种主要包括:大麦、水稻、高粱、玉米、燕麦等,它们都是良好的主食食品原料[2]。随着人们对营养与保健功能的关注,谷物蛋白质功能预测已经成为当前研究的热点问题之一[3]。面对大量的已完成测序的谷物蛋白质数据,其所对应的生物学功能仍然未知。采用传统的手工注释费时费力,已经无法满足需求,而以计算方法来预测蛋白质的生物学功能已经渐渐成为了主流。
近年来,国内外对运用计算智能技术解决此类问题已经建立了较为有效的方法。2007年,Zhou等[4]最早提出了多示例多标记(multi-instance multilabel learning,MIML)框架,随后又基于退化的思想提出了MIMLBOOST和MIMLSVM[5]算法应用于图像场景的识别。在生物信息学方面,Li等[6]提出了MIMLSVM+算法能够很好地解决果蝇基因表达模式注释的问题。Wu等[7]在MIMLNN算法的基础上,提出了En-MIMLNN算法用于全基因组的蛋白质功能预测,通过在En-MIMLNN算法中加入径向基函数来激活神经网络,使得其在主流的评价标准上预测结果有显著提升。Zhang等[8]也提出了一种基于RBF神经网络的MIMLRBF算法应用于图像场景的识别,该方法选择平均Hausdorff距离度量图像之间的相似性,从而在图像场景识别的应用中优于传统方法。在多示例多标记框架的基础上,衍生了多种方法应用到各个不同的领域中,同时在各种算法的基础上,主要通过对Hausdorff距离进行改进来度量各种场景中物体的相似性以及采用不同的径向基函数进行激活来提升算法的预测结果。
在本文中,首次将MIMLRBF算法应用于谷物蛋白质功能预测,并在此基础上,提出了多种改进的谷物蛋白质功能预测模型。首先,针对平均Hausdorff距离削弱了两种蛋白质之间最短结构域距离所起作用的问题,为平均Hausdorff距离引入一个自动调节系数[9]。同时,为提高MIMLRBF算法的预测效果,采用改进后的混合径向基核函数进行激活。通过所搭建模型分别在大麦、籼稻两种谷物蛋白质数据上进行蛋白质功能预测,并使用三种主流的评价标准进行度量,可以发现,改进后的MIMLRBF算法比传统的预测效果更好。再将作者提出的改进算法与传统的MIML算法 (En-MIMLNN、MIMLSVM)进行比较,可以发现,作者提出的改进后的算法在Hamming Loss、Macro-F1、Micro-F1这3项度量指标下具有较好的预测结果。
1 蛋白质功能预测原理
在1961年,Anfinsen[10]提出蛋白质序列、结构、功能之间存在某种相互作用关系,即通过蛋白质氨基酸序列特征可以决定其三维结构特征,同时在结构特征的基础上,可以确定所具有的某种功能。蛋白质功能预测在此理论的基础上,不断地发展和完善,提出了一系列蛋白质功能预测的方法。
在蛋白质中,其结构和功能与结构域密切相关[11],通过对氨基酸序列信息进行特征提取,所得到的由结构域信息所组成的特征向量与蛋白质的功能之间存在某种映射关系。因此可以将蛋白质功能预测转化成一个多示例多标记问题,即一个蛋白质样本往往可以由多个结构域(示例)进行表示,同时具有多种基因本体(GO)生物学功能(标记)。其预测原理见图1。

图1 蛋白质功能预测原理Fig.1 Principles of protein function prediction
2 实验方法
2.1 RBF神经网络的建立
RBF(径向基函数)神经网络[12]是一种基于径向基函数的多层前向神经网络模型,其网络结构可以看成是输入层空间到隐含层空间的一种非线性映射,以及隐含层空间到输出层的线性映射。从其网络结构(见图2)可以看出,隐含层神经元数目、RBF的中心、RBF的宽度和隐含层与输出层之间的权值矩阵等,在RBF神经网络中都是非常重要的学习参数。

图2 RBF神经网络结构图Fig.2 RBF neural network structure diagram
而在隐含层中,通常采用径向基核函数进行激活,常用的径向基核函数有:
高斯(Gauss)核函数:

其中,δ为核函数中围绕中心点的宽度,δ越小,则核函数宽度越大,函数的选择性就越大。
2.2 基于RBF神经网络MIML算法
MIMLRBF(multi-instance multi-label radial basis function)神经网络是从传统RBF神经网络派生而来的,用于解决多示例多标记问题的一种神经网络的方法。而蛋白质功能预测恰恰可以转化为一个多示例多标记问题。通过从已知蛋白质氨基酸序列信息出发,经过特征提取,将一个蛋白质的结构由所提取多个结构域(示例)信息表示,然后和已知蛋白质中多个GO生物学功能(标记)进行关联。
MIMLRBF算法[8]使用双层架构进行训练。在第一层中,通过结合包间平均Hausdorff距离,利用k-Medoids算法[13]将训练样本进行聚类,保留每个聚类簇的中心点。在第二层中,通过径向基函数神经网络计算样本和中心点之间的基函数,然后利用最小化平方和误差函数所获取的一、二层之间的权重乘以基函数φ(·),从而得到模型的输出,其中输出值大于等于零的标记设为正标记,否则为负标记。
令S={(X i,Y i)|1≤i≤N}为训练样本集,其中N为蛋白质样本的数目,X i为样本集中的第i个蛋白质,用多个结构域转化而成的特征向量所表示,也就是一个包含多个示例的包(bag),Y i表示对应于X i的生物学功能,由其基因本体生物学功能方面的GO编号表示,也就是一个含有多个标记的集合。用U l={X i|(X i,Y i)∈S,l∈Y i}表示拥有第l个标记的蛋白质的集合,其中S为所有U l的合集,其标记总数为m。
为了测算蛋白质之间的相似性,通过计算由包组成的蛋白质之间的平均Hausdorff距离来表示。即对包A={a1,a2,…,ani}和包B={b1,b2,…bnj},两包之间的平均Hausdorff距离[14]为:

接着在第二层中,对于X i的第j个径向基核函数的激活定义如下:

其中额外的核函数φ0(X i)设定初始值为1。而对于所有的δj取相同的标准偏差δ,其为每对中心点包间平均Hausdorff距离的平均值的u倍。

其中u为缩放系数。
接着通过两层之间的权值矩阵W=[wlj],使用式(8)结合径向基核函数得到输出:

W通过最小化平方和误差函数来优化:

其中til是样本X i在对于第l个功能的标记(groundtruth),若l∈Y i取+1,否则取-1。
于是,对于一个样本X,其最终预测结果Y表示为:
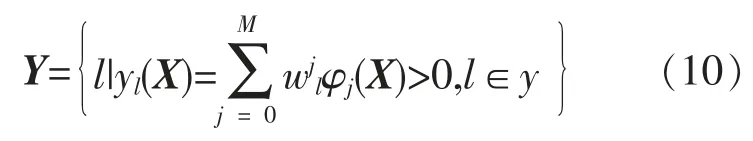
即对于一个给定的蛋白质,首先对氨基酸序列信息进行特征提取,得到由结构域信息所组成的特征向量;再通过MIMLRBF算法模型进行分析,最终可以预测其可能拥有的GO生物学功能。
2.3 基于RBF神经网络的MIML算法改进
2.3.1 包间距离的度量方式及改进 在本文中,蛋白质之间的相似性转化为求解两种蛋白质包与包之间的距离,而选取不同的距离公式所计算出的距离将直接决定整个算法的学习性能的好坏。在以往的研究中,Wu等[7]在MIMLNN算法的基础上,通过利用集成的Hausdorff距离进行改进,提出了En-MIMLNN算法用于预测全基因组的蛋白质功能。而集成的Hausdorff距离就是以包之间3种Hausdorff距离(最大Hausdorff[15]、最小Hausdorff[15]和平均Hausdorff[14])的平均值作为包间距离。其中最大Hausdorff距离对外围的噪声点比较敏感,而最小Hausdorff距离不会受到噪声点的影响,但是只考虑了最近结构域之间的距离。平均Hausdorff距离对最大Hausdorff距离和最小Hausdorff距离进行了修正,充分考虑了噪声点以及最近结构域之间的距离的影响。在本文中,MIMLRBF算法求解包之间的距离就是采用了平均Hausdorff距离。
然而,从公式(4)中可以看出,平均Hausdorff距离主要是一个蛋白质中每个结构域与其它蛋白质中最近的结构域的平均值。而一个蛋白质有可能由多个结构域所组成,个别较远的结构域可能增大了两种蛋白质之间的包间距离,从而削弱了两种蛋白质之间最近结构域的距离。在本文中,在原有的平均Hausdorff距离上进行改进,通过引用一个自动调节参数[9],将两种蛋白质之间所有结构域间的最短距离考虑进来,其定义如下:

则蛋白质之间的相似性通过蛋白质包A与包B之间距离求解,其定义如下:

即两种蛋白质之间最近结构域间的距离越小,则W值越小,蛋白质包间距离也就相对较小。
2.3.2 径向基核函数的改进 在本文中,MIMLRBF算法模型中隐含层激活函数通常采用径向基核函数,而不同的核函数的内推和外推能力不同。因此,选取不同的核函数,所搭建的算法模型的学习能力各有优劣。而为了得到一个更具有泛化能力的核函数,可以将不同的核函数进行混合得到新的核函数,即:
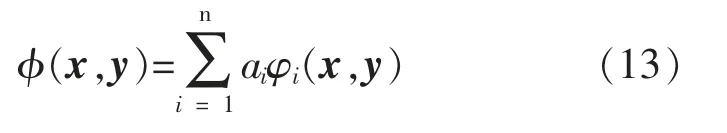
其中ai为各个核函数在混合核函数中所占的权重,n为核函数的数量。
当选取的单个核函数φi满足文献[16]提出的函数逼近定理,则混合核函数ϕ(x,y)一定满足,因此,可以用新的混合核函数在MIMLRBF算法模型隐含层中进行激活。而且混合核函数ϕ(x,y)是由多个核函数φi所张成的空间,所以其函数总体的逼近能力显然要强于其中任一单个核函数的逼近能力。
在当前的研究中,核函数的类型有许多,但是归结起来,可以分为全局性核函数与局部性核函数。在本文中,通过选取高斯核函数(局部性核函数)与多元二次核函数(全局性核函数)进行混合[17],得到新的混合径向基核函数ϕ(x,y),即:

其中:a为权重系数,0≤a≤1。
将新的混合核函数代替原有的MMLRBF算法中的核函数进行激活,通过所搭建的模型对蛋白质功能进行预测。
3 实验与结果
3.1 实验配置
在本文中,从UniProt蛋白质生物数据库中获取大麦(Hordeum vulgare)、籼稻(Oryza sativasubsp.indica)两种谷物蛋白质数据集。对于每个生物体,通过表1关键词进行检索,分别从UniProtKB/Swiss-Prot(包含检查过的、手工注释的条目)和UniProtKB/TrEMBL(包含未校验的、自动注释的条目)中得到蛋白质氨基酸序列元数据以及对应的基因本体(GO)生物学功能[18](包括分子功能、生物学过程、细胞组分)信息(2018年2月发布)。其中,通过关键词“molecular function”限定,使得所检索出的蛋白质必然包含分子功能方面相关信息。
然后,通过对所获取的氨基酸序列进行特征提取[19-20],可以得到由结构域信息所组成的216维的特征向量来表示任意一段氨基酸序列。而对于一个蛋白质往往具有一个或多个结构域,因此对于任意蛋白质可以由特征向量组成的示例所表示,同时对所有的蛋白质的GO生物学功能标记求合集,确定每个蛋白质的标记向量,从而得到了一个多示例多标记样本库。
作者选取了3种常见的多标记学习评价标准:Hamming Loss(HL)、Macro-F1(maF1)、Micro-F1(miF1)[21-23]。其中,HL指标通过计算预测的标记结果与实际样本标记之间的差距来衡量蛋白质功能模型的性能,其值越小则预测性能越好。其计算方法定义如下:

其中⊕表示两个集合的对称差分,N为样本的数量,y为标记向量,xi为预测值,yi为真实值。maF1、miF1指标表示分别对F1值(F1 Measure)应用宏平均(macro average)和微平均(micro average)。其中,maF1是基于统计量求得在各个类上的分类性能,然后把所有类上的均值作为最终结果。而miF1首先将各个类上的统计量相加,然后再将求得分类性能作为最终结果,两者指标值越大,其预测效果越好。两者的计算方法定义如下:

其中﹤>为数量积,N为样本的数量,xi为预测值,yi为真实值。
作者采用10折交叉验证,即通过UniProtKB/TrEMBL蛋白质中获取的样本数据进行10折交叉验证,获得了所建功能模型的内部测试结果。为了更好地说明所建模型的泛化能力,将UniProtKB/Swiss-Prot中的数据替换上述10折交叉的验证集进行验证,得到了所建功能模型的外部测试结果,更进一步证明所建模型的优越性。且每个算法都运行10次,计算“均值标准方差”作为最终结果。
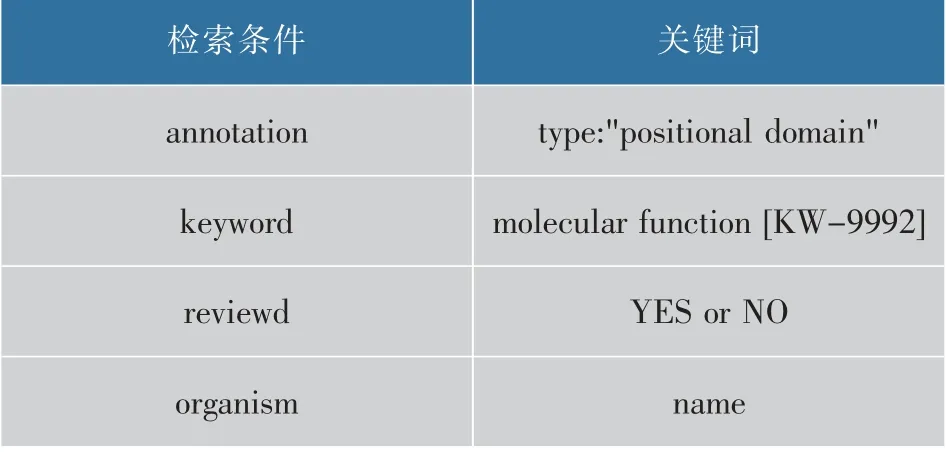
表1 UniProt检索条件Table 1 UniProt search criteria
3.2 参数选取与算法性能
在MIMLRBF蛋白质功能预测模型中,存在两个关键参数,分别是分数参数a和缩放系数u。在本文中,利用网格化方法进行搜索,其中a以步长0.02在区间[0.02,0.1]变化,而u则以步长0.2在区间[0.2,1]变化。获取所对应参数中Hamming Loss、maF1、miF1三个评价指标值的最优值,其最优结果见表2。对于大麦,可以发现,单独使用Hausdorff距离改进与核函数改进都取得了较好的结果,通过将两者组合进行改进,其改进效果比单独改进取得了更好的结果。而对于籼稻,两者组合改进的效果只是优于单独的某一种改进。为了更加合理地说明算法模型的可靠,采用了Swiss-Prot数据库(手工注释)中的数据进行外部测试,其改进的效果与内部测试所得的效果相符合,由于在外部测试中,其验证数据的数据量较小,所以改进幅度有所下降。总之,在本文中,通过对Hausdorff距离引入一个自动调节系数,然后采用改进后的混合径向基函数进行激活,所搭建的MIMLRBF蛋白质模型取得了更好的结果。

表2 改进前后MIMLRBF性能比较Table 2 Performance comparison of MIMLRBF before and after improvement
3.3 与其他算法性能比较
在本文中,将改进MIMLRBF算法与两种常见的MIML算法(即En-MIMLNN、MIMLSVM)进行性能比较,且算法的参数值都取最优值。将En-MIMLNN算法中a设为0.1,u设为0.8。而在MIMLSVM算法中,使用宽度为0.2的高斯核。实验结果见表3,可以发现:使用两种方法一起改进后的MIMLRBF算法,其性能在谷物蛋白质功能预测方面表现得比其他的MIML算法更好一些。

表3 各种算法性能比较Table 3 Performance comparison of various algorithms
3.4 预测功能与真实功能对比及分析
在表4中,选取了其中一种基于距离和核函数共同改进的MIMIRBF算法,将其对大麦蛋白质进行外部测试所得出的预测结果和真实功能比较。在UniProtKB/Swiss-Prot数据库中,MYB3_HORVU(P20027)蛋白质通过手工注释已经发现具有GO:0003677、GO:0005634、GO:0006351、GO:0006355功能。其中,GO:0003677主要是其分子功能,表示基因产物与DNA有选择性的结合。GO:0005634是其细胞组分属性,表示膜上一种细胞器,染色体可以在其中容纳和复制。而GO:0006351和GO:0006355是其参与的生物学过程,表示DNA模板上RNA合成以及调控转录频率及范围。通过对MYB3_HORVU(P20027)蛋白质进行预测,可以发现,GO:0006355功能没有预测出来,可能GO:0006351与GO:0006355功能相近,无法做出正确预测。同样地,对于IF1C_HORVU(A1E9M5)蛋白质,其手工注释的功能有GO:0003743、GO:0009507及GO:0019843。其中,GO:0003743,GO:0019843主要对应其分子功能,表示在mRNA转化为多肽的过程中起作用以及与rRNA有选择性的结合。而GO:0009507对应其细胞组分属性,表示主要是一种含有叶绿素的质体。在预测的过程中,还预测出了GO:0043022分子功能,即表现出与核糖体有选择性相互作用。而在文献[24]中,已经明确地表示出GO:0043022是其核心功能之一。
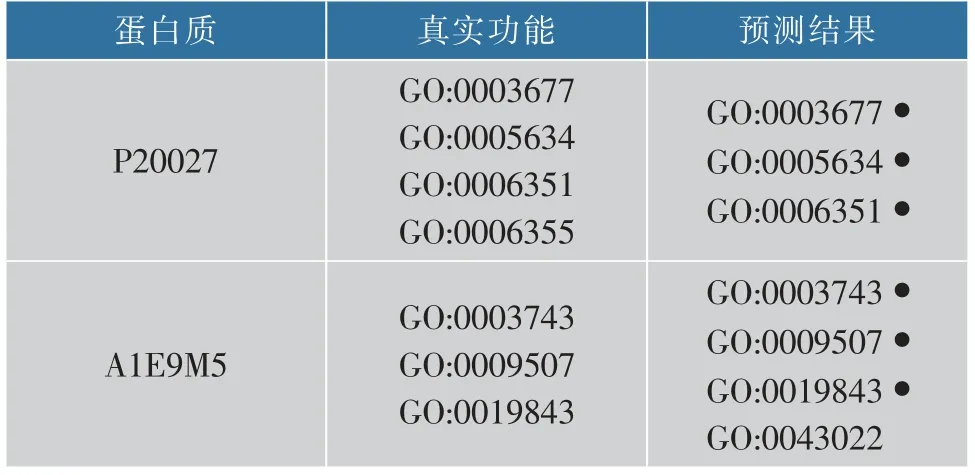
表4 大麦蛋白质预测结果举例Table 4 Examples of barley protein prediction results
4 结语
随着计算智能技术的发展,对谷物及其营养的研究越来越被重视。而在谷物食品开发的过程中,存在一系列亟待解决的关键技术难题,其中,对谷物蛋白质功能预测的研究目前还较为缺乏。在本文中,首次将MIMLRBF算法运用到谷物的蛋白质功能预测,然后在此基础上,提出了多种改进后的MIMLRBF算法模型。其中,针对平均Hausdorff距离削弱了两种蛋白质之间最短结构域距离所起作用的问题,为平均Hausdorff距离引入一个自动调节系数来计算蛋白质之间的相似性。同时,为提高MIMLRBF算法的预测能力,采用改进后的混合径向基核函数进行激活,建立了改进后的谷物蛋白质功能模型。通过使用交叉验证以及利用主流的评价标准进行评价,最后可以发现,作者提出的改进的MIMLRBF算法综合预测效果最佳,即所建模型具有较好的泛化能力。
