计算设计改造Thermobifida fusca 5-羧基-2-戊烯酰-辅酶A还原酶促进己二酸生产
2021-04-27杨菊毛银黄晓强周胜虎邓禹
杨菊,毛银,黄晓强,周胜虎,邓禹*
1(江南大学 生物工程学院,江苏 无锡,214122)2(密歇根大学 计算医学与生物信息系,密歇根 安娜堡,48109)
己二酸是一种重要的二元羧酸,广泛应用于医疗卫生、食品、化工等行业[1],据统计,超过60%的己二酸被用于生产尼龙类纤维(如尼龙6-6)[2-3]。目前,己二酸的大规模生产仍依赖化学合成,主要通过硝酸对环己醇-环己酮的混合物(醇酮油)进行氧化反应制取[4-5]。化学合成法存在着工艺流程长、副产物(尤其是氮氧化物)排放多、产品收率低等问题。因此,人们迫切希望寻找可替代化学合成己二酸的新方法。目前,全生物法合成己二酸已成为可能[6]。
本课题组于2015年发现天然菌株ThermobifidafuscaB6中存在一条全生物合成己二酸的代谢途径(图1),并且将该途径可移植到大肠杆菌中[7]。该途径涉及5个酶:β-酮硫解酶(β-ketothiolase)、3-羟酰基-辅酶A脱氢酶(3-hydroxyacyl-CoA dehydrogenase)、3-羟基己二酰-辅酶A脱氢酶(3-hydroxyadipyl-CoA dehydrogenase)、5-羰基-2-戊烯酰基-辅酶A还原酶(5-carboxy-2-pentenoyl-CoA reductase)和己二酰辅酶A合成酶(adipyl-CoA synthetase)[7]。总体来讲,该代谢过程能通过消耗D-葡萄糖来合成己二酸,但产量较低(0.2 g/L)[8]。其中,5-羰基-2-戊烯酰基-辅酶A还原酶是该途径的限速酶,其催化5-羰基-2-戊烯酰基-辅酶A(5-carboxy-2-pentenoyl-CoA)反应生成己二酰辅酶A(adipyl-CoA)的活性很低[7]。目前,关于改造5-羰基-2-戊烯酰基-辅酶A还原酶以促进己二酸合成的研究尚未见报道。
近年来,基于结构的计算酶设计方法被广泛应用于改造天然酶以适应工业需要[9-14],甚至是从头设计具有新功能的酶[15-17]。T.fusca5-羧基-2-戊烯酰-辅酶A还原酶的结构尚未通过实验解析出来。本研究首先通过同源建模构建5-羧基-2-戊烯酰-辅酶A还原酶的结构模型并对其进行分析,进而基于酶-配体复合物的结构,通过计算酶设计方法来改造5-羧基-2-戊烯酰-辅酶A还原酶的结合残基,尝试引入氢键网络来增强突变酶与目的底物5-羧基-2-戊烯酰-辅酶A的结合能力以提高酶催化效率,并通过分子动力学模拟来检验设计的合理性。
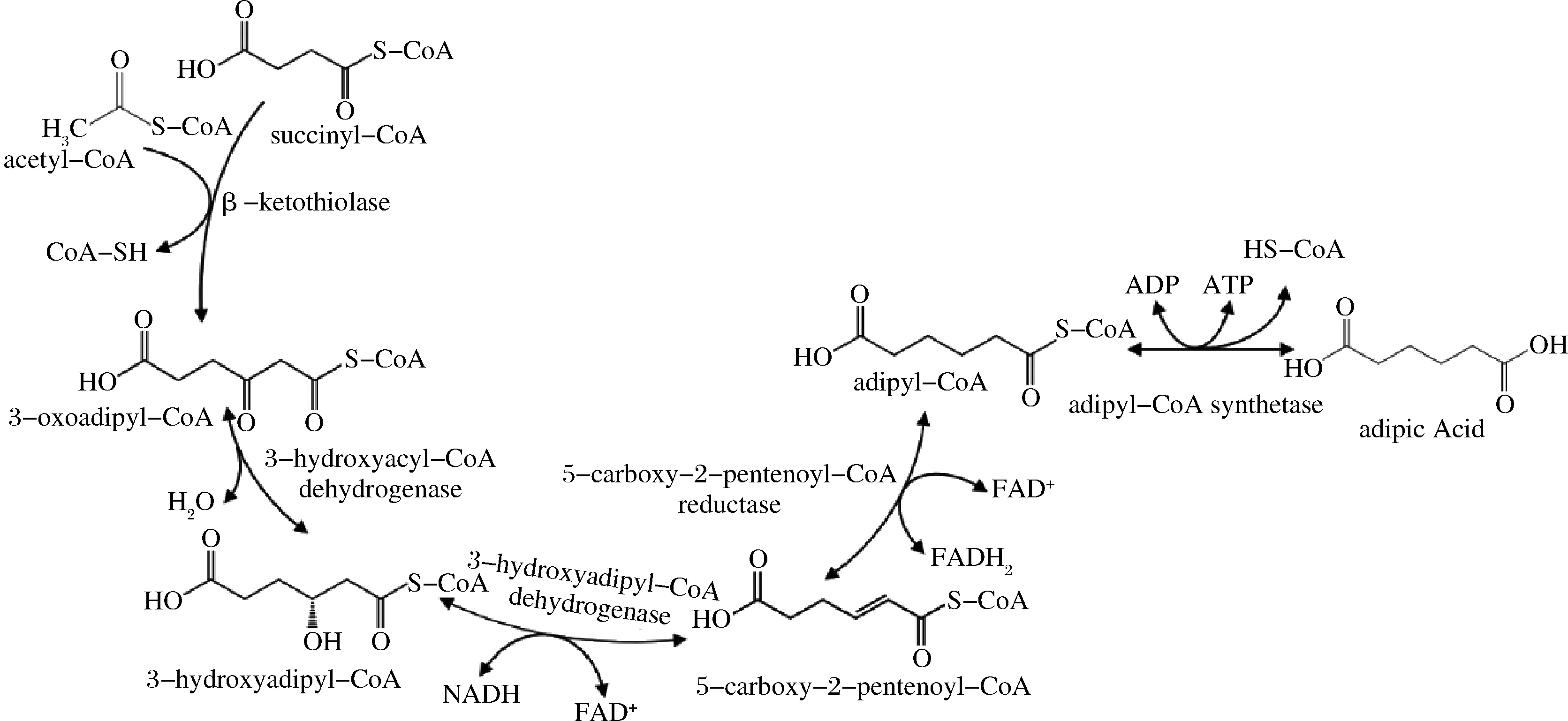
图1 己二酸的生物合成途径Fig.1 Biosynthetic pathway for producing adipic acid
1 材料和方法
1.1 蛋白质序列
5-羧基-2-戊烯酰-辅酶A还原酶包含385个氨基酸,其蛋白质序列为:
MSDFDLYRPTEEHEALREAIRSVAEDKIAPHAADVDEQ-SRFPQEAYEALRASDFHAPHVAEEYGGVGADALATCIVIEEIA-RVCASSSLIPAVNKLGSMPLILSGSDEVKQRYLPELASGEAMFS-YGLSEREAGSDTASMRTRAVRDGDDWILNGQKSWITNAGISK-YYTVMAVTDPDGPRGRNISAFVVHIDDPGFSFGEPERKLGIKG-SPTRELIFDNVRIPGDRLVGKVGEGLRTALRTLDHTRVTIGAQ-AVGIAQGALDYALGYVKERKQFGKAIADFQGIQFMLADMAM-KLEAARQMVYVAAAKSERDDADLSFYGAAAKCFASDVAMEI-TTDAVQLLGGYGYTRDYPVERMMRDAKITQIYEGTNQIQRVV-MARQLLKK
1.2 同源建模
蛋白质数据库(protein data bank,PDB)[18]中没有5-羧基-2-戊烯酰-辅酶A还原酶的实验结构,因此本文通过同源建模来构建其结构模型。对上述序列进行BLASTp[19]搜索(采用默认参数),发现PDB中存在多个同源结构。因为计算酶设计过程中需要同时对配体分子(FAD和5-羧基-2-戊烯酰-辅酶A)进行建模,所以本文选择包含了FAD和类似于5-羧基-2-戊烯酰-辅酶A配体的实验结构作为模板。将上述蛋白质序列提交到SWISS-MODEL服务器[20],搜索模板,结果显示有超过50个结构模板可供使用。本文选择分辨率1.8Å的晶体结构4L1F_A作为模板建模[21],因为该结构中包含了配体分子FAD和过硫化辅酶A(CoA persulfide,PDB文件中对应名字为COS,与目标底物5-羧基-2-戊烯酰-辅酶A结构相似),并且与5-羧基-2-戊烯酰-辅酶A还原酶具有46.28%的序列相同性。
1.3 分子动力学模拟
本文使用GROMACS 5.4.0软件进行分子动力学模拟研究[22]。首先使用其中的pdb2 gmx模块构建蛋白模型的拓扑文件,蛋白的力场选择GROMOS96 43A1联合原子力场。然后将蛋白-配体复合物模型置于一个充满TIP3P水模型的十二面体盒子中心,盒子边缘距离蛋白分子边缘为1 nm。向盒子中添加适量的Na+以平衡体系电荷。然后利用grompp模块完成体系的能量最小化。先将体系至于NVT(T=300 K)条件下平衡100 ps,然后置于NPT条件下平衡100 ps。在NVT和NPT过程中,对蛋白和配体分子上的重原子加入位置约束(1 000 kJ/mol)[23]。最后,参考文献[24-25],执行无约束分子动力学模拟,时长16 ns。
2 结果与分析
2.1 构建蛋白-配体复合物结构模型
由图2可知,本研究构建的5-羧基-2-戊烯酰-辅酶A还原酶模型(蓝绿色)具有典型的α/β折叠结构。因为该结构模型与模板4L1F_A处于相同的坐标系,将4L1F_A中的FAD和COS配体分子复制到5-羧基-2-戊烯酰-辅酶A还原酶结构模型中。需要指出的是,COS并不能参与反应,它不是一个可以参与反应的底物。如图3所示,COS与目的底物5-羧基-2-戊烯酰-辅酶A具有相同的母核结构,但其侧链不同。COS的侧链为巯基,而5-羧基-2-戊烯酰-辅酶A的侧链为5-羧基-2-戊烯酰基。本文使用蛋白质设计工具EvoEF2[26]的配体生成模块将配体COS修改为底物5-carboxy-2-pentenoyl-CoA(图2)。考虑到5-羧基-2-戊烯酰基侧链具有较大的柔性,配体侧链生成过程中对可自由旋转的二面角进行离散(-180°~180°,间隔30°离散),一共生成20 726个底物分子构象。放置过程中,同时计算出每个底物构象的内能及其与蛋白质骨架的范德华排斥能(两者均为正值,越小越好)。接下来对所有构象分别按照2种能量由低到高排序,共有1 728个构象,在2种能量排序中都处于前25%。最后,按照适当的均方根距离偏差(root mean squared error,RMSD,单位为Å)对1 728个构象进行筛选去重。本文采用的RMSD阈值为0.2 Å,筛选后得到48个底物分子构象;其中与酶骨架排斥能最小的分子如图2所示。

图2 5-羧基-2-戊烯酰-辅酶A还原酶的模型Fig.2 Structure model of 5-carboxy-2-pentenoyl-CoA reductase in complex with FAD and 5-carboxy-2-pentenoyl-CoA注:蓝绿色为5-羧基-2-戊烯酰-辅酶A还原酶,粉红色球棍模型为FAD配体,浅蓝色球棍模型为5-羧基-2-戊烯酰-辅酶A,绿色棍状模型为结合残基

图3 三种辅酶A配体的比较Fig.3 Comparison of three CoA ligands注:过硫化辅酶A(CoA persulfide,COS)为PDB结构4L1F中的配体;乙酰辅酶A(acetyl-CoA)为推测的天然底物;5-羧基-2-戊烯酰-辅酶A(5-carboxy-2-pentenoyl-CoA)为目的底物
同源建模过程中搜索到的结构模板绝大部分被注释为乙酰辅酶A脱氢酶(acyl-CoA dehydrogenase)。其中与5-羧基-2-戊烯酰-辅酶A还原酶序列最相近为Mycobacteriumthermoresistibile乙酰辅酶A脱氢酶(PDB ID:3PFD)[27];两者的序列相同性达到73.35%。据此推断,T.fusca5-羧基-2-戊烯酰-辅酶A还原酶的天然底物也可能为乙酰辅酶A或其类似物。从图3可以看出,目的底物5-羧基-2-戊烯酰-辅酶A与乙酰辅酶A的侧链有较大差异,5-羧基-2-戊烯酰基较长,有更强的柔性,且包含强极性的羧基。从图2可知,侧链附近的结合残基大多数为疏水残基。因此,可推测这些疏水基团不能与5-羧基-2-戊烯酰基侧链很好地结合,从而导致酶催化5-羧基-2-戊烯酰-辅酶A反应的活性较低。
2.2 计算设计改造5-羧基-2-戊烯酰-辅酶A还原酶
基于上述分析,本研究选择与5-羧基-2-戊烯酰-辅酶A侧链直接接触(5Å范围内)的氨基酸残基作为设计位点。这样的残基共有15个,分别为Thr 161、Ile 90、Leu 89、Ser 88、Pro 91、Ala 92、Val 93、Lys 95、Leu 96、Ile 250、Gln 253、Thr 249、Tyr 367、Thr 246和Glu 368(图4)。其中Glu 368为催化残基,设计过程中不改变其残基类型。此外,本文选择距离5-羧基-2-戊烯酰基7Å范围内的其他氨基酸残基作为侧链重新安装的位点(未显示);这些残基的侧链构象可改变,但氨基酸类型不变。从图4可以看出,14个设计位点中只有Ser 88、Lys 95、Thr 161、Thr 246、Thr 249 和 Gln 253为亲水性残基,其余9个位点为疏水性残基,并且所有残基均未能与5-羧基-2-戊烯酰基侧链的羧基官能团形成氢键或盐桥。

图4 5-羧基-2-戊烯酰-辅酶A侧链5Å范围内的氨基酸残基Fig.4 Amino acids within 5Å to the side chain of 5-carboxy-2-pentenoyl-CoA
鉴于5-羧基-2-戊烯酰-辅酶A与乙酰辅酶A的差异(图3),本研究尝试通过计算设计引入氢键网络来稳定极性的5-羧基-2-戊烯酰基。设计过程通过计算蛋白质设计工具EvoEF2[26, 28]完成。每个蛋白质设计位点均可在20种天然氨基酸类型中自由选择,每个氨基酸的侧链构象取自SHAPOVALOV等[29]开发的蛋白质骨架依赖旋转异构体库(backbone-dependent rotamer library);而5-羧基-2-戊烯酰-辅酶A配体的空间结构从前面生成的48个构象中自由选择。计算酶设计的过程相当于求解组合优化问题,即从每个位点(包括配体分子)选择一种残基类型(或构象),使得酶设计体系的整体能量达到最低。由于需要搜索的构象空间巨大,EvoEF2采用模拟退火优化算法来搜索低能量的氨基酸序列,但并不一定保证能取得全局能量最低序列。由于设计过程存在一定随机性,每次设计的低能量序列可能并不完全相同,因此可以通过多次独立运行EvoEF2计算程序来产生许多备选序列进行分析。EvoEF2自动输出每个设计的结构和体系能量(单位为EvoEF2 energy unit,简记为EEU)。
如表1所示,本研究生成10个不同的设计(记为Des0~Des9)作为对照,对天然序列(WT)的设计位点的侧链构象进行重新安装,并计算其体系能量。不同设计的能量值存在一定差异,说明EvoEF2每次优化求解的过程可能收敛于不同的解,但能量差异并不大,说明EvoEF2优化算法收敛较好。10个设计的体系能量(-1462.58~-1452.97 EEU)明显低于WT的能量(-1247.53 EEU)。这说明天然酶与底物5-羧基-2-戊烯酰-辅酶A的结合作用可能较弱,而通过设计产生突变则可能引入一些有利的结合作用力。从表1来看,除了92、95和246位点十分保守(始终选择对应的WT氨基酸类型)之外,其余位点都是可变的(可能选择非WT氨基酸类型)。

表1 基于计算改造5-羧基-2-戊烯酰-辅酶A还原酶产生的10个设计Table 1 Ten designs for computational engineering the 5-carboxy-2-pentenoyl-CoA reductase
仔细分析设计的结构发现Des0、Des3、Des4和Des9这4个设计中底物分子的羧基可与氨基酸残基形成氢键(或盐桥)网络。如图5所示,Des0中形成了如下氢键:R253的胍基和Q250侧链的胺基可与配体羧基的其中一个氧原子分别形成距离为3.0Å和2.8Å的盐桥或氢键,S89的羟基可与R253的胍基形成距离为2.6Å的氢键,并且R253胍基可与T364(非设计位点)的羟基形成一个较弱的氢键(3.7Å)。Des9中引入的氢键网络与Des0类似;形成氢键网络的氨基酸残基完全相同,配体5-carboxy-2-pentenoyl-CoA的羧基侧链构象稍有不同。Des3和Des4中,位点89选择为苏氨酸(T);与Des0和Des9相比,Des3和Des4中的T89取代S89与R253的胍基形成氢键。

图5 Des0设计中的氢键网络Fig.5 Hydrogen-bonding network in Des0
2.3 基于分子动力学模拟检验设计的氢键网络
计算酶设计过程中假定蛋白质骨架固定不变,但在实际酶反应过程中,酶、配体及其相互作用都可动态变化。本研究通过分子动力学模拟来检验设计的氢键在动态过程中能否稳定存在,以此来评估设计的优劣。统计分析设计的氢键在整个动力学模拟过程中的变化,氢键具有方向性,统计氢键一般考虑3个参数:XHY键角α,HY距离d1和XY距离d2(图6)。在一定范围内,α越大、d1和d2越小氢键越强。通常认为α>130°,d1<2.2Å,d2<3.2Å可算作稳定的氢键[24-25]。

图6 氢键及相关几何参数图示Fig.6 Schematic of hydrogen bond and related geometric parameters
对分子动力学模拟过程进行统计分析表明,Des0设计中构建的氢键网络较稳定。Des0中引入的4个氢键统计结果如图7所示。

a~d-不同氢键的分析结果:第一列描述所要分析的氢键,第二列描述氢键距离随模拟时间的变化情况,第三列描述氢键距离与角度的关系图7 Des0设计中引入的氢键在分子动力学模拟过程中的统计结果Fig.7 Statistical result of hydrogen bonds introduced in Des0 during 16 ns MD simulation
在16 ns的模拟过程中,5-carboxy-2-pentenoyl-CoA羧基的其中一个氧原子可以与R253的胍基以及Q250的胺基形成稳定的氢键(图7-a和b,d1平均值约为2Å,α平均值约为160°)。相较之下,S89的羟基与R253的胍基之间的氢键较弱,d1平均值约为3Å,α平均值约为110°(图7-c)。这可能是因为T364的羟基可竞争性地与R253的胍基形成较强的氢键(图7-d,d1平均值约为2Å,α平均值约为170°)。从图5来看,Des0设计中R253可与S89形成的氢键较强,而与T364的氢键较弱。在动力学模拟过程中,这2个氢键的强弱虽然发生了转换,但R253至少可与S89和T364其中之一形成较强的氢键。因此,分子动力学模拟结果表明部分设计(如Des0)中的氢键网络可以起到稳定5-羧基-2-戊烯酰-辅酶A柔性侧链的作用。这些设计可用于进一步实验验证。
3 结论
菌株ThermobifidafuscaB6中的野生型5-羰基-2-戊烯酰基-辅酶A还原酶(5-carboxy-2-pentenoyl-CoA reductase)为己二酸生物合成途径中的决速酶。其主要原因是目标底物5-羰基-2-戊烯酰基-辅酶A与其天然底物乙酰辅酶A相比柔性更大、极性更强。本文尝试采用基于结构的计算酶设计方法对野生型5-羰基-2-戊烯酰基-辅酶A还原酶的结合位点进行改造,试图通过设计引入氢键网络来更好地结合5-羰基-2-戊烯酰基-辅酶A的极性侧链,目的是降低酶促反应的活化能以提高催化效率。为检验设计的合理性,本研究通过分子动力学模拟来观察10个设计中氢键网络的变化情况,结果发现Des0设计中的氢键网络很稳定,与野生型酶相比,可增强与5-羰基-2-戊烯酰基-辅酶A的结合作用。因此,Des0突变可用于进一步实验验证。
