海藻糖合成酶结构及其抑制剂的研究进展
2021-04-06蒋志洋王金娥张婧瑜邓鸣飞黄家兴段红霞
蒋志洋, 韩 清, 王金娥, 朱 凯, 张婧瑜, 邓鸣飞, 黄家兴, 段红霞
(中国农业大学 理学院 应用化学系 农药创新中心,北京 100193)
海藻糖是由两个分子葡萄糖通过α-1,1 糖苷键相连的非还原性二糖[1],其广泛分布于昆虫、细菌、真菌以及植物等众多生物体内,发挥着重要的生物学功能。经研究表明,海藻糖不仅可作为储能物质参与生物体的能量代谢[2-3],亦可作为结构物质参与昆虫体壁和真菌细胞壁的形成过程[4-5],还可作为“毒力因子”决定病原菌的致病性[6-7],以及作为化学信号物质调控昆虫的生长与发育[8-9]。
海藻糖合成酶是生物体内参与海藻糖合成的重要功能酶。在昆虫体内,海藻糖-6-磷酸合成酶(trehalose-6-phosphate synthetase, TPS) 和海藻糖-6-磷酸磷酸酯酶 (trehalose-6-phosphate phosphatase,TPP) 是海藻糖合成酶的两个独立保守结构域;在细菌体内,分别对应为OtsA 和OtsB[10];在真菌酵母体内,海藻糖合成酶中的两个催化亚基TPS1和TPS2 分别执行TPS 和TPP 的功能[2]。目前,在不同的生物体内已有5 条海藻糖合成途径被阐明,分别为:海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶 (TPS-TPP/OtsA-OtsB/TPS1-TPS2) 途径、麦芽低聚糖海藻糖合成酶-麦芽低聚糖海藻糖水解酶 (TreY-TreZ) 途径、海藻糖合酶 (TreS) 途径、海藻糖磷酸化酶 (TreP) 途径及海藻糖糖基转移酶 (TreT) 途径[11-12]。其中海藻糖-6-磷酸磷酸合成酶和海藻糖-6-磷酸磷酸酯酶途径是普遍被认可的途径,该途径中海藻糖-6-磷酸合成酶是其限速关键酶。其海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶途径催化海藻糖生物合成的具体过程(图1) 为:核苷酸葡萄糖 (NDPG) 和葡萄糖-6-磷酸(G6P) 在海藻糖-6-磷酸合成酶的催化下首先合成6-磷酸海藻糖 (T6P),随后,由海藻糖-6-磷酸磷酸酯酶催化T6P 去磷酸化,最终生成海藻糖[13-15]。
有研究表明,海藻糖代谢通路在昆虫和植物病原真菌的重要生理过程中存在着调控机制,如昆虫海藻糖代谢可调控其几丁质代谢通路。几丁质是天然的线性糖胺聚糖[16],其作为昆虫体壁的主要成分,在昆虫生长发育过程中的旧表皮蜕去和新表皮形成中发挥着重要调控作用[17]。相关研究显示,昆虫海藻糖代谢路径位于其几丁质代谢通路上游,二者通过海藻糖酶相互偶联 (图2),即海藻糖在海藻糖酶催化下分解为葡萄糖,随后,在己糖激酶催化下生成G6P,G6P 焦磷酸化生成UDP-N-乙酰氨基葡萄糖,随即进入几丁质生物合成通路,即UDP-N-乙酰氨基葡萄糖经几丁质合成酶作用生成几丁质,最终经几丁质酶和β-N-乙酰己糖胺酶等作用,完成几丁质降解,生成N-乙酰氨基葡萄糖[18]。海藻糖作为沟通这两个通路的重要物质,其代谢过程可调控几丁质的合成与代谢通路,从而影响昆虫生长发育[19-20]。此外,海藻糖代谢还可调控植物病原真菌侵染过程。在对稻瘟病菌 Magnaporthe oryzae 侵染力调控中,存在着依赖于还原型烟酰胺腺嘌呤二核苷酸磷酸 (NADPH)的开关机制,该开关可被稻瘟病菌TPS1 调控。TPS1 缺失的稻瘟病菌其附着胞和侵染钉产生受阻,使其无法正常侵染[21-23]。有报道指出,真菌细胞壁的形成、菌丝的生长与其体内几丁质酶存在着密切联系,几丁质酶异常可以导致植物病菌生长受阻、其致病能力下降[24-25]。因此,笔者推测在真菌中海藻糖酶也可能与几丁质酶代谢相关,但尚未见相关文献报道。
海藻糖代谢途径凭借着其重要的生物学功能及其在哺乳动物体内缺失的优势[26],在新型杀虫剂、杀菌剂开发领域受到越来越广泛关注。例如,以海藻糖酶为靶标的杀菌剂井冈霉素A (validamycin A) 已成功上市,且对水稻纹枯病、棉花猝倒病等具有良好的防效[27-28]。生物体内参与海藻糖合成的重要功能酶,如海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶也陆续被认为是新型杀虫剂、杀菌剂研发的候选靶标[10,29],尤其是近年来有关海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶的结构生物学研究陆续取得较大进展,这为基于海藻糖相关酶靶标结构进行新农药分子合理设计,进而开发绿色安全的新农药品种奠定了重要生物学基础。但迄今为止,尚未见有关海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶晶体结构及其抑制剂的相关综述报道。鉴于此,本文主要综述近年来关于海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶的晶体结构、底物结合模式及其酶抑制剂的研究进展,以期为以海藻糖合成酶为靶标的先导化合物的发现和新农药活性分子合理设计提供指导和借鉴。
1 海藻糖合成酶晶体结构
1.1 海藻糖-6-磷酸合成酶晶体结构及其与底物结合模式分析
糖基转移酶 (GTs) 是生物体内参与糖代谢的重要功能酶,负责多糖及其他糖类复合物的合成[30]。截至2020 年10 月,碳水化合物活性酶数据库中(CAZy, http://www.cazy.org) 根据酶一级结构 (氨基酸序列) 保守性原则[31],将已知序列的GTs 划分为111 个家族,即GT1~GT111。此外,根据GTs高级结构相似性,又将其划分为GT-A、GT-B、GT-C 和GT-D 共4 个超家族[32-35]。通常大部分GTs 在形成三维结构时均采取GT-A 和GT-B 折叠型 (图3)。这两种折叠方式都可形成两个典型的Rossmann 结构域 (包含β/α/β 型超二级结构),其差异主要体现在GT-A 折叠形成的两个Rossmann结构域是彼此相邻的,且联系紧密;而GT-B 折叠形成的两个相似的Rossmann 结构域往往更加独立,联系也更为灵活,且在两结构域交界处常存在着缝隙[36-37]。
海藻糖-6-磷酸合成酶是GTs 中的重要成员。研究发现,TPS/OtsA 属于GT20 家族,且采取GTB 折叠型,其催化活性位点存在于由两个Rossmann结构域形成的缝隙中[36]。海藻糖-6-磷酸合成酶在海藻糖生物合成过程中负责将糖基供体底物NDPG的葡萄糖环部分转移至糖基受体底物G6P 的葡萄己糖环上,并催化二者间α-1,1 糖苷键的形成。随着蛋白质结晶学、X-射线晶体衍射以及核磁共振等技术日渐成熟,越来越多物种的海藻糖-6-磷酸合成酶晶体结构被解析。截至目前,真菌TPS1 和细菌OtsA 晶体结构均已有许多报道,但尚未见昆虫TPS 晶体结构的报道。
1.1.1 细菌OtsA 晶体结构 目前,已有6 种细菌的17 个OtsA 蛋白晶体结构被解析,如表1 所示。其中大部分为人类病原细菌的OtsA 晶体,如大肠杆菌Escherichia coli 和耐热分枝杆菌Mycobacterium thermoresistibile 的OtsA 晶体,分别简称为 EcOtsA和MtrOtsA 等,但尚未见植物病原细菌OtsA 晶体结构的报道。

表1 目前已报道的不同细菌OtsA 的晶体结构信息Table1 The reported crystal structures of different bacteria OtsAs
Gibson 等[14]于2002 年报道了首个海藻糖-6-磷酸合成酶三维晶体结构,即大肠杆菌OtsA 与小分子G6P、尿苷二磷酸 (UDP) 的复合物蛋白 (PDB号:1GZ5)。该研究指出:EcOtsA以四聚体形式存在,组成四聚体的单体结构之间是高度相似的,每个单体都包含有两个Rossmann结构域,即N-端结构域和C-端结构域,在两个结构域界面处有一明显裂缝,OtsA 的催化中心就位于这一裂缝中。其N-端结构域核心是由7 条平行的β 链形成的三维结构,其核心两侧各有一条反向平行的β 链及6 个α 螺旋,而其C-端结构域则是由6 条β 链形成的核心和外侧的7 个α 螺旋构成的。
Mendes 等[40]以EcOtsA 为模型,解析了耐热分枝杆菌MtrOtsA 的四聚体结构 (PDB 号:5JIJ),其单体拓扑结构与EcOtsA 较相似,即其N-端结构域核心是由7 条平行的β 链及两侧反平行的β 链构成,该核心区域被外侧8 个α 螺旋所包围,而其C-端结构域则包含6 个平行β 链和9 个α 螺旋。位于C-端结构域最末端的α 螺旋可延伸至N-端结构域,并在这两个结构域界面处产生纽结,这是GT-B 型折叠糖基转移酶的普遍结构特征。然而,该研究也指出:虽然MtrOtsA 与EcOtsA 蛋白单体三维结构显示出较高的相似性,但其相应序列比对结果显示,与其他非分枝杆菌物种相比,MtrOtsA 蛋白四聚体界面处氨基酸残基的保守性较低,而在分枝杆菌及其他近缘物种中,该位置残基则呈现出高度保守性,这可能是由于不同物种海藻糖-6-磷酸合成酶在形成高级结构时,多肽链空间折叠方式存在着细微差异导致的。
1.1.2 真菌TPS1 晶体结构 与细菌OtsA 相比,被报道的真菌TPS1 晶体数量较少,截至目前,仅有3 种真菌的9 个TPS1 晶体被解析 (表2)。值得关注的是,Wang 等[41]于2019 年报道了首个农业病害微生物稻瘟病菌Magnaporthe oryzae MoTPS1的晶体结构,这为基于稻瘟病菌MoTPS1 结构进行新型农用杀菌剂合理化设计奠定了生物学基础。
Miao 等[42]通过对已揭示三维结构的真菌TPS1 与细菌OtsA 多肽链的空间折叠方式进行比对发现:真菌TPS1 与细菌OtsA 高级结构总体相似度较高,即真菌TPS1 的C-端Rossmann 结构域内部也是由若干个平行β 链组成的β 折叠片构成的,其外侧则被数个长短不一的α 螺旋包围着,最末端α 螺旋也可跨越至N-端结构域继续形成α 螺旋,并在两结构域界面处产生纽结。如人类病原真菌白色念珠菌Candida albicans 的CaTPS1单体,C-端结构域就是由6 条β 链和8 个α 螺旋构成的,其α16 螺旋在氨基酸残基Y457 处产生纽结,而稻瘟病菌MoTPS1 单体的C-端结构域则包含6 条β 链和10 个α 螺旋,其α18 螺旋于G252、T464 处产生纽结[41]。
当然,真菌TPS1 和细菌OtsA 三维结构也存在着显著差异:如真菌TPS1 的N-端结构域表现为非典型Rossmann 结构域。有研究指出,CaTPS1单体N-端结构域中β1 链与一组伸出催化中心的反向平行β 折叠片 (β2-β3) 相连,而并非α 螺旋;同时该研究显示,烟曲霉Aspergillus fumigatus AfTPS1B 单体也展现出同样结构特点,而另一个AfTPS1A 单体中却未观察到该类似结构特征[42]。Wang 等[41]的研究同样表明,MoTPS1 单体的N-端结构域中也存在着由β2 链和β3 链反向折叠形成的β-发夹结构,由于在细菌OtsA 中尚未观察到类似发夹结构,因而推测该结构可能仅特异性地存在于真菌TPS1 中,并可作为区分真菌TPS1 和细菌OtsA 的显著特征之一。
1.1.3 海藻糖-6-磷酸合成酶与糖基供体底物NDPG 的结合模式分析 在海藻糖-6-磷酸合成酶催化过程中,NDPG 发挥着糖基供体的作用,其可以与海藻糖-6-磷酸合成酶C-端结构域活性位点的氨基酸残基结合 (图4)。生物体内共存在5 种NDPG,分别为:腺苷二磷酸葡萄糖 (APDG)、尿苷二磷酸葡萄糖 (UPDG)、鸟苷二磷酸葡萄糖(GDPG)、胞苷二磷酸葡萄糖 (CDPG) 和胸苷二磷酸葡萄糖 (TDPG)。研究表明,大多数生物海藻糖-6-磷酸合成酶是以UDPG 为底物的,但少数物种酶对底物也存在着其他偏好,如MtrOtsA 和委内瑞拉链霉菌Streptomyces venezuelae SvOtsA 就是分别以ADPG 和GDPG 为最适供体底物的[39-40,43]。
分析已被解析的TPS1 蛋白结构,发现在3 种真菌TPS1——CaTPS1、AfTPS1 及MoTPS1 与底物NDPG 复合物晶体中,NDPG 葡萄糖环部分电子密度极低,以致于其结构不可见,这可能是由于TPS1 中NDPG 葡萄糖结合腔较大,柔性过高导致的[41-42]。除上述3 种真菌外,在其他细菌OtsA与NDPG 结合的二元复合物晶体中均可见葡萄糖环,且葡萄糖环与OtsA 结合的关键氨基酸残基在不同生物体内是高度保守的,二者间分子互作模式也非常相似。如:大肠杆菌中,EcOtsA 以His154、Gln185、Asp361、Met363 和Asn364 等保守的氨基酸残基与UDPG 的葡萄糖环通过氢键产生结合作用[14],在耐热分枝杆菌MtrOtsA 中也可观察到NDPG 的葡萄糖基与该酶活性中心的His168、Asp385、Gly386、Met387 及Asn388 等残基形成氢键作用[40]。
NDPG 核糖部分及两个磷酸根与OtsA/TPS1催化中心结合也是十分保守的,在不同物种海藻糖-6-磷酸合成酶晶体结构中均可以观察到:NDPG 五元核糖环的羟基与该位点谷氨酸残基以氢键产生相互作用;NDPG 两个磷酸根结合于OtsA/TPS1 活性中心的正电结合腔中,该正电中心由一组保守的Lys-Arg 残基对构成;此外,也可观察到NDPG 磷酸根的羟基与该位点的亮氨酸残基通过其主链氨基形成氢键相互作用。
然而,NDPG 碱基部分与海藻糖-6-磷酸合成酶的结合却存在着一定物种间差异,如EcOtsA的Phe339 残基可与UDPG 尿嘧啶的O4 和N3 分别形成氢键作用力[14],而在白色念珠菌和烟曲霉菌体内,UDPG 的尿嘧啶可分别与二者TPS1 中的Ile357 或Val364 残基形成氢键[42]。Mendes 等[40]研究指出,MtrOtsA 在行使催化功能时,是不以UDPG 为糖基供体底物的,而对ADPG 存在明显偏好,因此,该研究团队分别解析了MtrOtsA 与ADPG 或GDPG 结合的二元复合物晶体,对比分析二者结构发现:ADPG 与MtrOtsA 氨基酸残基Val363 和Leu319 均可形成氢键,而GDPG 仅可与MtrOtsA 的Val363 残基形成氢键,这表明与GDPG 相比,APDG 可与MtrOtsA 形成更加紧密的相互作用,这为MtrOtsA 的底物偏好性提供了有力的结构生物学证据。此外,该研究还通过关键氨基酸残基突变等研究证明:MtrOtsA 对催化底物的偏好性主要是由Leu319、Val363 和Glu367 这3 个残基介导的。可见,不同物种海藻糖-6-磷酸合成酶的NDPG 结合口袋大致是保守的,仅在与碱基结合位点处表现出细微差异,这可能也解释了不同物种海藻糖-6-磷酸合成酶对供体底物存在着不同偏好的原因。
1.1.4 海藻糖-6-磷酸合成酶与糖基受体底物G6P 的结合模式分析 海藻糖-6-磷酸合成酶的糖基受体底物G6P 大部分结合于该酶N-端Rossmann结构域,可以通过氢键作用力、疏水作用力、静电作用力等与该酶催化活性中心的氨基酸残基产生相互作用 (图5)。
研究发现,海藻糖-6-磷酸合成酶N-端结构域内部存在着一个正电结合口袋,底物G6P 的磷酸酯基可通过静电相互作用与这一口袋产生结合。如EcOtsA 中这一正电口袋是由Arg9 和Arg300 两个残基形成的[14],分别对应着MtrOtsA 中Arg18和Arg324 以及MoTPS1 中Arg22 和Arg327[40-41]组成的各自正电结合口袋。由此可以看出:参与该静电特征结合口袋形成的氨基酸残基在不同生物体内是具有高度保守性的。除上述静电相互作用外,G6P 磷酸酯基还可以与海藻糖-6-磷酸合成酶N-末端保守的酪氨酸残基以氢键产生相互作用,进一步增强了底物与酶活性中心的亲和力。
受体底物G6P 的葡萄糖基被由若干个氨基酸残基形成的疏水口袋包围,同时也可通过形成氢键作用与海藻糖-6-磷酸合成酶活性中心的氨基酸残基产生相互作用。经比对分析发现:与G6P 葡萄糖基产生相互作用的氨基酸残基虽然表现出细微的物种特异性,但大部分氨基酸残基在不同物种间仍是相对保守的。Mendes 等[40]通过氨基酸序列同源性分析发现:MtrOtsA 中与G6P 葡萄糖基结合的关键残基Asp144 等在真菌TPS1 和细菌OtsA 中也是保守的,而残基Gln146 仅在耐热分枝杆菌及其近缘物种中是保守的,该残基对应其他物种海藻糖-6-磷酸合成酶中保守的组氨酸残基。Miao 等[42]也证实,CaTPS1 中与G6P 葡萄糖基相互作用的氨基酸残基Asp143 和His145 等在物种间也是高度保守的。
分析与G6P 结合的海藻糖-6-磷酸合成酶三维空间结构,除可得到“配体-受体”相互作用模式外,还可以为进一步阐明酶催化机制提供科学性证据。目前,已有糖基转移酶两种催化机制被阐明,分别为保留催化机制和反转催化机制。海藻糖-6-磷酸合成酶则采取保留催化机制,即在催化葡萄糖基转移过程中不发生异头碳原子构型的改变,且该催化机制在不同生物体内也是相当保守的[30,37]。Gibson 等[14]通过解析以G6P 和UDP 为配体的EcOtsA 三维结构发现:EcOtsA 内部包围G6P 葡萄糖基部分的疏水结合口袋中存在着两个保守氨基酸残基:Trp85 和Ile155,二者形成的疏水环境仅可使呈α-构型的葡萄糖O1 羟基处于某一特定位置,以便于其亲核攻击相应的供体葡萄糖基,最终只能形成α,α-1,1 糖苷键。该结论首次以海藻糖-6-磷酸合成酶三维结构为依据佐证了海藻糖-6-磷酸合成酶是采取保留催化机制的观点。此外,Gibson 等[14,36]还研究报道了在EcOtsA的N-端结构域存在着保守残基Trp40,该残基可能是发挥着G6P 进出蛋白活性空腔的关键。即当G6P 进入EcOtsA 活性中心后,其关闭该位点,而催化完成后,则开启该位点以释放产物。Wang 等[41]通过叠合分析MoTPS1 单体、二元复合物 (MoTPS1+UDPG) 和三元复合物 (MoTPS1+T6P+UDP) 结构,进一步对G6P 进出海藻糖-6-磷酸合成酶的门控机制进行了阐释。该研究指出,MoTPS1 单体与其二元复合物结构类似,但相比于单体和二元复合物,其三元复合物构象却更为紧凑,基于此提出了海藻糖-6-磷酸合成酶催化“开-关”模型,即:UDPG 首先进入TPS1 催化中心并与之结合,但这一结合并未改变TPS1 分子整体构象,也就是此时TPS1 仍呈现“开放”状态;当G6P 随后进入TPS1 并结合至其位于N-末端催化中心时,会导致TPS1 构象发生改变,随即转入“关闭”状态,在此状态下G6P 与UDPG 相互靠近,并完成糖基转移的亲核反应;反应完成后,TPS1 构象又发生改变再度呈现为“开放”状态,分别释放反应产物T6P 和UDP,完成这一催化反应。此外,分析CaTPS1 和MtrOtsA 等多元复合物三维结构也发现了与MoTPS1 类似的构象变换特点,这进一步支持了海藻糖-6-磷酸合成酶在催化时存在门控“开-关”机制的假设。
综上所述,不同物种海藻糖-6-磷酸合成酶的三维结构虽略有差异,但总体结构仍具有较高相似性,通过对不同生物海藻糖-6-磷酸合成酶蛋白序列进行比对 (图6) 发现:该酶底物结合位点的关键氨基酸残基同源性较高,表明酶活性中心底物结合口袋构象相对保守,这为基于海藻糖-6-磷酸合成酶设计广谱性新农药提供了重要依据。此外,海藻糖-6-磷酸合成酶与底物结合的关键氨基酸残基和关键作用力的解析以及酶催化机制的阐明也为基于靶标结构的新农药分子设计开拓了新思路。
1.2 海藻糖-6-磷酸磷酸酯酶晶体结构及其与底物结合模式分析
除海藻糖-6-磷酸合成酶外,海藻糖-6-磷酸磷酸酯酶作为海藻糖合成酶中另一个重要功能域,其位于海藻糖合成途径下游,主要负责使T6P 去磷酸化最终生成海藻糖 (图1)。在生物体内,磷酸酯酶依赖镁离子催化磷酸根水解反应,是生物化学反应和能量代谢的重要参与者。生物体内磷酸酯酶种类众多,但其在高级结构上却表现出较高相似性。因此,学者们将具有类似结构的磷酸酯酶划分为卤酸脱卤酶 (HAD) 超家族,这些酶均具有两个结构域,其中核心结构域呈典型的Rossmann折叠,另一个结构域为帽型,这两个结构域在不同生物体内是高度保守的,但相比较帽型结构域大小往往会存在一定差异[44]。
本文介绍的海藻糖-6-磷酸磷酸酯酶就隶属于HAD 超家族,目前已有近10 种生物的海藻糖-6-磷酸磷酸酯酶晶体结构已陆续被解析 (表3),这些蛋白晶体结构均具有HAD 超家族典型结构特征。Rao 等[45]于2006 年首次报道了嗜酸热原体菌Thermoplasma acidophilum 海藻糖-6-磷酸磷酸酯酶TaOtsB 晶体结构,其核心结构域是一个经修饰的Rossmann 折叠结构域,它是由8 个β 折叠片层和两侧5 个α 螺旋构成的,其中β4 和β5 在β3 处形成了明显的发夹结构。而其帽型结构域包含4 个β 折叠片和2 个α 螺旋,主要是通过两条蛋白铰链与其核心结构域相连的,TaOtsB 活性中心主要位于Rossmann 折叠结构域和帽结构域之间界面处,由3 个保守氨基酸序列构成。Cross 等[46]报道了铜绿假单胞菌Pseudomonas aeruginosa PaOtsB蛋白结构,通过解析该结构发现:PaOtsB 结构域具有与TaOtsB 几乎完全相同的折叠模式,仅β-发夹处存在着细微差异,其PaOtsB 核心结构域的β-发夹是由β2 链和β3 链折叠而成的。细菌假伯克霍尔德氏菌Burkholderia pseudomallei 的海藻糖-6-磷酸磷酸酯酶BpOtsB 同样也具有HAD 超家族酶保守结构,其核心结构域同样为8β5α 型,其β4链与其他β 折叠片反平行,并无类似于TaOtsB中的发夹结构,其C2B 帽型结构域同样是由4 个β 折叠片和2 个α 螺旋构成的[47]。
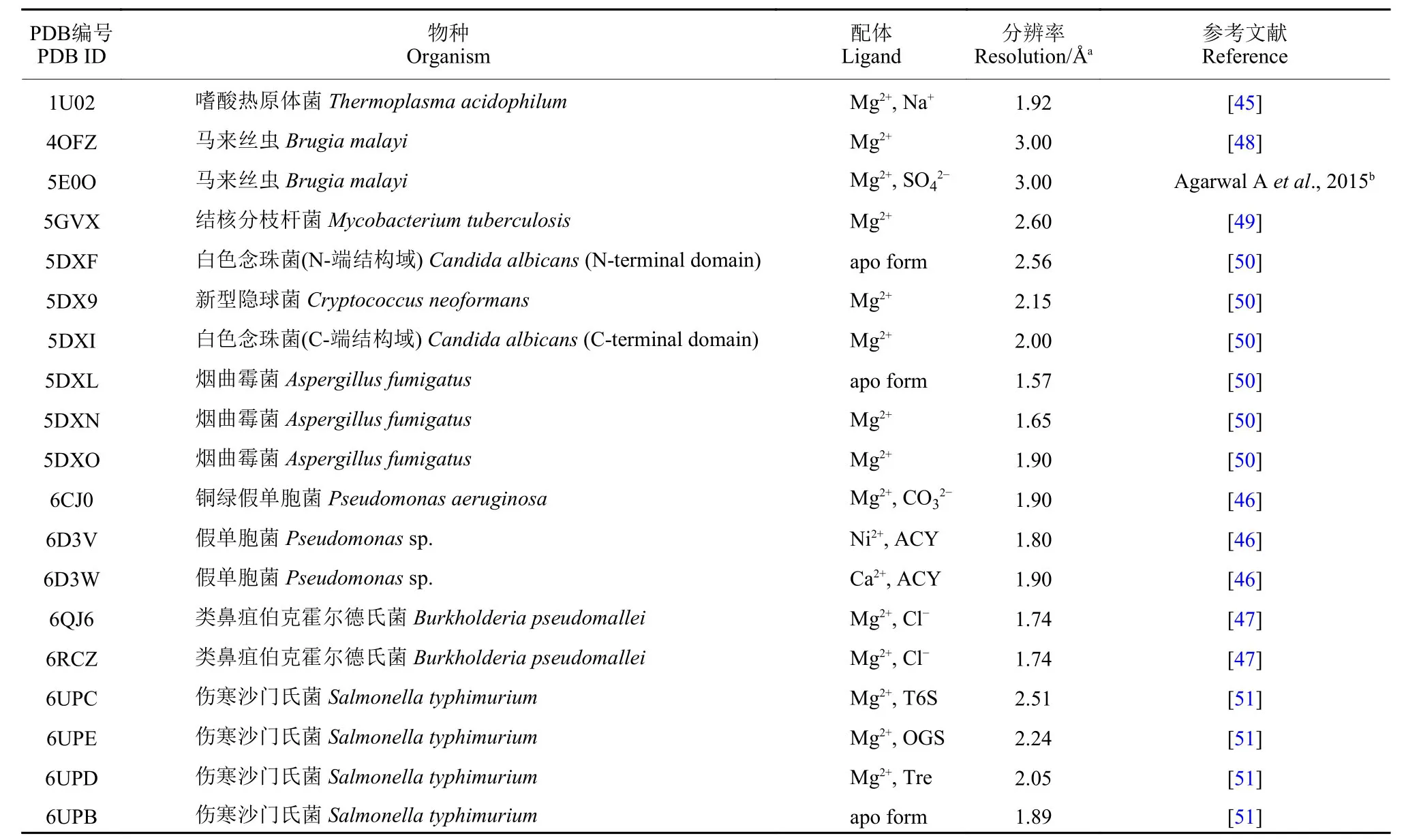
表3 目前已报道的不同海藻糖-6-磷酸磷酸酯酶晶体结构信息Table3 The reported crystal structures of different trehalose-6-phosphate phospholipase
不同生物的海藻糖-6-磷酸磷酸酯酶总体结构是呈现较高相似性的,但往往也存在物种间差异性。如马来丝虫 Brugia malayi TPP (BmTPP) 蛋白除拥有HAD 超家族酶普遍存在的核心结构域和帽型结构域外,还具有一个由3 股螺旋束构成的N-端结构域[48]。来自细菌Mycobacterium tuberculosis 和真菌C. albicans 的海藻糖-6-磷酸磷酸酯酶晶体结构显示:这两个海藻糖-6-磷酸磷酸酯酶空间结构也是由核心结构域、帽型和N-端结构域构成的,在3 个结构域之间通过一段或几段多肽链进行偶联。与马来丝虫BmTPP 结构不同的是,结核分枝杆菌MtbOtsB 和白色念珠菌CaTPS2 的N-端结构域空间折叠方式更为复杂,且均为典型的Rossmann结构域。Miao 等通过结构比对发现,CaTPS2 的N-端结构域与大肠杆菌EcOtsA 结构类似,但却缺少EcOtsA 中与底物结合的关键氨基酸残基,如Arg9、Tyr76 和Arg300 等[49-50]。因此,表明CaTPS2的N-端结构域不具有合成T6P 的功能,推测该结构域或许可与另一亚基CaTPS1 互作,并在TPS1折叠和催化过程中发挥着一定功能,但该假说至今尚未得到实验证实。
不同生物海藻糖-6-磷酸磷酸酯酶的晶体结构也揭示了其相当保守的底物结合模式。Rao 等[45]通过模拟TaOtsB 与其底物T6P 结合,发现Ile16、Thr45、Lys111、His118、Lys149、Glu153 和Lys161是TaOtsB 与T6P 产生互作的关键氨基酸残基,且上述氨基酸残基在不同物种的海藻糖-6-磷酸磷酸酯酶中保守性极高。Harvey 等[51]分别解析了伤寒杆菌Salmonella typhimurium 的StOtsB 与第1 代海藻糖-6-磷酸磷酸酯酶抑制剂海藻糖-6-硫酸 (T6S)和1-(4-己基苯氧基)-葡萄糖-6-硫酸 (OGS) 复合蛋白晶体结构,并报道了StOtsB 与底物结合方式,发现StOtsB 中Glu123、Lys125 和Glu167 等残基是与T6S 及OGS 结合的关键残基,且保守度极高。这些关键氨基酸残基的解析,为以海藻糖-6-磷酸磷酸酯酶为靶标的新一代抑制剂合成和先导化合物改造提供了重要依据,也为靶向海藻糖-6-磷酸磷酸酯酶的广谱性农药设计与开发提供了有力参考。
2 海藻糖合成酶抑制剂
在基于靶标开展新农药分子设计与研发过程中,已知靶标酶抑制剂化学结构既可作为其他潜在酶抑制剂筛选的依据,也可为先导化合物结构优化与改造提供思路。截至目前,国内外先后报道了10 余个海藻糖-6-磷酸合成酶抑制剂结构 (表4)和若干个海藻糖-6-磷酸磷酸酯酶抑制剂结构 (表5)。根据这些抑制剂分子发现途径不同,可将其划分为3 种类型:其一,根据靶标酶天然底物结构进行改造得到的底物类似物;其二,靶标酶的负反馈调节抑制剂及其类似物;其三,基于靶标酶结构开展虚拟筛选或高通量筛选获得的酶竞争性结合抑制剂。应当注意的是,虽已有一些海藻糖合成酶抑制剂研究报道,但具有较好农用酶抑制活性的抑制剂却寥寥无几。因此,笔者按照上述分类方式分别对海藻糖合成酶中两个重要功能域——海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶的抑制剂结构和活性进行较为全面综述,期望为靶向这两种酶的农用抑制剂合理设计和生物活性评价提供参考。
2.1 海藻糖-6-磷酸合成酶抑制剂
2.1.1 负反馈抑制剂及其类似物 负反馈抑制是生物体内普遍存在的酶活调控方式,是指酶催化产物作为抑制剂反过来调控酶活性,广义也可指系列反应中下游产物对上游催化酶活性的抑制。通常,在一定范围内,产物浓度与负反馈抑制效果间存在着明显正相关性[52]。
T6P 是海藻糖-6-磷酸合成酶催化产物,其被称为该酶的负反馈抑制剂,且其发挥酶负反馈抑制效应已被阐明。Magalhães 等[52]发现,在体外条件下,T6P 会显著抑制酿酒酵母 Saccharomyces cerevisiae ScTPS1、白色念珠菌CaTPS1 和热带念珠菌Candida tropicalis CtTPS1 活性:当T6P 浓度为125 μmol/L 时,CtTPS1 活性降低约50%,CaTPS1 活性降低约60%。结合酶促动力学分析结果表明:T6P 是海藻糖-6-磷酸合成酶的有效非竞争性抑制剂。此后,有研究者通过同源模建构建了ScTPS1 蛋白模型,并将经能量优化的T6P 分子与该模型进行两次对接。结果显示:T6P 分子结合于ScTPS1 中一固定活性腔内,并与高度保守的氨基酸残基Tyr40、Ala41、Met42 和Phe372 通过氢键产生相互作用[53],进一步揭示了T6P 可作为海藻糖-6-磷酸合成酶抑制剂用于新药研发,但T6P 分子由于其磷酸基团的存在,使其不容易穿越细胞膜被生物体吸收。
海藻糖是生物体内海藻糖合成代谢的终产物,经相关研究表明,在海藻糖-6-磷酸合成酶活性负反馈调控中,海藻糖也发挥了重要功能。约半世纪前,Murphy 等首次报道了在蚕蛾体内海藻糖对其TPS 活性存在明显负反馈调控功能[54],但此后就鲜有关于海藻糖参与调控海藻糖-6-磷酸合成酶活性的报道。直至2017 年,Oide等[55]通过对离体谷氨酸棒状杆菌Corynebacterium glutamicum CgTPS1 蛋白进行酶促动力学分析发现:对CgTPS1而言,海藻糖是其供体底物UDPG的竞争性抑制剂,是其受体底物G6P 的非竞争性抑制剂,海藻糖对CgTPS1 酶活性负反馈调节占主导地位,而非T6P。Mendes 等[40]也证实:在生理条件下,海藻糖对耐热分枝杆菌MtrOtsA 活性存在一定抑制效果,且其半抑制浓度约为24 mmol/L。
井冈霉素A (validamycin A) 是一种产量大且应用广的微生物源农药,对小麦、水稻纹枯病表现出较好防效,其水解产物井冈羟胺A (validoxylamine A) 作为海藻糖类似物,早已被证实可作为海藻糖酶有效抑制剂[56-57]。然而,近些年研究表明:井冈羟胺A 可作为海藻糖-6-磷酸合成酶的过渡态抑制
剂[38,42],且白色念珠菌CaTPS1 和烟曲霉菌AfTPS1与井冈羟胺A 结合的复合物蛋白结构已被解析[30]。Errey 等[38]基于井冈羟胺A 结构,合成了6′-井冈羟胺A 磷酸 (6′-validoxylamine A phosphate),该化合物对EcOtsA 表现出较强抑制效果,其IC50值约为5.3 mmol/L,且进一步研究发现:在UDP存在条件下,井冈羟胺A-6′-磷酸对EcOtsA 的抑制效应会明显增强。

表4 目前已报道的不同海藻糖-6-磷酸合成酶抑制剂Table4 The reported inhibitors of different trehalose-6-phosphate synthetase

续表 4Table4 (Continued)
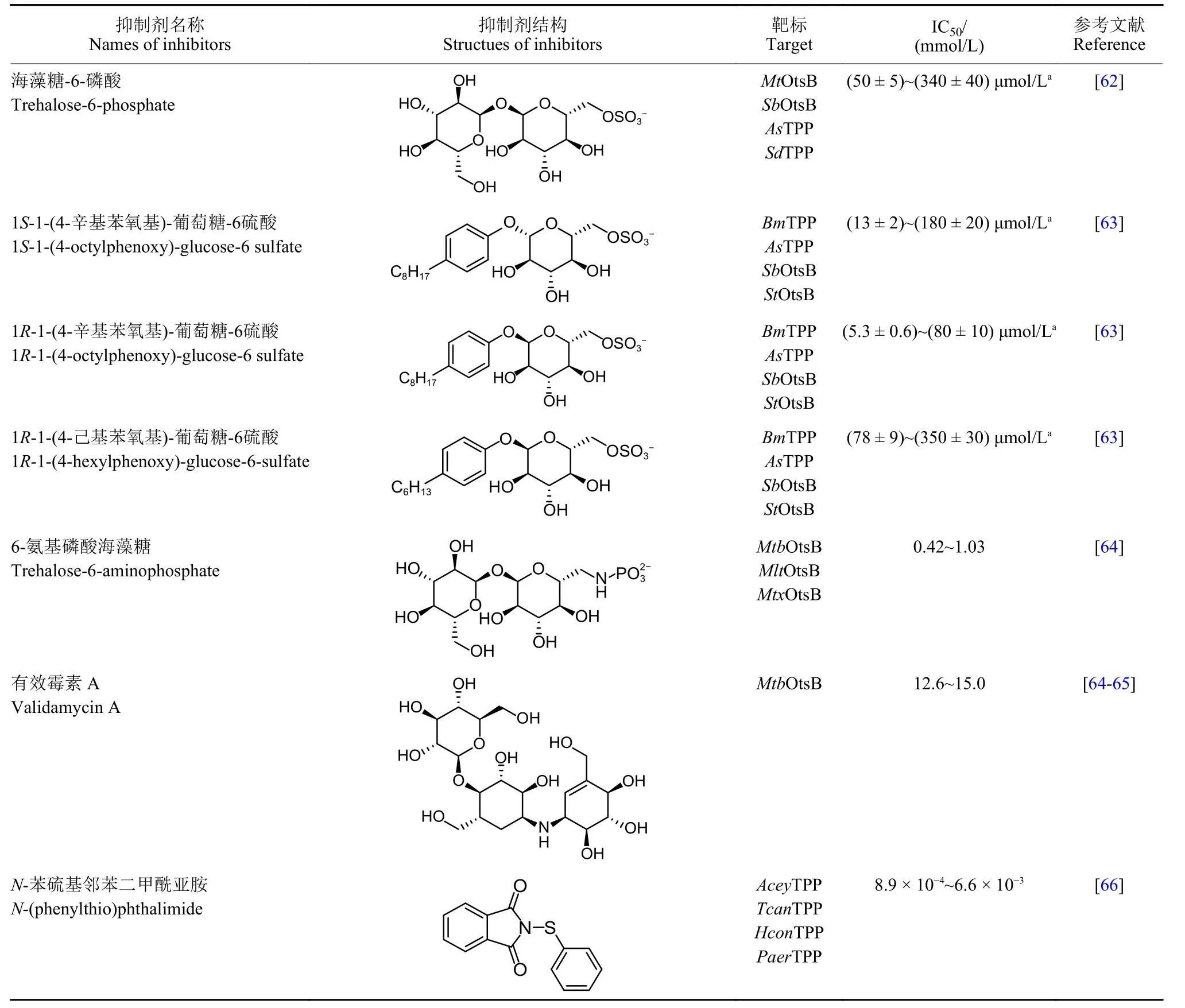
表5 目前已报道的不同海藻糖-6-磷酸磷酸酯酶抑制剂Table5 The reported inhibitors of different trehalose-6-phosphate phosphatase
除上述海藻糖代谢相关产物可对海藻糖-6-磷酸合成酶活性存在负反馈抑制外,与海藻糖代谢密切相关的其他糖代谢中间产物也可对该酶活性产生负反馈抑制效应。Mendes 等[40]通过测试若干糖酵解中间物对MtrOtsA 抑制活性研究发现:2-磷酸甘油酸 (2PG) 和酮戊二酸 (2OG) 可作为别构调节剂显著抑制MtrOtsA 活性,其IC50值分别为2.3 mmol/L 和1.8 mmol/L。
由此看来,双糖类和拟双糖类抑制剂在已报道的海藻糖-6-磷酸合成酶抑制剂中占比最大,且大部分抑制剂具有较出色的酶抑制活性。因此,在未来设计海藻糖-6-磷酸合成酶抑制剂时,可考虑引入糖环或双糖结构,期望有利于新合成化合物的酶抑制效果。同时应注意化合物相关理化性质并对其结构进行合理优化,以提高其在生物体内的吸收和利用率,使其既可达到良好的酶活性抑制效果,又可表现出优异的杀灭有害生物活性。
2.1.2 竞争性结合抑制剂 除生物体内存在的某些天然化合物可作为海藻糖-6-磷酸合成酶活性负反馈抑制剂外,通过应用计算机和分子生物学技术开展海藻糖-6-磷酸合成酶抑制剂的虚拟筛选或高通量筛选也已成为新型糖代谢相关酶抑制剂发现的另一重要途径,且筛选得到的化合物大多为酶的竞争性结合抑制剂。
Xue 等[58]通过同源模建方法构建得到了稻瘟病菌MoTPS1 三维结构模型,通过将该结构模型与MLSMR 化合物数据库中40 万个分子开展批量分子对接,筛选得到了与MoTPS1 亲和力最强的化合物2 478 993,并以其为母体进行结构优化,通过引入羰基得到了先导化合物Lead 25,该化合物既表现出较高的MoTPS1 亲和力 (亲和自由能为-13.8 kcal/mol,即 -57.76 kJ/mol),又具有较强水溶性。此外,作者还发现:与海藻糖-6-磷酸合成酶的天然底物UDPG 和G6P 相比,NADPH 与MoTPS1 具有更强的亲和力 (UDPG 亲和自由能:-10.4 kcal/mol,即 -43.53 kJ/mol;G6P 亲和自由能:-7.0 kcal/mol,即 -29.30 kJ/mol;NADPH 亲和自由能:-11.3 kca/lmol,即 -47.30 kJ/mol),这表明NADPH 也可作为海藻糖-6-磷酸合成酶的潜在抑制剂进行进一步开发。该研究是针对海藻糖-6-磷酸合成酶筛选和开发农用杀菌剂的首次成功试验,这为将来基于MoTPS1 结构开展新抑制剂的发现提供了借鉴和经验。
Verma[59]使用热位移分析法对耐热分枝杆菌MtrOtsA 抑制剂进行了Ⅰ、Ⅱ两种类型的高通量筛选,其中Ⅰ型筛选得到15 个MtrOtsA 潜在抑制剂分子,Ⅱ型筛选得到7 个在腺苷二磷酸 (ADP)存在条件下的MtrOtsA 潜在抑制剂分子,随后分别测定了两类型筛选所得化合物的IC50值。结果显示:5-(2-噻吩基) 烟酸对MtrOtsA 抑制效果最佳,其IC50值约为0.5 mmol/L,在ADP 存在条件下,4-羟基-6-(三氟甲基)-3-喹啉羧酸为抑制活性最强的MtrOtsA 抑制剂,其IC50值约为0.53 mmol/L。值得注意的是:经Ⅰ、Ⅱ两类型筛选得到一共同分子——5-氨基吲哚,该分子以0.65 mmol/L 的IC50值表现出显著的抑制MtrOtsA 活性,且化合物结构简单,改造潜力巨大。因此,在今后针对海藻糖-6-磷酸合成酶抑制剂进行结构优化时,应对5-氨基吲哚这一结构片段给予充分关注。
2017 年,来自杜克大学的Perfect 等[60]利用UDP 荧光偏振技术筛选得到了若干个白色念珠菌CaTPS1 和烟曲霉菌AfTPS1 抑制剂,其中克罗散泰 (Closanthal) 在体外条件下对隐球菌存在着显著抑制效应,其最小抑菌质量浓度为1 mg/L,但在体内条件下,克罗散泰并没有发挥良好的抑菌效果,这或许与药物在生物体内复杂的吸收和代谢机制有关。
Kern 等[61]通过克隆并表达黑腹果蝇Drosophila melanogaster 和猫蚤TPS 基因进行了首次靶向昆虫TPS 的抑制剂高通量筛选尝试,并得到了一系列对黑腹果蝇DmTPS 和猫蚤CfTPS有显著抑制效果的化合物,结构如表4 所示。其中2,6-二氨基-4-(3,4-二氯苯基)-3,5-二氰基-4-H-噻喃对DmTPS的抑制效果最佳,其IC50值仅为0.2 mmol/L,且以该结构为骨架合成的若干化合物对DmTPS 和CfTPS 也表现出优良抑制效果。此外,2,6-二氨基-4-(3-环己烯基)-3,5-二氰基-4-H-噻喃、2,6-二氨基-4-(2-硝基噻吩基)-3,5-二氰基-4-H-噻喃等结构及其衍生物也对昆虫TPS 存在一定抑制效应,这些化合物的IC50值范围分布在1.4~6.2 mmol/L 之间。这一研究为基于海藻糖-6-磷酸合成酶开发新型农用杀虫剂奠定了基础。
虚拟筛选和高通量筛选是酶抑制剂筛选和先导化合物发现的高效途径,许多研究者通过分子对接、药效团筛选、等温量热、荧光偏振和荧光热位移分析等方法均在短时间内实现了从大规模化合物库中筛选获得潜在的海藻糖-6-磷酸合成酶抑制剂,这提示我们在今后开展酶抑制剂筛选工作时,要注意整合优化筛选模式,综合运用多种筛选方法,有望更加高效地筛选出有更优活性的新型酶抑制剂。
2.2 海藻糖-6-磷酸磷酸酯酶抑制剂
有关海藻糖-6-磷酸磷酸酯酶抑制剂的研究于近年来刚刚起步,相关研究较少,但也报道了一些对海藻糖-6-磷酸磷酸酯酶有较好抑制效果的活性化合物,其中绝大部分为该酶天然底物T6P 类似物,仅有一种酶抑制剂为通过高通量筛选方法所得。
2.2.1 底物T6P 及其类似物 2017 年,Liu 等[62]首次尝试了海藻糖-6-磷酸磷酸酯酶抑制剂的合成,他们基于底物T6P 结构设计并合成了4 种底物类似物,这4 种底物类似物均保留了T6P 双糖结构,且都含有一个C6 位取代的类似磷酸根负电基团。然而,在上述4 种化合物中,仅海藻糖-6-硫酸 (T6S) 表现出对不同生物TPP 或OtsB 的一定抑制活性,经测定其Ki值主要分布在 (50 ± 5) ~ (340 ±40) μmol/L 之间,且具有较天然底物T6P 更强的结合活性 (T6P 的Km值为 (230 ± 70) ~ (690 ± 70)μmol/L)。
随后,该团队又开展了对T6S 结构优化工作,以苯环代替T6S 中葡萄糖环,并以此为母体合成了一系列化合物,其中1R-1-(4-辛基苯氧基)-葡萄糖-6-硫酸对不同生物海藻糖-6-磷酸磷酸酯酶的抑制活性最高,其Ki值仅为 (5.3 ± 0.6) ~ (80 ±10) μmol/L 之间。此外,1S-1-(4-辛基苯氧基) -葡萄糖-6-硫酸和1-(4-己基苯氧基)-葡萄糖-6 硫酸也表现出较好的酶抑制活性,二者Ki值分别为 (13 ± 2) ~(180 ± 20) μmol/L 和 (78 ± 9) ~ (350 ± 30) μmol/L[63],进一步构效关系分析发现,含有较长烃链且为α 异头型的T6S 衍生物普遍表现出较高的酶抑制活性。
2019 年,Kapil 等[64]也进行了以T6P 为母体的酶抑制剂结构改造,其合成的化合物6-氨基磷酸海藻糖对3 种分枝杆菌OtsB 均有较好抑制效果,其IC50值在0.42~1.03 mmol/L 之间。同时,鉴于有研究报道,有效霉素A 可作为EcOtsB[65]的有效抑制剂,因此上述研究中[64]也测定了有效霉素A 对结核分枝杆菌OtsB 的抑制活性,结果显示:有效霉素A 虽可对结核杆菌OtsB 产生一定抑制效应,但其抑制效果并不乐观,其IC50值为12.6~15.0 mmol/L。
2.2.2 竞争性结合抑制剂 海藻糖-6-磷酸磷酸酯酶是近年来新发现的杀虫剂、杀菌剂候选靶标,具有较大开发潜力,但目前有关该酶抑制剂的筛选工作研究仍较少。Cross 等[66]制定了两步筛选策略,从4 个化合物文库 (Natural products, Pathogen Box, CSIRO synthetic library 和Open Scaffolds Library) 的 5 000 余种化合物中筛选得到了N-苯硫基邻苯二甲酰亚胺,如表5 所示。该化合物对锡兰钩口线虫Ancylostoma ceylanicum、犬弓蛔虫Toxocara canis 等4 种线虫的TPP 均表现出强抑制性,其IC50值低至0.89~6.6 μmol/L。且经过体外试验研究发现:该化合物会使线虫成虫成活率和幼虫迁移率明显下降,证明该化合物既表现出很高的酶抑制活性,又具有良好的杀虫活性和驱虫活性,但其对细菌OtsB 几乎无抑制效果,这可能是由于细菌与线虫海藻糖-6-磷酸磷酸酯酶在空间结构上存在差异所致。
3 结论与展望
当前,利用农药进行化学防治是有害生物综合治理的主要方法,但农药过量和不合理使用导致了药剂防效降低、有害生物抗药性出现以及生态环境被破坏等一系列问题,甚至严重威胁了非靶标生物生存和正常生理过程,因此,探索新农药靶标并基于新靶标进行合理药物设计是解决这一问题的主要手段。海藻糖在病原真菌、细菌侵染及昆虫生长发育等生理过程中发挥着重要功能,更为重要的是与海藻糖代谢相关酶在人等哺乳动物体内缺失,这表明海藻糖合成酶可作为抗病原生物药物开发的潜在靶标,用于非靶标安全型新农药的开发。本文主要综述了参与海藻糖合成的海藻糖-6-磷酸合成酶和海藻糖-6-磷酸磷酸酯酶的晶体结构特征、酶与底物结合的关键氨基酸残基,为基于靶标的新农药分子设计提供结构依据,随后对已报道的酶抑制剂化学结构、生物活性及构效关系进行评述,以期对今后靶向海藻糖合成相关酶抑制剂的筛选、先导结构的发现与优化提供有意义指导。
虽然有关海藻糖合成酶结构及其抑制剂研究已取得一定进展,但仍存在许多问题,如海藻糖合成酶晶体结构较少,且尚无昆虫海藻糖合成酶晶体结构报道;现有酶抑制剂结构较为复杂且单一,已报道酶抑制剂虽具有较好离体活性,但其活体活性较差或鲜有报道。综上所述,未来研究应聚焦解析更多农业有害生物的海藻糖合成相关酶晶体结构,尤其是针对昆虫海藻糖合成酶结构的获取,以实现基于酶结构进行新农药分子的合理设计。在以海藻糖合成酶为靶标的抑制剂筛选过程中,应不断优化改进以实现多种筛选方法融合,预期发现酶抑制活性高的先导化合物,并进行进一步结构改造。随着相关研究的不断深入,可以有力推动以海藻糖合成酶为靶标的新农药设计与开发,为现代农业有害生物防治提供全新解决方案。
