陆地棉MADS-box 家族基因鉴定及组织特异性表达分析
2020-10-10张爱王彩香宿俊吉张先亮史春辉刘娟娟彭云玲马雄风
张爱,王彩香,宿俊吉,张先亮,史春辉,刘娟娟,彭云玲,马雄风,4*
(1.甘肃农业大学农学院,兰州730070;2.甘肃省干旱生境作物学重点实验室/ 甘肃农业大学生命科学技术学院,兰州730070;3.中国农业科学院棉花研究所,河南 安阳455000;4.郑州大学农学院,郑州450001)
MADS-box 基因广泛存在于植物、动物和真菌中, 所编码的蛋白为转录因子。 植物MADS-box 基因被分为Type Ⅰ型和MIKC 型两类[1]。 Type Ⅰ型基因通常有1~2 个外显子,编码蛋白质含高度保守的MADS 结构域, 依据MADS 结构域的差异进一步分为Mα、Mβ 和Mγ三类[2]。 MIKC 型基因有6~8 个外显子,编码蛋白包含MADS-box(M 结构域)、Intervening 结构域(I 结构域)、Keratin-like 结构域(K 结构域)和C 端结构域4 种保守结构域, 依据I 结构域的不同进一步分为MIKCC型和MIKC*型。 根据基因功能和序列特征差异,MIKCC型基因又可以分为14 个 亚 类:AGAMOUS-LIKE 17 (AGL17)、AGL15、AGL6、AGL12、SHORT VEGETATIVE PHASE (SVP)、SEPALLATA(SEP)、AGAMOUS(AG)、APETALA1 (AP1)、APETALA3/PISTILATA(AP3/PI)、GGM13 (BS)、FLOWERING LOCUSC(FLC)、TM8、TM3 和GpMADS4[3],MIKC*型基因又分为P 类基因和S 类基因[4-6]。
在拟南芥、 水稻等模式植物的研究中发现,MADS-box 基因所编码的转录因子能够通过形成多聚复合体的方式参与植物生长发育的生命进程。 例如,Type Ⅰ基因AGL23 参与胚乳发育、雌配子体发育和叶绿体形成[7],AGL62 基因在种子发育中起重要作用[8]。 MIKC 型基因FLC 是拟南芥春化的主要决定因素,AG 基因影响胚珠发育和花形态建成,SEP3 与生长素信号转导有关[9-11]。此外,Zhang 等[12]在桃(Prunus persica)中发现PpMADS1 可以促进果实成熟,刘国琴等[13]发现梨(Pyrus pyrifolia white pear group)芽内休眠的解除受PpMADS1 和PpMADS2 基因调控。 前人对MADS-box 基因的研究探索了其在植物生长发育中的作用机制, 验证了其功能多样化,也反映出MADS-box 基因在植物中的重要作用。
MADS-box 基因的编码蛋白作为1 类重要转录因子,参与调控棉花开花和纤维生长等发育进程。 有研究认为,棉花MADS-box 基因与细胞的伸长相关, 这类基因可能参与赤霉素(Gibberellins,GAs)合成途径,影响棉纤维伸长[14-15]。也有研究发现,GhMADS22 可能具有促进棉花开花、延缓衰老、通过脱落酸(Abscisic acid,ABA)信号途径提高植物对非生物胁迫抗性的作用[16]。另有研究证明,MADS-box 家族基因GhSOC1 能促使棉花提前开花,茎生叶数量增加,过表达该基因能使棉花花器官发生变异[17]。 陆地棉(Gossypium hirsutum L.)作为棉花4 大栽培种之一,其种植面积占棉花总种植面积90%以上[18],研究陆地棉MADS-box 家族基因对改良陆地棉早熟性、纤维品质等性状具有重要意义。 周娜等[19]利用2015年最初完成的陆地棉参考基因组序列[20],通过生物信息学方法鉴定到100 个陆地棉MIKCC基因, 并认为MIKCC基因可能受miRNA 调控,从而调控陆地棉生长发育。 Ren 等[21]也鉴定出110个MIKC 型基因, 通过遗传转化拟南芥证明GhAGL17.9(MIKCC基因)正调控LFY 基因促进开花。 综上所述,我们发现目前MADS-box 基因家族的生物信息学研究主要集中于MIKC 型基因,而对于Type Ⅰ型基因研究很少。 本研究在前人基础上, 利用2019 年最新组装的陆地棉参考基因组[22],从全基因组水平鉴定陆地棉Type Ⅰ型及MIKC 型MADS-box 家族全部基因,对该家族基因开展染色体定位、 多序列比对聚类分析、Motif 预测和基因结构鉴定等多方面研究, 并对该家族Type Ⅰ型及MIKC 型基因进行组织特异性表达分析,以期为棉花分子育种提供基因资源和理论依据。
1 材料和方法
1.1 陆地棉MADS-box 基因家族成员鉴定及染色体定位
陆地棉全基因组蛋白序列数据来自Cotton-FGD 网站[23](https://cottonfgd.org/about/download.html),从Pfam 网站[24](https://pfam.xfam.org/)下载MADS-box 家族HMM 模型文件SRF-TF(PF00319)和K-box(PF01486),使 用HMMER 3.0 软件[25]鉴定陆地棉MADS-box 家族基因,阈值E<1e-5。 利用Pfam 网站[24](https://pfam.xfam.org/)和SMART 网 站[26](http://smart.embl-heidelberg.de/) 进一步确认所鉴定MADS-box 基因编码蛋白是否含有其保守结构域。从CottonFGD 网站[23](https://cottonfgd.org/jbrowse/) 获取陆地棉MADS-box 家族基因物理位置信息文件,利用软件MapInspect(http://www.mybiosoftware.com/mapinspect-compare-display-linkage-maps.html)绘制基因在染色体上的位置图。
1.2 系统进化树构建分析
从TAIR 网站[27](https://www.arabidopsis.org/)获得拟南芥MADS-box 蛋白质序列,通过基因组注释信息 (http://rice.plantbiology.msu.edu/)[28]获得水稻MADS-box 蛋白序列, 利用Pfam 网站[29](https://www.ebi.ac.uk/Tools/pfa/pfamscan/)确认所获得拟南芥和水稻MADS-box 蛋白含有的结构域。 用ClustalW 将棉花MADS-box 蛋白序列与已经分型的拟南芥和水稻MADS-box 蛋白进行序列比对分析,利用MEGA 7.0[30]构建系统进化树,使用邻近法(Neighbor-joining method),选择成对删除(Pairwise deletion),设置Bootstrap 值为1 000。
1.3 陆地棉MADS-box 家族保守基序(motif)预测及基因结构分析
使用MEME 在线工具[31](http://meme-suite.org/) 进行motif 预测,motif 最大发现数设为5,利用浙江大学Gossypium new sequence data release 网 站 (http://ibi.zju.edu.cn/cotton/) 获 取MADS-box 基因结构注释gff3 文件。 运用TBtools[32]对motif 分析结果及基因结构分析结果进行可视化。
1.4 MADS-box 基因组织特异性表达分析
从NCBI SRA 数 据 库(https://www.ncbi.nlm.nih.gov/sra/?term=PRJNA248163) 获得陆地棉标准系TM-1 根、茎、叶、花托、雌蕊、雄蕊、花萼、花瓣、花后5 d 纤维、花后10 d 纤维、花后20 d 纤维以及花后25 d 纤维12 个组织的RNA-seq 数据[20]。通过TopHat[33]和Cufflinks[33]比对到陆地棉基因组并组装样本转录本。通过计算标准化FPKM 值显示基因的表达水平。 表达模式热图通过omicshare 网 站(https://www.omicshare.com/tools/Home/Soft/heatmap)呈现。
2 结果与分析
2.1 陆地棉MADS-box 家族基因鉴定
为了全面准确鉴定陆地棉MADS-box 家族基因,利用最新组装的陆地棉参考基因组[22],最终鉴定到陆地棉MADS-box 基因家族成员181 个,其中,Type Ⅰ基因占36.5%(66 个),MIKC 基因占63.5%(共115 个, 包括90 个MIKCC型和25个MIKC*型基因)(表1)。 运用CottonFGD 网站的信息, 分析MADS-box 基因理化性质, 发现85%的Type Ⅰ型基因具有1~2 个外显子,编码蛋白质最小包含74 个氨基酸残基(GH_D06G0314), 最 大 包 含593 个 氨 基 酸 残 基(GH_A01G1960);97%的MIKCC基因含有6~8个外显子, 编码蛋白质长度从174 氨基酸残 基 (GH_D03G0830) 到315 氨 基 酸 残 基(GH_A08G1545); 而MIKC*型基因的外显子数目范围较大(1~12),保守性相对较差,所编码蛋白质长度分布在63(GH_D04G0829)~373 氨基酸残基(GH_D12G1969)。 分析显示:相对TypeⅠ型和MIKC*型基因,MIKCC型基因外显子数目及基因所编码蛋白质长度均比较保守。
2.2 MADS-box 基因在染色体上的位置
为了确定MADS-box 基因在陆地棉染色体上的分布,通过CottonFGD 网站获得所鉴定181个MADS-box 基因的物理位置信息文件, 利用MapInspect 进行可视化展示,发现A 组染色体上分布78 个MADS-box 基因,D 组染色体上分布103 个,D 组染色体上分布的基因较多(图1)。 进
一步分析发现, 除了A02、A07 和D07 染色体,Type Ⅰ型基因在其余23 条染色体上均有分布,其中染色体D13 上最多(8 个),在染色体A01、A03、A05、A06 和A10 上各有1 个。 MIKCC型基因在陆地棉26 条染色体上均匀分布, 其中在A01 和D01、A08 和D08、A09 和D09、A10 和D10 共4 对同源染色体上各有2 个MIKCC基因。MIKC*型基因分布于15 条染色体上,其中染色体D02 和D04 上分布最多,分别有4 个基因;A03、A05、A11 和D11 染色体上各有2 个MIKC*基 因;A02、A06、A07、A12、A13、D05、D06、D07 和D12 染色体上各含1 个MIKC*基因。
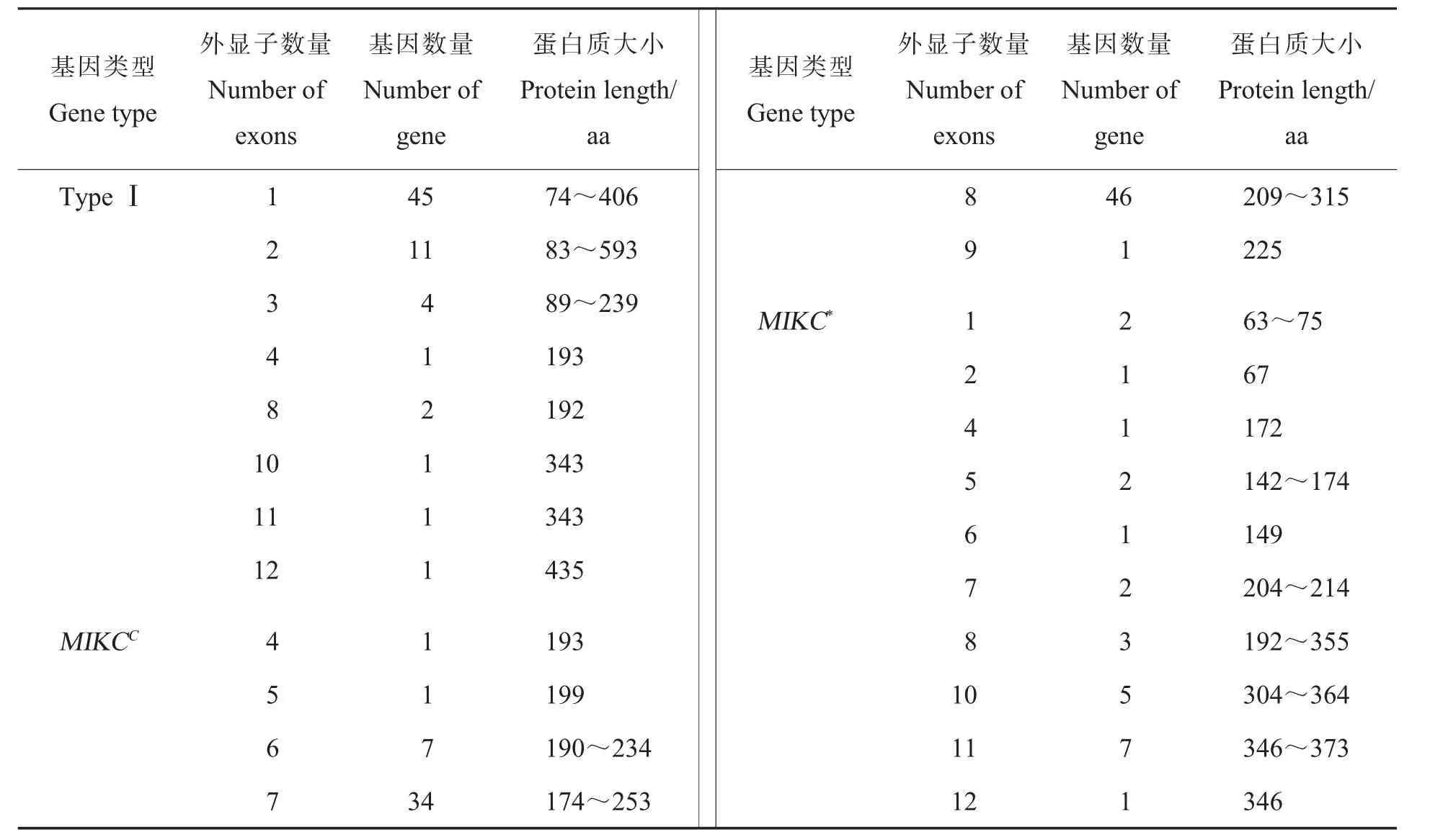
表1 陆地棉MADS-box 基因外显子数量及蛋白质长度Table 1 The Number of exons and protein length of MADS-box genes in upland cotton
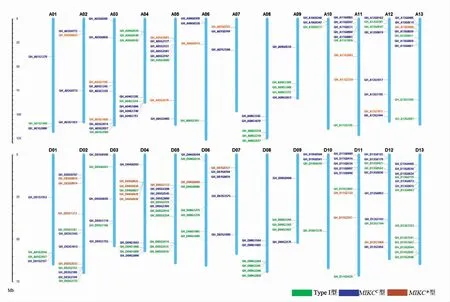
图1 MADS-box 基因在陆地棉染色体上的分布Fig. 1 Chromosomal distribution of MADS-box genes in upland cotton
2.3 多序列比对及进化分析
为了确定陆地棉MADS-box 基因同源进化关系,分别筛选100 个拟南芥MADS-box 蛋白序列(57 个Type Ⅰ型,43 个MIKC 型),71 个水稻MADS-box 蛋 白 序 列 (36 个Type I 型,35 个MIKC 型)(表2)。 使用MEGA 7.0 构建拟南芥、水稻和陆地棉Type I 型MADS-box 蛋白系统进化树, 结果显示陆地棉66 个Type I 型蛋白共被分为3 个亚家族,Mα、Mβ 和Mγ (表2 和图2A), 其中40 个Type I 为Mα 类,21 个为Mγ类,二者数量均大于拟南芥和水稻Mα 及Mγ 蛋白数量;而陆地棉Mβ 蛋白仅有5 个,少于拟南芥和水稻Mβ 类蛋白数量。 构建拟南芥、水稻和陆地棉MIKC 型MADS-box 蛋白系统进化树,发现陆地棉MIKC 型蛋白包含MIKCC型10 个亚家族 (AGL17、AG、SVP、AGL15、BS、AP3/PI、SOC1、AP1、AGL6 和SEP)以及MIKC* 型(图2B)。进一步比较发现, 拟南芥和水稻中分别有5 个和6个AP1 蛋白,而陆地棉中有11 个;对于AG 亚家族,拟南芥、水稻、陆地棉中成员数目分别是4、5和10 个;AP3/PI 亚家族中,拟南芥和水稻分别有2 个和3 个,而陆地棉有12 个该亚家族蛋白。 结果显示,在AP1、AG、AP3/PI 亚家族中,陆地棉包含成员数量均超过拟南芥和水稻。 此外,陆地棉中与开花时间相关的SVP、AGL17、AGL6 亚家族成员数量是水稻或拟南芥蛋白数量的2 倍多;且在陆地棉中未发现FLC 蛋白,该类蛋白在拟南芥春化开花调控途径中起关键作用(表2 和图2B)。
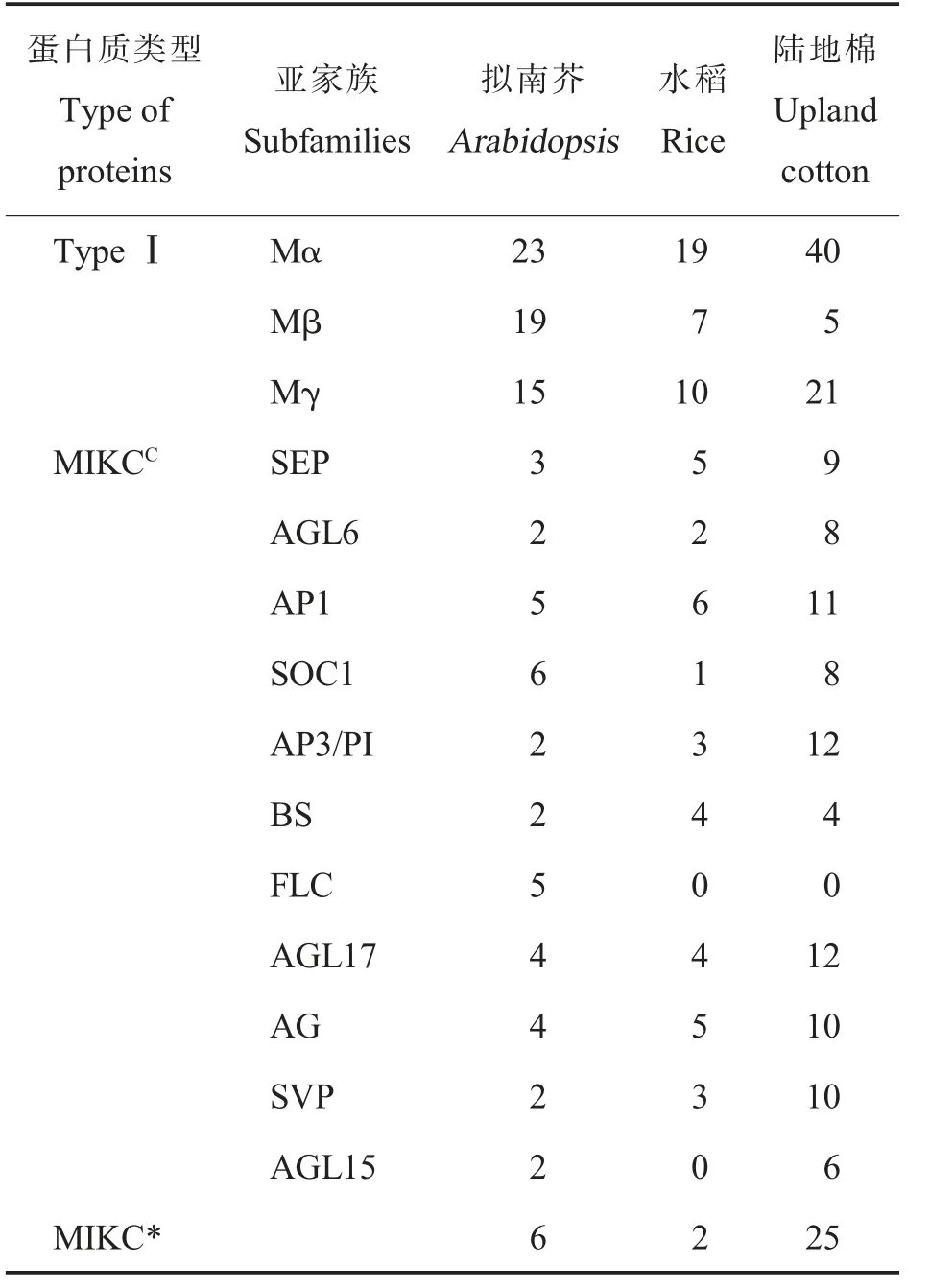
表2 拟南芥、水稻和陆地棉MADS-box 蛋白质数目比较Table 2 Comparison of MADS-box proteins number in Arabidopsis, rice and upland cotton
2.4 MADS-box 家族成员保守基序(motif)预测及基因结构分析
为了确定MADS-box 基因编码蛋白所含基序,通过MEME 在线工具预测分析发现,Type Ⅰ型66 个基因所编码蛋白均含有MADS 结构域(motif 2)(图3A)。 MIKC 型基因编码蛋白中,90个MIKCC蛋白均含有MADS 结构域(motif b)和K 结构域(motif e)(图3B);25 个MIKC* 型蛋白均含有MADS 结构域(motif 7),含有K 结构域(motif 10)的有22 个(图3C)。 综上,通过保守基序分析可知,在MADS-box 家族中,所有成员均具有MADS 结构域,K 结构域仅在MIKC 类蛋白中存在,MADS-box 家族蛋白I 结构域和C 结构域的序列长度及结构保守程度较低。

图2 拟南芥、水稻、陆地棉MADS-box 家族成员的系统进化分析Fig. 2 Phylogenetic analysis of members of MADS-box family in Arabidopsis, rice and upland cotton

图3 陆地棉MADS-box 蛋白motif 预测结果Fig. 3 Motif prediction results of MADS-box proteins in upland cotton
分析MADS-box 家族基因结构发现:TypeⅠ型基因中GH_D04G0827、GH_D02G1381、GH_D06G0900、GH_A13G0811 和GH_D13G0779 包含8 ~12 个 外 显 子,GH_D13G1667 、GH_D13G1333、GH_D13G0907、GH_D02G2153 和GH_D12G1194 分布3~4 个外显子, 其余56 个Type Ⅰ型基因均有1~2 个外显子(45 个基因含有1 个外显子,11 个基因含有2 个外显子)(图4A);而MIKC 型基因中,同一亚家族成员外显子结构和数量具有相似性, 例如,SEP、AGL6、AG和AP1 亚族的大多数成员均含有8 个外显子,SOC1 和AP3/PI 基因含有7 个外显子,BS 基因含有6 个外显子(图4B)。 基因长度分析结果显示,Type Ⅰ型基因均未超过5 000 bp (base pair,碱基对);MIKC 型基因中, 基因长度是各亚族间最明显的区别,例如:SEP 和MIKC* 类基因长度均不超过5 000 bp,AGL17 类基因中8 个基因的长度超过13 000 bp。 上述结果表明,Type Ⅰ型基因的外显子数目和基因长度均小于MIKCC型基因。

图4 陆地棉MADS-box 基因结构Fig. 4 Gene structure of MADS-box genes in upland cotton
2.5 陆地棉MADS-box 基因组织特异性表达分析
根据基因表达分析结果可将Type Ⅰ型MADS-box 基因分为8 类。 第1 类基因在茎、叶、花托中表达, 第3、8 类基因主要在纤维中表达,第2、4、5、6、7 类基因分别在萼片、雌蕊、根、雄蕊和花瓣中高水平表达(图5A)。
MIKC 型基因根据表达分析结果可分为10类,分别在纤维(第1、2 类基因)、花器官(第3、4、6、7、9 和10 类)、根和茎(第5、8 类基因)中表达量较高,而叶中的表达水平很低(图5B)。 对各组织中所表达基因进一步分析发现,有4 个AG 基因(GH_A05G2521、GH_A05G2747、GH_D05G2764和GH_D05G2543)在纤维中特异表达。 1 个AP1基因(GH_D03G1119)和1 个AGL6 基因(GH_D07G1573)仅在萼片中表达,3 个SVP 基因(GH_A06G0328、GH_A12G1017 和GH_D12G0863)在花托中优势表达,此外,有5 个基因在雄蕊中特异表达, 包括4 个MIKC* 基因、1 个AP3/PI 基因。 有3 个SOC1 基因和3 个AGL17 基因在茎中表达量较高。综上,MIKC 型基因主要在陆地棉花器官和纤维各发育阶段表达。
3 讨论
MADS-box 家族基因编码蛋白是植物生长发育过程中的重要转录因子。 随着多物种基因组测序完成,在拟南芥[34-35]、水稻[36-37]、小麦[38]、大豆[39]、番茄[40]、甘蓝[41]、葡萄[42]、向日葵[43]和银杏[3]等多种植物中对该家族基因进行了相关报道。 本研究利用最新组装的陆地棉参考基因组数据[22],通过生物信息学方法鉴定出181 个陆地棉MADS-box家族基因(66 个Type Ⅰ型,115 个MIKC 型)。前期Ren 等[21]鉴定到110 个陆地棉MIKC 型基因,分布于25 条染色体, 染色体D01 上无MIKC 型基因分布。 我们的研究结果鉴定出115 个MIKC型基因,分布于陆地棉26 条染色体上,其中D01染色体上有2 个MIKCC基因(GH_D01G1353 和GH_D01G2157); 鉴定出66 个Type Ⅰ型基因,分布于23 条染色体上, 鉴定结果不同可能与所使用参考基因组有关。
通过系统进化分析发现,陆地棉中与花形态建成相关基因(如:AG 和AP3/PI)、开花时间相关基因(如:SVP、AGL17 和AGL6)超过拟南芥或水稻相同亚家族基因数目的2 倍,这可能是由于陆地棉基因组较大[44-45],而拟南芥和水稻基因组较小[46-50]造成的。 我们推测陆地棉MADS-box 基因在调控开花进程中,比拟南芥和水稻更加精细而复杂。 FLC 是MADS-box 基因所编码蛋白,是1个开花抑制因子,高水平表达可抑制开花,低温负调控FLC 转录及蛋白表达水平促进植物开花[51]。分析发现,陆地棉缺失FLC 基因,这与前人研究结果一致[52],可能与陆地棉开花不需要春化有关。 此外,本次鉴定也未发现陆地棉AGL12 类基因, 且拟南芥与水稻的AGL12 基因均与陆地棉AP1 基因聚到1 个亚家族,这与前期Ren 等[21]的研究结果(陆地棉有2 个AGL12 基因)不同,可能与所使用参考基因组有关。
根据前人报道,MADS-box 基因参与雌配子体发育[7]、种子发育[53]、胚及胚乳发育[8]、控制雄蕊及花瓣发育[54]、心皮形态建成[55]、与不同物种间生殖器官分界决定有关[56],并且与棉花纤维细胞伸长有关[14-15]。 通过组织特异性表达分析发现,陆地棉MADS-box 基因在花器官和纤维中高水平表达, 例 如:Type Ⅰ型 基 因 中GH_D05G2910、GH_D09G1491 和GH_D02G2551 在纤维发育初期特异性表达,GH_A08G2258、GH_D06G1276、GH_D06G1275、GH_D10G1756 在纤维发育后期特异表达。MIKC 基因中AP3/PI 基因主要在花器官中表达,AP1 基因主要在萼片中表达,AG 基因主要在纤维中高水平表达,BS 和AGL15 类基因均可在纤维中被发现,这些基因可能参与陆地棉开花和纤维发育调控,具体功能还需通过遗传转化等进一步研究。
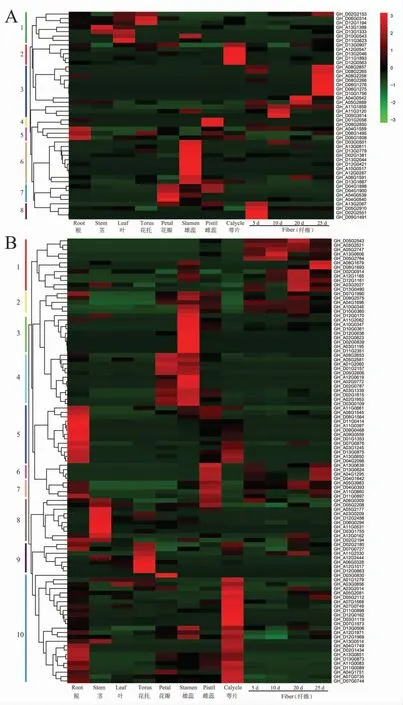
图5 MADS-box 基因在陆地棉不同组织中的表达分析Fig. 5 Expression analysis of MADS-box genes from different tissues in upland cotton
4 结论
通过生物信息学方法,鉴定了181 个陆地棉MADS-box 家族基因,包括Type Ⅰ和MIKC 型2大类基因。 染色体定位结果显示,Type Ⅰ基因分布于陆地棉23 条染色体,MIKC 型基因分布在陆地棉26 条染色体上。 聚类分析发现, 陆地棉Type I 型蛋白分为3 个亚家族,MIKC 型蛋白包括MIKCC型 (包含10 个亚家族) 以及MIKC*型。Motif 预测结果显示,陆地棉MADS-box 蛋白均含有MADS 结构域。 基因结构分析可知,MADS-box 基因外显子和内含子结构及长度在同一亚家族内具有相似性。 组织特异性表达分析显示,12 个组织中均有Type Ⅰ型基因参与表达,MIKC 基因主要在陆地棉花器官和纤维各发育阶段表达量较高。对陆地棉MADS-box 家族的生物信息学分析,为了解陆地棉开花和纤维发育的基因组学研究提供了有益的借鉴,对于揭示棉花纤维品质等重要性状的遗传调控机制及分子育种具有一定的理论意义和应用价值。
