野生树鼩与人工饲养树鼩消化道不同部位微生物组成的比较研究
2020-05-26李艳艳杨凤梅王俊斌陈丽雄段素琴杨亚平和占龙
李艳艳,杨凤梅,王俊斌,陈丽雄,段素琴,杨亚平,刘 雨,和占龙,马 进,赵 远
(中国医学科学院/北京协和医学院医学生物学研究所,云南省重大传染病疫苗研发重点实验室,昆明 650118)
树鼩(Tree shrew,Tupaia belangeri)属于哺乳动物、攀鼩目和树鼩科。它主要生活在东南亚,在中国树鼩主要分布在云南省和广西省,分为6个亚种(Tupaia belangeri chinensis,Tupaia belangeri gaoligongensis,Tupaia belangeri modesta,Tupaia belangeri tonquinia,Tupaia belangeri yunalis and Tupaia belangeri yaoshanensis)[1],其特点是生育和生命周期短,繁殖力高(4~6个月到成年期,每胎出生2~6个后代),中等大小(成年体重120~150 g),并且容易喂养。近年来越来越多地应用于科学和生物医学研究。树鼩可以感染甲型肝炎病毒、乙型肝炎病毒、丙型肝炎病毒、轮状病毒、腺病毒和疱疹病毒[2-6]。此外,树鼩还被用来建立耐药性细菌感染、败血症、心理压力和抑郁、近视和代谢疾病的动物模型[7-9]。这些研究极大地促进了中国树鼩在生物医学研究中的应用。
哺乳动物胃肠道拥有一个巨大的微生物生态系统,被称为微生物群,它们与宿主共生、共代谢和共进化,有利于宿主生物学[10]。有证据表明,不同的因素可以导致肠道菌群的变化,这些变化可以影响健康和疾病进程[11],并与多种疾病密切相关,如消化道疾病、代谢类疾病(如II型糖尿病)、免疫性疾病(如风湿性关节炎)、甚至心脑血管疾病(动脉粥样硬化),通过对肠道菌群的研究为多种疾病研究和防治提出了新的方向[12-14]。由于粪便采样方便且无创,目前大量的动物和人类肠道菌群的研究通常集中在以粪便菌群来代表肠道菌群[15-16]。然而,粪便菌群与肠道菌群有相关性,但并不能代表肠道菌群,研究表明粪便菌群成分与近端的结肠腔和黏膜高度相关,与远端小肠适度相关[17]。
然而作为最接近灵长类动物的树鼩各肠段微生物的组成至今未见报道。本研究采用16S rRNA基因测序技术,避开传统活菌培养耗时较长、细菌生长条件特殊、培养技术复杂等缺点,对野生树鼩与人工驯养树鼩不同肠道段菌群的种类和分布进行了比较研究,为树鼩不同肠段内菌群的种类、分布、功能及与宿主的关系提供参考,对理解不同部位菌群间的代表性也有参考价值。研究结果提示,在今后研究中应充分考虑粪便样品微生物的组成是否能够完全代表肠道微生物的组成。
1 材料和方法
1.1 实验材料
1.1.1 实验动物
野生健康树鼩3只,4月龄,体重120~200 g,2018年捕捉于中国云南省昆明市西山区谷律乡(25° 07′N,102° 26′E,海拔2221 m)。人工饲养健康树鼩3只,4月龄,体重120~200 g,来自于中国医学科学院医学生物学研究所饲养繁殖的仔一代树鼩(普通级)[SCXK(滇)K2018-0002],饲养于中国医学科学院医学生物学研究所普通级实验室[SYXK(滇)K2018-0002],饲喂全价颗粒饲料,并辅以水果。对以上两组动物肌注氯胺酮深度麻醉后,颈动脉放血处死,全身75%酒精消毒后在无菌手术室内取各肠段内容物0.5~1 g于无菌EP管(置于冰面上),并迅速转移至-80℃冰箱冻存至检测,样品编号如表1。实验经本单位伦理委员会审核批准(DWSP201810002),实验过程严格遵循实验动物使用的3R原则和福利伦理原则。
1.1.2 实验样品
实验所用材料详见表1。
1.2 主要试剂与仪器
Phusion®High-Fidelity PCR Master Mix with GC Buffer、高效高保真酶(New England Biolabs公司);TruSeq®DNA PCR-Free Sample Preparation Kit、HiSeq2500测序平台(Illumina公司);QIAquick Gel(QIAGEN公司);-80℃冰箱(Thermo Scientific公司)。
1.3 实验方法
按参考文献[18-22]的方法进行基因组DNA提取和PCR扩增文库构建、测序及生物信息学分析。采用CTAB方法对样本的基因组DNA提取后检测DNA的纯度和浓度。用无菌水稀释基因组DNA至1 ng/μL,以其作为模板,针对16S rDNA的V3-V4片段,515F(5’-GTGCCAGCMGCCGCGGTAA-3’)和806R(5’-GGACTACHVGGGTWTCTAAt-3’),使用带Barcode的特异引物,Phusion®High-Fidelity PCR Master Mix with GC Buffer,和高效高保真酶进行PCR,确保扩增效率和准确性。使用TruSeq®DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,使用HiSeq2500 PE250进行上机测序。根据Barcode序列和PCR扩增引物序列从下机数据中拆分出各样品数据,截去Barcode和引物序列后使用FLASH对每个样品的reads进行拼接得到原始Tags数据(Raw Tags),经严格过滤处理得到高质量 的Tags数 据(Clean Tags)。参 照Qiime(V1.9.1)的Tags质量控制流程得到最终的有效数据(Effective Tags)。利用Uparse软件对所有样品的全部Clean Reads进行聚类,默认以97%的一致性(identity)将序列聚类成为OTUs,对OTUs代表序列进行物种注释,用Mothur方法与SILVA的SSUrRNA数据库进行物种注释分析。对OTUs进行Alpha多样性分析和Beta多样性分析。最后,用PICRUSt根据Marker基因进行元基因组功能预测的生物信息软件包,根据测序数据进行基于KEGG数据库的功能预测。

表1 分组及样品编号Table 1 Grouping and sample number
1.4 统计学方法
2 结果
2.1 样品细菌丰富度和多样性评估
在检查了测序的质量后,从42个不同胃肠道样本中获得了2305843个有效序列。通过不同样品在97%一致性阈值下的α多样性指数(shannon、simpson、chao1、ACE、goods_coverage)分别对样本序列进行丰富度和多样性分析。结果显示覆盖范围从99.1%到99.7%,表明本实验每个样品的丰富度及多样性足以完整描述该样品菌群的组成;Shannon指数及Chao 1指数结果显示,野生组树鼩空肠和胃低于人工繁育组,其余肠段均高于人工繁育组,说明以上野生树鼩大部分肠段内菌群多样性高于人工繁育树鼩,且Shannon指数显示,野生组胃内菌群多样性与人工繁育组差异极显著(P<0.01),而盲肠内菌群多样性野生组明显高于人工繁育组(P<0.05)(见表2)。同时,从表2可以看出野生树鼩各肠段OTUs数从高到低依次是:十二指肠、结肠、回肠、直肠、空肠、盲肠、胃;而人工繁育组依次是:空肠、胃、直肠、回肠、结肠、盲肠、十二指肠。
2.2 消化道不同部位菌群组成
细菌群落的相对丰度结果显示,在门水平,野生组树鼩直肠、结肠和盲肠主要由变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)组成;而人工繁育树鼩直肠、结肠主要由变形菌门(Proteobacteria)、螺旋体门(Spirochaetes)和梭杆菌门(Fusobacteria)组成,盲肠主要由变形菌门(Proteobacteria)、螺旋体门(Spirochaetes)和拟杆菌门(Bacteroidetes)组成。野生树鼩回肠中主要由厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)组成,人工繁育树鼩回肠中主要由厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和柔膜菌门(Tenericutes)组成;野生树鼩空肠中主要由厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和柔膜菌门(Tenericutes)组成,而人工繁育树鼩空肠中厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)组成;野生树鼩十二指肠中主要由厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和Cyanobacteria菌门组成,而人工繁育树鼩十二指肠中厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和螺旋体门(Spirochaetes)组成;野生树鼩胃中主要由厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)组成,而人工繁育树鼩胃中主要厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、Cyanobacteria和拟杆菌门(Bacteroidetes)组成(见图1A)。
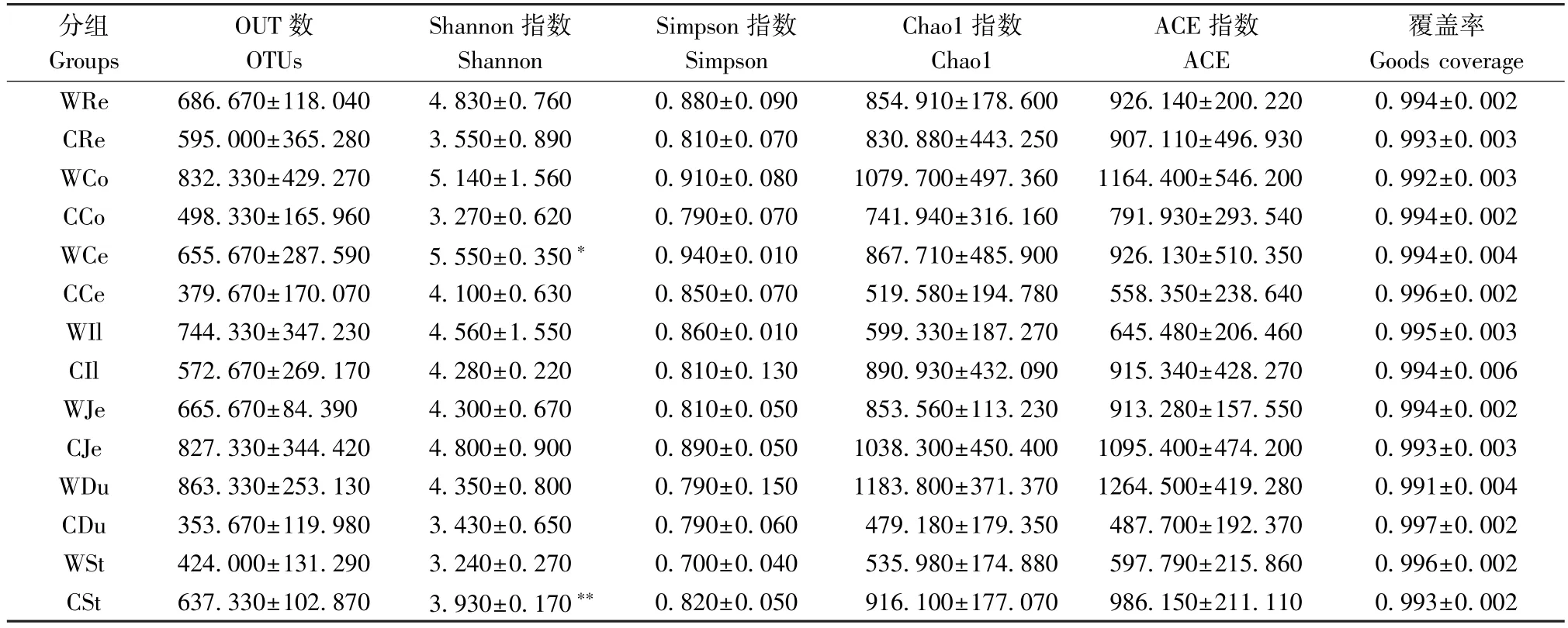
表2 野生和圈养繁殖群不同肠段微生物α多样性指数的比较Table 2 Comparison of Alpha diversity index of gut microbiota from the wild and captive bred groups
属水平结果显示,野生树鼩直肠和结肠内以Proteobacteria菌门的螺杆菌属(Helicobacter)、弯曲杆菌属(Campylobacter)为主,而人工繁育树鼩则以Proteobacteria菌门的螺杆菌属(Helicobacter)、弯曲杆菌属(Campylobacter)和Spirochaetes菌门的短螺菌属(Brachyspira)为主;盲肠内,野生树鼩主要以Proteobacteria菌门的螺杆菌属(Helicobacter)、弯曲杆菌属(Campylobacter)和Firmicutes菌门的魏氏菌属(Weissella)为主,而人工繁育树鼩则以Spirochaetes菌门的短螺菌属(Brachyspira)和Bacteroidete菌门的普雷沃菌属(Prevotella)为主;回肠内,野生树鼩主要以Firmicutes菌门的魏氏菌属(Weissella)、乳球菌属(Lactococcus)和链球菌属(Streptococcus)为主,而人工繁育树鼩则以Firmicutes菌门的链球菌属(Streptococcus)、Tenericutes菌门的Ureaplasma属、Firmicutes的乳酸杆菌属(Lactobacillus)和Proteobacteria菌门的螺杆菌属(Helicobacter)为主;空肠内,野生树鼩主要以Firmicutes菌门的魏氏菌属(Weissella)、Proteobacteria菌门的螺杆菌属(Helicobacter)和Tenericutes菌门的Ureaplasma属为主,而人工繁育树鼩则以Firmicutes门的乳酸杆菌属(Lactobacillus)、链球菌属(Streptococcus)和Proteobacteria菌门的螺杆菌属(Helicobacter)为主;十二指肠内,野生树鼩主要以Firmicutes菌门的魏氏菌属(Weissella)和Proteobacteria菌门的螺杆菌属(Helicobacter)为主,而人工繁育树鼩则以Spirochaetes菌门的短螺菌属(Brachyspira)、Firmicutes门的乳酸杆菌属(Lactobacillus)、Tenericutes菌门的Ureaplasma属和Proteobacteria菌门的螺杆菌属(Helicobacter)为主;胃内,野生树鼩主要以Firmicutes菌门的魏氏菌属(Weissella)为主,而人工繁育树鼩则以Bacteroidete菌门的普雷沃菌属(Prevotella)和Firmicutes门的乳酸杆菌属(Lactobacillus)为主(见图1B)。
2.3 消化道不同部位菌群比较
韦恩图被用来确认野生和人工繁育的树鼩各肠段的核心肠道微生物群。结果显示,野生和人工繁育的树鼩直肠、结肠、盲肠、回肠、空肠、十二指肠及胃中共有的OTUs数量分别为:648、663、459、620、692、401和453个,见图2。
LEfSe分析的意义是在组间寻找具有统计学差异的生物标记物,并强调其统计学意义与生物物种的相关性,用来识别组间不同物种丰度的特征及其关联的类别。
LEfSe(LDA effect size)分析为我们提供了野生组和人工繁育组类群之间有显著差异的分类群(图3)。结果显示,在野生组和人工繁育组直肠内共发现17个和8个相对丰度差异的分类群,在结肠内为5个和8个,在盲肠内为12个和10个,回肠内为11个和13个,空肠内为7个和9个,胃内为10个和5个。
从图3中,我们发现,在门水平上,野生组直肠、结肠、盲肠及胃四个部位中厚壁菌门(Firmicutes)的平均占比明显高于人工繁育组(P<0.05);人工繁育树鼩直肠、结肠、盲肠及回肠内螺旋体门(Spirochaetes)的平均占比明显高于野生树鼩(P<0.05);野生组直肠内拟杆菌门(Bacteroidetes)的平均占比明显高于人工繁育组(P<0.05),而该菌门在野生组回肠内的平均占比则明显低于人工繁育组(P<0.05)。属水平上,各肠段野生组明显高于人工繁育组的主要菌属为:回肠、空肠和胃内的魏氏菌属(Weissella),直肠内的拟杆菌属(Bacteroides)以及回肠段内的乳球菌属(Lactococcus);而人工繁育组明显高于野生组的主要菌属为:直肠、结肠、盲肠及回肠内的短螺菌属(Brachyspira),盲肠和胃内的普雷沃菌属(Prevotella_9),盲肠和空肠内的链球菌属(Streptococcus)以及回肠中的乳酸菌属(Lactobacillus)。
2.4 PICRUSt功能预测
KEGG功能预测结果显示,在1级水平中,人类疾病和组织系统不同肠段野生组与人工繁育组表现出基本相似的基因功能,其他基因功能在同组不同肠段或同肠段不同组间有一定程度的差异(图4)。通过进一步在2级水平上的比较,我们发现,野生组与人工繁育组共有5个解剖部位17个基因类别之间的差异有统计学意义(P<0.05),它们分别是:野生组直肠段的复制与修复和细胞生理过程及信号传导明显高于人工繁育组;结肠段的氨基酸代谢、细胞生理过程及信号传导野生组明显低于人工繁育组;野生组盲肠段外源物质的生物降解与代谢、能量代谢和转录明显高于人工繁育组,而其他次生代谢物的生物合成、感染性疾病和信号分子与相互作用野生组明显低于人工繁育组;回肠段的消化系统野生组明显高于人工繁育组;胃内的转录和脂代谢野生组明显高于人工繁育组,而酶家族、辅助因子和维生素代谢、遗传信息处理和免疫系统野生组明显低于人工繁育组(见图5)。
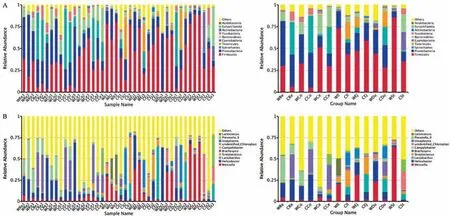
图1 野生和人工繁育的树鼩的不同肠段门水平(A)和属水平(B)微生物组成Figure 1 Phylum levels(A)and genus levels(B)of different intestinal segments of wild and captive tree shrews
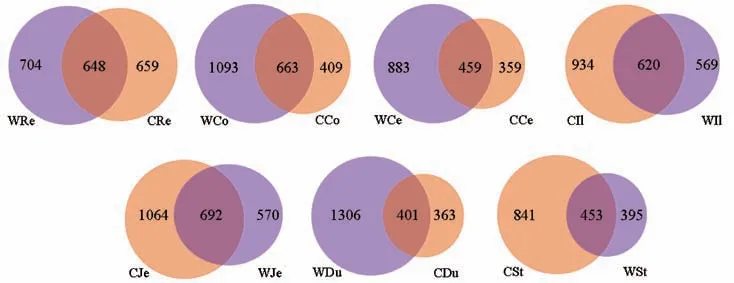
图2 野生组和人工繁育组同一肠段所共有的OTUs的数量Figure 2 The number of OTUs in the same intestinal segment Shared by the wild group and the captive group
3 讨论
树鼩是一种新兴的实验动物,近年来,在生物学和医学研究中都产生了大量的疾病树鼩模型[23]。然而,树鼩消化道中微生物群落的特征和分布尚不清楚,尤其是树鼩各个肠段微生物的组成情况,至今仍未见报道。本研究首次采用16S rRNA Illumina MiSeq高通量测序技术,比较了人工繁育树鼩和野生树鼩的不同胃肠段微生物的组成及差异。研究数据表明,饮食是影响哺乳动物肠道微生物群的主要因素[24]。在本研究中,人工繁育树鼩的食物主要由含有高蛋白和多糖的饲料和新鲜水果组成,野生树鼩主要食用野生高纤维植物叶片。因此,人工繁育和野生树鼩之间的微生物群的差异可能与饮食差异密切相关。
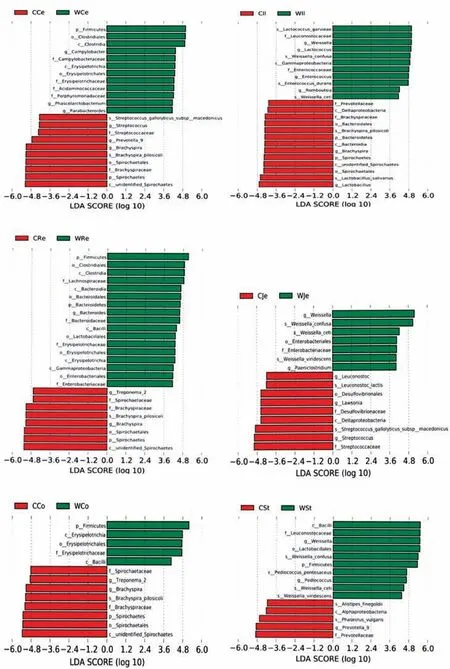
图3 组间菌群差异线性分析(LEfSe)Figure 3 Linear analysis of inter-group flora differences(LEfSe)

图4 不同肠段功能的相对丰度热图Figure 4 Relative abundance heat map of different intestinal segment functions

图5 2级水平不同肠段间差异功能基因的相对丰度变化(P<0.05)Figure 5 Significant differences in gene categories at level 2(P<0.05)between the different GI
长期以来研究认为,哺乳动物消化道内细菌多样性是从胃到粪便的增加,因为胃和小肠内环境过于严酷(由于pH值低)以至于很难保证微生物的生长和保持更大的多样性[25]。我们对野生树鼩的研究结果也支持这一传统观念,Alpha多样性指数显示野生组树鼩盲肠段物种丰富度最高,结肠次之(见表2)。同时,传统研究认为哺乳动物盲肠和结肠中,微生物负责分解在通过小肠运输过程中未被代谢的多糖,盲肠和结肠成为身体内拥有菌落密度最大、多样性最丰富的场所[26-27]。然而,人工繁育组树鼩消化道细菌多样性却不支持这一观念,在空肠、回肠和盲肠样本以及胃样本中都发现了更大的多样性,在十二指肠和大肠末端样品中留下了最少的多样性(见表2)。这一结果可能与树鼩肠段结构有关,空肠和回肠是树鼩肠段中最长的两段,为微生物的生长提供了更好的环境,这可能造成人工饲养树鼩空肠段和回肠段细菌多样性较其他肠段高的原因,然而,还需要进一步的研究来解释这些现象。
本研究发现野生树鼩胃肠道各解剖部位内门水平菌群组成上有很大差异,例如:在野生组直肠、结肠内以Proteobacteria菌门丰富度最高,盲肠、回肠、空肠、十二指肠及胃内都以Firmicutes菌门丰富度最高(图1A)。张飞燕等[28]对野生树鼩粪便菌群组成的研究认为门水平以Firmicutes和Bacteroidetes为主,属水平以乳球菌属、链球菌属、普雷沃菌属、拟杆菌属、魏斯氏菌属等为主,这一结果与本研究胃肠道各肠段微生物组成有所不同,由此可见,粪便菌群与肠道菌群有相关性,但并不能代表肠道内菌群。同时,我们还发现人工繁育组树鼩各肠段与野生组也有很大差别,人工繁育组直肠、结肠和盲肠中以Spirochaetes菌门丰富度最高(37.14%、42.29%、30.33%),且明显高于野生组(P<0.05);树鼩直肠、结肠和盲肠内的Firmicutes明显高于人工繁育组(P<0.05);回肠、空肠、十二指肠及胃内都以Firmicutes菌门丰富度最高(73.30%、47.23%、44.03%、85.52%)(图1A)。这一结果与人工饲养的SPF C57BL/6小鼠及我们之前对非人灵长类恒河猴的研究结果均不一致[29-30]。在我们中心人工繁育的树鼩与人工繁育恒河猴饲喂的饲料,其成分基本一致,然而对不同肠段菌群的研究结果的不一致是否说明肠段菌群的组成在很大程度上与物种有关。
从功能基因预测结果的分析,我们发现,野生树鼩与人工饲养树鼩之间的差异功能基因主要集中在直肠、结肠、盲肠和胃几个解剖部位,尤其以盲肠和胃之间的差异基因较多。已有的研究认为大肠和小肠之间在功能上有明显不同[31]。野生树鼩与人工饲养树鼩各肠段的功能差异主要集中在大肠各段,主要在细胞修复、信号传导、复制、消化、异源物质降解和代谢、能量代谢、转录等功能高于人工繁育组,说明野生树鼩大肠段承担了更多了能量代谢和外源物清除相关工作,这也与大肠段优势菌群组成相关。小肠段作为动物消化系统主要的吸收器官,两组动物功能基因并没有显著差异。而在胃内人工饲养组树鼩承担了比野生组更多的酶家族、辅酶、维生素代谢以及遗传与免疫功能。这些功能上的差异也提示不同食物来源可能造成了野生树鼩与人工繁育树鼩不同肠段微生物组成的差异从而导致功能上的差异。
由于标本取材的特殊性,目前国内外研究均以粪便中微生物代替肠道微生物,尚无健康野生树鼩不同肠段微生物组成的报道。本研究采用宏基因组测序技术,避开传统活菌培养耗时较长、细菌生长条件特殊、培养技术复杂等缺点,对树鼩不同肠段菌群组成进行分析研究。从研究中发现,野生树鼩与人工饲养树鼩在不同肠段菌群组成无论在门水平还是属水平均有差异,通过微生物功能预测发现,各肠段差异较大,对于了解该物种肠道微生物群的组成具有重要意义,也为树鼩作为新型实验动物提供基础数据。本研究基于成年树鼩不同胃肠道段样本,不能完全揭示整个生长周期胃肠道菌群的情况,将开展进一步的研究以进行完善。
