大腹园蛛主壶腹腺蛛丝蛋白末端结构域的克隆表达
2019-06-14贾秋品
贾秋品,温 睿,李 雪,孟 清
(东华大学生物科学与技术研究所,中国上海210620)
蜘蛛丝是具有极佳材料学性能的天然高分子材料,其延展性和强度可与已知合成材料相媲美,并且具有非常好的生物相容性和生物可降解性,在纺织、军事军工及生物医学领域具有重要的潜在应用价值[1~6]。由于蜘蛛同类相食的天性,不能通过养殖来获取大规模的蛛丝纤维,因此研究者转向通过基因工程的方法来获取人造蛛丝纤维[7]。自20世纪90年代以来,研究者对于蜘蛛丝的研究逐步深入并取得了一系列突破性的成果,比如:已成功获取多条蜘蛛丝的全基因编码序列;对蜘蛛成丝过程中的腺体环境变化进行了表征;对蜘蛛丝成丝机理提出了假设模型等[8~11],为蛛丝的仿生应用奠定了重要的理论基础。但是,人们在获取人造蛛丝纤维的过程中仍面临几个瓶颈问题:1)蛛丝蛋白编码序列的高度重复,造成全长基因获取困难[12~15],不能为蛛丝仿生提供足够的基因原材料,也为后续异源表达带来压力;2)蜘蛛丝成丝机理尚不明晰,目前认可的成丝模型有“小球模型”[9]和“液晶态模型”[16],但两者均未能完整解析成丝过程。这些问题是蛛丝仿生必备的理论基础,均需要进一步的深入探索。
主壶腹腺蛛丝(major ampullate silk)包括在圆网中起支撑作用的辐射状丝和作为蜘蛛生命绳索的牵引丝(dragline silk)。其中,牵引丝强度可达4 GPa,延展性高达35%,是蜘蛛丝研究中的热点[17]。主壶腹腺蛛丝的组成蛋白质为主壶腹腺蛛丝蛋白(major ampullate spidroin,MaSp),包括两个组分MaSp1和MaSp2[17]。现有研究表明,MaSp1和MaSp2在主壶腹腺蛛丝中的组成比例由蜘蛛的营养状况决定,并调控蛛丝的性能[18]。MaSp蛋白的相对分子质量可达250~350 kD[19]。目前已获得的MaSp全长编码基因有:黑寡妇蜘蛛(Latrodectus hesperus)的MaSp1和MaSp2[15](两序列相似性为64%)、横纹金蛛(Argiope bruennichi)的MaSp2[20]以及摩鹿加云斑蛛(Cyrtophora moluccensis)的 MaSp1s[21]。这些全长编码序列的获得为探索新蛛丝蛋白种类的结构和功能提供了新的方向。
完整的MaSp蛋白可划分为3个部分:末端结构域 N-terminal domain(NT)、C-terminal domain(CT))和中间高度重复区repetitive domain(RP)。RP对蛛丝的材料学性能起决定性作用[3],MaSp1的重复区序列中含有大量小重复单元(GA)n、(GGX)n、(GX)n及(A)n(X可以是A、L、Q等),而MaSp2的重复区因Pro的存在,使其除上述重复单元之外,还有(GPGXX)n重复单元[17]。在MaSp中大量(A)n所形成的β-折叠结构赋予了主壶腹腺蛛丝极高的强度[22]。越来越多的研究结果显示,NT和CT在蛛丝蛋白成丝过程中扮演着极其重要的角色,其结构功能解析是阐述成丝机理的关键步骤。2010年,Nature发表的两篇文章分别报道了NT和CT在成丝过程中的作用,一篇研究南非盗蛛(Euprosthenops australis)MaSp NT的3D结构和功能,显示NT以pH依赖的方式,通过三步二聚化机制,保证MaSp的快速成丝[23];另一篇聚焦十字园蛛(Araneus diadematus)MaSp CT的3D结构和功能,研究表明CT以二聚体方式存在,调控MaSp的成丝品质[24]。虽然不同蜘蛛种类的MaSp的NT和CT具有一定保守性,但仍存在一些差异,从而可能影响蛛丝纤维的性能,例如食用金丝蜘蛛(Nephila edulis)和E.australis的牵引丝相比,前者的弹性性能更好而后者具有更高强度[25~26]。因此,不同蜘蛛种属的MaSp NT和CT的编码基因获取及结构功能研究对生产获得高性能人造蛛丝是十分重要的。
1 材料与方法
1.1 材料和试剂
本研究使用的大腹园蛛基因从蜘蛛体内提取,蜘蛛采于上海东华大学松江校区园内,-80℃冻存蛛体。基因组DNA提取试剂、质粒DNA提取试剂、PCR产物回收试剂、胶回收试剂、SDS-PAGE电泳试剂、DNA电泳试剂均购自生工生物工程(上海)股份有限公司;Q5超保真DNA聚合酶购于美国NEB公司;酶切和连接反应相关试剂购自赛默飞世尔科技(中国)公司;Taq DNA聚合酶购自南京诺唯赞生物科技有限公司;Ni-NTA亲和纯化柱填料购自德国Qiagen公司;DNA引物合成和基因测序均在上海睿迪生物科技有限公司进行。
1.2 克隆构建
1.2.1 大腹圆蛛基因组DNA的提取
取冻存蜘蛛的胸部组织捣碎,参照改良的CTAB法[27]提取基因组DNA,将提取的核酸冻存以待PCR扩增实验使用。
1.2.2 A.v.NTMa1基因序列的获取
通过对NCBI基因库中的部分已知NT的蛋白质序列进行比对,找到其中一段含有11个氨基酸的保守序列(MAFASSXAEIA,其中X为V或M),取其中7个氨基酸(FASSVAE)对应的DNA序列设计5′方向引物 P5(引物序列见表1);根据NCBI已知MaSp的重复区 RP序列(GenBank:JN857964.2)设计3′方向引物 P3。以提取的大腹圆蛛基因组为模板,P5和P3为引物,通过PCR反应得到产物DNA1,回收并进行基因测序,此段基因序列中含有部分NT和部分RP编码基因序列。根据DNA1,设计3′方向的两个同向引物R1(靠近N端末尾)和 R2(在P5和R1之间)。以基因组DNA为模板,R1为单引物,进行PCR反应,得到包含全部NT编码序列的单链ssDNA2,随后回收产物并将其作为模板重新PCR,以获得足够的DNA产物;将ssDNA2进行末端脱氧核苷酸转移酶处理,以dCTP为原料进行polyC加尾处理,得到ssDNA2-CCCCCCC-3′;设计末端含polyG和其余随机碱基序列(来自其他物种,不与蛛丝基因序列互补)的5′方向引物 AL,以ssDNA2-CCCCCCC-3′为模板,以AL和R2为引物,进行锚定PCR反应,得到DNA3,回收并进行基因测序,此段基因序列中含有引物R2之前的NT编码基因剩余DNA序列。将DNA1和DNA3基因序列进行拼接,通过识别启动子密码和开放阅读框(open reading frame,ORF)的上游特征序列得到大腹圆蛛MaSp1 NT的完整基因序列A.v.NTMa1。
根据拼接得到的A.v.NTMa1基因序列设计5′和3′端引物PN5和PN3,以基因组DNA为模板,通过PCR反应得到不含信号肽的A.v.NTMa1DNA片段,回收产物并进行BamHⅠ和HindⅢ双酶切,得到含有两个黏性末端的NT-bh基因片段。
1.2.3 A.v.CTMa1基因序列的获取
依据NCBI上发表的大腹圆蛛MaSp1的CT序列(GenBank:AEV46833.2)设计 5′和 3′端引物PC5和PC3,通过PCR反应得到含有完整A.v.CTMa1基因的DNA片段,回收产物并进行BamHⅠ和HindⅢ双酶切,得到含有两个黏性末端的CT-bh基因片段(引物序列参见表1)。
“怀旧”,一方面是两岸听众的审美特征,而另一方面也反映出当代华语流行乐坛存在的问题。如当前各个经纪公司在筹划歌手个人演唱会时,都在打“怀旧”牌,列出该歌手多年来唱过的经典曲目以增加演唱会的市场号召力,让歌迷到现场“见证青春岁月”。[29]这一方面证明了这些流行歌手的影响力,另一个方面也显示了整个流行乐坛一定程度上的青黄不接。[29]
1.2.4 A.v.NTMa1和A.v.CTMa1的表达克隆构建
对改造后的pET-32a质粒(Novagen)进行BamHⅠ和HindⅢ双酶切,回收载体大片段,并与NT-bh和CT-bh分别在T4 DNA连接酶作用下进行连接反应,得到含有完整A.v.NTMa1和A.v.CTMa1基因序列的重组克隆质粒,通过酶切和基因测序验证基因序列正确。
1.3 序列比对及二级结构预测
在NCBI网站(https://www.ncbi.nlm.nih.gov/nuc-core/)搜索已发布的不同蜘蛛种类的NT氨基酸序列进行比对分析;通过PSIPRED网站(http://bioinf.cs.ucl.ac.uk/psipred/)对已获得的A.v.NTMa1和A.v.CTMa1蛋白序列进行二级结构预测。
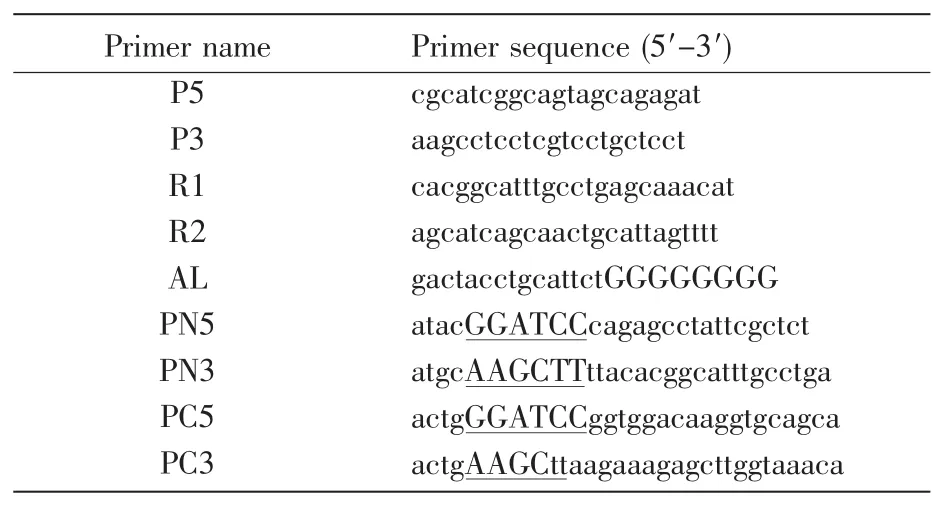
表1 引物序列Table 1 Primer sequences
1.4 重组蛋白的表达与纯化
将含有正确基因序列的质粒转化到大肠杆菌E.coli BL21(DE3)表达宿主菌株(北京天根生化科技有限公司)。30℃200 r/min条件下,在含有70 μg/mL卡那霉素的LB培养基中将大肠杆菌培养至OD600为0.8~1.0,加入终浓度为0.3 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropylthio-β-D-galactoside,IPTG),在20℃180 r/min条件下过夜培养诱导蛋白质表达。4℃条件下6 000 r/min离心20 min收集菌体,20 mmol/L Tris-HCl缓冲液(pH 8.0)重悬,随后使用高压细胞破碎仪JN-3000(广州聚能纳米生物科技股份有限公司)破碎,将裂解物离心(6 000 r/min,20 min,4℃),收集上清。含有目的蛋白的上清液通过Ni-NTA柱(Qiagen公司,德国)进行亲和层析纯化。将蛋白质溶液装入镍柱内,低温孵育1 h后穿柱,使用3倍柱体积的洗涤缓冲液(含10 mmol/L咪唑的20 mmol/L Tris-HCl缓冲液,pH 8.0)洗柱两次,适量洗脱缓冲液(含300 mmol/L咪唑的20 mmol/L Tris-HCl缓冲液,pH 8.0)洗脱柱上蛋白质,4℃过夜透析(20 mmol/L Tris-HCl缓冲液,pH 8.0),得到纯化后的重组蛋白。
1.5 圆二色谱分析
将目的蛋白稀释到pH 7.5的20 mmol/L磷酸缓冲液中,至终浓度为0.5 mg/mL左右。在25℃室温条件下,通过CD光谱仪(Applied Photophysics Ltd.,英国),收集扫描波长190~260 nm的光谱信号。具体参数设置:光程0.1 cm,扫描步长0.5 nm,响应时间1 s,带宽1 nm。CD光谱取3次扫描的平均值。
2 结果
2.1 A.v.NTMa1和A.v.CTMa1序列获取及序列分析
通过锚定和常规PCR的方法,依次得到DNA1、ssDNA2、DNA3(图 1A),最后将 DNA1 和DNA3进行拼接得到包含完整NT和部分重复区RP编码基因序列在内的片段。通过设计两对特定上下游引物,并分别通过常规PCR扩增的方法得到两端含有BamHⅠ、HindⅢ限制性酶切位点的A.v.NTMa1(图 1A-d)和 A.v.CTMa1(图 1B)片段,以用于后续表达克隆的构建。
在A.v.NTMa1基因的获取过程中,以基因组为模板和特异性下游引物进行单引物扩增获取的目标基因ssDNA2的量非常低,因此以该扩增产物为模板进行两轮常规PCR(图1A-b),以实现目标基因的富集。此外,根据拼接后序列设计特异性引物,进行基因组PCR验证,保证了该拼接编码序列的准确性,翻译后氨基酸序列如图2所示。分析该序列的重复区RP序列可见,其中基本不含有GPGXX模块,因此初步确定该序列属于A.v.MaSp1。
为进一步确定上述MaSp1序列的可靠性,将A.v.NTMa1(图3中第一条序列)与已发表的13条MaSp NT的氨基酸序列(表2)进行序列比对分析。结果表明:不同蜘蛛种属间,MaSp NT序列相似性可达50%以上,具有较高的保守性;MaSp1 NT之间的保守性可达60%。同时,对上述序列进行系统进化树分析,发现A.v.NTMa1与其他已报道的MaSp NT具有较近的亲缘关系(图4)。由此可见,本次获取的A.v.NTMa1编码基因具有很高可靠性,适用于后续的研究。利用SignalP 4.1对A.v.NTMa1序列进行信号肽预测,发现该序列中存在信号肽序列(MTWTARLALSLLAVICS),因此在后续的克隆阶段,将信号肽去除,得到A.v.NTMa1的氨基酸序列(图2红色序列)以用于后续实验。
A.v.CTMa1基因翻译后得到的氨基酸序列为GGQGAASSAAAASAAASRLSSPSAASRVSSAVSSLVSSGGPSSPAALSSTISNVVSQISASNPGLSGCDVLVQALLEIVSALVHILGSANIGQVNSSAAGQSASLVGQSVYQALS。
2.2 A.v.NTMa1和A.v.CTMa1的表达纯化
分别将正确的A.v.NTMa1和A.v.CTMa1基因序列插入到pET-M质粒载体中,该载体是改造过的pET-32a系列载体(含有用于后续亲和纯化的6×His标签)。随后,在大肠杆菌中分别对两种蛋白质进行外源表达,利用镍柱对表达产物进行亲和纯化,结果如图5所示。其中CT为A.v.CTMa1蛋白(11.8 kD),NT为A.v.NTMa1蛋白(15.2 kD)。两个重组蛋白的纯度均远高于90%,产量分别为80 mg/L和60 mg/L。

图1 A.v.NTMa1和A.v.CTMa1基因序列的PCR扩增(A)A.v.NTMa1的PCR扩增结果。DNA1:多条带,其中包含部分NT和部分RP的编码基因序列。泳道1~3:以基因组为模板的PCR产物。ssDNA2:1~3产物回收后重新扩增的结果,单链DNA含有全部NT编码基因序列。DNA3:含有部分NT编码基因序列,530 bp。NT:A.v.NTMa1基因,425 bp;(B)A.v.CTMa1的PCR扩增结果(CT:366 bp)。泳道M均为DNA ladder。Fig.1 The PCR amplification of A.v.NTMa1and A.v.CTMa1gene sequences(A)The PCR amplification of A.v.NTMa1.DNA1:The DNA sequences of the bands contain partial NT and a part of RP.Lanes 1~3:PCR products from genomic DNA.ssDNA2:The results re-amplified from the original lanes 1/2/3 products,whose singlestrained DNA contains the whole NT gene sequence.DNA3:The 530 bp-band has a part of NT gene sequence.NT:The 425 bpband A.v.NTMa1;(B)The PCR amplification of A.v.CTMa1(CT:366 bp).All the M lanes represent DNA ladder.

图2 大腹园蛛MaSp1的部分氨基酸序列氨基酸总长为584 aa。BN:14 aa,为N端之前的序列;NT:151 aa,为A.v.NTMa1序列(红色),下划线部分为信号肽(17 aa);RP:419 aa,为部分重复区序列。Fig.2 The partial amino acid sequence of the A.v.MaSp1The overall A.v.MaSp1 is 584 amino acids.BN:The first 14 aa in N-terminal domain;NT:151 aa including the 134 aa A.v.NTMa1(in red)and the 17 aa signal peptide(in indigo blue and underlined);RP:Partial repetitive domain(419 aa).
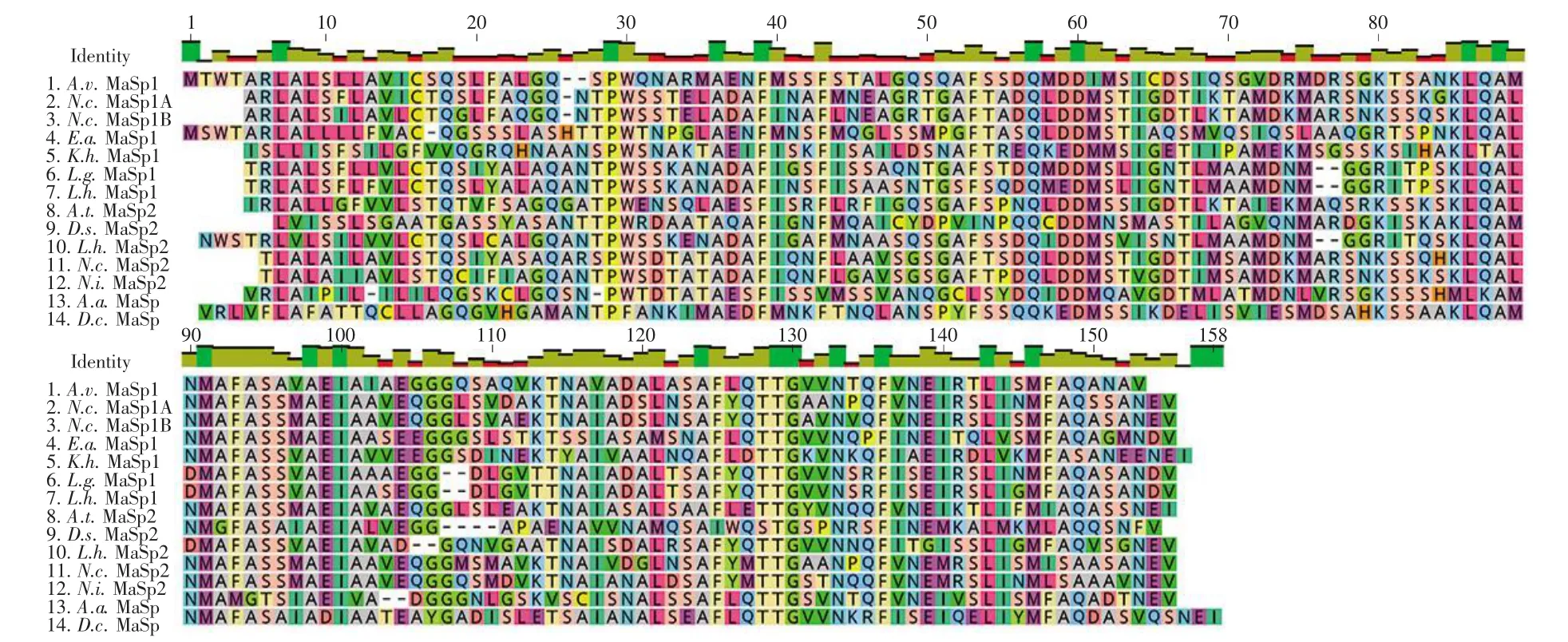
图3 不同蜘蛛种属MaSp NT氨基酸序列的比对分析Fig.3 The sequence alignment of MaSp NT from different spider species
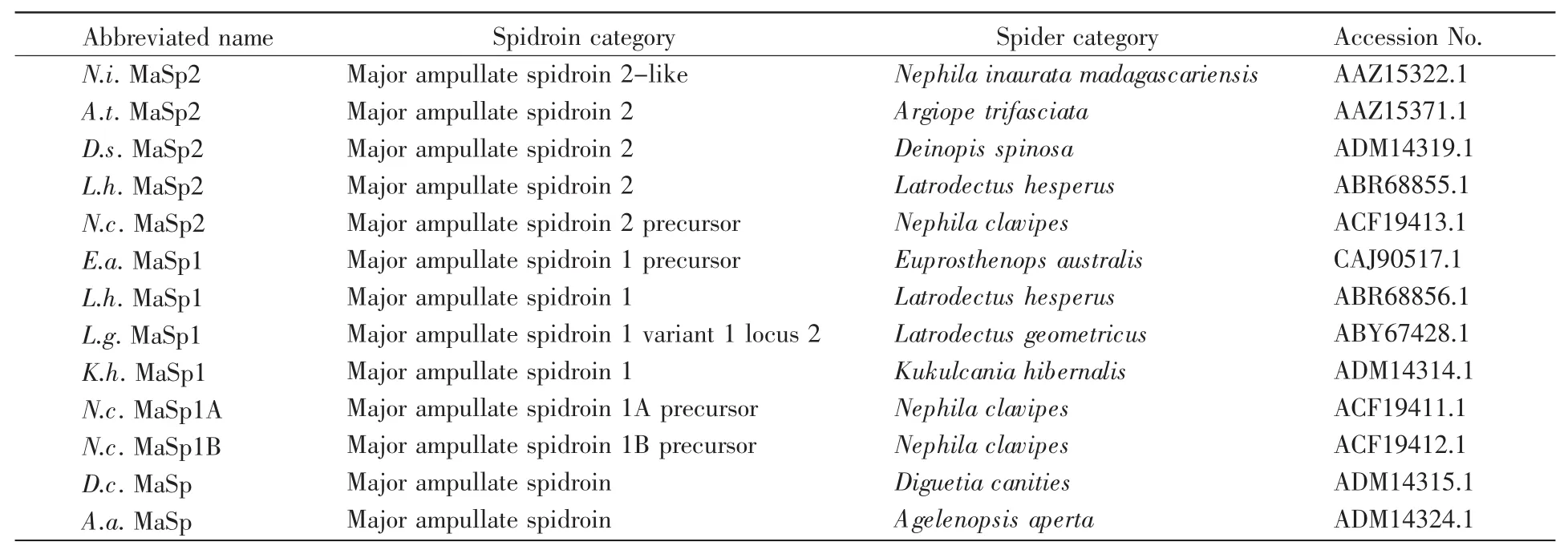
表2 已报道的MaSp NT序列Table 2 The reported MaSp NT sequences

图4 不同种属蜘蛛MaSp NT的种系发生树Fig.4 The phylogenetic tree of MaSp NT from different spider species
2.3 A.v.MaSp1 NT和CT的二级结构预测及CD色谱分析
利用PSIPRED网站对A.v.NTMa1和A.v.CTMa1的二级结构进行预测,结果显示两者均以α-螺旋二级结构为主要构象,可形成5个α-螺旋(图6),这与已发表的MaSp NT和CT的结构是一致的。利用CD光谱对两者的二级结构进行分析,结果如图7所示,在25℃、pH 7.5条件下,A.v.NTMa1和A.v.CTMa1的CD曲线均在208 nm和222 nm附近有两个负峰,表明两者的二级结构组成均以α-螺旋为主要构象,与PSIPRED网站的二级结构预测结果相符合。
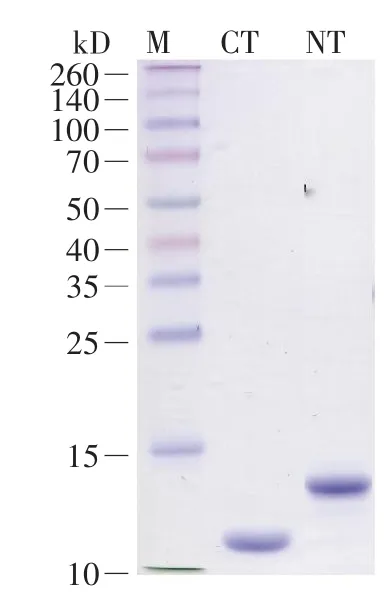
图5 纯化的大腹园蛛MaSp1重组NT和CT蛋白的SDS-PAGE图谱M:蛋白质marker;NT:纯化后的A.v.NTMa1蛋白;CT:纯化后的A.v.CTMa1蛋白。Fig.5 SDS-PAGE analysis of the purified target proteinsM:Protein marker;NT:Purified A.v.NTMa1;CT:Purified A.v.CTMa1.
3 讨论
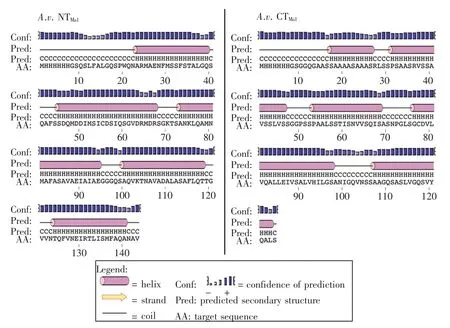
图6 A.v.NTMa1和A.v.CTMa1的二级结构预测Fig.6 The secondary structure prediction of A.v.NTMa1and A.v.CTMa1
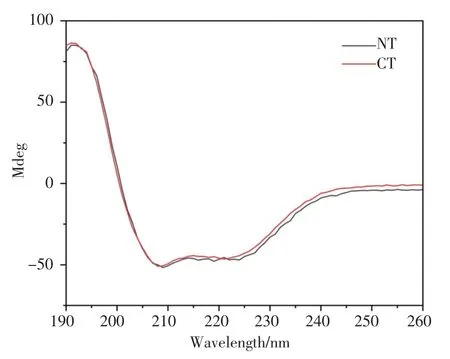
图7 A.v.NTMa1和A.v.CTMa1的CD 图谱Fig.7 The CD spectra of A.v.NTMa1and A.v.CTMa1
蛛丝蛋白的NT和CT结构域在其成丝过程中扮演着重要的角色,对两者结构和功能的探索是阐述成丝机理的先决条件。目前仅有E.australis和络新妇蛛(Nephila clavipes)MaSp NT的高级结构被报道[23,26],相关结果表明MaSp NT单体的二级结构包含5个α-螺旋,且蛛丝蛋白在腺体内以单体可溶状态高浓度储存。而对蜘蛛丝成丝过程的研究显示,随着pH逐渐降低和其他理化条件的改变,蛛丝蛋白的单体以同向平行的方式形成二聚体,并经由保守氨基酸发生“三步”质子化,最终形成稳定的二聚体结构,帮助蛛丝蛋白快速成丝[28~29]。对于 MaSp CT,目前有 A.diadematus的MaSp1 CT被报道,研究表明MaSp1 CT依赖疏水作用力形成二聚体,以反向平行方式存在,调控MaSp的成丝过程,使丝纤维具有更为规则的结构[24]。而对于A.v.NTMa1和A.v.CTMa1结构与功能方面的研究却非常少,这限制了人们对大腹园蛛MaSp成丝机理的探索和仿生应用。
为解决这一问题,我们通过设计特定引物,使用锚定PCR和常规PCR反应的方法成功获取了大腹园蛛MaSp1的A.v.NTMa1和A.v.CTMa1的完整编码基因。其中,在利用锚定PCR方法获取A.v.NTMa1完整编码基因时,为了确认A.v.NTMa1的准确性,我们进行了基因组PCR验证,保证了后续蛋白质表达和结构表征的准确性;同时,利用序列比对分析和进化树分析,确定了A.v.NTMa1基因序列的可靠性。A.v.NTMa1和A.v.CTMa1的CD图谱表明,其二级结构均以α-螺旋为主,与已发表的MaSp的NT和CT的二级结构保持一致。此外,在不同 pH(7.5、6.5、5.5)条件下,A.v.NTMa1和A.v.CTMa1的CD曲线并没有检测到明显变化(图片未展示),但是其高级结构很可能发生改变,需要进一步的实验证明。本文对A.v.NTMa1和A.v.CTMa1的二级结构进行了初步的探索,分析了其二级结构的主要组分,可为后续A.v.NTMa1和A.v.CTMa1的结构和功能探索提供一定的数据支持和理论依据。
通过序列比对分析可知,A.v.NTMa1与其他MaSp NT具有保守性,同样包含一个W残基,因此可通过检测W荧光变化[30]分析A.v.NTMa1的二聚化机制,这是后续探索A.v.NTMa1结构功能的有效途径。然而与其他已知MaSp NT序列不同的是,A.v.NTMa1仅含有一个Cys残基,由此Cys巯基形成的分子间二硫键对于A.v.NTMa1的二聚体结构稳定或在成丝过程中可能具有一定的影响,需要进一步研究。另外,若该二硫键仅用于辅助二聚化结构的稳定性,则可将该Cys进行荧光分子标记,该Cys位于A.v.NTMa1的H2螺旋中,而H2属于NT二聚体结构的相互作用界面,因此可通过检测荧光信号的动态变化,分析不同条件下A.v.NTMa1的二聚体结构变化,探索A.v.NTMa1的二聚化机制或在成丝过程中所扮演的角色[29]。总的来说,A.v.NTMa1的二聚化机制需要更加深入的探索,以帮助阐述MaSp1的成丝机理。
已有文献报道蛛丝蛋白的CT具有较高的序列保守性,即同源性高[31]。但研究显示,不同种类蜘蛛的同种蛛丝蛋白的CT,虽具有较高的保守性和相似的3D结构,其功能却有所区别。例如:金圆网蛛(Nephila antipodiana)的葡萄状腺蛛丝蛋白(aciniform spidroin,AcSp)的CT对其重复区RP在成丝过程中的结构转变无明显影响;而三带金蛛(Argiope trifasciata)的AcSp CT则在成丝过程中对重复区RP的β-折叠的形成起到晶核作用,并可提高丝纤维的机械性能[32~33]。所以,对 A.v.CTMa1的结构和功能进行探索仍是必需的,也利于系统研究A.v.MaSp的成丝机理。另外,有研究表明,N.clavipes的MaSp具有热敏感性,在低温2℃或高温65℃下可形成凝胶,并具有一定的可逆性[34]。据此,我们可以在后续的研究中对A.v.CTMa1的热稳定性和形成凝胶特性进行探索,以拓宽其应用。
综上所述,本文通过锚定PCR成功获取了大腹园蛛MaSp1 NT的编码基因,为研究MaSp提供了基因素材,并成功进行了外源表达纯化,表达量可达60 mg/L;同时克隆表达了大腹园蛛的MaSp1 CT,其表达量可达80 mg/L。A.v.NTMa1和A.v.CTMa1的CD图谱显示,两者的二级结构以α-螺旋为主要组分。上述研究结果为大腹园蛛MaSp1 NT和CT的结构及功能研究提供了基因素材和一定数据支持,也为大腹园蛛MaSp1的成丝机理研究奠定了基础。
