一个CRISPR/Cas9-VQR基因编辑系统的构建
2019-05-31孙国梁宋高原李爱丽谢传晓耿帅锋
陈 凯 孙国梁 宋高原 李爱丽 谢传晓 毛 龙 耿帅锋
中国农业科学院作物科学研究所, 北京 100081
作为新一代基因组编辑技术, 与 ZFN (Zinc Finger Nucleases)、TALEN (Transcriptional Action Like Effector Nucleases)技术相比, 具有操作简便、切割效率高、靶位点多等优点, 在模式植物和作物中得到广泛的应用[1]。CRISPR/Cas9系统在 gRNA的引导之下, 将Cas9带到目的基因上, 利用RuvC和HNH切割DNA双链, 形成双链断裂结构(Double Strand Break), 然后生物体内会在修复DSB过程中引起一些碱基的随机插入或缺失, 从而产生移码突变[2-3]。虽然CRISPR/Cas9相对于其他基因编辑技术具有较高的编辑效率, 但对不同物种或同一物种中不同基因的编辑效率却不相同[4-5]。在拟南芥中, 研究人员以AtFT、AtSPL4、AtBRI1、AtJAZ1、AtADH1、AtFLS2和AtPDS3等为目的基因验证 CRISPR/Cas9的功能,其切割效率为1.10%~84.78%[6-10]。在烟草、水稻、大豆、西红柿、高粱、玉米、棉花、小麦和药用植物丹参中的切割效率分别为 1.8%~87.5%[6,11-13]、2.1%~100.0%[8,14-21]、14.7%~20.2%[22]、83.56%[23]、33.33%[24]、13.1%[25]、47.6%~81.8%[26]、5.6%[27]和 42.3%[28]。
根据现有文献报道, 影响 CRISPR/Cas9系统编辑效率的因素, 主要包括Cas9基因密码子的优化、Cas9基因启动子的替换、gRNA启动子的替换和gRNA的靶标数目[29-32]。在拟南芥、烟草、番茄、大豆、水稻和玉米中, 都有优化Cas9基因密码子的报道, 但只有单子叶植物中的优化能够提高编辑效率[29,31]。利用植物内源组成型启动子, 如 ZmUbi、OsUBQ和OsActin1, 启动Cas9基因, 也能获得较高的编辑效率[15,30,33]。在单子叶植物中, 利用植物内源的U6或者U3启动子启动gRNA更有利于基因编辑[31,34]。在拟南芥和玉米中, 串联 2个gRNA比单个 gRNA的编辑效率高[31-32]。在水稻中, 分别设计 1、2、3个 gRNA靶向一个基因, 结果表明靶向同一个基因的多个位点时编辑效率提高[35]。另外,CRISPR/Cas9的PAM位点NGG, 也在一定程度上限制了其使用范围。虽然在哺乳动物细胞中 CRISPR/StCas9可识别NNAGAA PAM位点, CRISPR/SaCas9可识别 NNGGGT、NNGAAT和 NNGAGT PAM 位点[36-37], 但这些CRISPR系统在植物中报道较少。在水稻中, 通过将 SpCas9突变, 可以识别 NGA、NGAG和 NGNG PAM 位点, 但是 CRISPR/Cas9-VQR识别 NGA PAM 位点的切割效率仅为 2.1%~23.4%, 低于原有 CRISPR/Cas9系统[38]。通过优化gRNA结构和使用水稻内源强启动子启动gRNA可以提高CRISPR/Cas9-VQR的切割效率, 但对影响编辑效率的其他因素的改造还未见报道[39]。
本研究通过突变SpCas9编码氨基酸、使用玉米Ubi启动子启动Cas9-VQR蛋白、优化SpCas9蛋白、加入核定位信号序列、增加单子叶中保守的 3′ UTR序列、使用水稻U6 启动子启动gRNA, 构建了一个能够识别 NGA PAM 位点, 并进行有效切割的CRISPR/Cas9-VQR基因编辑系统, 扩大了CRISPR/Cas9的使用范围。
1 材料与方法
1.1 植物材料
水稻(Oryza sativa)材料日本晴(Nipponbare) 由中国农业科学院作物科学研究所吴传银老师提供。
1.2 实验试剂
DNA 限制性内切酶、PCR扩增酶、T4-DNA连接酶、qRT-PCR荧光酶均购自TaKaRa公司; 反转录试剂盒 SuperScript III Reverse Transcriptase、RNA 提取试剂TRIzol Reagent均购自Invitrogen公司; 卡那霉素、X-gal、IPTG等购自Simga公司。其他化学试剂如氯仿、异丙醇等购自北京化工公司。实验中所用到引物均由生工生物工程(上海)股份有限公司合成。
1.3 CRISPR/Cas9密码子改造及优化
CRISPR/Cas9-VQR质粒由实验室和北京唯尚立德生物科技有限公司合作构建, 主要利用PCR定点突变技术, 将SpCas9序列进行突变, 进而改变编码氨基酸, 从而改变CRISPR/Cas9载体识别的PAM位点。同时通过基因合成将哺乳动物里应用的CRISPR/Cas9载体进行植物密码子优化, 并加入核定位序列、增加单子叶中 3′ UTR序列、使用玉米Ubi启动Cas9-VQR转录、水稻U6启动gRNA转录,构建双元表达载体系统。
1.4 gRNA靶位点引物设计
以水稻 (Oryza sativa)八氢番茄红素脱氢酶基因(phytoene desaturase, PDS)为靶基因[27], 根据靶位点设计的原则, 在OsPDS基因不同外显子上筛选并设计了4个长度为 23 bp的靶位点序列, 分别命名为 gRNA1、gRNA2、gRNA3 和 gRNA4 (表 1)。
1.5 Cas9/VQR-gRNA体外酶切活性检测
利用 Cas9/VQR-gRNA靶点效率检测试剂盒(北京唯尚立德生物科技有限公司)检测 4个 gRNA对靶位点的切割效率。首先, 参考试剂盒分别合成gRNA1、gRNA2、gRNA3 和 gRNA4 引物(表 2); 然后以标准 gRNA模板为模板, 分别通过 PCR扩增gRNA1、gRNA2、gRNA3和gRNA4及试剂盒提供的标准gRNA (S1和S2), 回收纯化PCR产物作为DNA模板分别进行体外转录, 回收并定量转录 gRNA;其次, 通过搭桥PCR的方式, 将gRNA靶位点(包括PAM 序列) 构建到 PCR产物中, 获得用于酶切的DNA; 最后, 参照试剂盒提供的 Cas9/VQR 酶切体系, 加入 2 μL 10 × Cas9 buffer、1 μL Cas9/VQR 酶、50 ng体外转录的 gRNA、50 ng酶切的 DNA、补DEPC H2O至20 μL, 充分混合后, 37℃反应 30 min,然后加入3 μL DNA Loading buffer, 混合后65℃ 5 min, 2%琼脂糖凝胶电泳检测分析酶切结果。此外,每次酶切实验的同时做标准靶位点的gRNA酶切实验, 以标准靶位点酶切实验结果作为阳性对照比较活性。通过与2个阳性参照标准S1和S2估算gRNA的活性。根据SSA活性, 标准S1=3, 标准S2=10, 通过酶切条带的灰度与标准S1和标准 S2对比, 换算出 gRNA1 (g1)、gRNA2 (g2)、gRNA3 (g3)、gRNA4(g4)的活性(若检测gRNA酶切条带的灰度小于标准S1的灰度, 则活性估值小于 3; 若检测 gRNA酶切条带的灰度大于标准S1的灰度, 且小于标准S2的灰度则活性估值大于3小于10; 若检测gRNA酶切条带的灰度大于标准 S2的灰度, 则活性估值大于 10)。
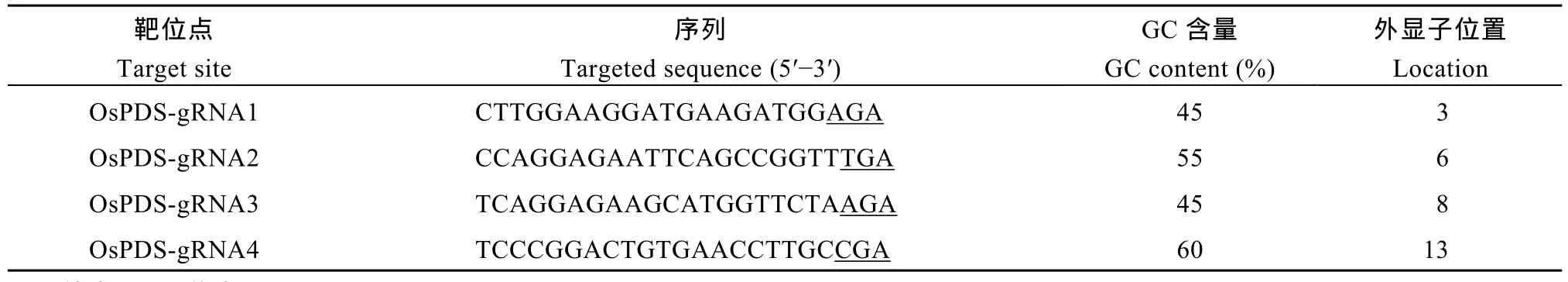
表1 OsPDS-gRNAs序列Table 1 Sequence of OsPDS-gRNAs

表2 本研究所用引物Table 2 Primers used in this research
1.6 水稻转化
依据4对gRNA体外酶切活性检测结果, 选取3对分别设计 oligo引物(表 2), 通过正义链和反义链gRNA的oligo引物的退火反应, 形成oligo二聚体,并插入CRISPR/Cas9-VQR载体, 转化大肠杆菌, 进而挑选阳性克隆测序, 获得构建好的 CRISPR/Cas9-VQR-gRNA载体, 参照Hiei等的方法[40], 通过液氮冻融法将 CRISPR/Cas9-VQR-gRNA载体转入农杆菌菌株 EHA105感受态细胞中, 涂板挑取单克隆并进行鉴定, 将阳性克隆用于水稻转化。选用日本晴作为转化受体, 将预培养的水稻愈伤组织与农杆菌共培养, 并将共培养后的愈伤组织转移至NB1S1筛选培养基上继续培养, 获得转基因植株。
1.7 阳性植株鉴定
采用 CTAB 法提取转化水稻叶片全基因组DNA。通过特异性引物 PCR, 以提取的转基因材料DNA为模板, 鉴定阳性转基因植株。引物见表2。
2 结果与分析
2.1 CRISPR/Cas9 密码子优化
植物体内翻译中对G和C有一定的偏好性, 以水稻为模型, 对野生型SpCas9基因进行密码子优化,优化后Cas9中碱基G含量由原来的733个增加到1094个, 碱基C由原来的566个增加到931个, GC总含量由31.56%增长到49.20%, 提高了SpCas9 GC碱基的含量(表3)。
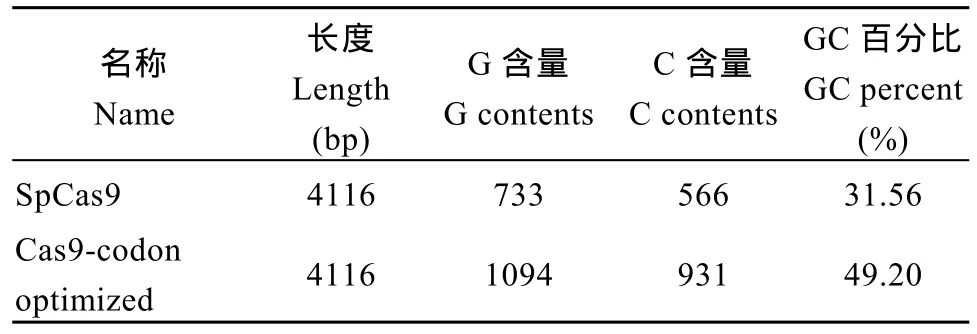
表3 Cas9密码子优化结果Table 3 Result of Cas9 codon optimized
2.2 CRISPR/Cas9 PAM位点分布
利用生物信息学技术, 通过对水稻和小麦的CDS区域NGG、NGA、NGC和NGT PAM位点的统计分析, 发现水稻和小麦中 NGA PAM 位点数量均多于传统NGG PAM位点。水稻中NGA PAM位点比NGG位点多出大约330,000个, 约占NGG位点的12.34%。小麦中NGA PAM位点比NGG多出大约1,300,000个位点, 约占NGG位点的24.78%。因此通过改造SpCas9, 使其能够识别NGA PAM位点, 可以有效增加该基因编辑技术的使用范围, 尤其是在水稻和小麦中(图1)。
2.3 CRISPR/Cas9载体改造
利用PCR定点突变技术, 将传统的SpCas9基因序列进行突变, 改变编码氨基酸: 由 1135位的天冬氨酸(D)突变为缬氨酸(V), 由1335位的精氨酸(R)突变为谷胱氨酸(Q), 由 1337位的苏氨酸(T)突变为精氨酸(R), 命名为Cas9-VQR (图2)。从而使CRISPR/Cas9-VQR的PAM识别位点变为NGA。
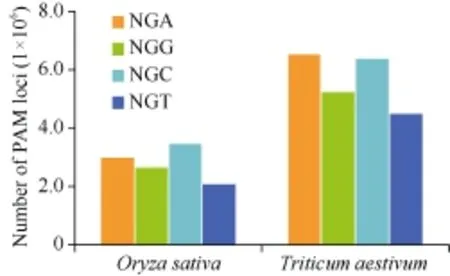
图1 水稻和小麦全基因组CDS中PAM位点的数量Fig. 1 Number of PAM loci in whole genome CDS of rice and wheat
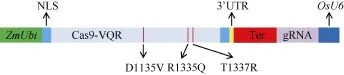
图2 CRISPR/Cas9-VQR载体Fig. 2 Construction of CRISPR/Cas9-VQR vector
同时, 根据影响基因编辑效率的因素, 对载体进行修饰, 在 Cas9-VQR序列两端加上保守的核定位序列, 增加单子叶中保守的 3′UTR序列, 使用玉米 Ubi启动子启动 Cas9-VQR蛋白, 优化的Cas9-VQR蛋白和使用水稻 U6启动子表达 gRNA(图 2)。
2.4 CRISPR/Cas9-VQR 蛋白活性和体外酶切活性检测
为验证Cas9-VQR的蛋白活性, 我们通过Cas9-VQR蛋白切割标准样(S1, S2)检测, 发现该 Cas9-VQR具有酶切活性, 可以有效地切割标准样(S1,S2)(图3), CK1和CK2为未加gRNA的对照。经过SSA luciferase检测活性, 发现S1和S2的SSA活性分别为3和10。
在水稻PDS基因第3、第6、第8和第13外显子上, 分别设计gRNA (表1), 然后分别进行体外酶切活性检测。与对照S1和S2相比, g1、g2、g3、g4靶位点均有活性, 但活性强度有所不同, g1和g2靶位点最好, 基本和阳性对照一致, 而g4靶位点较弱,g3靶位点最差(图3)。根据酶切条带灰度换算gRNA的活性, g1和g2体外酶切活性约为70%, g3体外酶切活性约为5%, g4体外酶切活性约为30% (图4)。
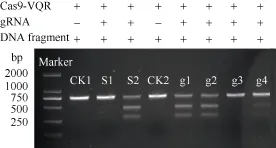
图3 体外酶切检测活性结果Fig. 3 Cas9-VQR enzyme digestion activity in vitro
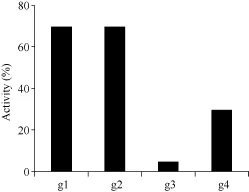
图4 OsPDS-gRNA体外活性检测结果Fig. 4 OsPDS-gRNAs activity in vitro
2.5 转化植株的表型观察及突变位点检测
根据 4个 gRNA的体外酶切活性, 挑选其中 3个(gRNA1、gRNA2和 gRNA4)插入 CRISPR/Cas9-VQR载体, 转化水稻。获得OsPDS-gRNA1表达载体转化再生植株 24个, 其中 17株阳性植株, 约占70.8%; 获得 OsPDS-gRNA2表达载体再生植株 45个, 其中阳性植株为 40株, 约占 88.89%; 获得 53株 OsPDS-gRNA4水稻再生植株, 其中阳性植株为49株, 约占92.45%。
由于OsPDS基因为剂量基因, 所以突变植株因突变发生时期不同而表现不同的表型。若Cas9-VQR切割OsPDS基因是在早期第一个胚胎细胞发育还没分化之前, 就会产生纯合白化苗(图 5-A)。若Cas9-VQR切割OsPDS基因是在分化后的细胞中,则会产生绿色和白色相间表型, 该表型在叶片、叶鞘和穗部都会出现(图5-B, C)。
通过对不同的靶位点区域片段的测序, 发现OsPDS-gRNA1产生缺失-28 bp到插入+19 bp等不同类型的突变, 共检测到 12株突变植株, 突变效率为 70.59%, 其中 4株为纯合突变, 约占 33.33%;OsPDS-gRNA2产生缺失-11 bp到插入+10 bp等不同类型的突变, 共检测到11株突变植株, 突变效率为27.5%, 其中纯合突变植株为5株, 约占45.45%;OsPDS-gRNA4产生缺失-13 bp到插入+9 bp等多种类型的突变, 共检测到 20株突变植株, 突变效率为40.8%, 其中7株为纯合植株, 约占35% (图6和表5)。
3 讨论
传统的 CRISPR/Cas9系统虽然应用广泛, 但受NGG PAM位点的限制。CRISPR/Cas9-VQR能够识别NGA PAM位点, 并进行有效切割, 极大地扩大了CRISPR/Cas9系统的使用范围。本研究通过突变Cas9序列和修饰基因编辑载体, 获得了一个能够识别 NGA PAM 位点, 并有效切割靶基因的基因编辑系统, 为该编辑系统在 NGA PAM 位点较多作物中的应用提供了借鉴。

图5 表型观察Fig. 5 Phenotype observation

图6 靶位点突变类型Fig. 6 Target site mutation
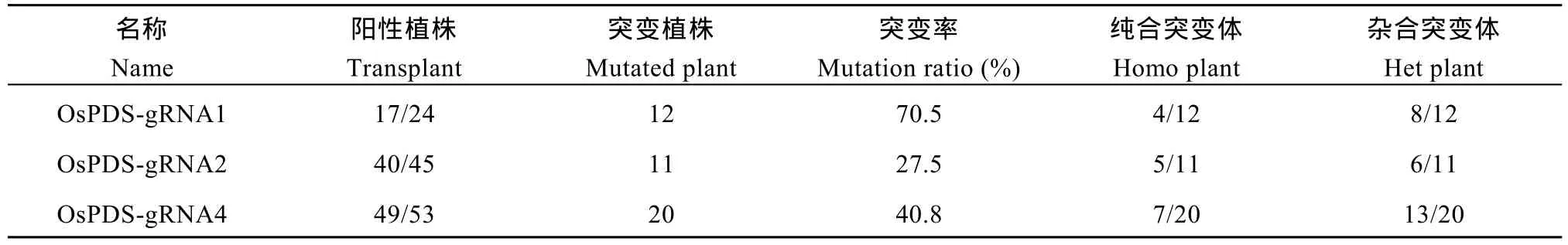
表5 转基因植株突变体数量统计Table 5 Number of mutants among transgenic plants
在哺乳动物细胞中, 虽然有 CRISPR/StCas9识别NNAGAA PAM位点及CRISPR/SaCas9识别NNGGGT、NNGAAT和NNGAGT PAM位点的报道[36-37], 但在植物中却较少。在水稻中, 通过突变SpCas9改造的CRISPR/Cas9-VQR, 其切割效率约为2.1%~23.4%, 相对较低[38]。通过优化CRISPR/Cas9-VQR的gRNA结构和使用内源强启动子启动gRNA,提高了 CRISPR/Cas9-VQR的切割效率[39], 但并未考虑影响编辑效率的其他影响因素。本研究除了对Cas9蛋白进行改造, 还对影响编辑效率的因素进行修饰, 获得了一个能够识别NGA PAM位点, 并进行有效切割的基因编辑系统。
传统的CRISPR/Cas9系统在水稻的切割突变效率为2.1%~100.0%[8,14-21], 突变效率变化较大。其中使用玉米Ubi作为SpCas9的启动子, 其突变效率为4%~100%[19,21], 而使用 35S作为启动子, 目的基因的突变效率为2.1%~75.0%[8,14-18,20], 相对较低。针对相同基因, 例如OsSWEET11/14, 使用玉米Ubi作为启动子, 突变效率为 87%~100%, 平均突变效率为93.5%[19], 而使用 35S启动子, 突变效率为66.67%[24]。这可能是由于35S启动子在单子叶植物中容易被甲基化修饰[41], 从而影响SpCas9的转录水平。本实验中使用玉米Ubi作为SpCas9-VQR的启动子, 虽然获得46.23%的平均突变效率, 但玉米Ubi启动子的作用还需设置相应的对照进行深入研究。
另外, 我们在载体构建中的修饰, 如对 Cas9-VQR进行密码子优化、增加单子叶植物中保守的 3′UTR 序列和使用 OsU6 启动子启动 gRNA转录,都会对系统的编辑效率产生影响, 我们将进一步研究这些修饰对编辑效率的影响。
4 结论
建立了有效识别 NGA PAM 位点的 CRISPR/Cas9-VQR编辑系统, 并扩大了其使用范围, 为后期在水稻等高NGA PAM位点作物中的应用提供了参考。
