基于90K芯片SNP标记的小麦遗传图谱构建及抗纹枯病QTL定位
2019-04-06崔德周李永波樊庆琦隋新霞黄承彦楚秀生
崔德周 李永波 樊庆琦 隋新霞 黄承彦 楚秀生


摘要:为挖掘定位小麦抗纹枯病QTL,以莱州953×山农辐63的F 2∶ 3为作图群体,用Illumina Wheat 90K芯片检测F 2单株的基因型,并用QTL IciMapping 4.1软件绘制该群体的遗传连锁图谱;在自然发病条件下,鉴定分离群体的抗、感表型,利用QTL IciMapping 4.1进行抗纹枯病QTL定位分析。结果表明,构建的小麦遗传连锁图谱包含21个连锁群,覆盖了小麦的21条染色体,图谱总长度5 528.12 cM,平均图距5.25 cM;共检测到6个分布于小麦1A、1B、2A、3A、7A和7D染色体上的加性QTL位点,单个QTL的贡献率为3.24%~10.37%。该结果可为小麦抗纹枯病QTL精细定位与相关基因克隆奠定基础,也为小麦抗纹枯病分子标记辅助选择育种提供了理论依据。
关键词:小麦;遗传连锁图谱构建;纹枯病;QTL定位;90K芯片SNP标记
中图分类号:S512.103.53文献标识号:A文章编号:1001-4942(2019)02-0013-05
Construction of Wheat Genetic Linkage Map Based on 90K SNP
Array and Mapping QTLs for Sharp Eyespot Resistance
Cui Dezhou Li Yongbo Fan Qingqi Sui Xinxia Huang Chengyan Chu Xiusheng1,2
(1. Crop Research Institute, Shandong Academy of Agricultural Sciences/Key Laboratory of
Wheat Biology and Genetic Improvement in the North Yellow﹠Huaihe River Valley, Ministry of Agriculture/
National Engineering Laboratory for Wheat and Maize, Jinan 250100, China;
2. College of Life Sciences, Shandong Normal University, Jinan 250014, China)
AbstractIn order to map the QTLs associated with sharp eyespot resistance in wheat, the F 2∶ 3 population was derived from a combination of Laizhou 953 × Shannongfu 63. The genotype of F2 single plant was obtained through the Illumina Wheat 90K SNP array, and the genetic linkage map of the population was constructed using QTL IciMapping 4.1 software. The phenotype of the population was identified under natural conditions, and the QTL mapping was performed by using QTL IciMapping 4.1 software. The genetic linkage map was composed of 21 linkage groups, covering 21 chromosomes of wheat, with a total length of 5 528.12 cM and an average spacing of 5.25 cM. A total of 6 additive QTLs were detected out, which distributed on chromosome 1A, 1B, 2A, 3A, 7A and 7D with the contribution rate of 3.24%~10.37%. The results would lay a foundation for the fine mapping and cloning of QTLs associated with wheat sharp eyespot resistance, and provide basis for molecular marker assisted selection breeding in wheat.
KeywordsWheat; Genetic linkage map construction; Sharp eyespot disease; QTL mapping; 90K SNP array
小麥纹枯病又称白穗病(white head)、尖眼斑病(sharp eyespot),主要是由禾谷丝核菌(Rhizoctonia cerealis)的第一菌丝融合群(CAG-1)以及立枯丝核菌(Rhizoctonia solani)的第四、五融合群(AG-4、AG-5)引起的一种土传病害,已成为我国长江流域和黄淮麦区的主要病害之一,对山东省小麦的危害已超过白粉病,由次要病害升级为主要病害。该病主要破坏小麦的茎、叶鞘等输导组织和机械组织,阻碍营养物质的运输,降低植株的抗倒伏能力,严重时还会引起籽粒皱缩和白穗,一般造成减产10%~30%,严重地块减产可超过50%[1]。因此,选育和推广抗纹枯病小麦新品种,是解决小麦纹枯病危害最直接、最有效的途径,而抗病品种的培育得益于纹枯病抗性基因的挖掘与利用。
从遗传学上来讲,作物抗病性是由多个数量性状位点(quantitative trait loci,QTL)控制的性状,遗传机理复杂。通过构建分子标记遗传连锁图谱定位抗病QTL,进而明确其在相应遗传背景下的作用强度和作用方式,对植物抗病育种有着十分重要的意义。国内外学者在水稻、大麦、小麦、玉米等多种作物上均定位到抗病QTL[2-5]。就小麦抗纹枯病QTL而言,最早见于霍纳新的相关报道,其利用SSR与AFLP标记共检测到3个成株期QTL和6个苗期QTL [6]。而后,蔡士宾等[7]用ARz×扬麦158的RIL群体为材料,釆用单标记方差分析法,发现11个SSR标记与抗病表型显著相关,能解释表型变异的5.0%~13.0%。Chen等[8]用美国抗病品种Luke与国内抗病品系AQ24788-83构建RIL群体,在温室和大田两种环境下,共检测到11个抗纹枯病QTL,其中7个在不同环境条件下均稳定地表现抗病作用;然而该研究利用两个抗病亲本组配分离群体,在后续QTL分析过程中容易漏掉在双亲中共同表达的抗病QTL。Wu等[9]以抗病材料CI12633和感病品种扬麦9号为亲本,构建分离群体,共鉴定到5个抗病QTL,同时发现小麦纹枯病抗性与抽穗期以及分蘖角度呈显著负相关,与基部第一节间和第二节间的直径呈显著正相关。
虽然前人研究中已得到了一批抗性QTL,但检测到的QTL存在连锁图谱密度低、QTL区段大以及有些QTL供体亲本在生产上难以直接利用等问题,导致目前还没有抗性QTL在小麦育种中成功利用的报道。本研究以前期鉴定到的农艺性状优良的抗纹枯病材料莱州953和感病材料山农辐63为亲本组配分离群体,以高通量的SNP标记构建连锁图谱,定位控制纹枯病抗性的QTL,为小麦抗纹枯病QTL定位和基因克隆奠定基础,同时为小麦抗纹枯病分子标记辅助选择育种及种质创新提供理论依据。
1材料与方法
1.1供试材料
以農艺性状优良的抗纹枯病品种莱州953为父本、高感纹枯病品种山农辐63为母本进行杂交,构建含170个株系的F 2∶ 3群体,由山东省农业科学院作物研究所小麦种质创新与利用团队创制并保存。
1.2田间种植及抗性鉴定
2016—2017年度将F 2∶ 3分离群体及其亲本种植于山东省临沂市纹枯病鉴定圃,每个株系种植2行,2次重复,行长2 m,行距0.25 m,株距3.0 cm。田间管理参照当地一般大田。4月中旬调查发病情况,病级划分按照0~5级分级标准[10]。每重复随机取20个茎调查纹枯病感病性,并计算平均病级。
1.3基因型分析
用植物基因组DNA提取试剂盒(康为世纪生物科技有限公司)提取亲本和F 2各单株的DNA。小麦90K SNP标记,由北京康普森生物技术有限公司利用Illumina SNPGenotyping技术测试平台、依靠微珠芯片技术(Bead Array)进行检测,并利用GenomeStudio v1.0软件进行SNP多态性分析。
1.4构建遗传连锁图谱
筛选多态性标记后,将分离群体基因型数据导入QTL IciMapping 4.1软件,利用BIN程序去除冗余标记,然后利用MAP程序构建该群体的遗传连锁图谱。
1.5QTL定位
结合连锁图谱与抗病表型数据,采用基于完备区间作图法QTL IciMapping 4.1软件的BIP程序进行加性QTL检测,设LOD阈值为2.5。QTL命名方法按照QTL+性状+染色体命名,其中,QTL以Q表示,性状以英文缩写表示且仅首字母大写(Ses)。
2结果与分析
2.1小麦遗传连锁图谱的构建
本研究采用的90K芯片包含81 577个SNP标记,覆盖小麦21条染色体。其中,在双亲间共检测到4 378个多态性位点,利用IciMapping 4.1的BIN程序删除冗余标记后,共有1 052个标记用于遗传连锁图谱的绘制。该图谱全长5 528.12 cM,平均图距5.25 cM。分布于小麦A、B和D染色体组的标记数分别为412、530、110个,连锁长度分别为1 737.73、2 412.62、1 377.77 cM,D染色体组标记数最少、平均图距最长。同源群中,第1同源群遗传长度最长,第4同源群标记数目最少、平均图距最短(表1)。
2.2小麦作图群体及其亲本的纹枯病抗性分析
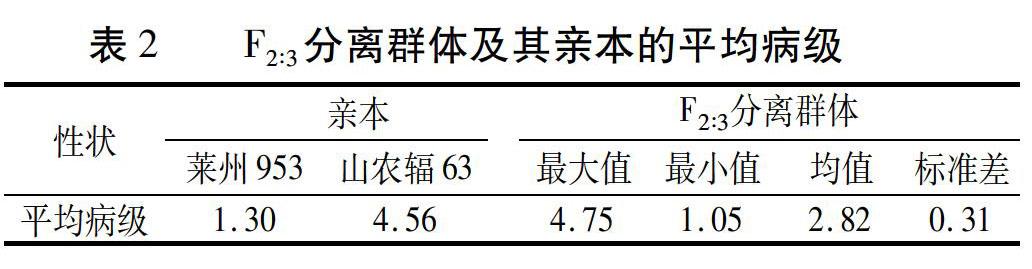
2016—2017年度,在纹枯病鉴定圃中,对F 2∶ 3分离群体及其亲本进行田间抗性鉴定,记录病级。结果(表2)表明,抗病亲本莱州953对纹枯病抗性较强(平均病级为1.30),感病亲本山农辐63高感纹枯病(平均病级为4.56),F 3家系的平均病级范围为1.05~4.75,呈连续性分布,符合数量遗传特征。
2.3小麦抗纹枯病QTL分析
利用QTL IciMapping 4.1软件的完备区间作图法进行QTL检测,在LOD值>2.5水平下,共定位到6个QTL位点,分布于小麦的1A、1B、2A、3A、7A和7D染色体上(表3和图1),单个QTL可解释表型变异的3.24%~10.37%。其中,1A染色体上检测到的QTL QSes1A 位于标记IAAV3695-BS00103166_51之间,贡献率大于10%,为主效QTL,其加性效应值为-0.60,表明该QTL抗纹枯病的等位基因来自抗病亲本莱州953。QTL QSes3A 和 QSes7D 分别定位于3A和7D染色体上,可解释抗病表型变异分别为9.55%和9.49%,加性效应值均为负值,表明其控制纹枯病抗性的等位基因来源于亲本莱州953。另外3个QTL, QSes1B 、 QSes2A 和 QSes7A ,贡献率相对较低,且抗性等位基因来源于山农辐63。
3討论
QTL定位结果准确可靠的前提条件是构建高质量的遗传连锁图谱。与传统的分子标记RFLP、AFLP、RAPD和SSR相比,SNP标记具有数量多、分布广、通量高等优点,是目前认可度最高的分子标记[10]。本研究利用小麦Illumina 90K SNP芯片,检测出双亲间具有多态性的位点4 378个,删除冗余标记后,用1 052个标记构建遗传连锁图谱,构建的图谱长度高于用传统的分子标记构建的图谱。随着小麦基因组数据库的完善,SNP标记将在小麦基因定位、候选基因预测、基因克隆等方面扮演更重要的角色[11,12]。
在本图谱1 052个标记中,B基因组标记数目最多、图谱最长,D基因组标记数目最少、图谱最短。这与张传量等[13]以小偃81 × 周8425B和小偃81 × 西农1376两个组合的RIL群体构建的图谱趋势一致,表明90K芯片SNP标记在小麦染色体组A、B和D间分布不均衡。D染色体组标记数目少,主要是因为在小麦进化过程中,D基因组加入较晚,没有产生充分的基因交流,故而保守性较高[14,15]。
前人研究表明,小麦抗纹枯病QTL主要分布在1A、2B、6B和7D染色体上[6-9, 16,17]。本研究共定位到6个QTL位点,分别位于小麦的1A、1B、2A、3A、7A和7D染色体上。其中,仅有位于1A染色体上的 QSes1A 为主效QTL。其他QTL贡献率都在10%以下,进一步表明小麦对纹枯病的抗性是由多个微效基因共同控制的,属于典型的数量性状。
本研究发现, QSes1B、QSes2A和QSes7A 这3个QTL的贡献率分别为8.70%、3.76%和3.24%,其抗性等位基因来源于感病亲本山农辐63。这种现象在作物抗旱性QTL定位研究中也有报道[18,19],如在潍麦8号与济麦20组配的RIL群体中,位于4A染色体上的两个QTL QSLDWR-WJ-4A和QCLR-WJ-4A ,其抗旱增效基因来源于抗旱性差的潍麦8号,表明抗性差的材料在某些位点上也可能存在相应的抗性增效基因,这也正是通过杂交聚合能够培育抗性优良作物新品种的理论基础,同时为抗病遗传改良中杂交亲本的选择拓宽了新的思路。
4结论
本研究基于90K芯片SNP标记,绘制了一张长度5 528.12 cM、具有1 052个标记的小麦遗传连锁图谱。在此基础上,定位到分布于小麦1A、1B、2A、3A、7A和7D染色体上的6个加性抗纹枯病QTL位点,其中来源于抗病亲本莱州953、位于1A染色体的 QSes1A 为抗纹枯病主效QTL。本研究结果可为小麦抗纹枯病QTL精细定位、相关基因克隆以及小麦抗纹枯病遗传改良奠定基础。
参考文献:
[1]路妍, 张增艳, 任丽娟, 等. 转 Rs-AFP2 基因小麦的分子分析及其纹枯病抗性[J]. 作物学报, 2009, 35(4): 640-646.
[2]Ashkani S, Rafii M Y, Shabanimofrad M, et al. Molecular breeding strategy and challenges towards improvement of blast disease resistance in rice crop[J]. Front. Plant Sci., 2015, 6:886.
[3]Huang Y, Haas M, Heinen S, et al. QTL mapping of fusarium head blight and correlated agromorphological traits in an elite barley cultivar Rasmusson [J]. Front. Plant Sci., 2018, 9:1260.
[4]Lin X, NDiaye A, Walkowiak S, et al. Genetic analysis of resistance to stripe rust in durum wheat (Triticum turgidum L. var. durum) [J]. PLoS ONE, 2018, 13(9): e0203283.
[5]Lanubile A, Maschietto V, Borrelli V M, et al. Molecular basis of resistance to fusarium ear rot in maize [J]. Front. Plant Sci., 2017, 8:1774.
[6]霍纳新. 小麦纹枯病、白粉病抗性QTL分析[D]. 北京:中国农业科学院, 2002.
[7]蔡士宾, 任丽娟, 颜伟, 等. 小麦纹枯病种质创新及QTL定位的初步研究[J]. 中国农业科学, 2006, 39(5): 928-934.
[8]Chen J, Li G, Du Z, et al. Mapping of QTL conferring resistance to sharp eyespot (Rhizoctonia cerealis) in bread wheat at the adult plant growth stage [J]. Theor. Appl. Genet., 2013, 126(11): 2865-2878.
[9]Wu X, Cheng K, Zhao R, et al. Quantitative trait loci responsible for sharp eyespot resistance in common wheat CI12633 [J]. Sci. Rep., 2017, 7(1): 11799.
[10] Wang S, Wong D, Forrest K, et al. Characterization of polyploid wheat genomic diversity using a high-density 90,000 single nucleotide polymorphism array [J]. Plant Biotechnol. J., 2014, 12(6): 787-796.
[11] Rasheed A, Hao Y, Xia X, et al. Crop breeding chips and genotyping platforms: progress, challenges, and perspectives [J]. Mol. Plant, 2017, 10(8): 1047-1064.
[12] 许陶瑜, 唐朝晖, 王长彪, 等. SNP标记在小麦遗传育种中的应用研究进展[J]. 山西农业科学, 2017, 45(9): 1549-1552.
[13] 张传量, 简俊涛, 冯洁, 等. 基于90K芯片标记的小麦芒长QTL定位[J]. 中国农业科学, 2018, 51(1): 17-25.
[14] 肖静, 田纪春. 小麦(T. aestivum L.) D基因组的研究进展[J]. 分子植物育种, 2008, 6(3): 537-541.
[15] 贾继增, 张正斌, Devos K, 等. 小麦21条染色体RFLP作图位点遗传多样性分析[J]. 中国科学: 生命科学, 200 31(1): 13-21.
[16] 任丽娟, 张旭, 周淼平, 等. 小麦抗纹枯病和赤霉病QTL定位研究[J]. 麦类作物学报, 2007, 27(3): 416-420.
[17] 蒋彦婕, 朱芳芳, 蔡士宾, 等. 小麦纹枯病抗性QTL遗传分析[J]. 江苏农业学报, 2014, 30(6): 1222-1226.
[18] Wang Y, Zhang Q, Zheng T,et al. Drought-tolerance QTLs commonly detected in two sets of reciprocal introgression lines in rice [J]. Crop Pasture Sci., 2014, 65(2): 171-184.
[19] 刘新元, 张红, 胡雪薇, 等. 小麦萌发期幼苗相关性状耐旱系數的QTL定位[J]. 核农学报, 2017, 31(2): 209-217.
