基于全基因组的卷柏复苏相关AP2/ERF转录因子基因家族分析△
2018-05-21徐志超浦香东宋经元
徐志超,浦香东,宋经元
(中国医学科学院 北京协和医学院 药用植物研究所/国家中医药管理局 中药资源保护重点研究室,北京 100193)
复苏植物能够在脱水95%以上的生理情况下生存且遇水复苏,在蕨类植物和苔藓植物中常见,被子植物中数量极少[1-2]。复苏植物极度耐旱的特点成为国际研究的热点之一,被认为是抗旱基因挖掘及利用的模式植物。截至目前多个复苏植物的基因组已公布,如单子叶植物复活草Oropetiumthomaeum[3]、维柯萨Xerophytaviscosa[4]和双子叶植物旋蒴苣苔Boeahygrometrica[5],为抗旱基因的挖掘提供资源。
卷柏Selaginellatamariscina,又称“九死还魂草”,为石松纲卷柏科卷柏属植物,旱时卷曲,遇水而荣,是典型的复苏植物[6-7]。卷柏属包括约700个种,《中国植物志》中记录有66种卷柏[8],2015版《中华人民共和国药典》以卷柏和垫状卷柏S.pulvinata入药,其干燥全草炒炭后具有活血通经的功能[9]。卷柏属不同物种间遗传分化极其显著,其生理特点也显著不同,如卷柏为耐旱型[10],而江南卷柏为干旱敏感型[11]。
AP2/ERF转录因子在植物生长发育、胁迫响应中发挥重要的调控作用,包括干旱、高盐、低温等逆境胁迫响应[12]。AP2/ERF转录因子家族至少含有一个高度保守的AP2结构域,根据结构域的数量和识别序列不同,分为AP2、ERF、RAV、DREB、SOLOIST等5个亚家族[13]。已报道多个基于全基因组分析AP2/ERF转录因子家族的研究,如拟南芥[14]、水稻[15]、葡萄[16]、丹参[17]等,然而低等植物AP2/ERF转录因子的挖掘研究较少。本研究基于课题组前期卷柏基因组的研究数据,挖掘卷柏的AP2/ERF转录因子,筛选与卷柏复苏密切相关的转录调控因子。
1 材料和方法
1.1 卷柏基因组、转录组数据
卷柏基因组组装、注释及转录组测序工作由本课题组完成。卷柏基因组测序原始数据(PacBio RSII测序平台)以及卷柏干旱及复水的转录组数据(Illumina HiSeq4000测序平台)已上传至NCBI(National Center for Biotechnology Information)的SRA(Sequence Read Archive)数据库,登录号:SAMN07840812(BioSample ID)、PRJNA416064(BioProject ID)。
1.2 基于蛋白序列比对的卷柏AP2/ERF转录因子鉴定
下载拟南芥AP2/ERF转录因子的AP2、ERF、DREB、RAV、SOLOIST亚家族成员,采用BLASTP比对卷柏基因组蛋白序列,Evalue设置为1E-5;采用PFAM数据库隐马尔科夫模型HMM:PF00847搜索卷柏基因组数据库,Evalue设置为1E-5。以gff文件格式从基因组数据库中提取所有AP2/ERF序列,根据NCBI网站BLASTP比对的结果,利用APOLLO软件手动校正序列。校正后,保存每个AP2/ERF转录因子的CDS、DNA、蛋白序列信息。在蛋白质数据库PROSITE(http://prosite.expasy.org/)中确认每个成员的保守结构域序列,用DNAMAN软件比对分析其保守性。
1.3 AP2/ERF系统进化、基因结构和保守基序分析
将卷柏和拟南芥AP2/ERF家族成员的蛋白序列采用MUSCLE做多序列比对,构建NJ(Neighbour-Joining)树,选择Jones-Taylor-Thornton(JTT)模型,重复次数1000次。基于CDS序列及对应基因组序列,利用在线服务器Gene Structure Display Server(GSDS 2.0)(http://gsds.cbi.pku.edu.cn/index.php)对基因结构进行分析。利用MEME(Suite version 4.9.1)(http://meme-suite.org/)对AP2/ERF转录因子保守基序进行分析,具体原则如下:E值小于2×10-30,基序重复次数不限,氨基酸个数10~200个。
1.4 AP2/ERF转录因子家族基因的差异表达分析
本研究分析卷柏干旱及复水处理的9个转录组数据,分别为干旱(hydration,HD)、70%相对含水量(Relative water content,RWC)、50% RWC、30% RWC、15% RWC、5% RWC、复水2 h(rehydration,RH)、RH 4 h、RH 6 h。基于转录组数据,通过HiSAT2和Cufflinks软件计算AP2/ERF转录因子的FPKM值[18]。按公式(1)计算RWC。
(1)
式中:Fwt为干旱时间点的卷柏质量;Dwt为卷柏在65 ℃烘干2 d后的质量;FTwt为卷柏复水24 h后的质量[11]。
2 结果和分析
2.1 卷柏AP2/ERF转录因子家族预测及系统进化分析
在卷柏基因组中共预测到68个AP2/ERF转录因子基因,基因数量远低于高等植物中AP2/ERF的基因数量,如拟南芥(147)、葡萄(132)、水稻(164)、丹参(170)。卷柏AP2/ERF转录因子的长度范围在72 aa(ST44G415)到587 aa(ST311G14)之间,其cDNA长度在219 bp到1764 bp之间。
用卷柏和拟南芥AP2保守结构域建进化树,AP2/ERF转录因子家族被分为5个亚家族,分别为AP2、DREB、ERF、RAV和SOLOIST,各亚家族成员个数分别为23、16、18、8和 3,如图1所示。拟南芥、葡萄和丹参的SOLOIST基因只有一个,水稻中未发现SOLOIST基因,但卷柏中发现3个,认为SOLOIST基因在进化过程中发生基因数量的收缩。高等植物AP2、ERF、DREB的亚家族成员远远多于卷柏中,证明进化过程中该亚家族存在巨大的基因扩张。利用DNAMAN软件对拟南芥和卷柏的DREB亚家族成员保守结构域序列进行比对分析,发现AP2/ERF保守结构域(YRG和RAYD元件)在卷柏和拟南芥中高度保守,如图2所示。
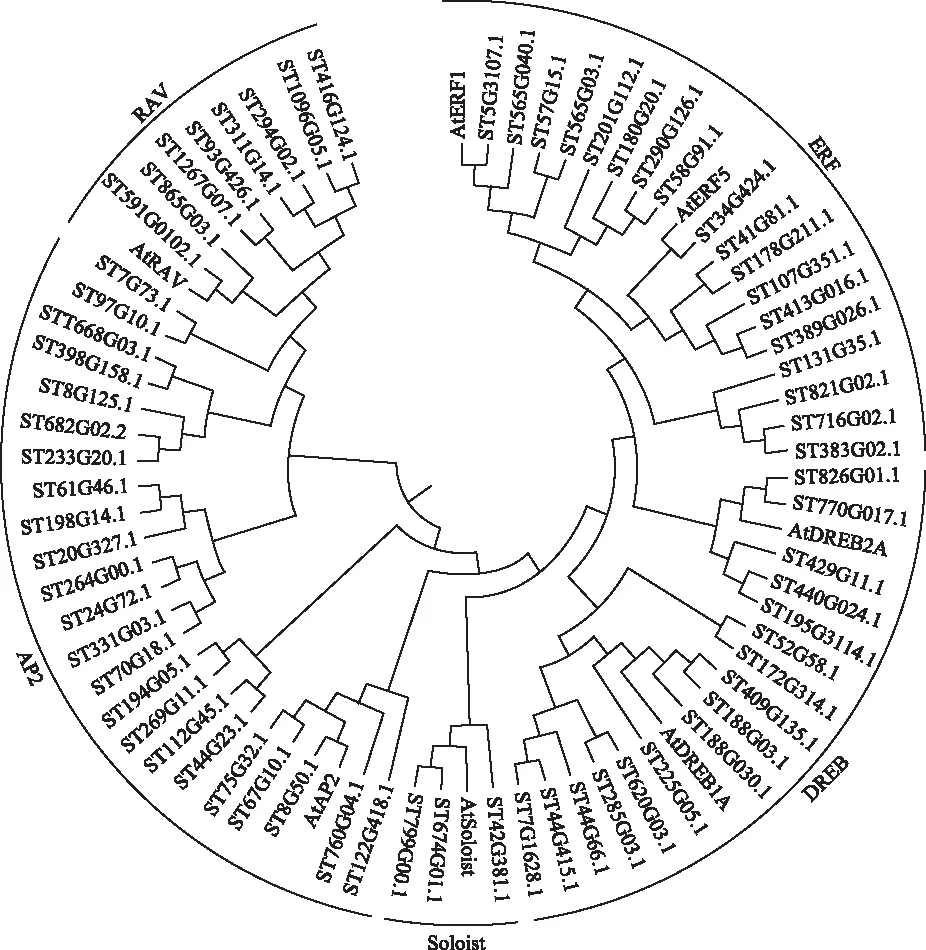
图1 卷柏AP2/ERF基因家族系统进化分析
2.2 卷柏AP2/ERF基因家族的基因结构分析
AP2/ERF基因家族的基因结构显示不同亚家族基因结构差异较大,两个RAV亚家族基因(ST294G02和ST311G14)所含内含子数最多(14个),而大部分DREB、ERF亚家族成员不含内含子,如图3所示。AP2/ERF家族成员的保守基序显示AP2/ERF各亚家族之间序列相对保守,亚家族内部有一定差异。位于同一个亚家族内含有相同基序的成员,可能具有相似功能。
2.3 AP2/ERF转录因子基因的差异表达分析
AP2/ERF转录因子的DREB亚家族是干旱应答元件的结合蛋白,与植物干旱、高盐等逆境密切相关。本研究对卷柏进行干旱胁迫及复水处理,HD(hydration)、70% RWC、50% RWC、30% RWC、15% RWC、5% RWC、RH 2 h(rehydration)、RH 4 h、

图2 卷柏AP2/ERF亚家族数量分布(A)及DREB亚家族AP2/ERF保守结构域比对(B)
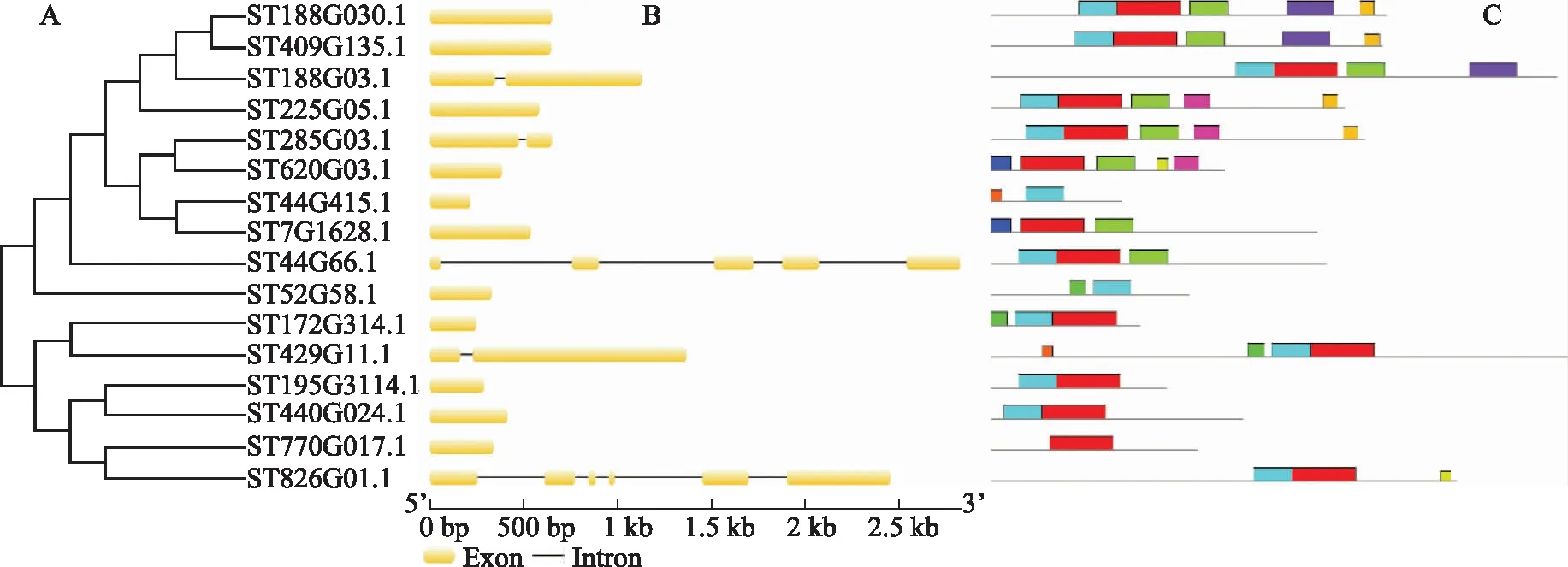
注:A.亚家族成员建树;B.亚家族成员基因结构;C.亚家族成员蛋白保守基序分布。图3 卷柏DREB亚家族基因结构特征和保守基序分布
RH 6 h处理的9个转录组数据(共100 G)比对到卷柏基因组,计算FPKM值,筛选AP2/ERF基因的表达谱,如图4所示。FPKM值越大,说明基因表达丰度越高,反之则说明基因表达丰度越低。由于基因表达FPKM值差异显著,将FPKM值用scale函数做Z-score标准化处理,显示基因在不同处理下的表达差异并聚类。从基因表达模式来看,HD、70% RWC和RH 6 h的AP2/ERF转录因子基因表达聚在一起,而干旱胁迫严重的50% RWC、30% RWC、15% RWC、5% RWC、RH 2 h(rehydration)、RH 4 h的基因表达聚为另一枝。ST429G11、ST44G415和ST565G040基因的表达聚在一起,响应干旱胁迫最为敏感的一枝,其中ST429G11和ST44G415属于DREB亚家族,ST565G040为ERF亚家族。
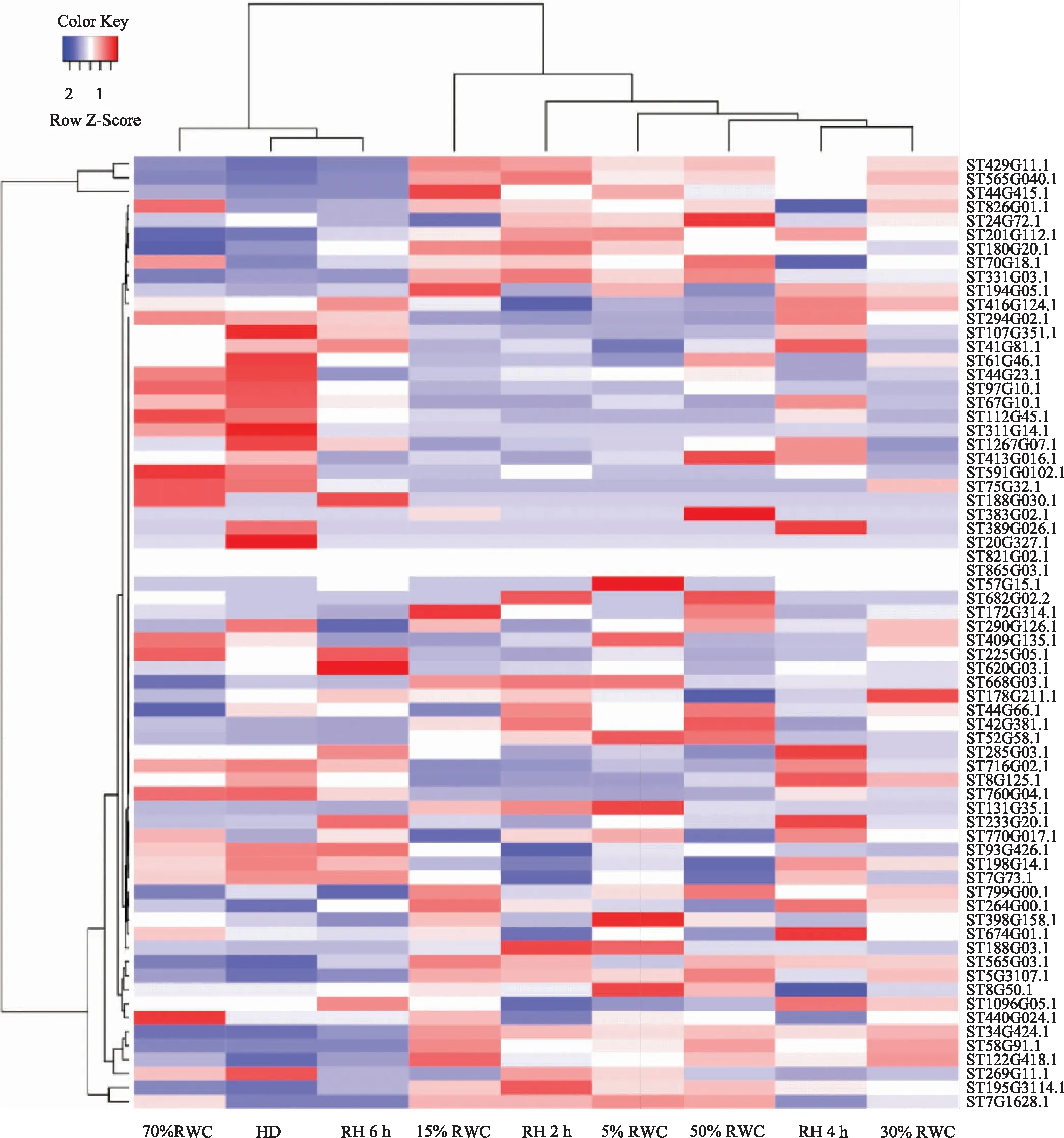
图4 AP2/ERF转录因子在卷柏干旱胁迫及复水处理下的表达图谱
47%的AP2/ERF基因家族成员在干旱胁迫及复水处理下显示高表达(FPKM>10),15%的AP2/ERF基因家族成员表达沉默(FPKM<1),推测为沉默基因或假基因。17个基因与卷柏干旱及复水表型显著正相关,呈共表达,在干旱胁迫后显著高表达[log2(50%RWC/HD)>1],且复水后基因表达降低,其中包括7个DREB亚家族、6个ERF亚家族和4个AP2家族成员,如图5所示。推测在卷柏干旱胁迫后显著高表达的AP2/ERF转录因子在卷柏生长发育及胁迫响应进程中起重要的调控作用。
卷柏中表达最高的两个AP2/ERF转录因子成员均属于DREB亚家族,分别为ST429G11和ST44G415,其FPKM值在5%RWC状态下高达2833和3977,分别是HD状态下的12倍和74倍。启动子结合元件显示ST429G11的启动子序列含有AREB转录因子结合的ABA响应元件(ABRE,ACGTGG)、GRF7转录因子结合的GTE元件(TGTCAGG)和热激元件(HSE,AAAAAATTAC)的结合位点;ST44G415的启动子序列含有 GRF7转录因子结合的GTE元件(TGTCAGG)的结合位点。研究推测ST429G11为ABA非依赖的调控途径中响应干旱胁迫的重要调控因子。

注:不同的线条颜色代表17个干旱胁迫相关的AP2/ERF编码基因。图5 候选参与卷柏干旱胁迫响应的AP2/ERF基因共表达
3 讨论
本研究基于卷柏全基因组测序,对其AP2/ERF家族成员进行系统鉴定,筛选参与卷柏干旱胁迫进程密切相关的AP2/ERF转录因子。研究发现卷柏DREB亚家族成员主要参与其非生物胁迫响应进程如干旱胁迫,与已报道的其他植物的DREB功能相一致。
在卷柏基因组中共鉴定到68个AP2/ERF转录因子,远远低于其他高等植物AP2/ERF家族成员数量,与低等植物齿肋赤藓Syntrichiacaninervis的数量(80)接近[19],低等植物到高等植物AP2/ERF基因数量的扩张反映了植物在进化过程中响应非生物胁迫和生物胁迫的转录因子调控的重要性及复杂性。此外,通过多序列比对及保守基序分析发现,卷柏AP2/ERF转录因子的AP2/ERF结构域与高等植物拟南芥AP2/ERF的存在差异,但核心保守结构域一致,表明该转录因子在物种进化中基因结构具有较高的保守性。
DREB亚家族和ERF亚家族结构相似,均具有一个AP2/ERF亚家族,且一般不含有内含子,主要区别在于保守AP2结构域的第14和19位氨基酸不同。两个亚家族功能亦相似,在植物响应生物及非生物胁迫时发挥重要的调控作用。在药用植物领域,干旱、高盐等非生物胁迫或生物胁迫显著影响药用植物品质,AP2/ERF转录因子的调控作用也陆续被发现,同样主要来自于DREB和ERF亚家族成员的调控,如长春花ERF亚家族成员ORCA3可以调控萜类吲哚生物碱代谢途径中的多个关键酶,影响长春碱的产量[20]。本研究发现:卷柏的DREB和ERF亚家族系统进化关系较近,聚在一大枝;卷柏的干旱胁迫响应极为敏感的AP2/ERF转录因子基因绝大部分来自于DREB和ERF亚家族。从复苏植物卷柏干旱响应调控因子AP2/ERF成员的结构保守性推测其在卷柏干旱胁迫进程中的调控机制与高等植物相似,但仍需要进一步的功能鉴定。卷柏AP2/ERF转录因子的挖掘将丰富植物干旱响应的基因资源库,为卷柏复苏分子机制的解析奠定基础,同时有助于研究植物进化过程中AP2/ERF转录因子的基因扩张或功能分化。
[1] Giarola V,Hou Q,Bartels D.Angiosperm plant desiccation tolerance:hints from transcriptomics and genome sequencing[J].Trends Plant Sci,2017,22(8):705-717.
[2] Farrant J M,Moore J P.Programming desiccation-tolerance:from plants to seeds to resurrection plants[J].Curr Opin Plant Bio,2011,14(3):340-345.
[3] Vanburen R,Bryant D,Edger P P,et al.Single-molecule sequencing of the desiccation-tolerant grassOropetiumthomaeum[J].Nature,2015,527:508.
[4] Costa M C D,Artur M A S,Maia J,et al.A footprint of desiccation tolerance in the genome ofXerophytaviscosa[J].Nat Plants,2017,3(4):17038.
[5] Xiao L,Yang G,Zhang L,et al.The resurrection genome ofBoeahygrometrica:A blueprint for survival of dehydration[J].Proc Natl Acad Sci USA,2015,112(18):5833-5837.
[6] Banks J A.Selaginellaand 400 Million Years of Separation[J].Annu Rev Plant Biol,2009,60:223.
[7] Korall P,Kenrick P.The phylogenetic history of Selaginellaceae based on DNA sequences from the plastid and nucleus:extreme substitution rates and rate heterogeneity[J].Mol Phylogenet Evol,2004,31(3):852-864.
[8] 中国科学院《中国植物志》编委会.中国植物志:第6卷[M].北京:科学出版社,2004:313
[9] 国家药典委员会.中华人民共和国药典:一部[S].北京:中国医药科技出版社,2015:226
[10] Wang X,Chen S,Zhang H,et al.Desiccation Tolerance Mechanism in Resurrection Fern-AllySelaginellatamariscinaRevealed by Physiological and Proteomic Analysis[J].J Proteome Res,2010,9(12):6561-6577.
[11] Yobi A,Wong B W,Xu W,et al.Comparative metabolic profiling between desiccation-sensitive and desiccation-tolerant species ofSelaginellareveals insights into the resurrection trait[J].Plant J,2012,72(6):983-999.
[12] Licausi F,Ohme-Takagi M,Perata P.APETALA2/Ethylene Responsive Factor(AP2/ERF)transcription factors:mediators of stress responses and developmental programs[J].New Phytol,2013,199(3):639-649.
[13] 季爱加,罗红梅,徐志超,等.药用植物转录因子AP2/ERF研究与展望[J].科学通报,2015(14):1272-1284.
[14] Sakuma Y,Liu Q,Dubouzet J G,et al.DNA-binding specificity of the ERF/AP2 domain ofArabidopsisDREBs,transcription factors involved in dehydration-and cold-inducible gene expression[J].Biochem Biophys Res Commun,2002,290(3):998-1009.
[15] Nakano T,Suzuki K,Fujimura T,et al.Genome-wide analysis of the ERF gene family inArabidopsisand rice[J].Plant Physiol,2006,140:411-432.
[16] Licausi F,Giorgi F M ,Zenoni S,et al.Genomic and transcriptomic analysis of the AP2/ERF superfamily inVitisvinifera[J].BMC Genomics,2010,11:719.
[17] Ji A J,Luo H M,Xu Z C,et al.Genome-wide identification of the AP2/ERF gene family involved in active constituent biosynthesis inSalviamiltiorrhiza[J].Plant Genome,2016,9(2):1-11.
[18] Trapnell C,Roberts A,Goff L,et al.Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J].Nat Protoc,2012,7:562-578.
[19] Li X,Zhang D,Gao B,et al.Transcriptome-wide identification,classification,and characterization of AP2/ERF family genes in the desert mossSyntrichiacaninervis[J].Front in Plant Sci,2017,8(47):262.
[20] Wang C T,Liu H,Gao X S,et al.Overexpression of G10H and ORCA3 in the hairy roots ofCatharanthusroseusimproves catharanthine production[J].Plant Cell Rep,2010,29:887-894.