实验室饲养和野外生存条件下东方田鼠肠道菌群的多样性
2018-05-04冯洁沈志敏王胜昌林金杏柏熊谢建云
冯洁,沈志敏,王胜昌,林金杏,柏熊,谢建云*
(1. 上海实验动物研究中心,上海 201203; 2. 上海西普尔-必凯实验动物有限公司,上海 201203)
东方田鼠(Microtusfortis)在分类学上隶属于啮齿目、仓鼠科、田鼠亚科、田鼠属,主要分布于我国西北、东北及南方的16个省区,在我国存在5个亚种。东方田鼠是迄今为止发现的唯一具有天然抗血吸虫病的哺乳动物,其中分布于长江中下游的长江亚种是血吸虫病流行区洞庭湖湖区的优势鼠种,具有重要的应用价值[1,2]。20世纪90年代末,上海实验动物研究中心和湖南湘雅医学院开始对东方田鼠进行实验室繁育。饲养的过程中研究人员发现,实验室饲养条件下的东方田鼠易发生脂肪肝和卵巢癌,可诱发导致糖尿病等情况[3-5]。为探讨不同生存模式下东方田鼠肠道菌群的分布和差异,本文首次采用基于细菌16S rDNA的高通量测序技术,对实验室饲养和野外捕捉的东方田鼠肠道菌群多样性进行比较分析,为充实东方田鼠基础生物学数据积累资料,为东方田鼠资源的进一步开发和利用提供参考。
1 材料与方法
1.1 材料
1.1.1 实验动物
实验室饲养洞庭湖种群东方田鼠12只,由上海西普尔-必凯实验动物有限公司提供【SCXK(沪)2013-0016】,8周龄,体重50~80 g,1998年捕获于洞庭湖湖区饲养至今,饲养于屏障设施环境中,室内的温度常年控制在(22±2)℃,相对湿度控制在40%~70%,人工采光,昼夜交替12 h∶12 h。饲料为大小鼠通用饲料,由上海西普尔-必凯实验动物有限公司生产并提供,自由采食和饮水。野生洞庭湖种群东方田鼠20只,2016年12月捕获于湖南洞庭湖区,成年,健康状况良好,体重约40~70 g,捕获现场采集肠道标本,置于干冰中带至实验室。
1.1.2 主要试剂和仪器
细菌基因组快速提取试剂盒购自Qiagen公司,DNA凝胶回收试剂盒购自Axygen公司,PCR扩增仪为ABI GeneAmp® 9700 型,测序仪为美国Illumina公司hiseq 2500型。
1.2 方法
1.2.1 DNA抽提
处死动物,用酒精消毒皮肤后解剖,暴露回盲部,取0.2 g回盲部内容物,悬浮于1 mL的PBS(pH7.4)中850 g离心5 min,取上清,制成细菌悬液用于DNA提取。基因组DNA抽提操作步骤参照试剂盒说明书。DNA抽提后经1%琼脂糖凝胶电泳检测DNA质量。
1.2.2 PCR扩增
设计引物接头对细菌的16S rDNA-V4-V5区片段进行PCR扩增。将同一样本的PCR产物混合后经2%琼脂糖凝胶电泳检测,切胶回收产物后进行定量,按相应比例进行混合。
1.2.3 Illumina PE250文库构建
连接“Y”字形接头,使用磁珠筛选去除接头自连片段,利用PCR扩增进行文库模板的富集。经氢氧化钠变性产生单链DNA片段。
1.2.4 高通量测序和生物信息学分析
高通量测序和生物信息学分析由上海凌恩生物科技有限公司完成。
测序流程如下:DNA片段一端与引物碱基互补,固定于芯片上,另一端随机与附近的另外一条引物互补,也被固定形成“桥”。PCR扩增产生DNA簇,激光扫描反应板表面,读取每条模板序列所聚合上的核苷酸种类。统计每轮收集到的荧光信号,即可获知模板DNA片段序列。
对所有有效序列进行质控过滤,舍弃低质量序列。利用软件将相似度大于0.97的序列归为1个操作分类单元(Operational taxonomic unit,OTU),统计各样品所含OTU的数量,去除无法分类的OTU后进行后续分析,绘制稀疏曲线。基于OTU聚类分析结果进行多样性指数分析。基于群落结构分析,在门和属两个分类水平统计多样品的物种分布和丰度差异。
2 结果
2.1 回盲部内容物细菌物种丰度分析
将序列按相似性进行OTU划分,绘制稀疏曲线(图1)。总体趋势体现为:序列数<10 000时OTU数量随着样本序列数的增加而迅速增加,序列数在10 000~30 000之间时OTU数量缓慢增加,之后则趋于平台期,表明取样数量合理,测序充分。
通过样本的多样性分析可反映出微生物群落的丰度和多样性,常用丰富度指数表示。在OTU水平,各样本的OTU数目、丰富度指数(Chao)、覆盖率指数(Coverage)及多样性指数(Shannon、Simpson)见表1。Coverage值越高,代表样本中序列被测出的概率越高。Shannon值越高,代表群落多样性越高,而Simpson 指数值越大,说明群落多样性越低。
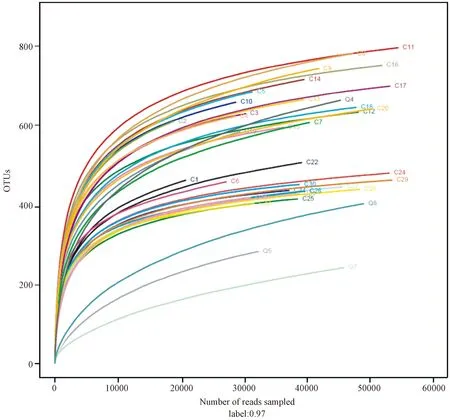
图1 各样本的稀疏曲线Fig.1 Rarefaction curves of the samples
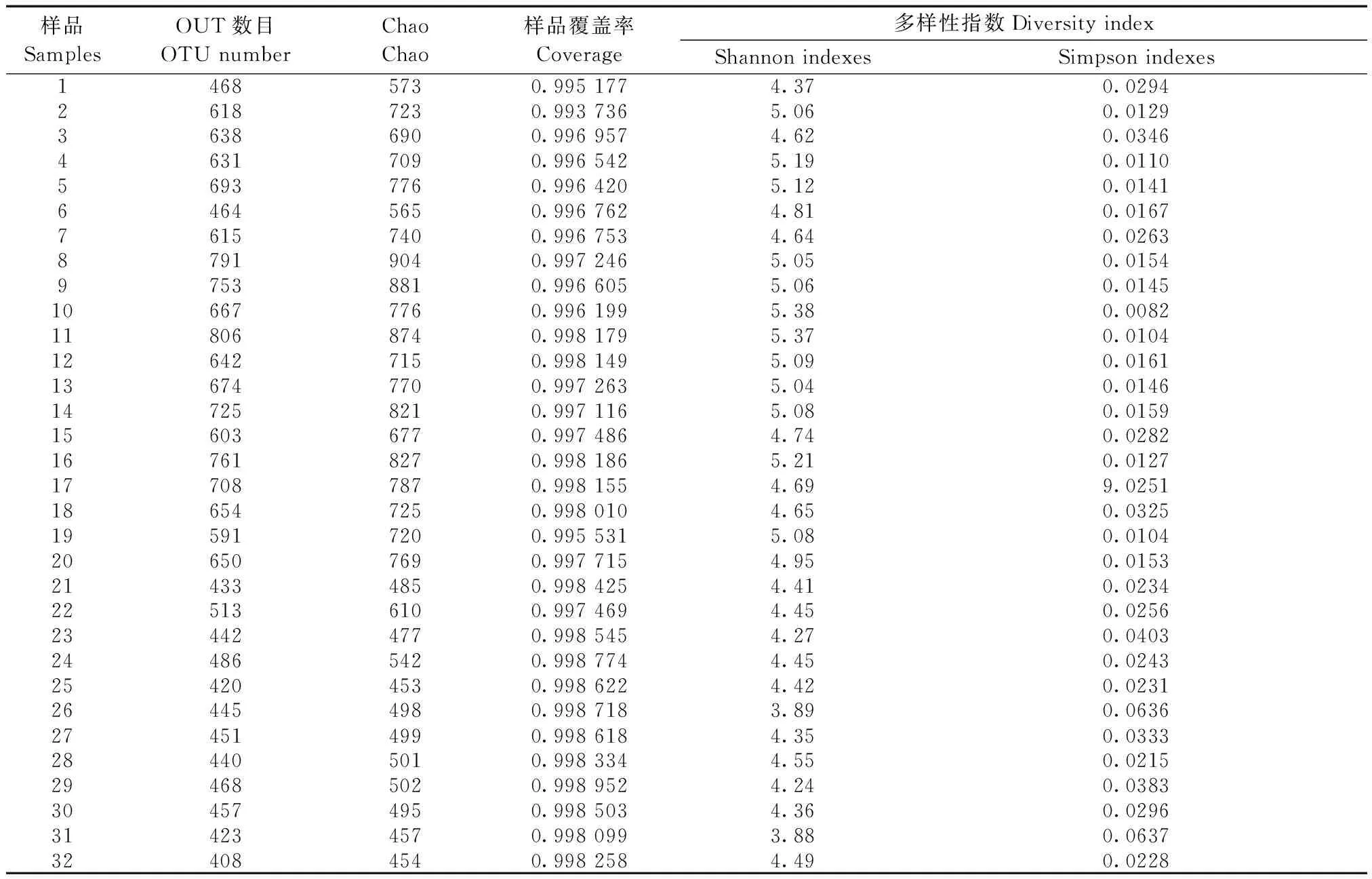
样品SamplesOUT数目OTUnumberChaoChao样品覆盖率Coverage多样性指数DiversityindexShannonindexesSimpsonindexes14685730.9951774.370.029426187230.9937365.060.012936386900.9969574.620.034646317090.9965425.190.011056937760.9964205.120.014164645650.9967624.810.016776157400.9967534.640.026387919040.9972465.050.015497538810.9966055.060.0145106677760.9961995.380.0082118068740.9981795.370.0104126427150.9981495.090.0161136747700.9972635.040.0146147258210.9971165.080.0159156036770.9974864.740.0282167618270.9981865.210.0127177087870.9981554.699.0251186547250.9980104.650.0325195917200.9955315.080.0104206507690.9977154.950.0153214334850.9984254.410.0234225136100.9974694.450.0256234424770.9985454.270.0403244865420.9987744.450.0243254204530.9986224.420.0231264454980.9987183.890.0636274514990.9986184.350.0333284405010.9983344.550.0215294685020.9989524.240.0383304574950.9985034.360.0296314234570.9980993.880.0637324084540.9982584.490.0228
2.2 细菌群落结构分析
将实验室饲养和野外生存的动物样本OTU在门和属的水平进行归类分析。如图2所示,两组样本共有的OUT有455个,各自独有的OTU个数分别是143和699。
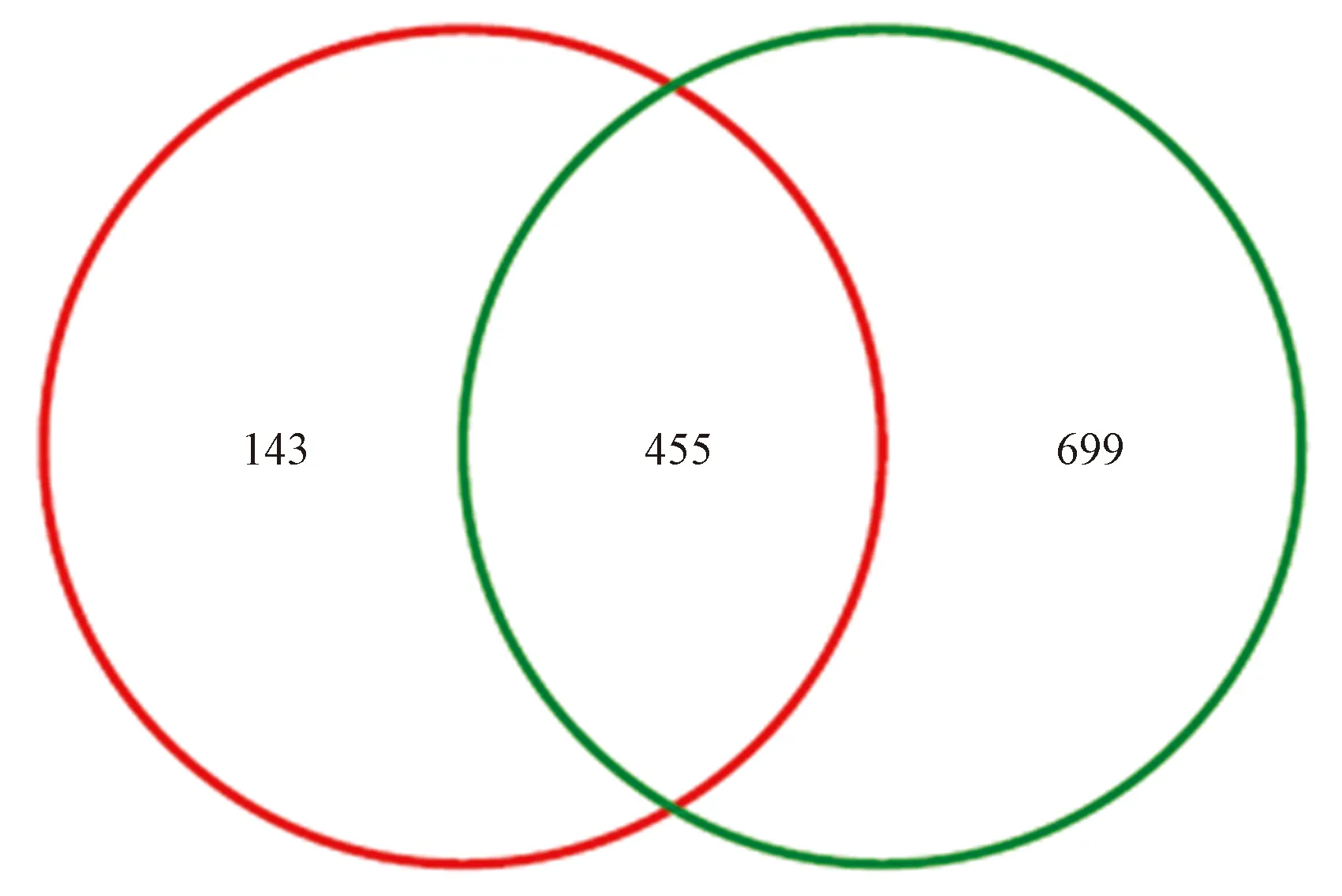
注:红色:实验室饲养组;绿色:野外生存组。图2 两组样本OTU归类图Note. Red: Laboratory feeding group. Green: Wild survival group.Fig.2 OTU classification of the two group samples
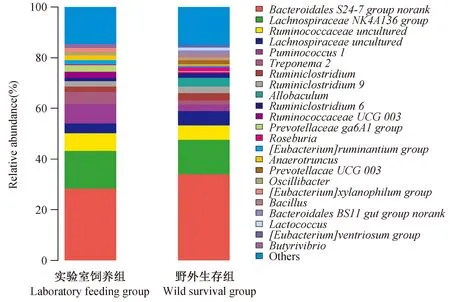
图4 两组样本细菌在属水平的分布Fig.4 Bacteria composition at genus level of the two groups of samples
在门水平,实验室饲养样本细菌隶属12个门,其中厚壁菌门(Firmicutes)占58.43%、拟杆菌门(Bacteroidetes)占34.76%、螺旋菌门(Spirochaetae)占4.87%,其余9个门合计仅占1.95%。野外采集样本细菌隶属14个门,其中厚壁菌门(Firmicutes)占54.29%、拟杆菌门(Bacteroidetes)占41.56%、螺旋菌门(Spirochaetae)占2.15%,其余10个门合计仅占2.00%(图3)。两组样本共有11个门重叠,实验室饲养样本有1个独有的门,即黏胶球形菌门(Lentisphaerae),野外生存样本有3个独有的门,分别是梭杆菌门(Fusobacteria)、奇古菌门(Thaumarchaeota)和未分类的门。在属水平,两组样本共检测到205个属,实验室饲养样本细菌隶属125个属,野外生存样本细菌隶属192个属。其中112个属为两者共有,13个属为实验组独有,80个属为野生组独有(图4)。

实验室饲养组 野外生存组Laboratory feeding group Wild survival group图3 两组样本细菌在门水平的分布Fig.3 Bacteria composition at phylum level of the two groups of samples
2.3 物种丰度差异性分析
根据分类学组成,对微生物类群进行组间比较分析(LEfSe),即LDA分析(线性回归分析),并绘制进化分支图以了解两组样本微生物物种的差异[6](图5)。红色区域和绿色区域分别表示实验组和野生组,红色节点表示在实验组中起重要作用的微生物类群,绿色节点表示在野生组中起重要作用的微生物类群,黄色节点表示在两组别中均未起到重要作用的微生物类群。当LDA值>2,P值<0.05时认为该物种为差异物种,差异组别为两组中丰度高的一组。结果显示,在门的水平,共有5个门存在差异(表2);在属的水平,共有64个属存在差异,差异最大的为瘤胃球菌属。因存在差异的属较多,仅列出差异值前10的属(表3)。
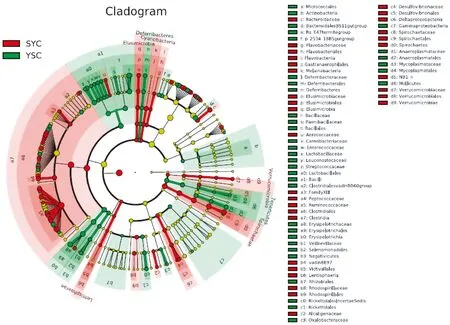
图5 两组样本LEfSe分析进化分支图Fig.5 Evolutionary branch cladogram of LEfSe analysis of the two groups of samples
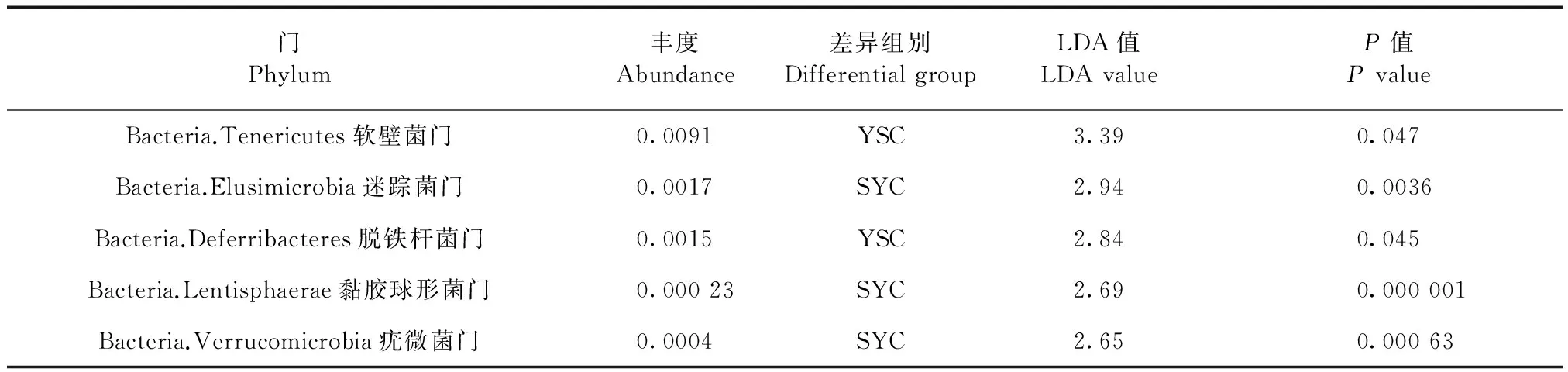
门Phylum丰度Abundance差异组别DifferentialgroupLDA值LDAvalueP值PvalueBacteria.Tenericutes软壁菌门0.0091YSC3.390.047Bacteria.Elusimicrobia迷踪菌门0.0017SYC2.940.0036Bacteria.Deferribacteres脱铁杆菌门0.0015YSC2.840.045Bacteria.Lentisphaerae黏胶球形菌门0.00023SYC2.690.000001Bacteria.Verrucomicrobia疣微菌门0.0004SYC2.650.00063
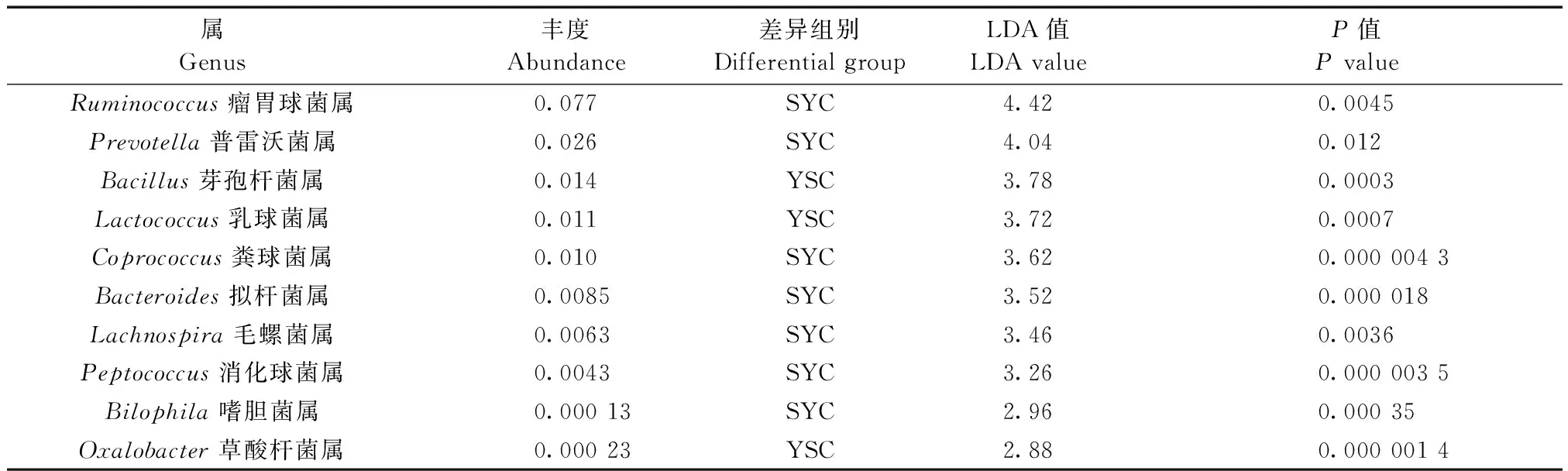
表3 两组样本在属水平的丰度差异分析Tab.3 Differential analysis of genus abundanceof the two groups of samples
3 讨论
随着近年来对肠道菌群研究的不断深入,人们发现肠道微生物对宿主健康起重要作用。临床上许多重要疾病的发生、发展都与肠道微生物的组成以及数量的增减有密切的关系。结构异常的肠道菌群很可能是肥胖、高血压、糖尿病等慢性病的直接诱因。研究表明,动物消化道中存在着种类繁多、数量庞大的微生物,它们是宿主机体重要的组成部分,参与肠道发育、营养吸收、能量代谢、免疫应答等生理活动[7-9]。肠道菌群组成的影响因素众多,主要包括宿主自身的因素和外部环境的影响。16S rDNA基因存在于所有细菌的基因组中,具有高度的保守性和特异性,并且该基因序列足够长(包含约50个功能域)。随着PCR技术的出现及核酸研究技术的不断完善,16S rDNA基因检测技术已成为病原菌检测和鉴定的一种强有力工具[10-11]。
本研究通过对洞庭湖种群东方田鼠肠道样本的细菌16S rDNA V4-V5可变区序列进行扩增,并利用高通量测序技术和序列比对,对实验室饲养和野外生存的东方田鼠肠道菌群的分布特点和差异进行分析。两组样本的稀疏曲线表明取样合理,高通量测序深度充分。在门水平比较发现,肠道菌群的分布和占比基本相似,丰度最高的为厚壁菌门,其次为拟杆菌门,这两类微生物对于机体健康有深远的影响,在纤维物质消化和脂肪沉积中起主要作用。丰度差异上比较发现,野外的东方田鼠软壁菌门、脱铁杆菌门的丰度较实验组高。在属水平上,野生的东方田鼠肠道细菌较实验室饲养的丰富。两组动物差异显著的菌群主要为参与消化道能量代谢和吸收的细菌,如瘤胃球菌与纤维素的附着程度和消化率有重要关联,普雷沃菌是一种纤维降解菌,能产生木聚糖酶,拟杆菌在分解多糖提高营养利用率、加快肠黏膜的血管形成、维持肠道微生态平衡等方面发挥重要作用。研究表明,普雷沃菌和拟杆菌可驱动胰岛素抵抗和葡萄糖耐受不良,引起糖尿病、高血压等疾病[12]。而嗜胆菌属在健康机体肠道中丰度极低,动物性食物、脂肪含量高的饮食可增加胆汁的产生,进而导致嗜胆菌属的增加。非酒精性脂肪肝引起的代谢综合征也有类似情况出现。上述发现为进一步探讨实验室饲养东方田鼠易发脂肪肝、糖尿病的机制提供了一个思路。
通过对食草动物、食肉动物和杂食动物的肠道菌群组成比较发现,食草动物为了消化复杂的含纤维素食物,肠道菌群多样性程度远高于杂食和食肉动物,说明在宿主和肠道菌群协同进化过程中,饮食结构对菌群结构影响非常明显[13]。东方田鼠是以取食植物绿色部分为主的草食性啮齿动物,野外生存时植物茎、叶在东方田鼠的食物组成中占绝对优势[14]。实验室条件下人工喂养的鼠饲料营养成分虽参照动物的生理特性进行制作,但与野生鼠觅食的情况仍有差异,且动物活动受限,能量需求较野生鼠也发生变化。我们推测,为了适应不同的环境,动物的生物学特性及代谢规律形成各自的规律和特点,这也是导致肠道菌群差异的主要原因。
本文对东方田鼠肠道菌群进行分析研究,发现不同的生活环境、饮食结构会对机体肠道微生物的组成产生影响,研究数据进一步充实了东方田鼠基础生物学数据,也为东方田鼠在比较医学及肠道微生态领域的研究开发提供参考。
(致谢:感谢中国科学院亚热带生态研究所王勇研究员、李波研究员、张美文研究员以及张琛同学在东方田鼠采样过程中提供的帮助!)
参考文献(References)
[1] Jiang WB, Hong Y, Peng JB, et al. Study on differences in the pathology, T cell subsets and gene expression in susceptible and non-susceptible hosts infected withSchistosomajaponicum[J]. PLoS One, 2010, 5(10): e13494.
[2] 阎玉涛,刘述先,宋光承,等.东方田鼠天然抗体相关的日本血吸虫抗原基因筛选和克隆[J].中国寄生学与寄生虫病杂志,2001,19(3):153-156.
Yan YT,Liu SX,Song GC, et al. Screening and cloning of genes encodingSchistosomajaponicumantigens related to the serun antibodies inMirotusfortis[J]. Chin J Parasitol Parasit Dis, 2001,19(3): 153-156.
[3] 俞远京, 苏志杰, 马亚东. 诱发性东方田鼠糖尿病动物模型的初步研究[J].湖南畜牧兽医,2003,4:8-9.
Yu YJ, Su ZJ, Ma YD, et al. The preliminary research on the induced diabetes animal model ofMirotusfortis[J]. Hunan J Animal Sci Vet Mede,2003,4:8-9.
[4] 冯洁, 柏熊, 沈志敏, 等. 不同生存条件下东方田鼠肝脏基因表达谱分析[J].中国实验动物学报, 2010,18(6):498-502.
Feng J, Bo X, Shen ZM, et al. Gene expression analysis in the liver ofMirotusfortisliving under different conditions[J].Acta Lab Anim Sci Sin, 2010,18(6): 498-502.
[5] 马亚东, 苏志杰, 俞远京.东方田鼠自发性卵巢癌及病理学特征初步观察[J].中国现代医学杂志, 2004,14(12):156.
Ma YD, Su ZJ, Yu YJ. Preliminary observation on spontaneous ovarian and pathology inMirotusfortis[J]. China J Modern Med, 2004, 14(12):156.
[6] Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biol, 2011,12(6):1-18.
[7] Ley RE, Peterson DA, Gordon JI. Ecological and evolutionary forces shaping microbial diversity in the human intestine [J]. Cell, 2006, 124(4):837-848.
[8] Backhed F, Ley RE, Sonnenburg JL, et al. Host-bacterial mutualism in the human intestine[J].Science, 2005, 307(5717):1915-1920.
[9] O’Hara AM, Shanahan F. The gut flora as a forgotten organ[J].EMBO Rep, 2006, 7(7):688-693.
[10] Shi P, Jia S, Zhang T, et al. Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water[J]. Water Res, 2013, 47(1):111-120.
[11] Maughan H, Wang PW, Diaz Caballero J, et al. Analysis of the cystic fibrosis lung Microbiota via serial illumina sequencing of bacterial 16S rRNA hypervariable regions[J].PloS One, 2012,7(10):e45791.
[12] Pedersen HK, Gudmundsdottir V, Nielsen HB, et al. Human gut microbes impact host serum metabolome and insulin sensitivity[J].Nature,2016,535 (7612):376-381
[13] Ley RE, Hamady M, Lozupone C, et al. Evolution of mammals and their gut microbes[J]. Science, 2008, 320(5883):1647-1651.
[14] 吴林,张美文,李波.洞庭湖区东方田鼠的食物组成调查[J].兽类学报,1998,18(4):282-291.
Wu L, Zhang MW, Li B. Studies on the food composition ofMirotusfortisin Dongting lake area[J]. Acta Theriol Sin, 1998, 18(4):282-291.
