多年连作土壤中棉花根际细菌群落结构及其动态
2018-04-09乔清华张传云袁哲诚王芙蓉张军
乔清华 ,张传云 ,袁哲诚 ,王芙蓉 *,张军
(1.山东师范大学生命科学学院,济南250100;2.山东棉花研究中心/农业部黄淮海棉花遗传改良与栽培生理重点实验室,济南250100)
1904年德国生物学家Hiltner首先提出根际的概念[1],根际是根周围受根影响的1个小区域内的土壤[2-3]。根际是植物和数百万计的土壤微生物相互作用的主要场所[4],是植物-土壤-微生物相互作用,并且进行物质能量交换和信号交流的1个重要的界面。根际微生物可以通过改变植物细胞代谢过程中的渗透作用、信号交流、酶的活性等影响植物的生长发育和生物非生物胁迫抗性[5-8]。例如根际促生菌可以直接或者间接为植物提供生长发育所需的磷和氮[9-13],分泌植物激素调节植物生长发育[14-17],提高植物对生物和非生物胁迫的抵抗能力[18-20]。另一方面,植物本身通过落叶、根系分泌物等形式向土壤中输入物质能量和信号调节根际环境,从而改变根际微生物的群落结构[21]。研究表明,植物通过光合作用固定的碳至少有21%通过根系分泌物等方式输入到土壤中,从而影响根际微生物的群落结构[22]。已经有大量的研究表明,植物根际微生物群落结构受生长环境、品种特性和发育时期等多种因素的影响[23-25]。
棉花是重要的经济作物,是天然纤维的主要来源。连作障碍引发的棉花大面积减产在我国主产棉区普遍存在。连作障碍是植物-土壤-微生物之间长期相互作用的结果。了解连作土壤中棉花根际微生物群落结构的动态变化,对人工干预棉花根际土壤微生物群落结构、适当调整棉花的生理周期、提高棉花对土传性致病菌的抗性或抑制土传致病菌的定殖具有重要意义。本研究利用高通量测序技术对多年棉花连作土中棉花不同发育时期的根际细菌群落结构进行研究,分析连作土壤中不同发育时期的棉花根际细菌群落结构特点,探明连作土壤中棉花根际细菌群落的动态变化规律,为克服连作障碍、促进棉花生产奠定理论基础。
1 材料与方法
1.1 材料
棉花材料选用陆地棉遗传标准系TM-1,由南京农业大学作物遗传与种质创新国家重点实验室张天真教授提供。
棉花多年连作土取样于山东棉花研究中心临清试验站棉田地表以下15~30 cm处土层。为了使大田土土质疏松易于取样,将大田土与灭菌后的细砂以质量比2∶1混匀后使用。
1.2 棉花种植和取样
种子脱绒后用10%(质量分数)的过氧化氢浸泡1 h进行消毒,然后用灭菌水反复冲洗3~5次。再用灭菌水浸泡12 h左右,种子吸足水后将其移至铺有灭菌湿润滤纸的培养皿里,28℃避光培养12~24 h。种子萌发后种植到装有大田土的花盆中放于28℃组培室育苗。待幼苗破土后转移到预先消毒过的温室中培养。每3 d浇1次灭菌水,幼苗期每次每盆500 mL,蕾期和花期每次每盆1 000 mL。
分别在幼苗期、蕾期、花期取样。将花盆倒扣,倒出土后轻轻抖动棉株,抖落多余的土壤,留下附着在根表面1 mm左右的根际土。将根剪下放于装有灭菌水的50 mL离心管中,用灭菌水反复冲洗后取出根,将根际土混悬液以104r·min-1离心15 min,倒掉上清液,沉淀液经液氮速冻,于-80℃冷冻备用。非根际土为不种植棉花的花盆中距土表6~15cm的土壤。每个处理3个重复。
1.3 DNA提取与测序分析
所获取的土壤样品DNA提取、检测和样品检测合格后建库测序以及下机数据过滤分析由深圳华大基因科技服务有限公司完成。
数据过滤采取按窗口去低质量的方法,设置30 bp为窗口长度,如果窗口平均质量值低于20,从窗口开始截除末端序列,移除最终长度低于原始长度75%的片段;去除接头污染的片段(接头序列与片段序列有15 bp的重叠,允许错配数为3;去除含N的片段;去除低复杂度的片段(片段中某个碱基连续出现的长度≥10 bp)。
序列拼接使用软件FLASH (Fast length adjustment of short reads,V1.2.11),利用重叠关系将双末端测序得到的成对片段组装成1条序列。最小匹配长度为15 bp,重叠区域允许错配率为0.1。
利用软件 USEARCH(V7.0.1090)将拼接好的片段聚类为运算分类单位 (Operational taxonomic unit,OTU)。得到OTU代表序列后,通过RDP classifer(v2.2)软件将OTU代表序列与数据库(Greengene)比对进行物种注释,置信度阈值设置为0.5。根据OTU在每个样品的丰度文件,计算每个样品或组别具有的OTU(不考虑OTU丰度,只考虑 OTU有无),通过 R语言(V3.0.3)中的VennDiagram包制作Venn图,并找出样品间或组间共有与特有的OTU。
α多样性指数 Observed species(Sobs)、Chao、Shannon稀释曲线是利用已测得序列中已知的各种OTU的相对比例,来计算抽取n个(n小于测得Reads序列总数)Tags时各Alpha指数的期望值,然后根据1组n值(一般为1组小于总序列数的等差数列,本项目公差为1 000)与其相对应的Alpha指数的期望值绘制曲线。通过mothur软件(V1.31.2)计算样品的Alpha多样性指数值并用R软件做出相应的稀释曲线图。计算公式参考:http://www.mothur.org/wiki/Calculators.
Beta多样性通过QIIME(V1.80)进行。由于不同样品的测序深度不一样,需要对每个样品的序列数进行统一。每个样品按所有样品中序列数最少的样品的序列数随机抽取序列,生成新的OTU table biom文件,并用该文件计算Beta多样性距离。
2 结果与分析
2.1 测序结果

图1 根际和非根际细菌共有和特有的OTU数目Fig.1 Common and special OTU numbers between rhizosphere and bulk soil bacterial community
在苗期、蕾期、花期收集棉花根际土和非根际土,利用Illumina MiSeq平台对土壤中的细菌16S rDNA进行扩增并测序。共得到2 037 056对高质量的reads,组装成2 025 475条tags。拼接后的序列经过优化后在97%的相似度下聚类为11 336个用于物种分类的OTU (表S1;注:表S1~7和图S1~2请参见本刊网站电子版,印刷版省略)。根际细菌OTU数量极显著高于非根际(P<0.01),并且随着发育时期的变化,棉花根际细菌的特异性OTU比例逐渐增加(图1),花期根际特有的OTU数量最高。不同发育时期之间的根际细菌OTU数量没有显著差异。
2.2 棉花不同发育时期根际微生物的群落组成
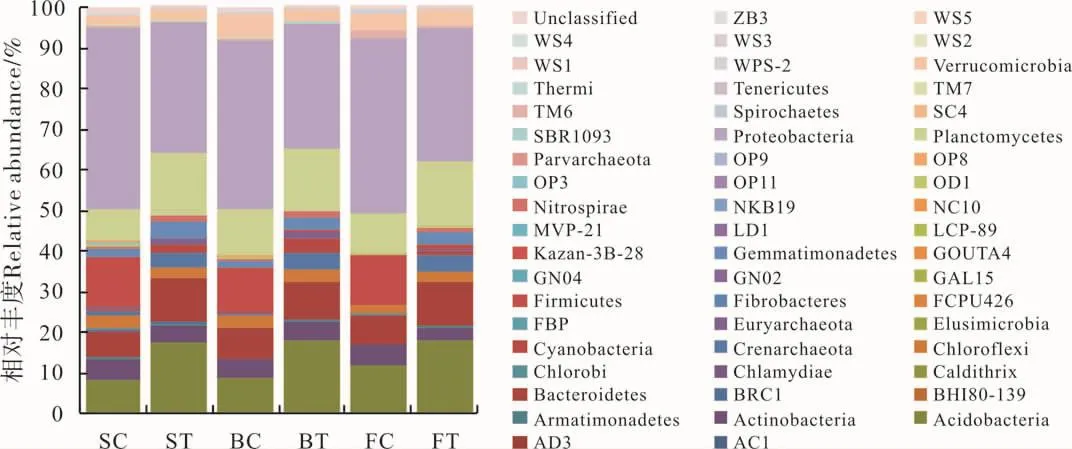
图2 棉花不同发育时期根际和非根际样品中细菌群落组成Fig.2 Bacterial community composition in rhizosphere and bulk soil during different development stage
棉花不同发育时期根际细菌各门的相对丰度存在差异,变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、浮霉 菌 门 (Planctomycetes)、拟杆菌门(Bacteroidetes)这4门细菌的丰度占整个细菌群落的73.25%~77.20%,在各时期棉花根际细菌群落中占主导地位,其中变形菌门的相对丰度远高于其他菌门,为30.44%~32.34%(图2、表S2)。每个菌门的主导菌纲或主导菌目(相对丰度>1%)在不同发育时期的根际土中相同。其中酸杆菌门的主导菌纲为Chloracidobacteria、Acidobacteria-6,约占酸杆菌门的 14.28%~15.00%;拟杆菌门的主导菌纲为Saprospirae、Cytophagia,约占拟杆菌门的8.94%~10.23%;浮霉菌门的主导菌纲为 Planctomycetia、Phycisphaerae,约占浮霉菌门的13.97%~15.21%;变形菌门的主导菌纲为 Alphaproteobacteria、Betaproteobacteria、Gammaproteobacteria、Deltaproteobacteria,约为变形菌门的30.22%~32.18%(表 S3)。
酸杆菌门、拟杆菌门、厚壁菌门(Firmicutes)、浮霉菌门、变形菌门在根际与非根际土中的相对丰度存在显著差异且差异较大(P<0.05,平均差值>0.03),为根际和非根际细菌群落中的主要差异菌门(表S2)。与非根际土相比,厚壁菌门、变形菌门在根际土中的相对丰度显著降低,其他菌门相对丰度显著升高。并且各菌门相对丰度升高或降低程度不同。例如3个时期平均酸杆菌门在根际土中的相对丰度比非根际土中高0.083 4±0.018 6,而拟杆菌门在根际土中的相对丰度比非根际土中高0.031 4±0.012 1。厚壁菌门在根际土中的相对丰度比非根际土中低0.1117±0.0070,变形菌门在根际土中的相对丰度比非根际土中低0.114 0±0.008 4(表 S2)。
与非根际土相比,棉花根际土中各菌属相对丰度增加或降低量存在差异。我们对与非根际土相比相对丰度显著增加或降低的菌属进行分析,发现Candidatus_Nitrososphaera、Steroidobacte等菌属在根际土中的相对丰度显著高于非根际土(P<0.05), 而Lactococcus、Rhodoplanes等菌属在根际土中的相对丰度显著低于非根际土。我们分析了在不同发育时期根际土中受到棉花根系的促进且与非根际土相比相对丰度差值较大的菌属(表1),发现3个发育时期受到显著促进作用的菌属基本相同。但是不同菌属受到的促进作用随发育时期的变化不同,例如Candidatus_Nitrososphaera、Steroidobacter、Planctomyces等菌属在根际与非根际土中的相对丰度差值随发育时期的变化逐渐升高,而Rubrivivax、Niastella在根际与非根际土中的相对丰度差值则随发育时期的变化逐渐降低,Novosphingobium、Chitinophaga在根际与非根际土中的相对丰度差值在蕾期最高。棉花对细菌的抑制作用在花期相对较弱。在花期根际土中相对丰度升高的菌属数量高于相对丰度降低的菌属数量(表S4)。
2.3 棉花不同发育时期的根际细菌α-多样性变化
用 Observed species(Sobs)、Chao 以及 Shannon指数衡量不同土壤的α-多样性。其中Sobs、Chao可以反映群落中物种的丰富度,而Shannon受群落中物种丰富度(Species richness)和物种均匀度 (Species evenness)的影响。此外,Sobs和Chao指数对应的稀释曲线还可以反映样品测序深度是否已经基本覆盖样品中所有的物种。由稀释曲线可以看出本试验的测序深度符合要求(表S5,图 S1、S2)。
由结果可以看出,棉花根际细菌群落的α-多样性极显著大于非根际土(P<0.01)。棉花不同发育时期的根际细菌α-多样性随着发育时期变化逐渐降低,但是差异不显著(P>0.05)。棉花根际和非根际细菌的Sobs和Chao指数在花期低于蕾期,而根际细菌群落的Shannon指数花期高于蕾期,非根际细菌Shannon指数低于蕾期,推测棉花在花期对根际细菌的均匀度有促进作用(表 S6)。
2.4 棉花不同发育时期根际细菌的β-多样性
为了比较不同土壤样品间的细菌群落多样性差异,我们用Bray-Curtis指数对样品的β-多样性进行分析。Bray-Curtis距离是反映2个群落之间差异性的常用指标,不考虑序列间的进化距离,只考虑2个样品中物种的存在性。Bray-Curtis值越大,表示2个样品间β-多样性越大。
对样品间多样性分析表明,不同发育时期根际细菌群落间存在差异(表S7)。棉花根际与非根际之间的细菌群落多样性差异随着发育时期的变化极显著升高(P<0.01,图4A),花期与蕾期的根际细菌群落差异显著大于蕾期与苗期(P<0.05,图4B)。说明棉花对根际微生物的影响随着发育时期的变化逐渐增加,在花期对根际细菌群落的影响比苗期和蕾期大。
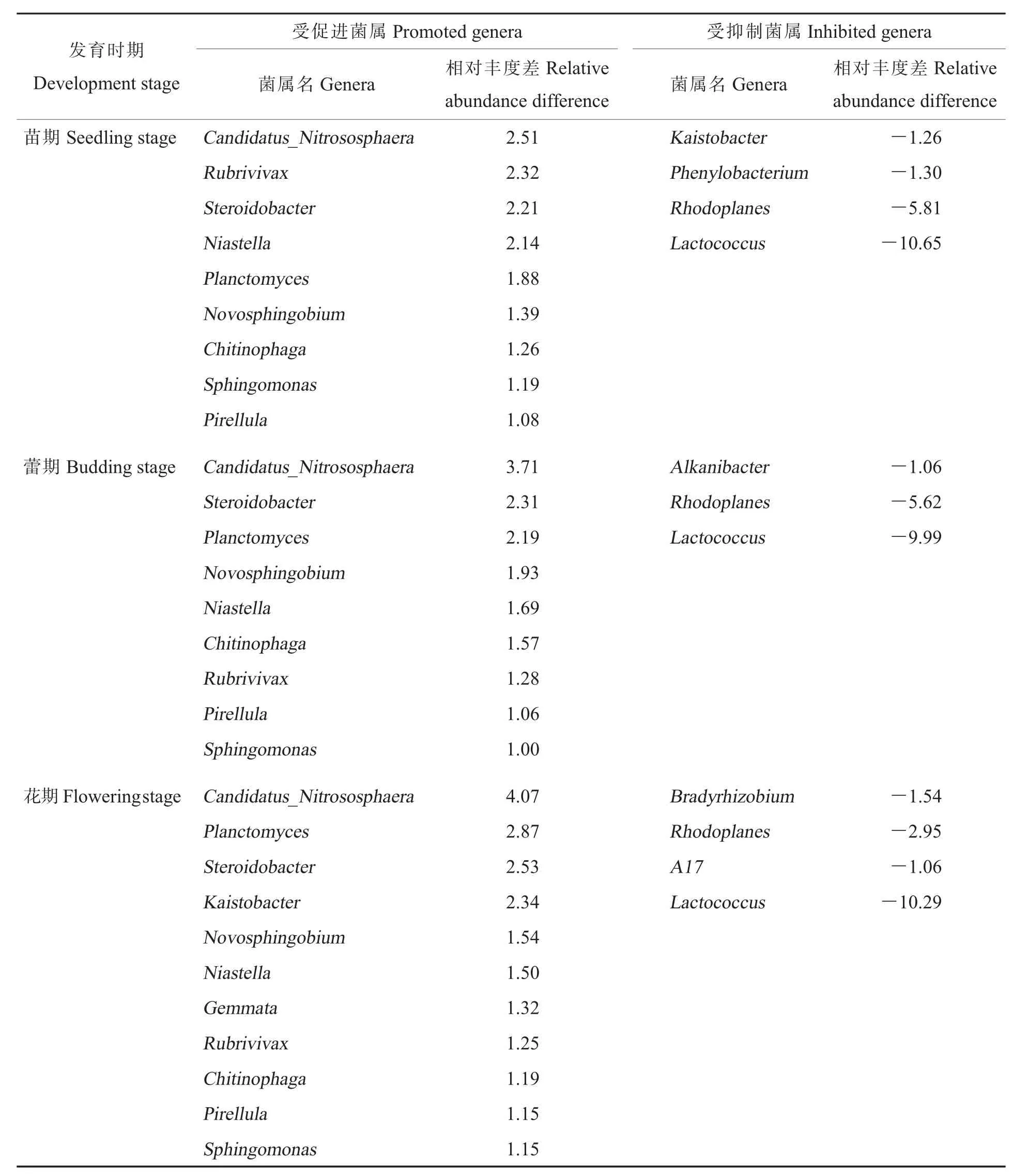
表1 各发育时期显著受棉花根系促进和抑制的菌属Table 1 Bacterial genera inhibited or promoted by cotton root in each development stages %
3 讨论
3.1 棉花根际细菌群落组成及其与非根际细菌群落之间的差异

图3 棉花不同发育时期根际和非根际土壤中Sobs、Chao以及Shannon指数变化Fig.3 Change of Sobs,Chao and Shannon indexes in rhizosphere and bulk soil during different development stages
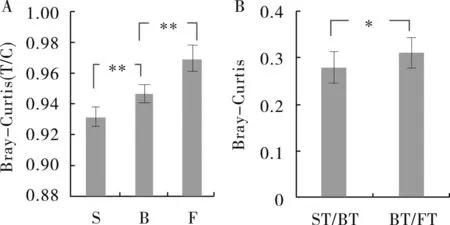
图4 棉花不同发育时期根际与非根际细菌群落间β-多样性和不同发育时期间的β-多样性比较Fig.4 β-diversity between rhizosphere and bulk soil in different development stages and β-diversity between different development stages
已有研究表明,植物可以与土壤和微生物相互作用,通过改变土壤的理化性质,释放根系分泌物等方式与根际微生物进行复杂的相互作用,调节微生物群落结构,形成其特有的根际微生物。本研究结果表明,在棉花多年连作土壤中变形菌门、酸杆菌门、浮霉菌门、拟杆菌门这4大菌门为棉花根际细菌群落的主导菌门,并且在不同发育时期每个菌门的主导菌纲相同。其中变形菌门为常见的土壤中相对丰度较高的菌门,其相对丰度远高于其他菌门。酸杆菌门是广泛存在于植物根际的主导菌门,能够降解多糖,所以可能在植物根际的碳循环中发挥重要作用[26]。浮霉菌门可能与氨的氧化有关[27],但是并不是植物根际细菌群落中常见的主导菌门。与其他植物相比,浮霉菌门在棉花根际土壤中的相对丰度较高,可能与棉花的某些生理特性有关。棉花根际与非根际细菌群落差异主要集中在4个主导菌门和厚壁菌门这5个菌门,并且酸杆菌门、拟杆菌门、浮霉菌门受到棉花根系促进,在根际土中的相对丰度高于非根际土,而厚壁菌门、变形菌门受到棉花根系抑制,在根际土中的相对丰度低于非根际土。这与之前的研究结果——长期连作土壤中厚壁菌门受到植物根系的显著抑制一致[28]。Dombrowski猜测,在寄主植物根系中丰度普遍升高的菌种是对寄主植物有特殊促进作用的菌种[29]。因此,我们推测酸杆菌门、拟杆菌门、浮霉菌门中的某些菌群对棉花生长发育有促进作用。
3.2 多年棉花连作大田土中棉花根际细菌群落的多样性高于非根际
已有研究表明,棉花连作导致土壤肥力下降,土壤酶活力、细菌群落物种丰富度降低[30-31]。张伟等的研究表明,棉花长期连作使土壤中的细菌群落物种丰富度降低,而多样性和均匀度指数增大[32]。本研究表明,与非根际土相比,棉花根际细菌群落的物种丰富度显著增加,并且丰度受到促进的菌属数量远高于受到抑制的菌属数量。推测多年棉花连作土壤中的微生物由于受到棉花根系长期的选择作用,其物种丰富度降低;多年棉花连作大田土中的细菌群落主要由受棉花根系促进的菌属组成,所以多年棉花连作大田土中的棉花根际土壤中细菌群落多样性高于非根际土,这与Li等对黑胡椒连作土壤中根际和非根际微生物群落结构变化的研究结果一致[28]。
3.3 发育时期对棉花根际细菌群落结构的影响
Baudoin等认为随着植物发育时期的变化,植物向土壤中输入的根系分泌物的数量和质量存在差异,导致植物不同发育时期根际微生物的差异[33]。随后不断有研究结果证明植物根际微生物受发育时期显著影响[34-36]。我们的研究结果表明,多年连作大田土中棉花根际微生物群落结构受棉花发育时期显著影响。不同菌属在棉花根际受到促进或者抑制,其在根际与非根际土中的相对丰度差值大小随发育时期的变化而不同。推测每个发育时期受到促进或抑制作用较大的菌属可能与这个时期特殊的棉花生长发育生理特性有关。β-多样性分析表明,随着发育时期的变化,棉花对根际微生物群落结构的影响越来越大,并且棉花蕾期与花期根际细菌群落结构的差异大于苗期与蕾期根际细菌群落结构的差异。我们推测棉花花期的根际细菌群落结构与其他2个时期相比差异较大,可能是该时期棉花根系对细菌的选择作用降低造成的。
棉花连作是我国棉区的主要耕作方式。土壤中微生物群落结构和功能的多样性降低程度是衡量连作障碍的重要指标。土壤微生物在调节植物的生长发育、营养物质吸收、信号转导、抵御生物和非生物胁迫等过程中都有非常重要的作用。所以了解棉花在连作条件下的根际微生物群落结构动态变化可为将来人为干涉棉田微生物群落结构和功能奠定基础,且对生物有机肥料的开发和利用、减少化学农药和化肥的使用、保护农田生态系统有重要意义。
参考文献:
[1]Hiltner L.U¨ber neuere Erfahrungen und probleme auf dem gebiete der bodenbakteriologie unter besonderer berücksichtigung dergründüngungundbrache[J].Arb Dtsch LandwGesBerl,1904,98:59-78.
[2]Hartmann A,Rothballer M,Schmid M.Lorenz Hiltner,a pioneer in rhizosphere microbial ecology and soil bacteriology research[J].Plant and Soil,2007,312(1):7-14.
[3]Darrah P R.The rhizosphere and plant nutrition:A quantitative approach[J].Plant and Soil,1993,155/156(1):1-20.
[4]Mendes R,Garbeva P,Raaijmakers J M.The rhizosphere microbiome:Significance of plant beneficial,plant pathogenic and human pathogenic microorganisms[J].FEMS Microbiology Reviews,2013,37:634-663.
[5]Berendsen R L,Pieterse C M,Bakker P A.The rhizosphere microbiome and plant health[J].Trends in Plant Science,2012,17(8):478-486.
[6]Soussi A,Ferjani R,Marasco R,et al.Plant-associated microbiomes in arid lands:Diversity,ecology and biotechnological potential[J].Plant and Soil,2016,405(1):357-370.
[7]Lugtenberg B,Kamilova F.Plant-growth-promoting rhizobacteria[J].Annual Review of Microbiology,2009,63:541-556.
[8]Lareen A,Burton F,Scha¨fer P.Plant root-microbe communication in shaping root microbiomes[J].Plant Molecular Biology,2016,90(6):575-587.
[9]Smith S E,Smith F A.Roles of arbuscular mycorrhizas in plant nutrition and growth:New paradigms from cellular to ecosystem scales[J].Annual Review of Plant Biology,2011,62:227-250.
[10]Karandashov V,Bucher M.Symbiotic phosphate transport in arbuscular mycorrhizas[J].Trends in Plant Science,2005,10(1):22-29.
[11]Ling N,Deng K,Song Y,et al.Variation of rhizosphere bacterial community in watermelon continuous mono-cropping soil by long-term application of a novel bioorganic fertilizer[J].Microbiological Research,2014,169(7/8):570-578.
[12]Prasad R,Kumar M,Varma A.Role of PGPR in soil fertility and plant health[M]//Egamberdieva D,Shrivastava S,Varma A.Plant-growth-promoting rhizobacteria(PGPR)and medicinal plants.Cham,Switzerland:Springer International Publishing,2015:247-260.
[13]Carvalhais L C,Muzzi F,Tan C H,et al.Plant growth inArabidopsisis assisted by compost soil-derived microbial communities[J/OL].Frontiers in Plant Science,2013,4(1):235(2013-07-04)[2017-01-01].https://doi.org/10.3389/fpls.2013.00235.
[14]Wagner M R,Lundberg D S,Coleman-Derr D,et al.Natural soil microbes alter flowering phenology and the intensity of selection on flowering time in a wildArabidopsisrelative[J].Ecology Letters,2014,17(6):717-726.
[15]Panke-Buisse K,Poole A C,Goodrich J K,et al.Selection on soil microbiomes reveals reproducible impacts on plant func-tion[J].The ISME Journal,2015,9(4):980-989.
[16]WintermansPC,BakkerPA,PieterseCM.NaturalgeneticvariationinArabidopsisforresponsivenesstoplantgrowth-promoting rhizobacteria[J].PlantMolecularBiology,2016,90(6):623-634.
[17]do Amaral F P,Pankievicz V C,Arisi A C,et al.Differential growth responses of Brachypodium distachyon genotypes to inoculation with plant growth promoting rhizobacteria[J].Plant Molecular Biology,2016,90(6):689-697.
[18]Bloemberg G V,Lugtenberg B J.Molecular basis of plant growth promotion and biocontrol by rhizobacteria[J].Current Opinion in Plant Biology,2001,4(4):343-350.
[19]Van der Ent S,Van Hulten M,Pozo M J,et al.Priming of plant innate immunity by rhizobacteria and beta-aminobutyric acid:differences and similarities in regulation[J].The New Phytologist,2009,183(2):419-431.
[20]Lau J A,Lennon J T.Rapid responses of soil microorganisms improve plant fitness in novel environments[J].Proceedings of theNationalAcademyofSciences,2012,109(35):14058-14062.
[21]Haichar F Z,Santaella C,Heulin T,et al.Root exudates mediated interactions belowground[J].Soil Biology and Biochemistry,2014,77(7):69-80.
[22]Marschner H.Mineral nutrition of higher plants[M].2ndEd.London,England:Academic Press,1995:229-312.
[23]Smalla K W G,Buchner A,Zock A,et al.Bulk and rhizosphere soil bacterial communities studied by denaturing gradient gel electrophoresis:Plant-dependent enrichment and seasonal shifts revealed[J].Applied and Environmental Microbiology,2001,67(10):4742-4751.
[24]Marques J M,da Silva T F,Vollu R E,et al.Plant age and genotype affect the bacterial community composition in the tuber rhizosphere of field-grown sweet potato plants[J].FEMS Microbiology Ecology,2014,88(2):424-435.
[25]Ibekwea A M,Orsb S,Ferreiraa J F S,et al.Seasonal induced changes in spinach rhizosphere microbial community structure with varying salinity and drought[J].Science of the Total Environment,2017,579:1485-1495.
[26]Ward N L,Challacombe J F,Janssen P H,et al.Three genomes from the phylum Acidobacteria provide insight into the lifestyles of these microorganisms in soils[J].Applied and environmental microbiology,2009,75(7):2046-2056.
[27]Strous M,Fuerst J A,Kramer E H,et al.Missing lithotroph identified as new planctomycete[J].Nature,1999,400(6743):446-449.
[28]Li Zhigang,Zu Chao,Wang Can,et al.Different responses of rhizosphere and non-rhizosphere soil microbial communities to consecutivePiper nigrumL.monoculture[J/oL].Scientific Reports,2016,6:35825(2016-10-24)[2017-01-01].https://doi.org/10.1038/srep35825.
[29]Dombrowski N.Structural and functional analysis of the bacterial root microbiota ofArabidopsis thalianaand relative species[D].Ko¨ln:Universita¨t zu Ko¨ln,2015.
[30]李锐,刘瑜,褚贵新,等.棉花连作对北疆土壤酶活性、致病菌及拮抗菌多样性的影响[J].中国生态农业学报,2015,23(4):432-440.Li Rui,Liu Yu,Chu Guixin,et al.Cotton continuous cropping influence to soil enzyme activity,pathogens,and antagonistic bacterial diversity in northern Xinjiang[J].Journal of Chinese Ecological Agriculture,2015,23(4):432-440.
[31]刘小龙,马建江,管吉钊,等.连作对棉田土壤枯、黄萎病菌数量及细菌群落的影响[J].棉花学报,2015,27(1):62-70.Liu Xiaolong,Ma Jianjiang,Guan Jizhao,et al.Effects of continuous cropping on numbers ofFusarium oxysporum,Verticillium dahliaeand soil bacterial community in cotton field[J].Cotton Science,2015,27(1):62-70.
[32]张伟,陈一峰.棉花长期连作对新疆土壤细菌群落结构的影响[J].生态学报,2014,36(16):4682-4689.Zhang Wei,Chen Yifeng.Effect of cotton long term continuous cropping to soil bacterial community structure in XinJiang[J].Acta Ecologica Sinica,2014,36(16):4682-4689.
[33]Baudoin E,Benizri E,Guckert A.Impact of growth stage on the bacterial community structure along maize roots,as determined by metabolic and genetic fingerprinting[J].Applied Soil Ecology,2002,19(2):135-145.
[34]Okubo T,Tokida T,Ikeda S,et al.Effects of elevated carbon dioxide,elevated temperature,and rice growth stage on the community structure of rice root-associated bacteria[J].Microbes and Environments,2014,29(2):184-190.
[35]Inceoglu O,Salles J F,van Overbeek L,et al.Effects of plant genotype and growth stage on the betaproteobacterial communities associated with different potato cultivars in two fields[J].Applied and Environmental Microbiology,2010,76(11):3675-3684.
[36]Li Xiangzhen,Rui Junpeng,Mao Yuejian,et al.Dynamics of the bacterial community structure in the rhizosphere of a maize cultivar[J].Soil Biology and Biochemistry,2014,68:392-401.
