PCR-DGGE分析不同品牌腐乳中细菌的多样性
2017-08-07陈颖慧
陈颖慧
(永城职业学院 食品化工系,河南 永城 476600)
PCR-DGGE分析不同品牌腐乳中细菌的多样性
陈颖慧
(永城职业学院 食品化工系,河南 永城 476600)
以不同品牌腐乳为研究对象,利用聚合酶链式反应的变性梯度凝胶电泳技术对腐乳中的细菌进行多样性分析。结果表明:克东牌腐乳的细菌多样性最低,广合牌腐乳的细菌多样性最高。乳酸杆菌属是腐乳中细菌的优势菌群。乳酸菌属、藤黄微球菌和粪肠球菌为4种品牌腐乳的共有菌株。融合魏斯氏菌、瑞士乳杆菌和乳酸链球菌存在于老才臣牌腐乳、广合牌腐乳和王致和牌腐乳中。植物乳杆菌和发酵乳杆菌存在于广合牌腐乳和王致和牌腐乳中。玫瑰考克氏菌只存在于克东牌腐乳中,而醋酸钙不动杆菌只存在于王致和牌腐乳中。可知不同品牌腐乳中细菌的多样性存在着差异。
腐乳;细菌;多样性;聚合酶链式反应的变性梯度凝胶电泳
腐乳,又被称为豆腐乳,是我国重要的调味品之一,其风味鲜咸、口感软滑、营养价值较高,深受我国消费者的喜爱[1,2]。在腐乳的发酵过程中细菌扮演了至关重要的角色,与腐乳的风味、口感和营养物质的变化密切相关,研究已经表明玫瑰耐盐球菌、植物乳杆菌和藤黄微球菌等微生物在腐乳发酵过程中都发挥着重要作用[3-5]。市场上腐乳产品多种多样,也会呈现出不同的风味,因此对不同品牌腐乳产品进行细菌多样性分析十分必要。
目前,大多数的研究只停留在腐乳中微生物的分离和鉴定,但是传统分离鉴定的方法不仅费时费力,而且容易受到培养条件的限制,很难达到分析样品细菌多样性的目的[6,7]。聚合酶链式反应的变性梯度凝胶电泳(Polymerase Chain Reaction-Denaturing Gradient Gel Electrophoresis, PCR-DGGE)技术能够不经过分离鉴定,通过样品的DNA指纹图谱反映多样性情况,并结合基因测序技术对目的条带进行鉴定,能够满足揭示乳酸菌多样性的需要[8,9]。因此,本研究利用PCR-DGGE技术分析不同品牌腐乳中细菌群落特征的差异,为腐乳发酵剂的筛选提供参考,具有一定的应用价值。
1 材料与仪器
1.1 主要材料
1.1.1 样品来源
4种不同品牌腐乳产品:分别为A(克东牌腐乳),B(老才臣牌腐乳),C(广合牌腐乳),D(王致和牌腐乳),购买于沃尔玛超市。
1.1.2 主要试剂
dNTP(2.5 mmol/L)、10×rTaq Buffer(含15 mmol/L MgCl2)、TaqDNA聚合酶、6×Loading Buffer、Marker I和双蒸水 上海吉泰依科赛生物科技有限公司;TAE缓冲液、TE缓冲液、氯仿、冰乙酸、蛋白酶K、琼脂糖、丙烯酰胺、聚丙烯酰胺和过硫酸铵 无锡蓝波化学品有限公司;GeneFinder电泳核酸染料 北京金博益生物技术有限公司;引物Lac1和Lac2,40bp“GC” 苏州金唯智生物科技有限公司。
1.2 实验仪器
YP102N电子天平 成都一科仪器设备有限公司;H2100R台式高速冷冻离心机 湘仪离心机仪器有限公司;涡旋振荡器 上海纳兹仪器有限公司;JK-WB-2A数显恒温水浴锅 上海精学科学仪器有限公司;EPS-300稳压稳流电泳仪 上海天能科技有限公司;变性梯度凝胶电泳仪 美国Bio-Rad公司。
2 方法
2.1 样品DNA提取及PCR扩增
根据谢显华等[10]的报道,对腐乳样品中的总DNA进行提取。以提取的总DNA为模板,对腐乳中的细菌16S rRNA V3区进行扩增。所用的引物BA338f(5'-ACTCCTACGGGAGGCAGCAG-3'),UN518r(5'-ATTACCGCGGCTGCTCC-3'),BA338f引物的5'端加一个40 bp“GC”夹,扩增条件为92 ℃预变性2 min,92 ℃变性1 min,55 ℃退火30 s,72 ℃延伸1 min,循环30次,最后76 ℃延伸6 min[11]。
2.2 DGGE分析
腐乳中细菌DGGE分析的变性凝胶梯度为30%~55%,将32 μL乳酸菌PCR特异产物与8 μL 6×Loading Buffer混合后进行上样,加入TAE缓冲液并保持在60 ℃、电压70 V、电泳18 h[12]。电泳结束后小心将DGGE胶体取下,并用去离子水冲洗2次,配制1%的GeneFinder的染色液,浸没胶体,在摇床上避光染色30 min,最后利用UVP凝胶成像系统记录结果,得到DGGE图谱,并利用Shannon-Weaver指数分析腐乳中乳酸菌的多样性,公式如下:
Shannon-Weaver=-∑Pilnpk=-∑(Ni/n)ln(Ni/N)。
式中:Pi为样品中某一条带强度在该样品的所有条带总强度所占的比例;Ni为第i个条带的光密度;N为泳道中所有条带的光密度值之和。
2.3 基因测序
选取DGGE图谱不同位置的优势条带在紫外灯下进行切胶,将切下的凝胶转移到PCR管中,利用无菌水对其进行冲洗,捣碎后放入TE缓冲液中,并在4 ℃冰箱中放置12 h。取上清液5 μL,对其进行PCR特异扩增,PCR产物送往苏州金唯智生物科技有限公司进行基因测序。将测序结果进行Blast比对,从而完成菌株的鉴定。
3 结果与分析
3.1 不同品牌腐乳中细菌DNA-PCR扩增结果
利用1%的琼脂糖凝胶电泳对不同品牌腐乳中的细菌DNA扩增产物进行检验,结果见图1。
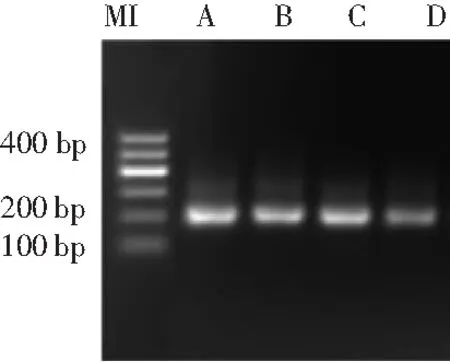
图1 不同品牌腐乳中细菌DNA的扩增产物
注:MI表示Marker I;A~D表示样品编号。
由图1可知,所有样品的条带大小在200 bp左右,条带均一清晰,可认为是细菌的目的条带。
3.2 不同品牌腐乳中乳酸菌的生物多样性分析
利用DGGE电泳技术分析不同品牌腐乳中细菌的生物多样性,结果见图2。

图2 不同品牌腐乳中细菌的DGGE图谱
注:A~D表示样品编号;1~13表示DGGE图谱中不同位置条带编号。
在此基础上,根据DGGE图谱中每个样品的条带数及Shannon-Weaver指数评估其多样性,结果见图3。
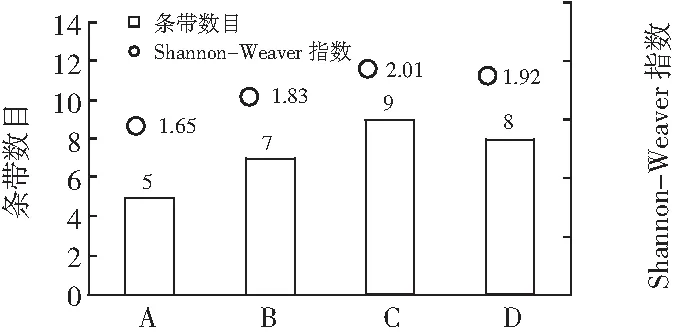
图3 不同品牌腐乳中细菌的条带数目及Shannon-Weaver指数
由图2可知,4个来自不同品牌腐乳中细菌的DGGE图谱中共有13个不同位置的条带,且不同样品的图谱也存在着一定的差异。由图3可知,4个样品的DGGE条带的个数分别为C(广合牌腐乳,9条)>D(王致和牌腐乳,8条)>B(老才臣牌腐乳,7条)>A(克东牌腐乳,5条)。Shannon-Wiener指数是评价样品生物多样性的重要参数,且Shannon-Wiener指数越高说明样品中微生物的种类越多,生物多样性就越高。Shannon-Weaver指数结果表明(见图3):C中细菌的生物多样性最高,而A中细菌的生物多样性最低,可见4种不同品牌的腐乳样品中细菌的生物多样性存在一定的差异。
3.3 DGGE图谱优势条带测序分析
将13个不同位置的条带进行割胶、PCR扩增及基因测序,条带编码见图2,测序结果见表1。
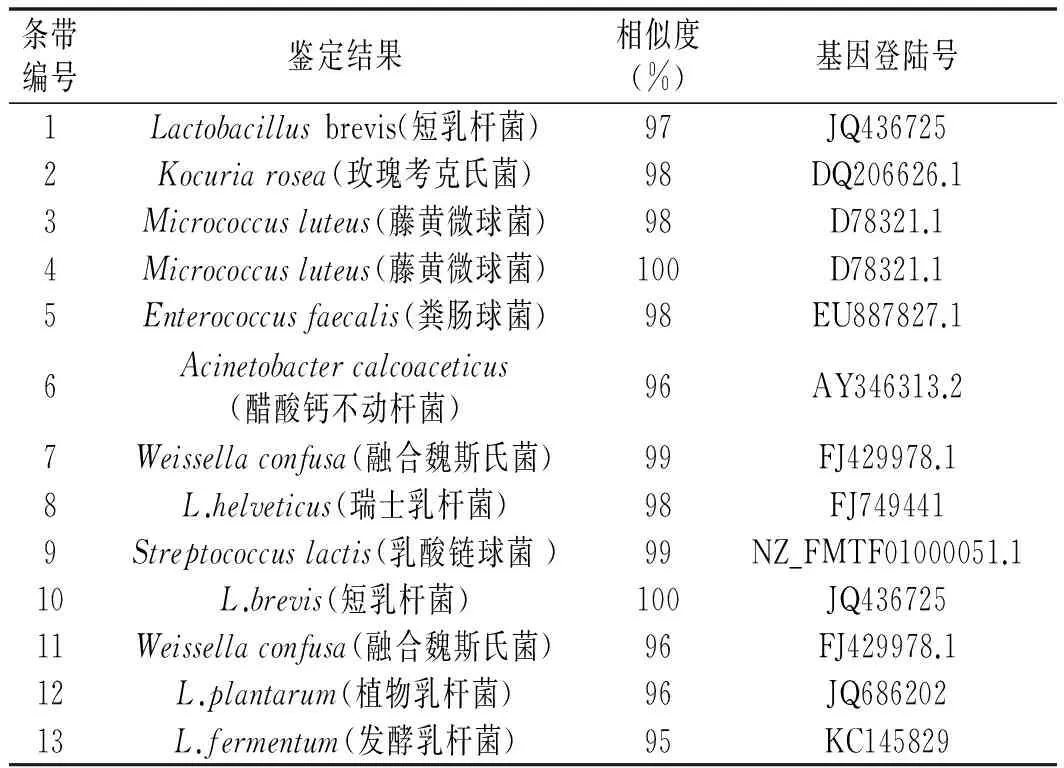
表1 DGGE图谱中条带Blast对比结果Table 1 Blast comparison results of brands in DGGE profiles
13个不同的条带代表了10种不同的细菌,其中乳酸杆菌属是腐乳中细菌的优势属。乳酸菌属、藤黄微球菌和粪肠球菌在4种不同品牌的腐乳中均有发现。融合魏斯氏菌、瑞士乳杆菌和乳酸链球菌存在于B,C,D的样品中。植物乳杆菌和发酵乳杆菌则存在于C和D中。玫瑰考克氏菌只存在于A样品中,而醋酸钙不动杆菌只存在于D样品中,可见4种不同品牌的腐乳样品中的细菌群落组成也存在着一定的差异。
4 结论
腐乳风味的形成主要取决于微生物的发酵,腐乳中细菌群落特征的差异是不同品牌风味不同的重要原因之一。在本研究中,4种品牌的腐乳中都含有乳酸杆菌属、藤黄微球菌和粪肠球菌,其中乳酸杆菌属是优势菌群。同时,不同样品中细菌多样性也存在着一定的差异,C(广合牌腐乳)中细菌的生物多样性最高,而A(克东牌腐乳)中细菌的生物多样性最低,此外4种不同品牌的腐乳样品中的细菌群落组成也存在着差异。本研究的结果揭示不同品牌腐乳细菌多样性的差异,为腐乳的工业生产及相关发酵剂的开发提供理论依据。
[1]李幼筠.中国腐乳的现代研究[J].中国酿造,2006,25(1):4-7.
[2]李理,罗立新,梁世中.腐乳的研究进展[J].食品与发酵工业,2002,28(10):70-74.
[3]鲁绯,张京生,刘子鹏,等.青方腐乳中乳酸菌的分离鉴定[J].食品与发酵工业,2006,32(4):38-41.
[4]陈曦.藤黄微球菌(Micrococcusluteus)KDF2及其胞外蛋白酶KRP2在腐乳中的应用研究[D].哈尔滨:东北农业大学,2014.
[5]李娟娟.玫瑰考克氏菌(Kocuriarosea)KDF3及其胞外蛋白酶KRP3在腐乳中的应用研究[D].哈尔滨: 东北农业大学,2014.
[6]费鹏.轮状病毒腹泻婴儿肠道微生态变化的研究[D].哈尔滨:东北农业大学,2013.
[7]Fei P,Li L,Cai X,et al.Differences in the biodiversity of the fecal microbiota of infants with rotaviral diarrhea and healthy infants[J].Jundishapur Journal of Microbiology,2016,9(4):32-36.
[8]费鹏,白洪健,程述震,等.PCR-DGGE法分析婴儿肠道菌群多样性[J].食品与机械,2013,29(2):60-63.
[9]Muyzer G,Smalla K.Application of denaturing gradient gel electrophoresis(DGGE)and temperature gradient gel electrophoresis (TGGE) in microbial ecology.[J].Antonie van Leeuwenhoek,1998,73(1):127-141.
[10]谢显华,李国基.ERIC-PCR在腐乳品质检测中的应用[J].食品研究与开发,2010,31(3):135-138.
[11]陶东娅,金银旗.不同地区粘豆包酸面团中微生物多样性分析[J].食品研究与开发,2016,37(13):156-159.
[12]张巧云,孟祥晨.几种发酵豆酱的微生物组成及理化性质分析[J].食品科技,2013(8):266-270.
Bacterial Diversity Analysis of Fermented Bean Curd with Different Brands
CHEN Ying-hui
(Department of Food and Chemical Engineering, Yongcheng Vocational College, Yongcheng 476600,China)
Fermented bean curd with different brands is as the research object in this study. The bacterial diversity of samples is analyzed using polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE) technology. The results show that the bacterial diversity of Kedong fermented bean curd is the lowest, in the meanwhile, the bacterial diversity of Guanghe fermented bean curd is the highest.Lactobacillusspp. is the dominant bacterial community.Lactobacillusspp.,MicrococcusluteusandEnterococcusfaecalisare found in all samples.Weissellaconfusa,L.helveticusandStreptococcuslactisare presented in Laocaichen, Guanghe and Wangzhihe fermented bean curd.L.plantarumandL.fermentumare identified in Guanghe and Wangzhihe fermented bean curd.Kocuriaroseais only found in Kedong fermented bean curd.Acinetobactercalcoaceticusis only identified in Wangzhihe fermented bean curd. Conclusively, there are some differences in bacterial diversity among fermented bean curd with different brands.
fermented bean curd;bacteria;diversity;polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE)
2017-02-01
陈颖慧(1981-),女,吉林白城人,讲师,硕士,研究方向:食品科学与工程。
TS214.2
A
10.3969/j.issn.1000-9973.2017.07.007
1000-9973(2017)07-0029-04
