防御假单胞菌Pf-5中Rsm调控系统对脂肪酶表达的影响
2017-07-07李猛刚闫云君
李猛刚,闫云君
(华中科技大学生命科学与技术学院,湖北 武汉,430074)
防御假单胞菌Pf-5中Rsm调控系统对脂肪酶表达的影响
李猛刚,闫云君
(华中科技大学生命科学与技术学院,湖北 武汉,430074)
Rsm系统在细菌基因表达调控中发挥着重要作用。以防御假单胞菌Pf-5为研究对象,将其中的rsmY、rsmZ基因敲除,构建rsmY、rsmZ单突变株和rsmY/rsmZ双突变株,然后分析野生菌和突变株中lipA的全细胞脂肪酶相对活性及染色体整合型lipA-lacZ、lipA′-′lacZ、rsmA′-′lacZ、rsmE′-′lacZ的β-半乳糖苷酶酶活。结果表明,rsmY、rsmZ主要在转录后水平影响lipA的表达;rsmY、rsmZ均抑制rsmA和rsmE的表达,在rsmY、rsmZ敲除后,rsmA的表达水平有所提高,而rsmE的表达水平则显著提高。因此,rsmY、rsmZ对rsmE的影响要明显高于对rsmA的影响,其主要是通过影响rsmE的表达从而调控Pf-5中lipA的表达。
基因表达调控;脂肪酶;假单胞菌;Rsm;基因敲除;小分子RNA
非编码RNA(non-coding RNA,ncRNA)是一段基因间区域,此区间段的基因无法翻译或是编码成蛋白质,基因序列缺乏开放阅读框,在原核细胞中通常称为sRNA(small RNA)[1]。研究发现[2-5],细菌中一些调控因子在调控基因表达的时候需要sRNA(例如RsmX、RsmY、RsmZ)的调节和参与。这些sRNA与蛋白竞争性地和RsmA结合从而调控基因表达。RsmA是假单胞菌属中高度保守的全局性调控因子,在荧光假单胞菌P.fluorescensCHA0中,它是一个负调控因子,能与RNA分子结合,在转录后水平控制着受GacA/GacS系统调节的次生代谢相关基因的表达。RsmA主要通过与调控基因的mRNA结合来抑制调控基因的表达[6],而这种结合会被rsmX/Y/Z与RsmA的结合所阻止[2,7]。在防御假单胞菌P.protegensPf-5中存在着rsmX、rsmY、rsmZ、rsmA、rsmE等非编码RNA,它们在细菌基因的表达调控中发挥着重要的作用[8],一起构成了P.protegensPf-5中的Rsm系统。为进一步了解P.protegensPf-5中调控脂肪酶lipA表达的分子机理,本文通过基因敲除、全细胞脂肪酶相对活性的测定以及qRT-PCR等方法重点研究了Rsm系统与lipA表达的关系。
1 实验
1.1 材料及试剂
1.1.1 菌株及质粒
防御假单胞菌P.protegensPf-5,野生菌株,本实验室保存。Pf6285,rsmZ突变菌株;PfΔrsmY,rsmY突变菌株;PfΔrsmYΔrsmZ,rsmY和rsmZ双突变菌株。突变菌株均由本研究所构建。
pRK2073,三亲杂交辅助质粒;pJQ200SK,敲除用质粒;穿梭质粒pBBRM1CS-5,过表达用质粒。
1.1.2 培养基
采用LB培养基(包括液态和固态)。配制培养基的蛋白胨、酵母提取物以及琼脂糖购自Oxoid公司。培养基中抗生素使用浓度:氨苄青霉素,100μg/mL;庆大霉素,50μg/mL;卡那霉素,50μg/mL;大观霉素,100μg/mL。
1.1.3 试剂
实验中所用的各种酶,包括限制性内切酶、Taq酶等都购自宝生物工程(大连)有限公司。质粒提取试剂盒以及细菌总DNA提取试剂盒购自OmegaBio-Tek公司,反转录试剂盒购自ThermoFisherScientific公司。
1.2 实验方法
1.2.1 rsmY及rsmZ基因敲除
利用二次同源重组的办法,以蔗糖作为负筛选标记,采用敲除质粒pJQ200SK进行无痕敲除,只将整个目标基因序列敲除,而不影响其他基因序列。首先采用融合PCR手段将rsmY上下游序列整合在一起,然后将缺失rsmY序列的ΔrsmY序列与pJQ200SK载体连接,通过三亲杂交方式转入野生型Pf-5,ΔrsmY序列通过同源重组的方法将野生型的rsmY序列置换,就构建成了ΔrsmY突变菌株(命名为PfΔrsmY)。用同样方法构建rsmZ突变菌株(命名为Pf6285)和rsmY/rsmZ双突变菌株(命名为PfΔrsmYΔrsmZ)。
1.2.2 菌株生长曲线的制作
分别取野生菌株Pf-5和上述3种突变菌株在5mL的LB液体培养基中28 ℃、200r/min培养16h,然后测其OD600值。将各菌液的OD600值调为一致,按照1%的接种量转接种于100mL的LB液体培养基中,28 ℃、200r/min培养,每隔2h取样,测OD600值。以培养时间为横坐标、菌液OD600值为纵坐标做生长曲线。
1.2.3 lacZ融合报告菌株的构建
lacZ融合载体的构建方法为:①以Pf-5基因组DNA为模板,进行PCR扩增lipA′-P(+6bp到+891bp);②将扩增的lipA′-P片段用KpnI-HindIII进行双酶切,然后与′lacZ(+22bp到+3110bp)片段进行酶连;③将lipA′-P与′lacZ连接成功的片段以及载体pJQ200SK进行SphI-BamHI双酶切后连接;④将连接产物转化至E.coliTop10后涂抗性平板;⑤通过菌落PCR鉴定阳性克隆子;⑥提取阳性克隆子的质粒并测序。将测序验证正确的重组质粒命名为pJQ003,也就是lipA′-′lacZ融合质粒。
采用同样的方法构建lipA-lacZ(lipA的序列为-16bp到+891bp,lacZ序列为-10bp到+3110bp)融合质粒pJQ004、rsmA′-′lacZ融合质粒pJQ005(rsmA′的序列为+6bp到+189bp)和rsmE′-′lacZ融合质粒pJQ006(rsmE′的序列为+6bp到+195bp)。通过三亲杂交将pJQ003、pJQ004、pJQ005和pJQ006分别导入到Pf-5以及Pf6285、PfΔrsmY、PfΔrsmYΔrsmZ中构建染色体整合型lipA′-′lacZ翻译融合报告菌株、lipA-lacZ转录融合报告菌株、rsmA′-′lacZ翻译融合报告菌株和rsmE′-′lacZ翻译融合报告菌株。
1.2.4 lipA的qRT-PCR
qRT-PCR实验步骤如下:①将野生菌Pf-5和3种突变菌株分别接种于50mL的LB培养基中,28 ℃摇床培养至OD600约为5.7;②用RNApureBacteriaKit(DNaseI)试剂盒抽提总RNA;③对RNA质量进行检测及定量;④根据RevertAid First Strand cDNA Synthesis Kit试剂盒的说明合成cDNA;⑤利用rpoD作为内参基因,分析lipA在不同突变株中的表达差异。
1.2.5 全细胞脂肪酶及β-半乳糖苷酶酶活测定
①将过夜培养的细菌培养物接种于50 mL LB培养基中,28 ℃或37 ℃培养12 h;②4 ℃、5000 r/min离心10 min收集菌体,用5 mL0.9%NaCl溶液重悬菌体,重复该步操作2次;③再用5 mL0.9%NaCl溶液重悬菌体,即得全细胞。全细胞脂肪酶酶活测定采用pNP法[9],β-半乳糖苷酶酶活测定参照Miller法[10]。
2 实验结果
2.1rsmY及rsmZ敲除结果
采用无痕敲除的办法将目标基因序列完全敲除,同时不引入外源基因序列,通过蔗糖负筛选标记对突变株进行筛选,效果较好,如图1所示。
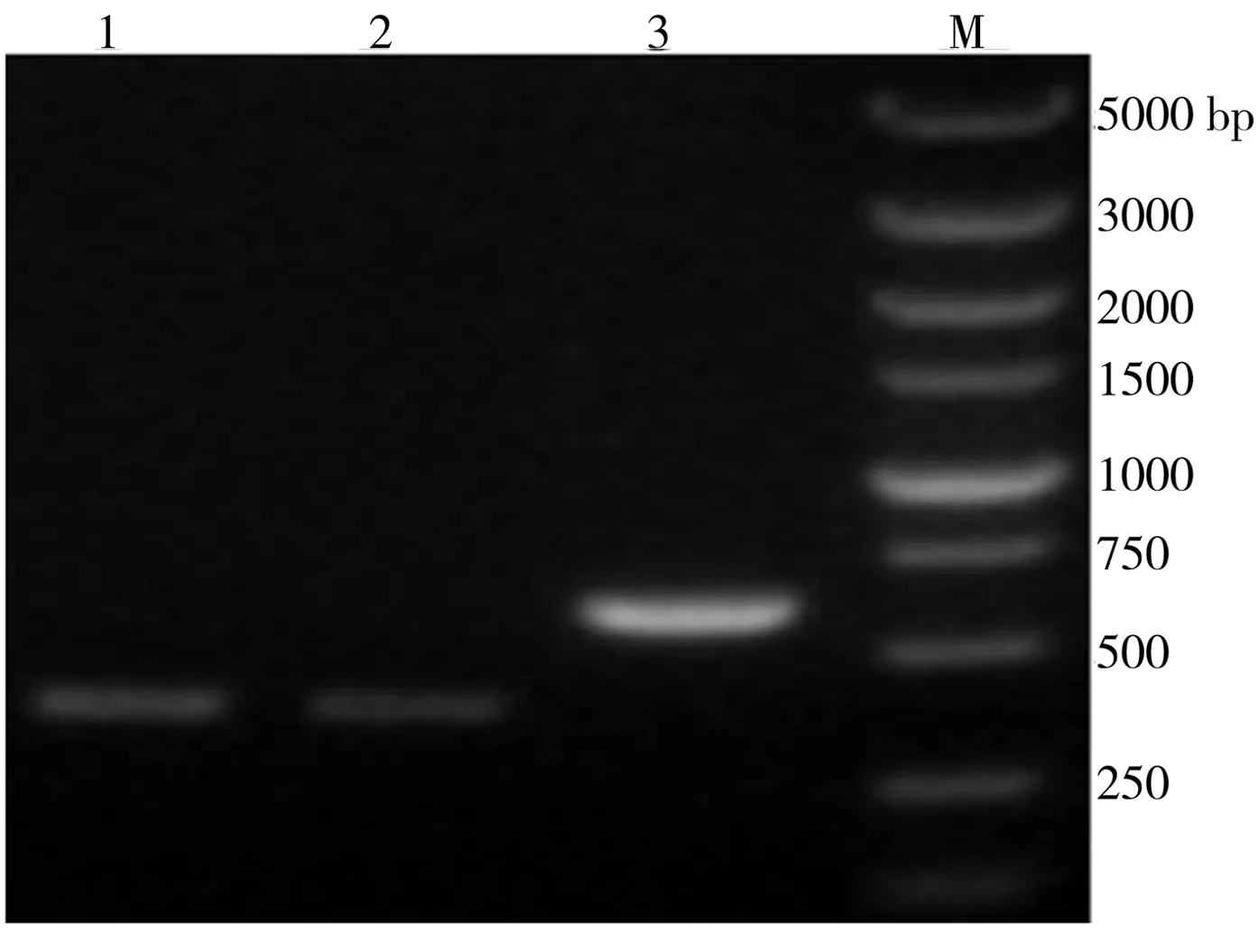
1,2:rsmZ突变株;3:野生菌株;M:DNA marker
(a)rsmZ敲除
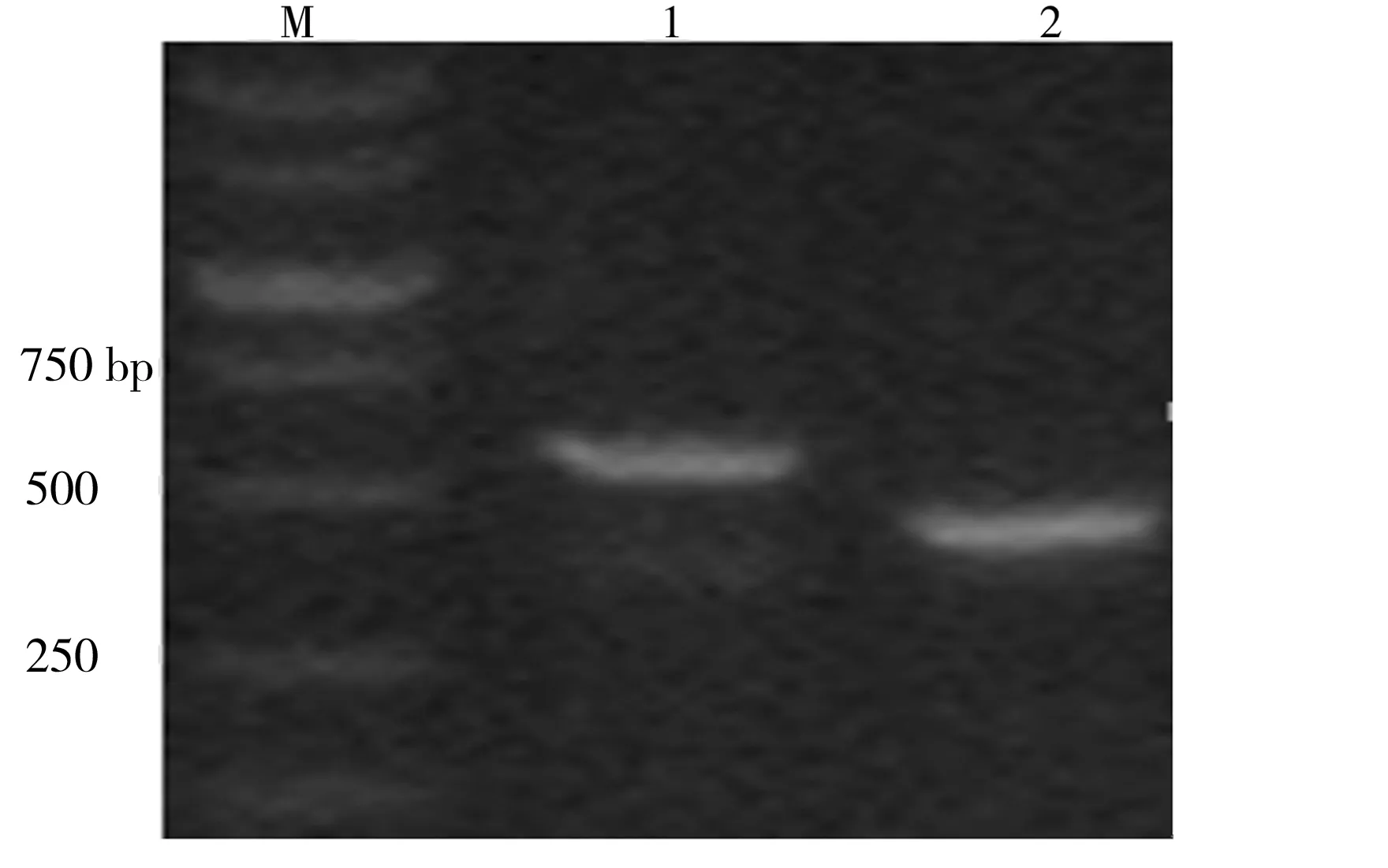
M:DNA marker;1:野生菌株;2:rsmY突变株
Fig.1ElectrophoretogramsofPCRproductsaftertheknock-out ofrsmZandrsmY
2.2rsmY及rsmZ敲除对细菌生长的影响
图2为野生菌株和突变菌株的生长曲线。从图2可以看出,rsmY、rsmZ单突变菌株的生长曲线和野生菌株的基本一致,而rsmY/rsmZ双突变后,细菌的生长曲线与野生菌株的相差较大。双突变菌株的生长速率变慢,稳定期后的OD600值也明显下降,表明rsmY及rsmZ双突变会影响细菌的生长,而rsmY和rsmZ相互之间可能存在互补的作用,因此,单突变任一基因对细菌的生长并未造成影响。
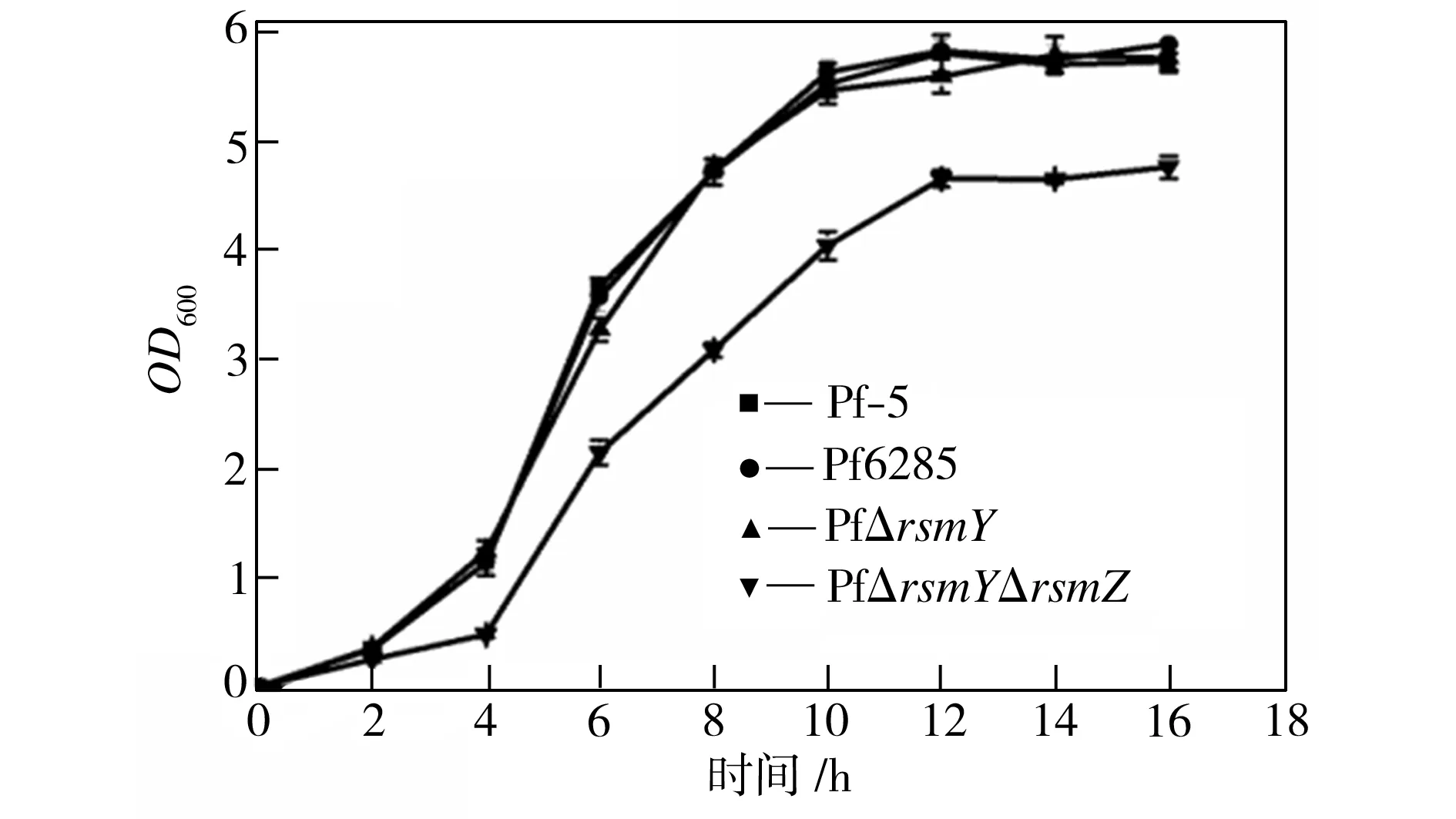
图2 野生菌株和突变菌株的生长曲线
2.3rsmY及rsmZ敲除对Pf-5lipA表达的影响
为研究rsmY、rsmZ对lipA表达的影响,分别测定了rsmY和rsmZ单敲除以及双敲除后全细胞脂肪酶的相对活性,结果见图3。
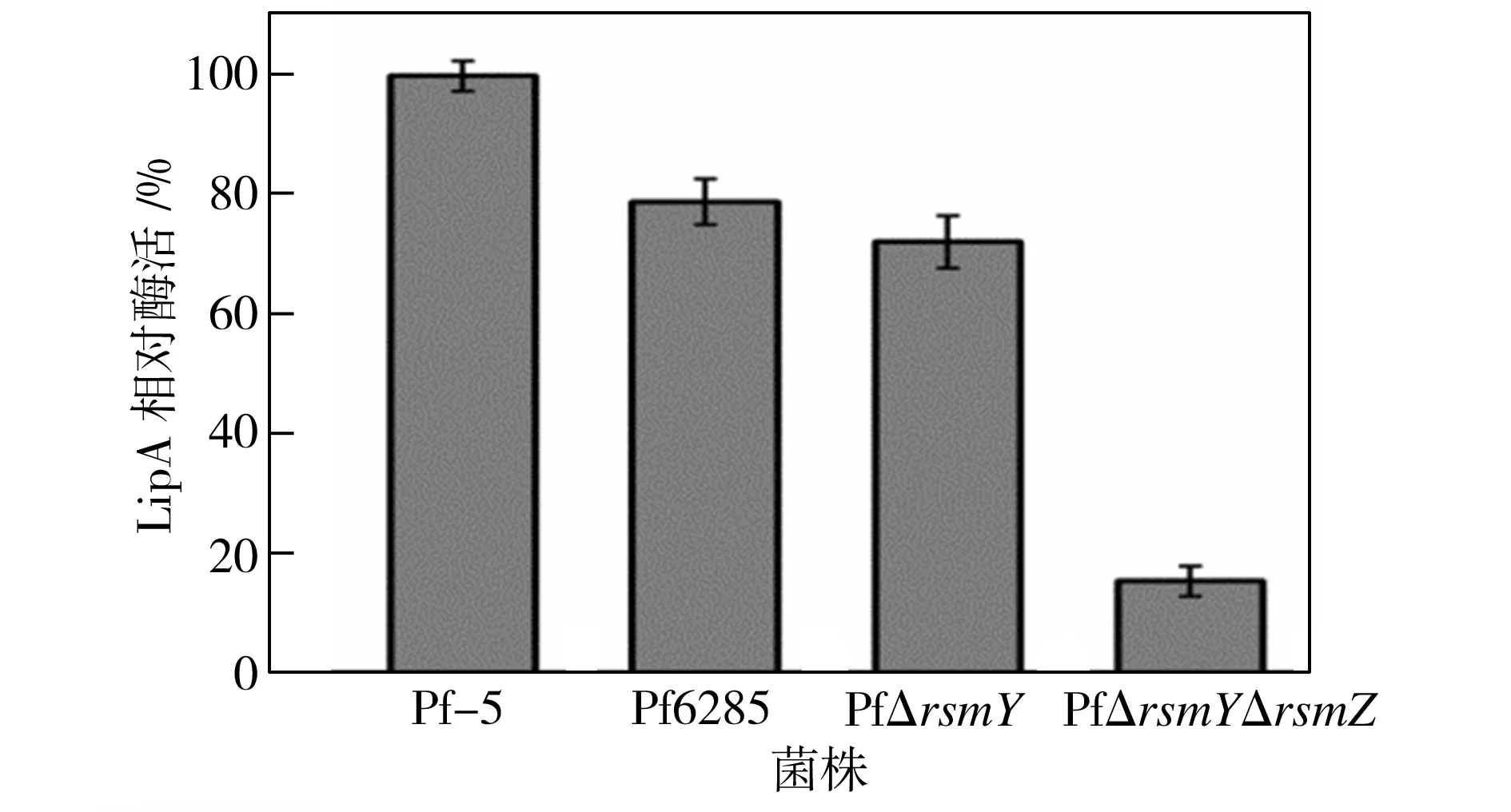
图3 野生菌株和突变株中全细胞脂肪酶的相对酶活
Fig.3 Relative enzyme activity of whole cell lipase in the wild and mutant strains
从图3中可以看出,与野生菌株相比,不管是rsmY还是rsmZ单敲除,lipA的全细胞脂肪酶活性均有所下降;将两个基因同时敲除后,lipA的全细胞脂肪酶活性急剧下降,其相对酶活只有野生菌株的17%左右。这是因为将rsmY和rsmZ敲除后,对细菌脂肪酶lipA表达的调控效应叠加,导致lipA的活性显著降低。这个结果也说明rsmY和rsmZ能够调控lipA的表达。
为了分析rsmY以及rsmZ是在转录水平还是在翻译水平影响lipA的表达,分别构建了染色体整合型lipA-lacZ和lipA′-′lacZ的报告菌株,并分别测定了稳定期的野生型和突变株的β-半乳糖苷酶的酶活,结果见图4和图5。由图可见,不管是在转录水平还是在翻译水平,rsmY及rsmZ的敲除都会影响lipA的表达,但是在翻译水平的影响更加明显。
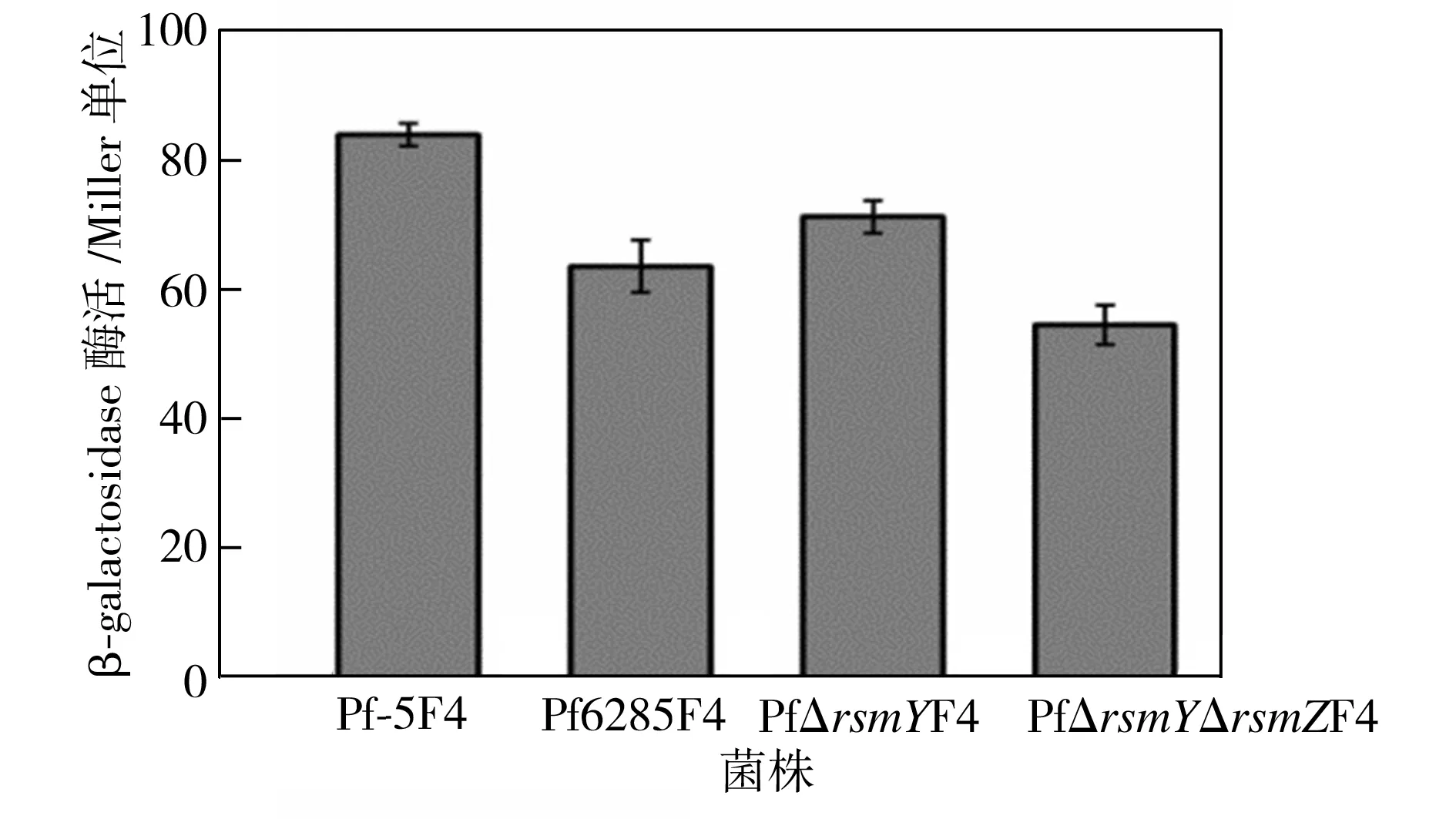
Pf-5F4:野生菌株融合lipA-lacZ; Pf6285F4:rsmZ突变株融合lipA-lacZ; PfΔrsmYF4:rsmY突变株融合lipA-lacZ; PfΔrsmYΔrsmZF4:rsmY/rsmZ双突变株融合lipA-lacZ
图4 染色体整合型lipA-lacZ转录融合报告菌株的β-半乳糖苷酶酶活
Fig.4β-galactosidaseactivityofchromosome-bornelipA-lacZtranscriptionalfusionreportstrains
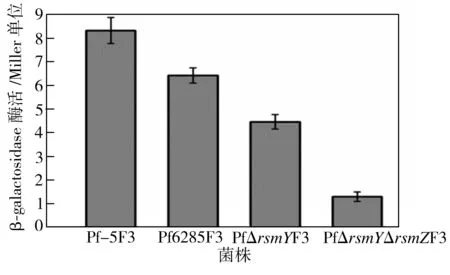
Pf-5F3:野生菌株融合lipA′-′lacZ;Pf6285F3:rsmZ突变株融合lipA′-′lacZ;PfΔrsmYF3:rsmY突变株融合lipA′-′lacZ; PfΔrsmYΔrsmZF3:rsmY/rsmZ双突变株融合lipA′-′lacZ
图5 染色体整合型lipA′-′lacZ翻译融合报告菌株的β-半乳糖苷酶酶活
Fig.5 β-galactosidase activity of chromosome-bornelipA′-′lacZtranslationalfusionreportstrains
为了进一步验证lacZ融合的结果,利用qRT-PCR来进行分析,结果见图6。由图6可以看出,rsmY及rsmZ的敲除会影响lipA的表达,但转录水平上的mRNA含量并没有急剧降低,这也说明了rsmY及rsmZ主要是在翻译水平影响lipA的表达。
2.4 rsmY及rsmZ敲除对rsmA、rsmE的影响
本课题组前期研究[11]表明,在Pf-5中,是rsmE而不是rsmA与lipA的启动子序列直接结合来调控lipA的表达;文献[12]亦报道,在荧光假单胞菌CHA0中,rsmX/Y/Z能够与RsmA及RsmE结合,阻断RsmA、RsmE与调控基因的mRNA结合,从而能够调控基因表达。因此下面进一步分析在Pf-5中rsmY、rsmZ主要是通过rsmA还是rsmE来调控lipA的表达。
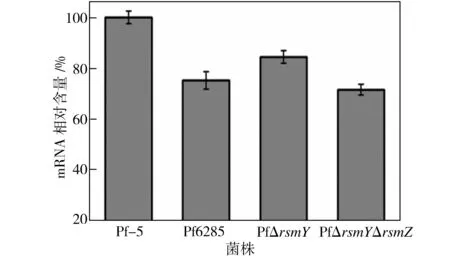
图6lipA在野生菌株和突变株中表达的qRT-PCR分析
Fig.6 qRT-PCR analysis oflipAexpression in the wild and mutant strains
染色体整合型rsmA′-′lacZ和rsmE′-′lacZ在野生菌株和突变菌株中的β-半乳糖苷酶酶活如图7所示。由图7可见,rsmY、rsmZ单敲除对rsmA和rsmE的表达影响均比较小,而双敲除后,rsmA、rsmE的表达均有所提高,并且在对数生长后期以及稳定期,双敲除突变株的rsmE表达急剧增加。
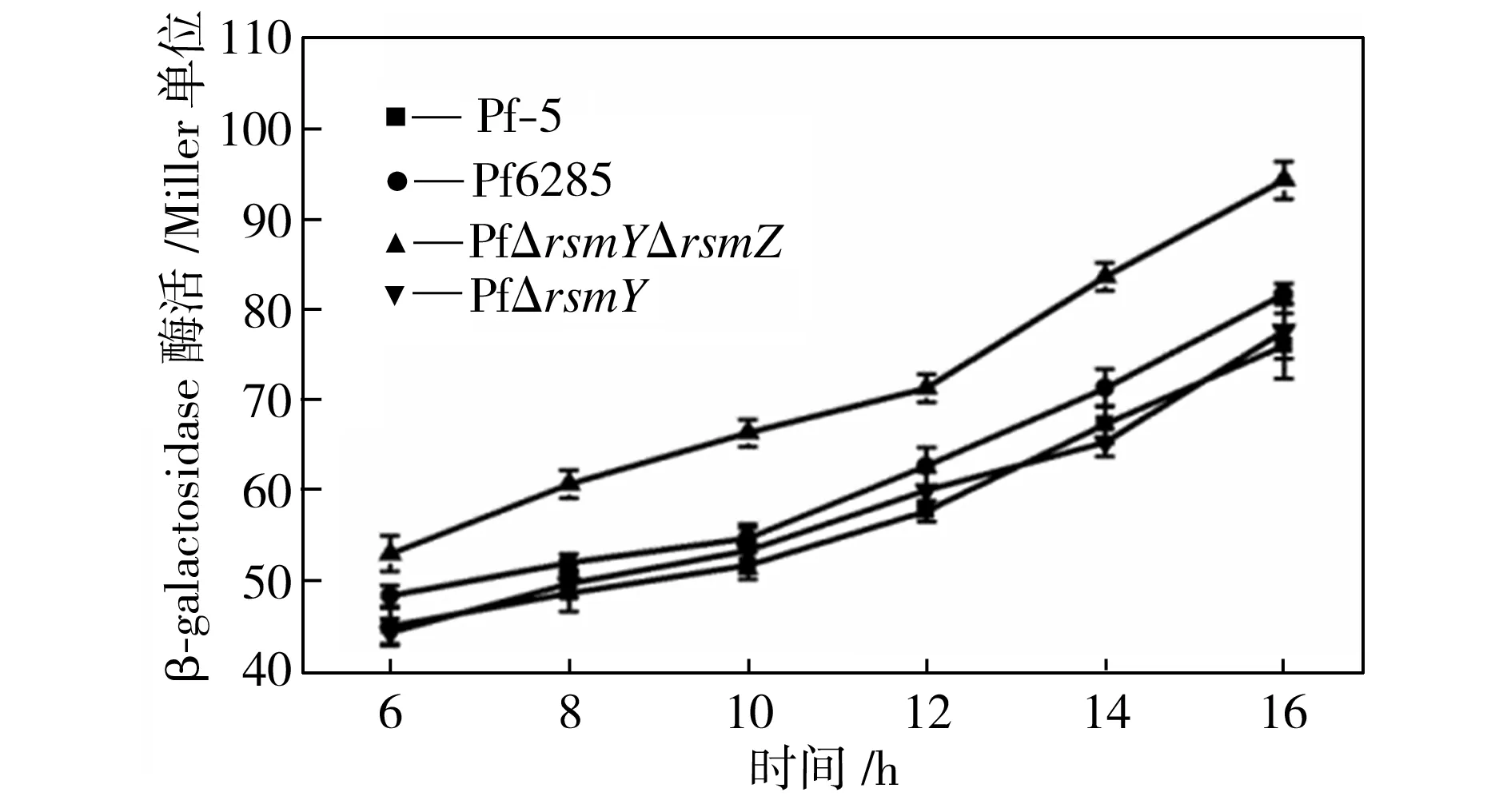
(a)rsmA′-′lacZ
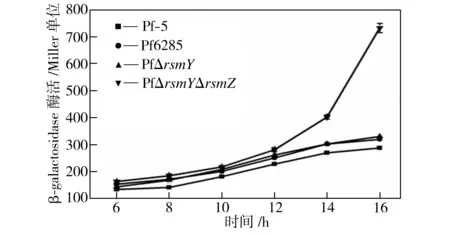
(b)rsmE′-′lacZ
图7rsmA′-′lacZ及rsmE′-′lacZ在野生菌株和突变菌株中的β-半乳糖苷酶酶活
Fig.7 β-galactosidase activity ofrsmA′-′lacZandrsmE′-′lacZinthewildandmutantstrains
3 讨论
Rsm调控系统在细菌基因的表达调控中发挥着重要的作用。在P.protegensCHA0中,RsmE和RsmA能与rsmX/Y/Z的GGA区域结合,从而阻断RsmA、RsmE与调控基因的mRNA结合[7]。因此,在Rsm调控系统中,rsmX/Y/Z是与RsmA以及RsmE协同发挥调控作用的。而在P.protegensPf-5中也包含RsmA和RsmE,而且RsmE与lipA的启动子区域结合来调控lipA的表达[11],因此,有必要深入研究rsmY、rsmZ影响lipA表达的分子机理。
本研究通过将rsmY、rsmZ基因敲除,并分析lipA的全细胞脂肪酶活性以及染色体整合型lipA-lacZ、lipA′-′lacZ、rsmA′-′lacZ、rsmE′-′lacZ的β-半乳糖苷酶酶活,发现rsmY、rsmZ主要在转录后水平影响lipA的表达。另外,rsmY、rsmZ均抑制rsmA、rsmE的表达。rsmY、rsmZ敲除后,rsmA、rsmE的表达水平均提高,而rsmE的表达则是显著提高,这些结果表明rsmY和rsmZ对rsmE的影响要明显高于其对rsmA的影响。结合前期研究结果可以推测,rsmY、rsmZ主要是通过影响rsmE的表达来调控lipA的表达。而在Pf-5的近缘菌株P.protegensCHA0中,rsmA、rsmE均可以与rsmY、rsmZ结合,但rsmA的表达强度要远高于rsmE的表达强度,而且rsmA的表达从对数生长期开始一直持续至稳定期,而rsmE的表达则是从对数生长后期开始直至稳定期。
另外,在Pf-5中,rsmE的表达强度和表达差异性高于rsmA,这也可能是rsmY/Z选择通过rsmYZ/RsmE/LipA途径激活lipA翻译而不选择通过rsmYZ/RsmA/RsmE/LipA途径来抑制lipA翻译的原因。结合已有的研究结果可以进一步证实rsmY、rsmZ与rsmA、rsmE的相互关系,更加明确Rsm调控基因表达的分子机理。
4 结语
本文通过rsmY、rsmZ基因敲除构建防御假单胞菌Pf-5的突变株,分析野生菌株和突变株中lipA的全细胞脂肪酶活性及染色体整合型lipA-lacZ、lipA′-′lacZ、rsmA′-′lacZ、rsmE′-′lacZ的β-半乳糖苷酶酶活,并采用qRT-PCR等分析手段,首次证明了在Pf-5中,rsmY、rsmZ在翻译水平抑制rsmA和rsmE的表达,而且主要是通过抑制rsmE的翻译来激活lipA翻译,从而调控lipA的表达。这些结果可为进一步研究脂肪酶lipA的表达调控机制、构建脂肪酶高效表达的基因工程菌提供新的思路和方法。
[1] Argaman L, Hershberg R, Vogel J, et al. Novel small RNA-encoding genes in the intergenic regions ofEscherichiacoli[J]. Current Biology, 2001, 11(12): 941-950.
[2] Reimmann C, Valverde C, Kay E,et al. Posttranscriptional repression of GacS/GacA-controlled genes by the RNA-binding protein RsmE acting together with RsmA in the biocontrol strainPseudomonasfluorescensCHA0[J]. Journal of Bacteriology, 2005, 187(1): 276-285.
[3] Heeb S, Blumer C, Haas D. Regulatory RNA as mediator in GacA/RsmA-dependent global control of exoproduct formation inPseudomonasfluorescensCHA0[J]. Journal of Bacteriology, 2002, 184(4): 1046-1056.
[4] Valverde C, Heeb S, Keel C,et al. RsmY, a small regulatory RNA, is required in concert with RsmZ for GacA-dependent expression of biocontrol traits inPseudomonasfluorescensCHA0[J]. Molecular Microbiology, 2003, 50(4): 1361-1379.
[5] Papenfort K, Vogel J. Small RNA functions in carbon metabolism and virulence of enteric pathogens[J]. Frontiers in Cellular and Infection Microbiology, 2014, 4:91.
[6] Kay E,Dubuis C, Haas D. Three small RNAs jointly ensure secondary metabolism and biocontrol inPseudomonasfluorescensCHA0[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(47): 17136-17141.
[7] Gottesman S, Storz G. Bacterial small RNA regulators: versatile roles and rapidly evolving variations[J]. Cold Spring Harbor Perspectives in Biology, 2011, 3(12): a003798.
[8] Chen R H, Weng Y D, Zhu F,et al. Polynucleotide phosphorylase regulates multiple virulence factors and the stabilities of small RNAs RsmY/Z inPseudomonasaeruginosa[J]. Frontiers in Microbiology, 2016, 7:247.
[9] Zha D M, Xu L, Zhang H J,et al. Molecular identification of lipase LipA fromPseudomonasprotegensPf-5 and characterization of two whole-cell biocatalysts Pf-5 and Top10lipA[J]. Journal of Microbiology and Biotechnology, 2014, 24(5): 619-628.
[10]Miller J H. A short course in bacterial genetics: a laboratory manual and handbook forEscherichiacoliand related bacteria[M]. New York: Cold Spring Harbor Laboratory Press, 1992.
[11]Zha D M, Xu L, Zhang H J,et al. The two-component GacS-GacA system activateslipAtranslation by RsmE but not RsmA inPseudomonasprotegensPf-5[J]. Applied and Environmental Microbiology, 2014, 80(21): 6627-6637.
[12]Sonnleitner E, Haas D. Small RNAs as regulators of primary and secondary metabolism inPseudomonasspecies[J]. Applied Microbiology and Biotechnology, 2011, 91(1): 63-79.
[责任编辑 尚 晶]
Effect of Rsm regulatory system on the expression of lipase inPseudomonasprotegensPf-5
LiMenggang,YanYunjun
(College of Life Science and Technology, Huazhong University of Science and Technology, Wuhan 430074, China)
Rsm system is a very important expression regulation factor for bacterial genes. This study focused onPseudomonasprotegensPf-5. Through the knockout ofrsmYandrsmZgenes, thersmYmutant,rsmZmutant andrsmY/rsmZdouble mutant were constructed. The relative whole-cell lipase activities oflipAand β-galactosidase activities of chromosome-bornelipA-lacZ,lipA′-′lacZ,rsmA′-′lacZandrsmE′-′lacZfusions in the wild and mutant strains were analyzed. The results show thatrsmYandrsmZmainly affect the expression oflipAat the post-transcriptional level. Moreover, bothrsmYandrsmZinhibit the expression ofrsmAandrsmE, and the knockout ofrsmYandrsmZcan slightly improve the expression level ofrsmAand significantly enhance that ofrsmE. In conclusion,rsmYandrsmZhave greater influence onrsmEthan onrsmA, and they regulate the expression oflipAin Pf-5 mainly through affecting the expression ofrsmE.
gene expression regulation; lipase;Pseudomonas; Rsm; gene knockout; small RNA
2017-04-20
国家自然科学基金资助项目(31070078);湖北省自然科学基金重点项目(2015CFA085).
李猛刚(1982-),男,华中科技大学博士生. E-mail:menggang2004@163.com
闫云君(1969-),男,华中科技大学教授,博士生导师. E-mail:yyyunjun@163.com
10.3969/j.issn.1674-3644.2017.04.007
Q784
A
1674-3644(2017)04-0274-05
