云杉矮槲寄生遗传多样性及其群体遗传结构分析
2016-05-06朱宁波李学武才让旦周吴有林田呈明
白 云,陈 磊,朱宁波,李学武,才让旦周,吴有林,田呈明*
(1 北京林业大学 林学院,北京 100083;2 青海省森林病虫害防治检疫站,西宁 810000;3 青海省海东地区北山林场,青海海东 810500)
云杉矮槲寄生遗传多样性及其群体遗传结构分析
白云1,陈磊1,朱宁波1,李学武1,才让旦周2,吴有林3,田呈明1*
(1 北京林业大学 林学院,北京 100083;2 青海省森林病虫害防治检疫站,西宁 810000;3 青海省海东地区北山林场,青海海东 810500)
摘要:该研究采用CTAB法提取云杉矮槲寄生(Arceuthobium sichuanense)的基因组DNA,利用ITS1片段的序列信息对中国9个不同地理群体的62份云杉矮槲寄生样本的遗传多样性及群体遗传结构进行分析。结果表明:(1)62条ITS1序列共定义16个单倍型(H1~H16),表现出较低的遗传多样性水平(h=0.678 5,π=0.005 9),而群体间的遗传多样性水平则表现出较大差异(h=0~1.000 0,π=0~0.009 4);AMOVA分析显示云杉矮槲寄生群体内的遗传变异占到51.37%,群体间为48.63%。(2)Network单倍型网络分析表明,单倍型H1和H12较为古老,且所有群体对2种单倍型无共享现象;单倍型H1是广布单倍型,存在于青海和甘肃的6个群体中,单倍型H12仅在四川的2个群体中有分布。(3)基于最大似然法(ML)构建的群体聚类和中介邻接法构建的单倍型网络图均显示,四川的3个群体为独立类群,区别于青海、甘肃群体,且甘肃和青海的群体之间没有明显分化。该研究首次报道了云杉矮槲寄生遗传多样性和遗传结构,为进一步研究其进化及后续的病害防控提供了一定的参考。
关键词:云杉矮槲寄生;遗传多样性;群体遗传结构;单倍型
云杉矮槲寄生(Arceuthobiumsichuanense)是槲寄生科(Viscaceae)油杉寄生属(Arceuthobium)的多年生半寄生性种子植物,分布于中国青海、甘肃、四川和西藏等高海拔地区[1]。云杉矮槲寄生主要寄生于云杉属植物[1],目前已报道的寄主有青海云杉(Piceacrassifolia)、紫果云杉(P.purpurea)、川西云杉(P.likiangensisvar.balfouriana)、西藏云杉(P.spinulosa)和青杄(P.wilsonii)[1-2]。云杉矮槲寄生侵染成功后,造成寄主枝条组织增生膨大,产生大量下垂的扫帚状丛枝,进而削减寄主生长量和材积产量,影响寄主的光合与蒸腾作用以及对水分和营养的吸收利用,同时削弱寄主抵御环境和其它病虫害的能力,最终随着感病级别的增加而死亡[3-5]。除此之外,云杉矮槲寄生还具有重要的生态学功能[3]。云杉矮槲寄生雌雄异株[1],主要依赖种子弹射进行近距离传播[6],在林间呈聚集发生[7]。尽管有报道称鸟类和一些哺乳动物可以携带矮槲寄生的种子进行远距离的传播[8],但在云杉矮槲寄生上尚未见报道。近年来,青海“三江源”地区的云杉天然林和人工林受到云杉矮槲寄生的严重危害,部分地区发病率高达90%以上,严重威胁了当地的森林健康和生态安全[9-10]。目前针对云杉矮槲寄生的研究主要集中于生物学特点、发病规律以及化学防治方面[2,4-7,11-12],缺乏对其群体遗传背景的分析,制约了对病害流行成灾原因的了解,从而影响了相关控制策略的制定。
了解寄生植物的遗传多样性及群体动态有利于理解寄生植物与寄主之间互作的生态、进化关系以及病原物的变异情况[13-14]。通常情况下,物种的生活史特点、分布以及与基因扩散有关的生态学过程均可以影响其遗传结构[15]。对于种子植物来说,其交配系统对群体的遗传分化也起着重要的作用[16-19]。而在寄生植物与寄主互作的系统中,寄生植物的生活史与其寄主植物、非寄主植物、花粉散播以及种子传播机制均有着密切的关系[3,20-22]。因此,矮槲寄生与其它生物之间的互作可能影响着矮槲寄生的遗传结构[13]。同时,由于寄生植物具有特殊的生活史策略,通常难以形成经典的遗传多样性和遗传结构模式[13]。鉴于需要适应不同的寄主基因型,克服寄主的抗性选择压力,寄生植物会产生较高的遗传多样性水平[23-25],甚至高于其寄主植物[26-27],那些依赖特定寄主或者缺乏长距离传播机制的寄生植物则会由于较低的基因频率而导致地理适应性或寄主依赖性小种的形成,表现出较强的遗传结构特征和地理距离隔绝[26,28]。事实上,不同的寄生植物会表现出不同的遗传分化水平[23,26-27,29-32]。
在物种遗传背景的研究过程中,不同的分子标记反映不同的影响因素。叶绿体基因标记反映了与地理相关的影响因素,而核基因标记则反映了与植物生育系统相关的影响因素[33]。核糖体基因(nrDNA)的内转录间隔区(internal transcribed spacer,ITS)以高度串联重复的方式存在于核基因组中,既具有核苷酸序列的高度变异性又有长度上的保守性[34],相比于编码基因序列,其变异速度相对较快;与中高度重复序列和非编码序列相比,又具有较高的保守性[35],已被广泛用于被子植物的系统发育、分子鉴定、遗传多样性、种内变异以及DNA条形码的相关研究[36-41]。本研究基于云杉矮槲寄生的ITS1序列,分析其群体遗传多样性及遗传结构,以揭示中国云杉矮槲寄生的遗传分化与其寄主种类、地理分布的关系,为进一步了解云杉矮槲寄生的传播以及制定病害控制策略提供参考。
1材料和方法
1.1样本采集
本研究于2013~2014年分别在青海、甘肃、四川等地采集9个地理群体的62份云杉矮槲寄生样本(表1)。样本采集地的主要生态因子数据来自中国气象数据网1981~2010年30年间的气象数据资料(http://data.cma.gov.cn/)。根据云杉矮槲寄生的种子弹射特点及其在林分内的空间分布格局[6-7],在受侵染林分的空间范围,每个群体采集2~10株云杉矮槲寄生,相邻云杉间隔10 m。采集时以被侵染的云杉小枝为单位,将云杉矮槲寄生植株装入离心管中带回实验室,经液氮速冻后保存在-80 ℃备用。
1.2基因组DNA提取和ITS序列PCR扩增及测序
采用CTAB法提取云杉矮槲寄生的总基因组DNA[42],经琼脂糖凝胶电泳检测后,保存于-20 ℃冰箱备用。用引物18S1830(5′-AACAAGGTTTCCGTAGGTGA-3′)和26S40(5′-TCCTCCGCTTATTGATATGC-3′)对云杉矮槲寄生的ITS区域进行扩增[43-44]。所用引物由上海英骏生物技术有限公司合成。25 μL PCR反应体系包含2.5 μL 10×PCR buffer、2.5 μL dNTPs(2.5 mmol/L),上下游引物(10 μmol/L)各0.5 μL,0.75 UTaqDNA聚合酶,DNA模板10~30 ng,加dd H2O至总体积25 μL。扩增反应程序为94 ℃预变性5 min,94 ℃变性30 s,52 ℃退火30 s,72 ℃延伸1 min,进行35个循环,最后72 ℃延伸10 min。按DNA凝胶回收试剂盒(天根生化科技北京有限公司)说明书对PCR产物进行回收纯化后,与pMD19-T载体连接,用大肠杆菌感受态细胞进行转化,挑取阳性克隆摇菌后进行PCR检测,转化成功的菌液由北京三博远志生物技术有限责任公司进行测序。
1.3数据处理与分析
利用DNAMAN软件对正反序列进行拼接,并用Chromas软件对序列峰图进行人工校对。多序列比对用MEGA 5中的Clustal W进行操作。GC含量的测定用DNAStar软件中的Editseq程序完成。利用DnaSP 5.1软件[45]对各个群体进行遗传多样性评价。
利用MEGA5软件构建最大似然法(maximum likelihood,ML)系统发育树,并通过1 000次重复的自展程序进行标准检测。用Arlequin 3.0软件[46]中的分子变异分析程序(AMOVA)计算群体遗传差异指数FST值,用于测量群体间的遗传分化程度[47]。利用软件Permut(http://www.pierrton.intra.fr/genetics/labo/software/permut)计算基于单倍型分化频率和程度的群体遗传分化系数NST和GST[48]。利用软件Network 4.2.0.1[49]的中介邻接法(Median-joining network)构建所有群体的单倍型网络图,以此进行ITS1单倍型分析。
2结果与分析
2.1ITS1序列多态性分析
云杉矮槲寄生的ITS1序列分析结果表明,ITS1序列长度为212 bp,没有检测到插入/缺失片段,包含了18个变异位点,其中9个为转换位点,9个为颠换位点,分别占总长度的4.25%和4.25%。不同群体的ITS1序列的碱基含量存在差异,GC含量为36.79%~38.68%(表2)。云杉矮槲寄生9个群体的62个样本共产生16个单倍型(表2)。其中单倍型H1出现频率最高,占总样本量的54.84%,仅在青海、甘肃6个群体的34个样本中有出现;单倍型H12的出现频率次之,占总样本量的14.52%,仅出现在四川的2个群体的9个样本中(表3)。
2.2云杉矮槲寄生群体遗传多样性分析
对所有群体的ITS1序列单倍型多样性(h)和核苷酸多样性(π)分析结果表明,云杉矮槲寄生表现出较低的遗传多样性水平(h=0.678 5,π=0.005 9),而单个群体间遗传多样性水平表现出较大的差异(h=0~1.000 0,π=0~0.009 4)。其中四川壤塘(SRT)群体具有最高的遗传多样性水平(h=1.000 0,π=0.009 4),四川炉霍(SLH)和甘肃夏河(GXH)的群体也具有较高的遗传多样性水平(h=0.800 0,π=0.006 0;h=0.800 0,π=0.004 7),而青海同仁(QTR)和甘肃合作(GHZ)的2个群体则表现出最低的遗传多样性水平(h=0,π=0;表3)。
各群体中的单倍型分布存在差异。其中,青海同仁(QTR)群体和甘肃合作(GHZ)群体只检测到一种类型的单倍型,其它7个群体均检测到2种或以上单倍型,四川炉霍(SLH)群体产生的单倍型最多,为5种。共享单倍型有4种(H1、H2、H11和H12),同时分布在多个群体中,其余12种单倍型仅在个别群体中出现。单倍型H1是广布单倍型,在青海、甘肃的6个群体中均有分布,单倍型H2、H11和H12仅分布在2个相邻的群体中。而在四川的3个群体中均未发现与青海、甘肃群体中相同的单倍型(表3,图1)。
2.3云杉矮槲寄生群体遗传结构分析
云杉矮槲寄生群体的分子变异(AMOVA)分析显示其群体内变异为51.37%,群体间变异为48.63%(表4)。群体间遗传差异指数FST值表现出较大的差异,分布在0~0.865 7之间,其中青海同仁(QTR)和四川壤塘(SRT)的2个群体之间表现出最高的遗传分化程度。另外,四川的3个群体(SRT、SDF和SLH)与青海、甘肃的6个群体(QTR、QHZ、QMY、GHZ、GXH和GLQ)之间均具有显著或极显著的遗传分化。同时,四川炉霍(SLH)、壤塘(SRT)、道孚(SDF)的群体之间也存在显著的遗传分化,而青海和甘肃的6个群体间的遗传分化均不显著(表5)。Permut软件分析云杉矮槲寄生群体的遗传分化系数结果为GST=0.421,NST=0.526,NST值显著高于GST值,表明在全部群体水平上,ITS1单倍型具有明显的分子谱系地理结构。

云杉矮槲寄生群体间的系统发育分析结果表明,四川的SRT、SDF、SLH群体聚在了一个小分支上,而青海(QTR、QHZ、QMY)、甘肃群体(GHZ、GXH、GLQ)没有明显分支(图2)。基于中介邻接法构建的单倍型网络图表现出与ML聚类类似的结果,以单倍型H1为节点形成了一个大的分支,以H11、H12为节点形成了2个小分支。H1位于整个单倍型网络图的最中心,而其它单倍型都是从单倍型H1经过一步或多步突变得到的,位列于整个单倍型网络图的外部节点上(图3)。

饼图代表各单倍型在该群体中的分布比例,
3讨论
本研究基于云杉矮槲寄生ITS1的序列信息,分析了云杉矮槲寄生的遗传多样性及群体遗传结构,表明云杉矮槲寄生整体上表现出较低的遗传多样性水平(h=0.6785,π=0.0059),这可能与其分

标尺代表进化距离;分支上数值表示自展支持率。
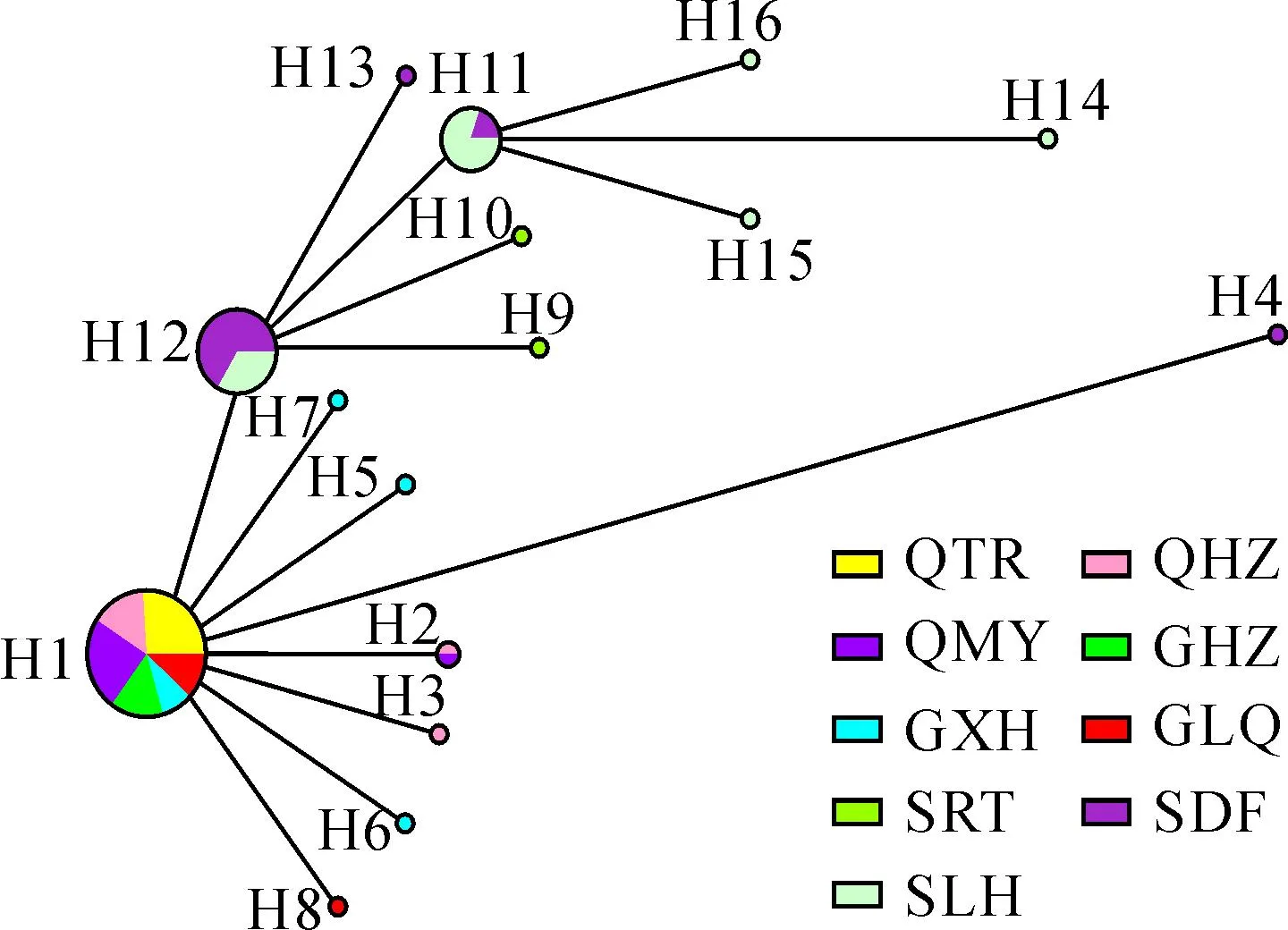
圆圈大小代表各单倍型频率,不同颜色代表不同群体

变异来源Sourceofvariation自由度Df平方和Sumofsquares变异组成Variancecomponent变异所占比例Percentageofvariation/%P群体间Amongpopulations820.100.3248.63<0.001群体内Withinpopulation5317.960.3451.37<0.001总变异Total6138.070.66
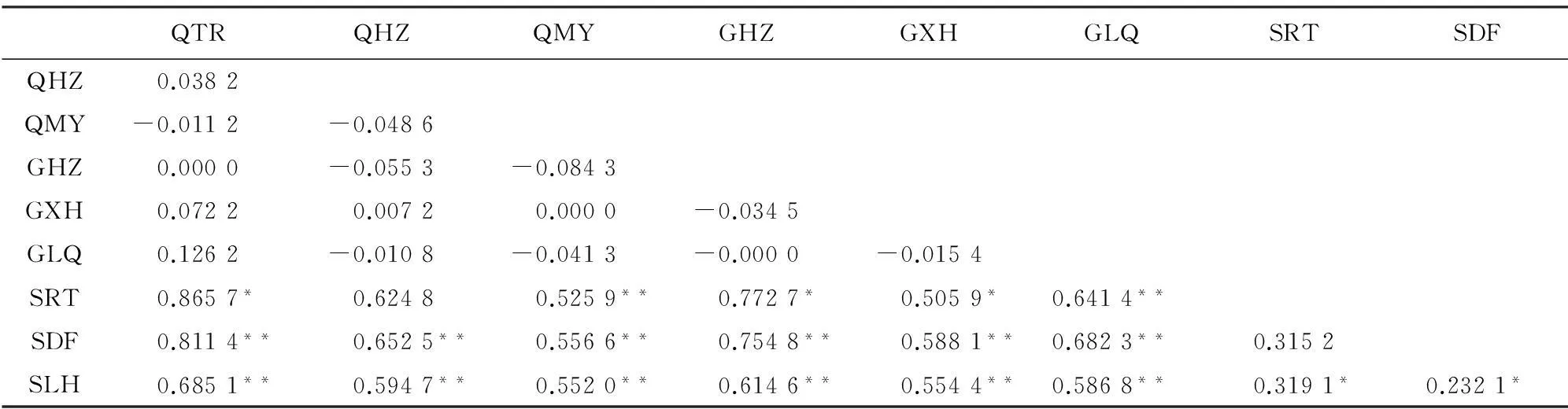
表5 云杉矮槲寄生各群体间的FST值
注:**差异达极显著水平(P<0.01);*差异达显著水平(P<0.05)。
Note:** indicate most significant difference(P<0.01);* indicates significant difference(P<0.05).
布范围有关。物种的遗传多样性受其生活史、种子散播和传粉机制、交配系统、生殖模式、突变率以及突变机制等生物学特性的影响[15]。不同因素对物种遗传变异大小的影响程度不同[50]。在物种水平上依次为分类地位>分布范围>生活型>繁育系统>种子散布机制,而在群体水平上则变为繁育系统>分布范围>生活型>分类地位>种子散布机制[50]。就分布范围而言,物种的分布范围越广,其遗传多样性越高,反之则越低[50]。云杉矮槲寄生分布于中国青海、甘肃、四川和西藏等高海拔地区[1],分布范围较为狭窄,个体和群体数量较少,适应幅度小,基因交流不频繁,遗传变异的能力也较差,因而产生新的遗传变异的机会也少,故有限的地理分布可能是其多样性偏低的主要原因。而对于寄生植物来说,寄主的多样性对寄生植物的遗传多样性水平也有重要的影响[13]。云杉矮槲寄生仅寄生于云杉属植物[1],寄主范围相对较窄,因此,对寄主的专化性也可能是导致云杉矮槲寄生遗传多样性水平较低的原因之一。
从本研究结果可以看出,云杉矮槲寄生群体具有明显的遗传分化,表现出了显著的谱系地理结构,其群体遗传分化系数(GST=0.421)均高于A.americanum的群体遗传分化系数(GST=0.286)[26]和A.cyanocarpum、A.apachecum、A.blumeri的群体遗传分化系数(GST=0.235)[51]。一方面,青海省南部的巴颜喀拉山是西北朝东南走向的山脉,其东面是若尔盖草原,西面同可可西里的东缘相接,将青海与四川群体完全隔开[52],岷山山脉位于甘肃南部,川西高原北部,阻断了四川与甘肃群体[53],从而阻碍了花粉的传播,限制了群体间的基因交流,进而造成了云杉矮槲寄生群体之间分化程度的加大。然而,青海与甘肃采样点间并无大的山系将其阻隔,且云杉苗木由于批量买卖可能导致基因交流较为频繁,故6个群体间未形成明显分化。另一方面,云杉矮槲寄生为雌雄异株植物,自然情况下可能通过风媒和昆虫进行花粉传播[3]。云杉矮槲寄生依赖种子弹射进行近距离传播,其种子弹射的最远距离为15 m[6],尽管矮槲寄生的种子可以通过鸟类和哺乳动物进行远距离传播[8],但尚未发现云杉矮槲寄生种子远距离传播的途径,因而造成其群体之间缺少基因交流,而导致群体间明显的遗传分化。从本研究的结果来看,群体间的地理隔绝是导致云杉矮槲寄生群体分化的主要原因。而在美洲矮槲寄生遗传结构的研究中也发现地理隔绝和不同寄主的共同作用促进了美洲矮槲寄生分化为3个小种[26]。然而,不同寄主来源的云杉矮槲寄生群体是否存在分化,还需要进一步研究探讨。
依据矮槲寄生群体的遗传多样性水平和单倍型分布可以推断其进化起源及迁移历史[26]。从单倍型网络图与ML聚类结果可以看出,单倍型H1和H12存在于网络图的内部,均为祖先单倍型。而青海和甘肃的6个群体中均包含了古老的H1单倍型,推测可能青海和甘肃的云杉矮槲寄生群体本来属于同一个古老的群体,随后在各自的区域内,由于地理环境和寄主的不同发生了独立进化,分化出新的单倍型。四川的3个群体中单倍型的演化网络也有类似的结果,由祖先单倍型H12逐渐衍生出其它单倍型。
综上所述,本研究基于ITS1序列,分析了云杉矮槲寄生的群体遗传多样性及遗传结构,揭示了云杉矮槲寄生群体较低的遗传多样性,且具有明显的地理谱系结构。本研究结果将对云杉矮槲寄生的进化、传播和扩散的深入研究以及病害控制提供参考。
参考文献:
[1]吴征镒.中国植物志[M].北京:科学出版社,2003.
[2]高发明,陈磊,等.云杉矮槲寄生的侵染对青杄光合与蒸腾作用的影响[J].植物病理学报,2015,45(1):14-21.
GAO F M,CHEN L,etal.Effect ofArceuthobiumsichuanenseinfection on photosynthesis and transpiration ofPiceawilsonii[J].ActaPhytopathologicaSinica,2015,45(1):14-21.
[3]HAWKSWORTH F G,WIENS D.Dwarf mistletoes:biology,pathology,and systematics[M].U.S.D.A.Forest Service Agriculture Handbook 709.Washington,DC.1996.
[4]XIA B,TIAN C M,LUO Y Q,etal.The effects ofArceuthobiumsichuanenseinfection on needles and current-year shoots of mature and young Qinghai spruce (Piceacrassifolia) trees[J].ForestPathology,2012,42(4):330-337.
[5]周在豹.三江源云杉矮槲寄生生物学特性及防治研究[D].北京:北京林业大学,2007.
[6]孙秀玲,许志春,才让丹周,等.云杉矮槲寄生种子雨的时空分布格局[J].西北林学院学报,2014,29(4):65-68.
SUN X L,XU Z C,CAIRANG D Z,etal.Spatiotemporal patterns of the seed rain ofArceuthobiumsichuanense[J].JournalofNothwestForestryUniversity,2014,29(4):65-68.
[7]胡阳,田呈明,才让旦周,等.青海仙米林区云杉矮槲寄生空间分布格局及其与环境的关系[J].北京林业大学学报,2014,36(1):102-108.
HU Y,TIAN C M,CAIRANG D Z,etal.Spatial distribution pattern ofArceuthobiumsichuanenseand its correlation with environment in Xianmi forest region of Qinghai,Northwestern China[J].JournalofBeijingForestryUniversity,2014,36(1):102-108.
[8]NICHOLLS T H,HAWKSWORTH F G,MERRILL L M.Animal vectors of dwarf mistletoe,with special reference toArceuthobiumamericanumon lodgepole pine[M].Gen.Tech.Rep.RM-111.Fort Collins,CO;U.S.Department of Agriculture,Forest Service,Rocky Mountain Forest and Range Experiment Station,1987:102-110.
[9]马建海,淮稳霞,赵丰钰.云杉矮槲寄生——危害青海云杉的寄生植物[J].中国森林病虫,2007,26(1):19-21.
MA J H,HUAI W X,ZHAO F Y.Arceuthobiumsichuanense,a parasitic plant attacking spruce in Qinghai Province[J].ForestPestandDisease,2007,26(1):19-21.
[10]李涛,马明呈,谭建萍.仙米林区云杉矮槲寄生为害状况及防治[J].青海大学学报,2010,28(3):69-72.
LI T,MA M C,TAN J P.Damage situation and controlArceuthobiumsichuanensein Xianmi forest area[J].JournalofQinghaiUniversity,2010,28(3):69-72.
[11]周在豹,许志春,田呈明,等.促使云杉矮槲寄生果实提前脱落药剂筛选[J].中国森林病虫,2007,26(5):39-41.
ZHOU Z B,XU Z C,TIAN C M,etal.Screening of the plant hormones causing the fruit pre-abscission ofArceuthobiumsichuanense[J].ForestPestandDisease,2007,26(5):39-41.
[12]周在豹,许志春,田呈明,等.除草剂防治云杉矮槲寄生效果初报[J].植物保护,2014,40(4):198-200.
ZHOU Z B,XU Z C,TIAN C M,etal.Preliminary report on effciency of herbicides on controllingArceuthobiumsichuanense[J].PlantProtection,2014,40(4):198-200.
[13]BARRETT L G,THRALL P H,BURDON J J,etal.Life history determines genetic structure and evolutionary potential of host-parasite interactions[J].TrendsinEcology&Evolution,2008,23(12):678-685.
[14]MCDONALD B A,LINDE C.Pathogen population genetics,evolutionary potential,and durable resistance[J].AnnualReviewofPhytopathology,2002,40:349-379.
[15]AMICO G C,VIDAL-RUSSELL R,AIZEN M A,etal.Genetic diversity and population structure of the mistletoeTristerixcorymbosus(Loranthaceae)[J].PlantSystematicsandEvolution,2014,300:153-162.
[16]HAMRICK J L,GODT M J W,SHERMAN-BROYLE S L.Factors influencing levels of genetic diversity in woody plant species[J].NewForests,1992,6:95-124.
[17]HAMRICK J L,GODT M J W.Effects of life history traits on genetic diversity in plant species[J].PhilosophicalTransactionsoftheRoyalSocietyB:BiologicalSciences,1996,351:1 291-1 298.
[18]NYBOM H.Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants[J].MolecularEcology,2004,13:1 143-1 155.
[19]NYBOM H,BARTISH I V.Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants[J].PerspectivesinPlantEcology,EvolutionandSystematics,2000,3:93-114.
[20]Mathiasen R L,NICKRENT D L,SHAW D C,etal.Mistletoes:pathology,systematics,ecology,and management[J].PlantDisease,2008:988-1 006.
[21]PHOENIX G K,PRESS M C.Linking physiological traits to impacts on community structure and function:the role of root hemiparasitic Orobanchaceae (ex-Scrophulariaceae)[J].JournalofEcology,2005,93:67-78.
[22]PRESS M C,PHOENIX G K.Impacts of parasitic plants on natural communities[J].NewPhytologist,2005,166:737-751.
[23]MUTIKAINEN P,KOSKELA T.Population structure of a parasitic plant and its perennial host[J].Heredity,2002,89:318-324.
[24]GAGNE G,ROECKEL-DREVET P,GREZES-BESSET B,etal.Study of the variability and evolution ofOrobanchecumanapopulations infesting sunflower in differentEuropeancountries[J].TheoreticalandAppliedGenetics,1998,96:1 216-1 222.
[26]JEROME C A,FORD B A.The discovery of three genetic races of the dwarf mistletoeArceuthobiumamericanum(Viscaceae) provides insight into the evolution of parasitic angiosperms[J].MolecularEcology,2002,11(3):387-405.
[27]ZUBER D,WIDMER A.Phylogeography and host race differentiation in the European mistletoe (ViscumalbumL.)[J].MolecularEcology,2009,18:1 946-1 962.
[28]STANTON S,HONNAY O,JACQUEMYN H,etal.A comparison of the population genetic structure of parasiticViscumalbumfrom two landscapes differing in degree of fragmentation[J].PlantSystematicsandEvolution,2009,281:161-169.
[29]DE VEGA C,BERJANO R,ARISTA M,etal.Genetic races associated with the genera and sections of host species in the holoparasitic plantCytinus(Cytinaceae) in the Western Mediterranean basin[J].NewPhytologist,2008,178:875-887.
[30]HOLZAPFEL S,FAVILLE M Z,GEMMILL C E C.Genetic variation of the endangered holoparasiteDactylanthustaylorii(Balanophoraceae) in New Zealand[J].JournalofBiogeography,2002,29:663-676.
[31]LINHART Y B,ELLLWOOD L M,KARRON J D,etal.Genetic differentiation in the dwarf mistletoesArceuthobiumvaginatumandArceuthobiumamericanumon their principal and secondary hosts[J].InternationalJournalofPlantSciences,2003,164:61-69.
[32]OLIVIER A,GLASZMANN J -C,LANAUD C,etal.Population structure,genetic diversity and host specificity of the parasitic weedStrigahermonthica(Scrophulariaceae) in Sahel[J].PlantSystematicsandEvolution,1998,209:33-45.
[33]DUMINIL J,FINESCHI S,HAMPE A,etal.Can population genetic structure be predicted from life-history traits[J].AmericanNaturalist,2007,169(5):662-672.
[34]田欣,李德铢.DNA序列在植物系统学研究中的应用[J].云南植物研究,2002,24(2):170-184.
TIAN X,LI D Z.Application of DNA sequences in plant phylogenetic study[J].ActaBotanicaYunnanica,2002,24(2):170-184.
[35]ALI M A,AL-HEMAID F,AL-QURAINY F,etal.Assessment of genetic diversity among Indian populations ofCuscutareflexabased on ITS sequences of nrDNA[J].JournalofMedicinalPlantsResearch,2011,5:1 217-1 223.
[36]PANDEY A K,ALI M A.Molecular markers in plant systematics I:nuclear sequences[J].PlantSciencesResearchinIndia:ChallengesandProspects,BotanicalSurveyofIndia,Dehradun,India,2006:21-36.
[37]ZUO Y J,CHEN Z J,KONDO K,etal.DNA barcoding ofPanaxspecies[J].Plantamedica,2011,77:182-187.
[38]狄红艳,骆凯,张吉宇,等.基于ITS和trnL-trnF序列的草木樨种群遗传多样性研究[J].西北植物学报,2014,34(2):265-269.
DI H Y,LUO K,ZHANG J Y,etal.Genetic diversity analysis ofMelilotuspupolationsbased on ITS and trnL-trnF sequences[J].ActaBotanicaBoreali-OccidentaliaSinica,2014,34(2):265-269.
[39]何文,张静,黄智林,等.基于ITS序列对栽培中国樱桃遗传多样性及其群体遗传结构的分析[J].西北植物学报,2014,34(3):463-472.
HE W,ZHANG J,HUANG Z L,etal.Genetic diversity and population genetic structure among local Chinese cherry varieties [Cerasuspseudorasus(Lindl.) G.Don] based on ITS sequence[J].ActaBotanicaBoreali-OccidentaliaSinica,2014,34(3):463-472.
[40]ZHANG Y B,SHAW P C,SZE C W,etal.Molecular Authentication of Chinese Herbal Materials (Review Article)[J].JournalofFoodandDrugAnalysis,2007:15.
[41]HAQUE I,BANDOPADHYAY R,MUKHOPADHYAY K.Intraspecific variation inCommiphorawightiipopulations based on internal transcribed spacer (ITS1-5.8S-ITS2) sequences of Rdna[J].Diversity,2009,1(2):89-101.
[42]DOYLE J J,DOYLE J L.Isolation of plant DNA from fresh tissue[J].Focus,1990,12:13-15.
[43]NICKRENT D L,GARCI′A M A,MARTI′N M P,etal.A phylogeny of all species ofArceuthobium(Viscaceae) using nuclear and chloroplast DNA sequences[J].AmericanJournalofBotany,2004,91(1):125-138.
[44]NICKRENT D L,GARCI′A M A.On the brink of holoparasitism:plastome evolution in dwarf mistletoes (Arceuthobium,Viscaceae)[J].JournalofMolecularEvolution,2009,68(6):603-615.
[45]LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1 451-1 452.
[46]EXCOFFIER L,LAVAL G,SCHNEIDER S.Arlequin (version 3.0):an integrated software package for population genetics data analysis[J].EvolutionaryBioinformaticsOnline,2005,1:47-50.
[47]曾真.基于cpDNA单倍型的银杏群体遗传与谱系分析[D].杭州:浙江大学,2008.
[48]PONS O,PETIT R J.Measuring and testing genetic differentiation with ordered versus unordered alleles[J].Genetics,1996,144(3):1 237-1 245.
[49]FORSTER P,BANDELT H,ROHL A.Network 4.2.0.1.Software available free at www.fluxus engineering.com[M/CD].Fluxus Technology Ltda.2004.
[50]金燕,卢宝荣.遗传多样性的取样策略[J].生物多样性,2003,11(2):155-161.
JIN Y,LU B R.Sampling strategy for genetic diversity[J].BiodiversityScience,2003,11(2):155-161.
[51]REIF B P,MATHIASEN R L,KENALEY S C,etal.Genetic structure and morphological diffenentiation of three western north american dwarf mistletoes (Arceuthobium,Viscaceae)[J].SystematicBotany,2015,40(1):191-207.
[52]吴玉虎.巴颜喀拉山地区植物区系研究[J].云南植物研究,2004,26(6):587-603.
WU Y H.The floristic characteristics in the region of Bayan Har Mountains[J].ActaBotanicaYunnanica,2004,26(6):587-603.
[53]杨明显,樊贤,谭垦.九寨沟-岷山山脉地区东方蜜蜂形态特征研究[J].西南农业学报,2007,20(3):542-546.
YANG M X,FAN X,TAN K.Morphometric study ofApisceranain Jiuzhaigou-Minshan moutain zones[J].SouthwestChinaJournalofAgriculturalSciences,2007,20(3):542-546.
(编辑:潘新社)
Genetic Diversity and Population Genetic Structure ofArceuthobiumsichuanense
BAI Yun1,CHEN Lei1,ZHU Ningbo1,LI Xuewu1,CAIRANG Danzhou2,WU Youlin3,TIAN Chengming1*
(1 College of Forestry,Beijing Forestry University,Beijing 100083,China;2 Forest Pest Control and Quarantine Station of Qinghai Province,Xining 810000,China;3 Beishan Farm of Qinghai Province,Haidong,Qinghai 810500,China)
Abstract:In this study,the total genomic DNA were extracted with CTAB method,and then the genetic structure of the Arceuthobium sichuanense from 9 different populations was analyzed according to the sequence of internal transcribed spacer 1 (ITS1).1)Sixteen haplotypes (H1-H16) were observed from 62 ITS1 sequences,showing a low level of genetic diversity (h=0.678 5,π=0.005 9) in the populations of A.sichuanense.However,there were larger differences of the level of genetic diversity (h=0-1.000 0,π=0-0.009 4) among different populations.The AMOVA analysis showed that the genetic variation within populations of A.sichuanense,accounted for 51.37%,while the genetic variation among populations was 48.63%.2)Haplotypes of network showed that H1 and H12 were relatively original in this study.Also,two haplotypes were not shared among all of populations.H1 exists widely in six populations from Qinghai and Gansu,and H12 only exists in two populations from Sichuan.3)Population clustering based on maximum likelihood and median-joining network based on maximum parsimony method indicated that the three populations from Sichuan are independent populations,while there is no significant differentiation between Gansu and Qinghai population.It is the first report about the genetic diversity and structure of A.sichuanense.The results will provide reference for the evolution and disease control of A.sichuanense.
Key words:Arceuthobium sichuanense;genetic diversity;population genetic structure;haplotype
中图分类号:Q789
文献标志码:A
作者简介:白云(1990-),女,在读硕士研究生,主要从事森林病理学研究。E-mail:yunbai2013@163.com*通信作者:田呈明,教授,博士生导师,主要从事森林病理学研究。E-mail:chengmt@bjfu.edu.cn
基金项目:国家“十二五”林业科技支撑项目(2012BAD19B0702);林业公益性行业科研专项(201204503)
收稿日期:2015-09-07;修改稿收到日期:2016-02-19
文章编号:1000-4025(2016)03-0458-09
doi:10.7606/j.issn.1000-4025.2016.03.0458
