实时荧光PCR在治理石油污染中的应用
2014-10-25喻婵,姚俊
喻 婵,姚 俊
(北京科技大学土木与环境工程学院环境与能源国际合作基地,北京 100083)
石油烃类化合物是环境中普遍存在的一类污染物[1-4],主要元素包括 C、H、O、N、P,是由烷烃、芳香烃、胶质及沥青组成的混合物。在石油的开采、加工、运输、处理等过程中常伴随有石油的泄漏,造成严重的石油污染[5]。治理石油污染的方法很多,主要包括物理方法、化学方法和生物方法[6],而生物方法以其操作简单、价格低廉及环境友好等优点逐渐成为目前的研究热点。同时,生物方法被认为是自然的、绿色的解决方案,能够加速石油污染物的降解,最大限度的减少生态效应问题[7]。近年来,越来越多的能够降解石油烃类化合物的微生物菌株被筛选鉴定出来[8-9],而研究菌株降解石油烃类化合物的代谢途径及降解过程中的功能基因为更好地了解它们的降解机制奠定了基础[10]。功能基因是控制物种的特定功能,并且其表达可能影响微生物本身活性的基因。在没有受到外界影响的情况下,相同种属物种中某个基因的表达量应该是相同的[11]。RT-PCR技术能够被用来检测基因的表达[12-13]。本文从大港油田原油样品中筛选获得12株能够降解原油的菌株,并对它们的降解特性进行了测定。对其中被命名为BS的1株对原油降解效果最好的菌株的代谢途径进行了分析,进一步利用RT-PCR技术对alkB基因的表达量进行了检测。
1 材料与方法
1.1 材料
1.1.1 样品 原油样品采集自大港油田港西区块58-8-3号油井;标准菌株Bacillus subtilis BS5由北京科技大学生物化学学院提供[14]。
1.1.2 培养基(g/L) LB培养基:酵母粉5,胰蛋白胨 10,NaCl 10,pH 7.0;无机盐培养基[15]:CoCl2·6H2O 0.366,KH2PO40.5,(NH4)2SO43.0,FeCl3·6H2O 0.05,Na2HPO4·12H2O 1.26,FeSO4·7H2O 0.03,MgSO4·7H2O 0.54,ZnSO4·7H2O 0.024,MnSO4·H2O 0.015,pH 7.2 ~7.4。添加2%的琼脂制成固体培养基,121℃,灭菌30 min。
1.2 方法
1.2.1 菌种筛选 将原油样品以2%的接种量接种于50 mL的无机盐培养基中,28℃好氧条件下180 r/min摇床培养7 d,相同条件下转接1次。然后采用稀释涂平板法分别涂于以十六烷、甲苯、萘、菲等为唯一碳源的无机盐固体培养基上,28℃恒温箱培养7 d形成单菌落。挑取边缘整齐、生长迅速的菌落接入无机盐培养基中,摇瓶复筛,得到12株目的菌株。
1.2.2 菌株降解特性的测定 石油在紫外光区的特征吸收波长为227 nm。分别配制1.8×10-3~5.4 ×10-3的系列标准原油样品,用 1 cm 的石英比色皿,以正己烷为对照,依次测定上述系列样品的吸光度,得到降解率与吸光度的标准方程,利用该方程得到各菌株的降解率。
标准方程:y=-0.00003x+2.340(R2=0.974),其中y表示吸光度,x表示样品稀释度,R2表示相关系数。
1.2.3 菌株的鉴定 筛选出对石油降解效果最好的菌株BS在28℃、LB培养基中震荡培养至对数生长期,取1.5 mL培养液,12000 r/min离心,收集菌体。采用细菌基因组DNA提取试剂盒进行该菌株总DNA。提取后的总DNA置于-20℃保存,备用。以总DNA为模版,正、反向引物分别为27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-GGTTACCTTGTTACGACTT-3')进行PCR扩增。反应体系为20 μL,其中总 DNA 0.5 μL,PCR mix 10 μL,正、反引物各 0.4 μL,ddH2O 8.7 μL;PCR 的扩增程序:94 ℃ 5 min,94 ℃ 30 s,55℃ 30 s,72 ℃ 90 s,72 ℃ 10 min,35个循环。华大基因公司进行16S rDNA序列的测定,测序结果在GenBank上与已知种属的16S rDNA进行BLAST比对,利用MEGA4构建系统进化树。
1.2.4 分析B.subtilis BSn5菌株的代谢途径SEED软件可被用来整合分析物种的代谢途径[16-17]。利用该软件不仅能够构建所有基因组已完成测序的物种的代谢途径,还能通过该软件找到编码某种酶的基因和此基因催化的代谢途径,比如降解石油烃类化合物的基因及催化途径。
1.2.5 RT-PCR 对 alkB基因的测定 alkB基因在石油降解的代谢途径中起重要作用,利用RT-PCR技术研究该基因经石油污染后表达量的变化,能够进一步了解微生物降解石油烃的机制。提取菌株BS和标准菌株B.stubilis BSn5的总RNA,反转录成cDNA,进行RT-PCR反应。
2 结果与分析
2.1 菌株对石油烃类化合物的降解
从原油样品中筛选得到的12株菌是严格的好氧菌株,属于假单胞杆菌属、金黄色葡萄球菌属、芽胞杆菌属、红球菌属和不动杆菌属。早期的报道指出,这些属的菌株在对烃类化合物、多环族化合物以及它们的衍生物的降解过程中有重要作用。12株菌对原油的降解如图1所示。
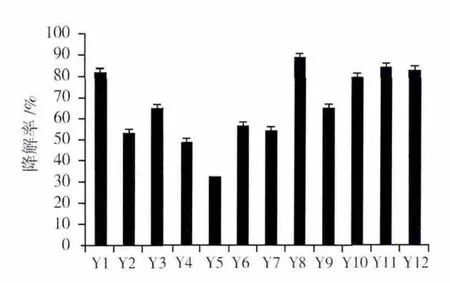
图1 12株菌对石油的降解率Fig.1 The oil degradation efficiency of the twelve strains
由图1可见,12株菌对原油的降解能力各不相同,其中Y8对原油的降解效果最好,降解率高达89.1%。由此推测,在此菌株中可能存在一些功能基因,而这些功能基因对石油降解途径有重要作用,当它们过量表达时,能够更加高效地降解原油。
2.2 菌株的鉴定
将对石油降解效果最好的菌株Y8命名为BS,利用16S rDNA测序,获得的同源性序列均为芽胞杆菌属的16S rDNA序列,其中与B.stubilis BSn5的同源性高达100%,被认为是同种菌株。菌株BS与标准菌株B.stubilis BSn5的基因在同一时刻的表达量是相同的。进一步构建系统进化树,确定该菌株在所属种属中的亲缘关系,如图2所示。从建立的系统进化树可以看出,菌株BS属于芽胞杆菌属,与枯草芽胞杆菌、甲基营养型芽胞杆菌等亲缘关系都较近。
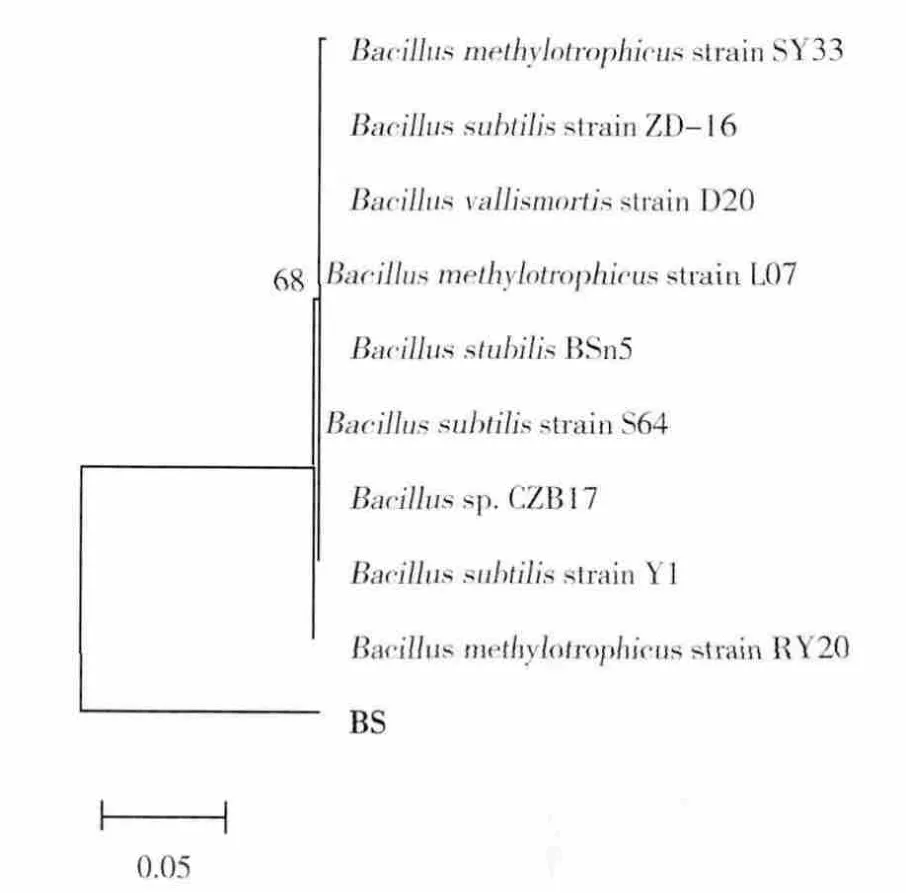
图2 菌株BS的系统进化地位Fig.2 The strain BS on 16S rRNA analysis
2.3 分析B.stubilis BSn5菌株的代谢途径
利用SEED软件重构B.stubilis BSn5的代谢途径图,分析该菌株中参与石油烃类化合物降解的功能基因,如图3所示。
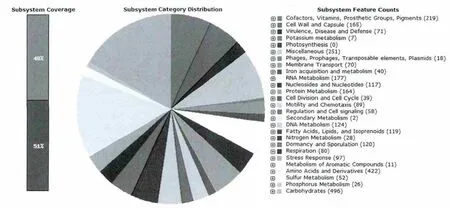
图3 B.stubilis菌株BSn5的亚系统分析Fig.3 The subsystem statistics of Bacillus stubilis BSn5
图3中显示出利用SEED软件构建出的B.stubilis BSn5菌株的整个亚系统分布图,在该菌的所有的编码基因中,有2048个基因参与这个亚系统,而另外2182个基因不参与此系统。利用这个软件重构的代谢途径中,可以找到菌株B.stubilis BSn5参与各类代谢途径的基因比例、基因编码的相关酶以及酶催化的代谢途径。从图3可以看出,菌株B.stubilis BSn5中有11个功能基因参与石油烃类化合物降解的代谢途径,本研究选择alkB基因测定其在经石油污染后表达量的变化。
2.4 RT-PCR测定alkB基因的表达
利用RT-PCR的方法定量了菌株BS和标准菌株B.stubilis BSn5的alkB基因的表达量,以16S核糖体基因为参照基因,结果见图4。
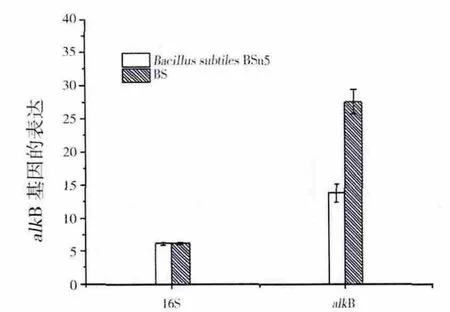
图4 RT-PCR测定alkB基因在菌株BS和B.subtilis BSn5的表达Fig.4 RT-PCR of alkB expression in BS and in B.subtilis BSn5
从图4可以看出,筛选到的菌株BS较标准菌株B.subtilis BSn5的编码alkB基因过量表达了约2倍,该酶被认为在降解原油组分的代谢途径中有重要作用,而作为内参基因的16S核糖体基因的表达量没有发生变化,说明菌株BS在石油污染胁迫条件下能够降解原油组分的有关基因的表达量上调,可能对降解原油组分有作用。
3 讨论
自大港油田港西区块58-8-3号油井原油样品中筛选得到12株能够降解原油的菌株细菌,经鉴定属于假单胞杆菌属、金黄色葡萄球菌属、芽胞杆菌属、红球菌属和不动杆菌属,均属于严格好氧型。对其降解特性进行测定,结果显示,12株菌株对原油的降解能力各不相同,其中菌株BS对原油的降解效果最好,降解率高达89.1%。进一步对菌株BS中alkB基因的表达进行研究发现,较标准菌株B.subtilis BSn5的编码alkB基因过量表达了约2倍,该酶被认为在降解原油组分的代谢途径中有重要作用,说明菌株BS在石油污染胁迫条件下能够降解原油组分的有关基因的表达量上调,可能对降解原油组分有作用。本研究为检测和分析石油污染、筛选含有降解石油功能基因的微生物及构建工程菌提供了新的手段。
[1]Watanabe K.Microorganism relevant to bioremediation[J].Curr Opin Biotechnol,2001,12(3):237-241.
[2]Margesin R,Labbe D,Schinner F,et al.Characterization of hydrocarbon-degrading microbial populations in contaminated and pristine alpine soils[J].Appl Environ Microbiol,2003,69(6):3085-3092.
[3]MacNaughton S J,Stephen J R,Venosa A D,et al.Microbial population changes during bioremediation of an experimental oil spill[J].Appl Environ Microbiol,1999,65(8):3566-3574.
[4]Roling W F M,Milner M G,Jones D M,et al.Bacterial community dynamics and hydrocarbon degradation during a fieldscale evaluation of bioremediation on a mudflat beach contaminated with buried oil[J].Appl Environ Microbiol,2004,70(5):2603-2613.
[5]Hitoshi I,Reia H,Masaaki M,et al.A turbine oil-degrading bacterial consortium from soils of oil fields and its characteristics[J].Int Biodeter Biodegr,2008,61(3):223-232.
[6]Castro-Gutiérrez V M,Rodríguez-Rodríguez C E,Vargas-Azofeifa I.Hydrocarbon degrading microflora in a tropical fuelcontaminated aquifer:assessing the feasibility of PAH bioremediation[J].Int J Environ Res,2012,6(1):345-352.
[7]Ronald M A.Bioremediation of petroleum pollutants[J].Int Biodter Biodegr,1995,35(1-3):317-327.
[8]Wook H W,Joo Y H,An Y J,et al.Isolation and characterization of psychrotrophic and halotolerant Rhodococcus sp.YHLT-2[J].J Microbiol Biotechnol,2006,16(4):605-612.
[9]Shen F T,Ho M J,Huang H R,et al.Molecular detection and phylogenetic characterization of Gordonia species in heavily oil-contaminated soils[J].Res Microbiol,2008,159(7-8):522-529.
[10]Chang Y J,John R S,Amy P R,et al.Phylogenetic analysis of aerobic freshwater and marine enrichment cultures efficient in hydrocarbon degradation:effect of profiling method[J].J Microbiol Meth,2000,40(1):19-31.
[11]Tanga F Y,Raghavendra V,DeLeo J A.Quantitative realtime RT-PCR assessment of spinal microglial and astrocytic activation markers in a rat model of neuropathic pain[J].Neurochem Int,2004,45(2-3):397-407.
[12]Achenbach J E,Topliff C L,Vassilev V B,et al.Detection and quantitation of bovine respiratory syncytial virus using realtime quantitative RT-PCR and quantitative competitive RT-PCR assays[J].J Virol Methods,2004,121(1):1-6.
[13]Yu C,Yao J,Cai M M,et al.Functional gene expression of oil-degrading bacteria resistant to Hexadecane toxicity[J].Chemosphere,2013,93:1424-1429.
[14]Deng Y,Zhu Y G,Wang P X,et al.Complete genome sequence of Bacillus subtilis BSn5,an endophytic bacterium of Amorphophallus konjac with antimicrobial activity for the plant pathogen Erwinia carotovora subsp.Carotovora[J].J Bacteriol,193(8):2070-2071.
[15]崔俊华,金文彪,孙宏量.高效石油降解菌筛选驯化研究[J].上海环境科学,2002,20(6):283-285.
[16]DeJongh M,Formsma K,Boillot P,et al.Toward the automated generation of genome-scale metabolic networks in the SEED[J].BMC Bioinformatics,2007,8(139):1-17.
[17]喻婵,王琰,徐承晨,等.蜡状芽胞杆菌群代谢途径的分析和比较[J].遗传,2011,33:1057-1066.
