原核生物16S rRNA基因多重拷贝及其序列异化
2013-12-03阎永伟张德民
阎永伟,张德民
(宁波大学应用海洋生物技术教育部重点实验室宁波大学海洋学院,宁波315211)
核糖体RNA(rRNA)分子存在于所有细胞生物中,在细胞中执行恒定的功能,其分子序列既有高度保守性片段,又有相对可变性部分,其本身的进化就反映了生物系统发育历史,因此可以用来重现所有细胞生物间的进化关系[1]。Carl Wose在对100多种细胞生物的小亚基RNA分子序列比较分析的基础上提出了生物三域说,使核糖体小亚基rRNA基因(Small subunit ribosomal rRNA gene,rrs)成了原核生物系统及分类学研究的核心标识基因,在原核生物分类、鉴定与系统发生[1-4]以及细菌群落结构和多样性分析[5-7]、细菌定量等方面得到广泛应用。随着对rrs研究的深入,人们逐渐发现,细菌基因组内的rrs是多重拷贝的,有些物种中rrs各拷贝间还存在差异[8],这种差异有时会大于不同菌株间甚至不同种间的差异。研究表明,细菌基因组内rrs拷贝数(Copy number of rrs,CN-rrs)和异化与其利用环境资源的生态策略和对环境的适应性有关。
1 原核生物基因组内的CN-rrs是原核生物种的基本特征之一
截至2012年10月31日,有1297个细菌菌株和89个古菌菌株的CN-rrs被测定,数量从1个到15个不等[8-9],不同拷贝数的菌株所占比例见图1,1~2个拷贝rrs的菌株最多,各占20%;3~7个rrs拷贝数的菌株数也比较多,各占10%左右;8个拷贝以上的菌株相对较少。在属以上分类阶元上CN-rrs与其发育学地位没有关系。但在属的水平上,CN-rrs相对稳定,如Borrelia属8个种23个菌株都是1个,Escherichia属的2个种21个菌株全部是7个;但也有一些属的CN-rrs有一定变化,有些属中变化还比较大,如Bacillus属7~14个拷贝,Clostridium属则从4个(如C.thermocellum)到15个(如C.paradoxum)不等。在种的水平上,绝大多数细菌的CN-rrs都一致,但也有个别种有变化,如Rhodopseudomonas palustris为 1~2个,B.cereus中 12~14个,而C.botulinum中变化最大,含有8、9和11个rrs的菌株都有发现。当然,目前绝大多数种只测了1个菌株的CN-rrs,相信随着每个种所测菌株数的增多,CN-rrs稍有波动的种的比例会有所增加,但不会太多,因为前面所述,有些属的CN-rrs就是一个恒定值。所以,可以说,CN-rrs是原核生物种的基本特征之一。
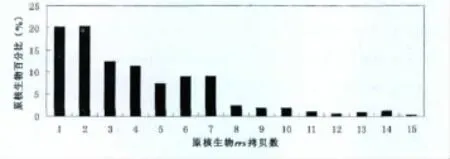
图1 具有不同拷贝数16S rRNA基因的原核生物比例分布图(数据来源于rrndb数据库[9])Fig 1 Distribution of frequency of prokaryotes with different copy number of 16S rRNA gene(Data retrived from rrndb database[9])
2 原核生物CN-rrs与其利用环境资源的生态策略有关
rRNA是调节核糖体合成的关键因子之一,而核糖体在细胞生长、繁殖等基本生命活动中占据核心地位,因此,一般认为,细胞内CN-rrs越多,其合成rRNA越多,进而促进了核糖体的合成,因而有利于细菌达到较高的生长速度。Stevenson和Schmidt[10]在敲除E.coli的不同rRNA操纵子后,发现细胞生长停滞期延长,生长速率降低,对营养物质的竞争能力下降,整体适应能力也随之降低。但是,另有实验表明,即使在富集培养基上快速生长时,细胞内rrs合成rRNA的潜力也没有充分表现出来,因为E.coli中的7个rrs有4个失活后,其他rrs的表达活性明显提高[11]。进一步工作表明,5个rrs的表达就足够支持E.coli在复合培养基上的快速生长,但是当E.coli由胁迫条件转入适宜条件时,如由合成培养基到复合培养基,或从28℃转入42℃,7个rrs对于其快速达到新的对数生长期都有作用,每失活1 个 rrs都伴随着响应时间的延长[12,13],说明多拷贝rrs是其适应进化的结果,赋予细菌面临可利用环境资源发生有利变化时的选择性优势[14]。但是,这种优势又带来不利的一面,就是当其处于恒定的、缓慢生长条件下,多拷贝的结构性表达产生过量的rRNA和核糖体,成为其代谢负担,反而引起生长速度的降低,且生长速度越慢,其影响越大[13,15]。
Klappenbach 等[16]提出,CN-rrs代表了细菌各发育谱系利用资源的生态策略,高CN-rrs菌对资源的输入快速响应[17],而低CN-rrs菌则在持续的缓慢生长时有效分配资源。因此,当土壤细菌暴露于复合营养培养基上时,能在1~2 d内快速形成菌落的细菌,具有的CN-rrs平均5.5个,而延迟到5 d后形成菌落的细菌,其 CN-rrs只有1.4 个[16]。Shrestha等[18]在研究土壤微生物群落演替时发现,细菌的生长反应时间和rrs拷贝数与群落演替阶段显著相关,早期细菌多数都在1 d,个别在2 d内形成肉眼可见菌落,且多数菌株的CN-rrs大于4,而后期细菌则大多在3~15 d内形成可见菌落,且CN-rrs小于2。另外,具有rrs拷贝较少的细菌通常是寡营养细菌,生长在低营养的环境中[19,20]。
3 原核生物基因组内rrs拷贝间的异化
因菌株而异,细菌基因组内rrs各拷贝序列间既可以完全相同[8],又可以完全不同,也可以有几个相同,又与另外几个不同[9]。如Streptococcus agalactiae 2603 V/R具有7个完全相同的 rrs,而 B.subtilis的菌株168,10个rrs序列各不相同[8]。rrs拷贝数较低的菌株中,序列相同的比例很高,如带有2个或3个rrs的菌株,序列完全相同的菌株超过70%,而8个拷贝以上的菌株都存在rrs序列的异化。通常,基因组内rrs序列间差异小于1%,但有时也会大于1%,其差异超过种内不同菌株,甚至不同种间的差异[21-23]。例如,V.splendidus菌株 PB1-10的13个 rrs拷贝分别与 V.splendidus,V.lentus,V.aestuarianus,V.tasmaniensis和 V.anguillarum等五个种聚在一起[22];V.alginolyti-cus ATCC 17749T的 2个 rrs序列(X74690,X56576)之间的相似性只有98.8%,但序列X74690与V.harveyi CECT 525T(X74706)、V.parahaemolyticus CECT 511T(X74720)、V.campbellii 523T(X74692)、V.natriegens CECT526T(X74714)和 V.rotiferianus CECT 577T(AJ316187)等种的模式株的相似性均高于98.8%,同时,序列 X56576与 V.campbellii 523T的另一条序列(X56575)相似性也高于98.8%。我们通过PCR产物直接测序得到的该模式株的序列(JN188438)与X56576和 X74690的序列均不同,相似度分别是98.5%与99.4%,而与V.parahaemolyticus CECT 511T(X74720)、V.campbellii 523T(X74692)、V.natriegens CECT526T(X74714)和 V.rotiferianus CECT 577T(AJ316187)等种模式株的相似性均高于99.5%[23]。在极端情况下,基因组rrs间的差异超过10%,甚至20%,如 Thermoanaerobacter tengcongensis的4个rrs间的最大差异可达到11.6%,Borrelia afzelii的两个rrs拷贝的差异为 20.38%[8]。
4 原核生物基因组内rrs拷贝间异化与其对环境的适应有关
Nilsson等[24]报道,V.vulnificus的 rrs可分成 A、B两种类型。B类型与临床菌株相关,而A与环境菌株相关。另外,Lin和Schwarz[25]发现A型菌株多分离于六七月份,而B型菌株多分离于9月份。已有一些证据将rrs序列的异化与细菌的生长温度联系起来。芽孢杆菌属的B.cereus群包括两个中温菌(B.cereus和B.thuringiensis)及两个低温菌(B.mycoides和 B.weihenstephanensis)。Pruss等[26]发现,中温菌和低温菌都有其特有的rrs印迹,在22个核苷酸长的片段上(180bp to 201bp,按B.cereus的16S rDNA计算),有3个碱基在中温菌是G或C,而在低温菌则是A或T。进一步研究发现,有些菌株2种印迹都包含,且适宜温度也介于中温菌和低温菌之间。实际上,B.cereus群的CN-rrs在6~10个之间,低温印迹和高温印迹在不同拷贝的rrs上。Guinebretiere等[27]依据AFLP指纹技术将425株B.cereus群分成7个组,其生长温度的上下限也相应地呈梯度分布,且在用rrs序列构建的系统发育树上也聚成相应的7个发育分支。嗜盐古菌Haloarcula marismortui具有3个rrs,其中2个拷贝(A和C)基本上是相同的,但另一个(B)的核苷酸序列则有较大的差异。研究发现,培养温度对rrs的表达产生了显著影响,基因B在50℃的表达量比另外一组的高4倍,而15℃时,其表达量则比后者低3倍。一个缺乏B基因的突变体在高温下生长速度较慢。但没有发现盐浓度对其表达量有影响[28]。Lauro等[29]的研究表明,Photobacterium profundum SS9的 rrs的2个螺旋臂的延伸与其嗜压生理特性有关。这些现象支持的观点是:差异的rRNA基因是对环境适应的需要,不同的基因在不同的条件下执行同样的功能。
5 展望
除了与其对环境资源利用策略有关外,原核生物的rrs拷贝数及其异化还和其细胞分化有关。例如,Schirrmeister[30]等人发现,蓝细菌细胞内 rrs的拷贝数的增加与其末端分化相关,蓝细菌门细胞内rrs的拷贝数变异(1~4个)和各拷贝间的异化度也明显低于其他门(绿细菌门、螺旋体门和拟杆菌门);Nanamiya[31]证实,含有单个rRNA操纵子拷贝的缺失突变体,其生长明显变慢,产芽胞率降低1至4个数量级。但总体上讲,目前对原核生物基因组内rrs拷贝数及其异化的生物学意义了解还是初步的,相信更多原核生物的基因组学研究的深入,将能全面揭示其对原核生物适应环境的意义[32]。另外,原核生物rrs拷贝数及其异化的研究对于避免菌种鉴定错误、纠正对原核生物丰度和多样性的高估也具有重要意义[2,33-36]。
[1]Rajendhran J,Gunasekaran P.Microbial phylogeny and diversity:small subunit ribiosomal RNA sequence analysis and beyond[J].Microbiological Research,2011,166:99-110.
[2]N rskov-Lauritsen N.Increased level of intragenomic 16S rRNA gene heterogeneity in commensal strains closely related to Haemophilus influenzae[J].Microbiology,2011,157:1050-1055.
[3]Brenner D J,Crieg N R,Staley J T.Polyphasic taxonomy.in:Garrity G M,et al,eds.Bergey’s manual of systermatic bacteriology[M].2nded.New York:Springer Press,2005,2:43-48.
[4]Balows A,Rusenberg E,Schleifer K H,et al.Defining taxonomic ranks.in:Dworkin M,eds.The prokaryotes[M].3rded.New York:Springer Press,2006,1:58-80.
[5]Lozupone C A,Stombaugh J I,Gordon J I,et al.Diversity,stability and resilience of the human gut microbiota[J].Nature,2011,489:220-230.
[6]Gilbert J A,Steele J A,Caporaso J G,et al.Defining seasonal marine microbial community dynamics[J].The ISME Journal,2012,6:298-308.
[7]Li X R,Ma E B,Yan L Z,et al.Bacterial and fungal diversity in the traditional Chinese liquor fermentation process[J].International Journal of Food Microbiology,2011,146:31-37.
[8]Pei A Y,Oberdorf W E,Nossa C W,et al.Diversity of 16S rRNA genes within individual prokaryotic genomes[J].Applied and Environmental Microbiology,2010,76:3886-3997.
[9]Lee Z M,Bussema C,Schmidt T M.rrnDB:documenting the number of rRNA and tRNA genes in bacteria and archaea[J].Nucleic Acids Research,2009,37:163-168.
[10]Stevenson B S,Schmidt T M.Life history implications of ribosomal RNA gene copy number in Escherichia coli[J].Applied and Environmental Microbiology,2004,70:6670-6677.
[11]Condon C,French S,Squires C,et al.Depletion of functional ribosomal RNA operons in Escherichia coli causes increased expression of the remaining intact copies[J].The EMBO Journal,1993,12:4305-4315.
[12]Condon C,Liveris D,Squires C,et al.rRNA operon multiplicity in Escherichia coli and the physiological implications of rrn inactivation[J].Journal of Bacteriology,1995,177:4152-4156.
[13]Asai T,Condon C,Voulgaris J,et al.Construction and initial characterization of Escherichia coli strains with few or no intact chromosomal rRNA operons[J].Journal of Bacteriology,1999,181:3803-3809.
[14]Weider L J,Elser J,Crease T J,et al.The functional significance of ribosomal(r)DNA variation:impacts on the evolutionary ecology of organisms[J].Annual Review of Ecology,Evolution and Systematics,2005,36:219-242.
[15]Stevenson B S,Schmidt T M.Growth rate-dependent expression of RNA from plasmid-borne rRNA operons in Escherichia coli[J].Journal of Bacteriology,1998,180:1970-1972.
[16]Klappenbach J A,Dunbar J M,Schmidt T M.rRNA operon copy number reflects ecological strategies of bacteria [J].Applied and Environmental Microbiology,2000,66:1328-1333.
[17]Dethlefsen L,Schmidt T M.The performance of the translational apparatus varies with the ecological strategies of bacteria[J].Journal of Bacteriology,2007,189:3237-3245.
[18]Shrestha P M,Noll M,Liesack W.Phylogenetic identity,growthresponse time and rRNA operon copy number of soil bacteria indicate different stages of community succession[J].Environmental Microbiology,2007,9:2464-2474.
[19]Cavicchioli R,Ostrowski M,Fegatella F.Life under nutrient limitation in oligotrophic marine environments:an eco/physiological perspective of Sphingopyxis alaskensis(formerly Sphingomonas alaskensis)[J].Microbial Ecology,2003,45:203-217.
[20]Eichorst S A,Breznak J A,Schmidt T M.Isolation and characterization of soil bacteria that define Terriglobus gen.nov.,in the phylum Acidobacteria [J].Applied and Environmental Microbiology,2007,73:2708-2717.
[21]Michon A,Aujoulat F,Roudiere L,et al.Intragenomic and intraspecific heterogeneity in rrs may surpass interspecific variability in a natural population Veillonella[J].Micriobiology,2010,156:2080-2091.
[22]Jensen S,Frost P,Torsvik V L.The non-random microheterogeneity of 16S rRNA genes in Vibrio splendidus may reflect adaptation to versatile lifestyles[J].FEMS Microbiology Letter,2009,294:207-215.
[23]郭 静,Gadafii L,郭安南,等.溶藻弧菌相关分离株的分子及VITEK鉴定[J].水产学报,2012,36(3):383-390.
[24]Nilsson W B,Parajpye R N,DePaola A,et al.Sequence polymorphisms of the 16S rRNA gene of Vibrio vulnificus is a possible indicator of strain virulence [J].Journal of Clinical Microbiology,2003,41:442-446.
[25]Lin M,Schwarz J R.Seasonal shifts in population structure of Vibrio vulnificus in an estuarine environment as revealed by partial 16S ribosomal DNA sequencing[J].FEMS Microbiology Ecology,2006,45:23-27.
[26]Pruss B M,Francis K P,Stetten F V,et al.Correlation of 16S ribosomal DNA signaturesequences with temperature-dependent growth rates of mesophilic and psychrotolerant strains of the Bacillus cereus group[J].Journal of Bacteriology,1999,181:624-630.
[27]Guinebretiere M H,Thompson F L,Sorokin A,et al.Ecological diversification in the Bacillus cereus group [J].Environmental Microbiology,2008,10:851-865.
[28]López-López A,Benlloch S,Bonfá M,et al.Intragenomic 16S rDNA divergence in Haloarcula marismortui is an adaptation to different temperatures[J].Journal of Molecular Evolution,2007,65:687-696.
[29]Lauro F M,Chastain R A,Blankenship L E,et al.The unique 16S rRNA genes of piezophiles reflect both phylogeny and adaptation[J].Applied and Environmental Microbiology,2007,73:838-845.
[30]Schirmeister B E,Dalquen D A,Anisimova M,et al.Gene copy number variation and its significance in cyanobacterial phylogeny[J].BMC Microbiology,2012,12:177.
[31]Nanamiya N,Sato M,Masuda K,et al.Bacillus subtilis mutants harbouring a single copy of the rRNA operon exhibit severe defects in growth and sporulation[J].Microbiology,2010,156:2944-2952.
[32]Schrider D R,Hahn M W.Gene copy-number polymorphism in nature[C].Proceedings of the Royal Society B:Biological Sciences,2010,277:3213-3221.
[33]Roux S,Enault F,Bronner G,et al.Comparison of 16S rRNA and protein-coding genes as molecular markers for assessing microbial diversity(bacteria and archaea)in ecosystem[J].FEMS Microbiology Ecology,2011,78:617-625.
[34]Kembel S W,Wu M,Eisen J A,et al.Incorporating 16S gene copy number information improves estimates of microbial diversity and abundance[J].PLOS Computational Biology,2012,8:1-11.
[35]Bodilis J,Nsigue-Meilo S,Besaury L,et al.Variable copy number,intra-genomic heterogeneities and lateral transfers of the 16S rRNA gene in Pseudomonas[J].PLoS ONE,2012,7:1-14.
[36]Ki J S,Zhang R,Zhang W,et al.Analysis of RNA polymerase beta subunit(rpoB)gene sequences for the discriminative power of marine Vibrio species[J].Microbiology Ecology,2009,58:679-691.
