四川牦牛奶和曲拉中九株乳酸菌的分子鉴定
2012-01-09包秋华吕嫱王宏梅张和平孙天松
包秋华, 吕嫱, 于 洁, 王宏梅, 张和平, 孙天松
(内蒙古农业大学 乳品生物技术与工程教育部重点实验室,内蒙古 呼和浩特 010018)
四川牦牛奶和曲拉中九株乳酸菌的分子鉴定
包秋华, 吕嫱, 于 洁, 王宏梅, 张和平, 孙天松*
(内蒙古农业大学 乳品生物技术与工程教育部重点实验室,内蒙古 呼和浩特 010018)
为准确鉴定分离自我国四川鲜牦牛奶和曲拉中9株乳酸菌到种水平,作者运用16S r DNA序列分析、rec A基因特异扩增和hsp60-RFLP等多种分子技术对分离自我国四川地区鲜牦牛奶和曲拉中的9株乳酸菌进行分类鉴定。结果证实,16S r DNA序列分析法可将9株乳酸菌初步归类为植物乳杆菌群(4株),肠膜明串珠菌(4株),瑞士乳杆菌(1株)。由于16S r DNA序列分析法不能区分植物乳杆菌和戊糖乳杆菌,为了进一步鉴定植物乳杆菌群中的4株菌,继续采用recA基因特异扩增和hsp60-RFLP技术对其细分,结果表明rec A基因特异扩增和hsp60-RFLP的方法均能很好地把植物乳杆菌群中的4株菌鉴定到种水平,且均为植物乳杆菌。
乳酸菌;分类鉴定;16S r DNA;recA基因;hsp60基因;RFLP
乳酸菌是一类革兰氏阳性、细胞形态为杆菌或球菌、过氧化氢酶为阴性、不形成内生孢子、利用可发酵糖产生50%以上乳酸的细菌的通称[1]。广泛地存在于自然界、动植物体、乳制品和发酵食品中,且大多数为非致病菌,属于有益微生物,具有改善肠道菌群、增强人体免疫力、抗肿瘤、降低血液胆固醇、降血压等功能[2],是人体正常菌群。牦牛乳和曲拉具有独特的生态学特性和较高的经济价值,同时含有种类丰富的微生物,如可检测到很多属、种的乳酸菌[3-5]。
乳酸菌的分类鉴定方法很多,总的来说可分为经典方法和分子生物学方法。经典方法包括形态特征、生理生化反应及血清学反应等,这些方法都是基于微生物表面受体的特异性,属于表型分类法。由于乳酸菌大多属于兼性厌氧菌,且其对营养要求具有复杂性;传统食品是一个复杂的环境系统,其中可能会存在许多人们尚未发过的乳酸菌菌种;传统的表型、生物化学方法鉴定微生物耗时耗力等原因,最终可能导致鉴定结果不稳定[6]。而分子生物学方法是在分子水平上对生物个体的核酸和蛋白质进行研究,根据获得的基因型信息对生物个体进行分类,从分子和基因水平来认识乳酸菌的遗传结构、组成和分类。目前,许多新的分子标记技术被用来进行乳酸菌的鉴定:如16S r DNA序列分析[7]和基于聚合酶链式反应(Polymerase Chain Reaction,PCR),即PCR技术的分子标记技术等,其中基于PCR技术的分子标记又可分为随机扩增多态性 DNA (Randomly Amplified Polymorphic DNA)、扩增片段长度多态性(Amplified Fragment Length Polymorphism)、限制性片段长度多态性(Restriction Fragment Length Polymorphism)等[8-10]。其中16S r DNA是细菌系统分类学中最有用的和最常用的分子钟,其进化具有良好的时钟性质,在结构和功能上具有高度的保守性,素有“细菌化石”之称。16S大小适中,约1.5 kb左右,方便用于PCR扩增和测序,故被细菌学家及分类学家所接受,成为系统发育关系分类的标准方法。16S rDNA结构由可变区和恒定区组成,不同细菌的恒定区序列基本保守,而可变区序列则因细菌而异。所以,可以根据其恒定区的序列设计引物将16S r DNA扩增出来,利用可变区序列的差异来对不同的。
,16S r D NA序列分析也是最常用的方法之一,对有些相近种需要采用其他分子标记技术做补充。
作者以分离自四川地区的鲜牦牛奶和采用传统方法制作曲拉样品中分离得到的乳酸菌为研究对象,首先运用16S r DNA序列分析将其初步鉴定,部分乳酸菌属于植物乳杆菌群,由于植物乳杆菌(Lactobacillus plantarum)和戊糖乳酸菌(Lactobacillus pentosus)之间的16S r DNA序列同源性为99%以上,难以从16S r DNA的系统发育树上区分,故继续采用recA基因特异扩增和hsp60-RFLP相结合的技术,对乳酸菌分离株进行鉴定。
1 材料与方法
1.1 材料
菌株:由内蒙古农业大学乳品生物技术与工程教育部重点实验室提供,是从四川牧民家庭采集的鲜牦牛奶和以传统方法制作的曲拉中分离得到的9株乳酸菌,菌号分别为IMAU80248,IMAU80249,IMAU80250,IMAU80251,IMAU80252,IMAU80286,IMAU80297,IMAU80323,IMAU80325。标准菌Lactobacillus plantarumATCC 14917,Lactobacillus pentosusATCC 8041:购于美国标准菌种收藏所(ATCC)。
1.2 试剂与仪器
10 g/d L脱脂乳培养基、TPY液体培养:参照文献[11]制备;Taq DNA聚合酶、限制性内切酶等:购自大连宝生物工程有限公司;5810R型Eppendorf冷冻离心机、ND-1000型微量紫外分光光度计、PTC-200型梯度基因扩增仪:均购自基因有限公司。
1.3 所用引物
PCR扩增乳酸菌的16S r RNA、recA和hsp60基因引物见表1,所有引物均由上海桑尼生物科技有限公司合成。
2 实验方法
2.1 乳酸菌基因组总DNA提取及纯度的检测
将分离纯化的真空冷冻干燥保存的9株乳酸菌接种于5 m L灭菌的10 g/d L脱脂乳培养基中,37℃、培养24~48 h,活化培养三代。经革兰氏染色镜检确定为纯培养物后,将分离株接种于3 m LTPY液体培养基中,置37℃培养24 h。离心收集菌体后按液氮冻融-CTAB法[8]提取基因组总DNA,并采用微量紫外分光光度计测定基因组DNA的浓度和纯度。

表1 扩增乳酸菌16S r RNA、rec A和hsp60基因扩增引物Tab.1 Primers used for amplification of 16S r RNA,rec A and hsp60 gene in Lactic acid bacteria
2.2 乳酸菌16S rDNA扩增、测序及系统发育树的构建
将提取的基因组DNA稀释到100 ng/μL左右作为PCR扩增的模板,采用通用引物27f和1495r[7,9]扩增9株乳酸菌的16S r RNA 基因。PCR反应体系(50μL)的配制:10×PCR Buffer 5μL,上下游引物各1.5μL (10μmol/μL),模板 DNA 1.5μL(100 ng/μL),d NTP Mix 4μL,r Taq酶0.5 μL,超纯水补充至50μL。扩增程序为:94℃5 min预变性;94℃1 min,58℃1 min,72℃ 2 min,30个循环;72℃延伸10 min。用1.0 g/d L的琼脂糖凝胶电泳检测,并将16S r DNA扩增产物纯化后送上海桑尼生物技术有限公司测序。
将 测 序 得 到 的 序 列 用 BLAST (http://www.ncbi.nlm.nih.gov/blast/)在 GenBank中搜索,与待测菌株同源性最高的已知分类地位的菌种初步确定待测菌株的种属。然后从GenBank数据库中下载已知的乳酸菌标准菌株的16S rDNA序列,使用软件Mega4.0作16S rDNA序列系统发育树。
2.3 rec A基因鉴定L.plantarum群的乳杆菌
RecA基因对于L.plantarum群的特异性较高,因此以相应的L.plantarum群的recA基因设计相应的引物[12],分别以planF、pentF为正引物,p REV为反引物。PCR反应体系(25μL)的配制:10×PCR Buffer 2.5μL,上下游引物各1.0μL(10 μmol/μL),模板 DNA 1μL (100 ng/μL),d NTP Mix 2μL,r Taq酶0.3μL,超纯水17.2μL。扩增循环参数为:94℃3 min预变性;94℃变性30 s,56℃复性10 s,72℃延伸30 s,共30个循环;循环结束后72℃延伸5 min。
2.4 hsp60 基 因 扩 增 产 物-RFLP 标 记L.plantarum 群的乳杆菌
采 用 表1中 引 物 H729和 H730[13]扩 增L.plantarum群的乳杆菌hsp60基因。PCR反应体系(25μL)的配制:10×PCR Buffer 2.5μL,上下游引物各1.0μL(10μmol/μL),模板DNA 1μL(100 ng/μL),d NTP Mix 2μL,r Taq酶0.3μL,超纯水17.2μL。扩增条件为:95℃3 min预变性;95℃变性30 s,40℃复性90 s,72℃延伸1 min,共40个循环。循环结束后72℃延伸10 min。采用NEBcutter 2分析已知L.plantarum和L.pentosus的hsp60序列的酶切位点,选用合适的限制性内切酶HhaI对L.plantarum群的乳杆菌hsp60基因扩增产物进行酶切即:酶切反应总体积为20μL,含2μL PCR反应产物,2 U内切酶和2μL10×反应缓冲液。37℃消化3 h,消化完毕在酶切产物中加0.5倍体积的10×Loading Buffer终止酶切,取6 μL酶切产物用5 g/d L聚丙烯酰胺胶(PAGE)凝胶电泳,银染并将凝胶用扫描仪成像。
3 结果与分析
3.1 DNA提取结果
通过微量紫外分光光度计测定9株待测乳酸菌DNA的浓度和纯度。A260/A280比值在1.8~2.0范围内,1.0 g/d L琼脂糖凝胶电泳结果见图1。基因组DNA在23 kb处,DNA条带清晰。从OD比值和DNA浓度可看出提取DNA质量较好,满足后续试验要求。
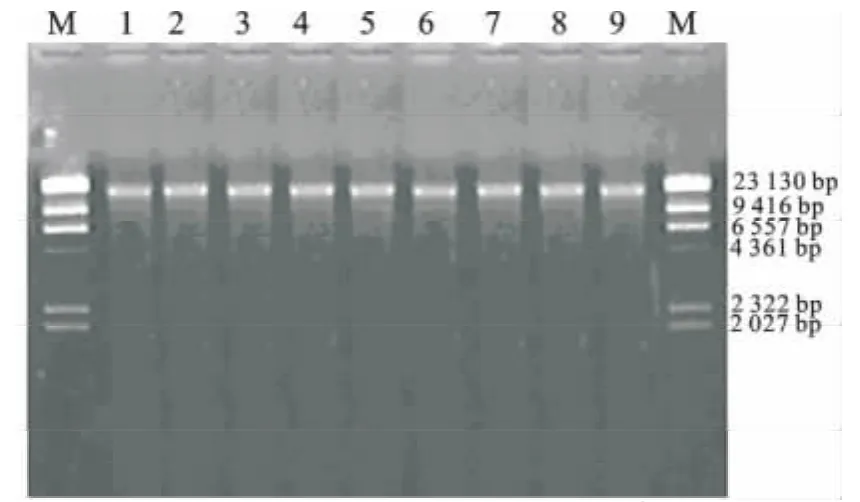
图1 DNA琼脂糖凝胶电泳图Fig.1 Agarose gel electrophoresis of DNA of Lactic acid bacteria
3.2 乳酸菌16S r DNA扩增产物电泳结果
16S r RNA基因的扩增产物于1 g/d L琼脂糖凝胶电泳后经溴化乙锭染色拍照,见图2。从图2中可以看出,PCR扩增比较成功,达到预期效果,即在1 500 bp附近均有清晰可见的明亮条带,并且没有非特异性扩增条带,故9株待测菌株的16S r DNA扩增产物可以测序。

图2 乳酸菌16S r DNA基因的PCR扩增产物电泳图Fig.2 Agarose gel electrophoresis of PCR amplification of 16S rDNA in Lactic acid bacteria
3.3 测序及构建系统发育树
通过对序列测定结果分析可知,9株待测乳酸菌的16S r DNA序列长度为1 500 bp左右。由待测9株乳酸菌的16S r DNA序列与8株参考菌株的16S r DNA序列用Mega4.0软件绘制系统发育树见图3。从总体上可将这9株待测乳酸菌分为3大群:即L.plantarumATCC 14917、L.pentosusATCC 8041、IMAU 80296、IMAU 80297、IMAU 80323和IMAU 80325为第一群;IMAU 80252和L.helveticusATCC 15009为第二群;Leuconostoc mesenteroidesATCC 8293、IMAU 80248、IMAU 80249、IMAU 80250和IMAU 80251为第三群。通过BLAST序列比对分析和系统进化树分析可知,第一类群菌株中IMAU 80296、IMAU 80297、IMAU 80323 和 IMAU 80325 与L.plantarumATCC 14917和L.pentosusATCC 8041相似性均达到99%以上,暂归为植物乳杆菌群,这4株菌还需要采用其他方法进一步鉴定;第二类群菌株中IMAU 80252与Lactobacillus helveticusATCC 15009的同源性在99%以上,从而将其归属为瑞士乳杆菌;第三类群菌株IMAU 80248、IMAU 80249、IMAU 80250和IMAU 80251与菌株Leuconostoc mesenteroidesATCC 8293的同源性达99%以上,故判定此4株菌为肠膜明串珠菌。
3.4 rec A基因鉴定
利用recA基因进一步鉴定植物乳杆菌群中的4株菌,经PCR反应可得到不同相对分子质量的PCR产物,L.pentosus的 PCR 产物 为218 bp,L.plantarum的PCR产物为318 bp[12,14]。由图4可得:菌株 IMAU 80325、IMAU 80297、IMAU 80296、IMAU 80323 与 标 准 菌 株L.plantarumATCC 14917条带一致,因此可将待测4菌株鉴定为植物乳杆菌。
3.5 hsp60酶切产物-RFLP电泳图谱及结果
为了更准确鉴定植物乳杆菌群中的4株菌,进一步采用hsp60/HhaI-RFLP法鉴定。由图5可知:待测菌株IMAU 80296、IMAU 80297、IMAU 80323和IMAU 80325与标准菌株L.plantarumATCC 14917条带一致,但与标准菌株L.pentosusATCC 8041存在差异。因此可将菌株IMAU 80296、IMAU 80297、IMAU 80323和IMAU 80325鉴定为植物乳杆菌,同时能够看出经限制性核酸内切酶HhaI可以完成对植物乳杆菌和戊糖乳杆菌 的区分。

图3 乳酸菌16S r DNA的系统发育树Fig.3 Phylogenetic tree based on 16S rDNA sequence analyses
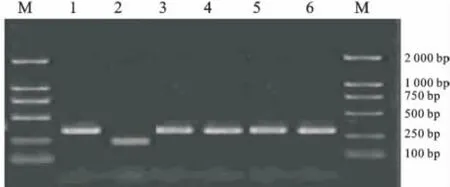
图4 rec A基因的PCR扩增产物电泳图Fig.4 Agarose gel electrophoresis of PCR amplification of rec A in Lactic acid bacteria
4 结语
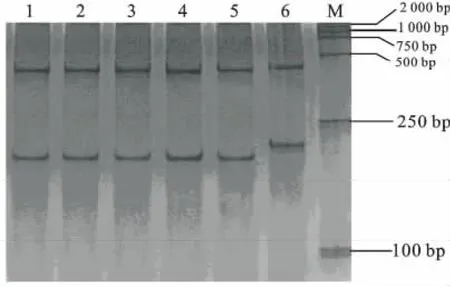
图5 hsp60/Hha I-RFLP的聚丙烯酰胺凝胶电泳图Fig.5 Polyacrylamide gel electrophoresis of Hsp60/Hha IRFLP
以乳酸菌16S r DNA序列测定为基础,并构建系统发育树,初步揭示9株乳酸菌的亲缘关系。由于植物乳杆菌群(植物乳杆菌和戊糖乳杆菌)的16S r DNA序列同源性为99%,难以从系统发育树上区分到种的水平。为了进一步鉴定植物乳杆菌群中的4株菌,采用recA-PCR和hsp60-RFLP相结合的技术,结果表明两种方法鉴定结果相同,即菌株IMAU 80296、IMAU 80297、IMAU 80323 和IMAU 80325全部为植物乳杆菌。综上所述,作者通过16S r DNA序列测定,并结合recA-PCR和hsp60-RFLP技术,成功将9株乳酸菌准确鉴定到种的水平,为其他部分乳酸菌的分子鉴定提供了简便快速的方法。
(References):
[1]凌代文,东秀珠.乳酸菌的分类鉴定及实验基础 [M].北京:中国轻工业出版社,1999:1-5.
[2]金世琳.乳酸菌的科学与技术[J].中国乳品工业,1998,26(2):14-17.
JIN Shi-lin.Science and technology of lactic acid bacteria[J].China Dairy Industry,1998,26(2):14-17.(in Chinese)
[3]ZHANG H P,Xu J,WANG J G,et al.A survey on chemical and microbiological composition of kurut,naturally fermented yak milk from Qinghai in China[J].Food Control,2008,19(6):578-586.
[4]王炜宏.蒙古国传统发酵乳制品中乳酸菌的分离鉴定 [D].呼和浩特:内蒙古农业大学,2010.
[5]DUAN Y H,Tan Z F,Wang Y P,et al.Identification and characterization of lactic acid bacteria isolated from Tibetan Qula cheese[J].Journal of General and Applied Microbiology,2008,54:51-60.
[6]段宇珩,谈重芳,王雁萍,等.乳酸菌鉴定方法在食品工业中的应用及研究 [J].食品工业科技,2007,28(2):242-244.
DUAN Yu-xian,TAN Chong-fang,WANG Yan-ping,et al.Research on the identification of lactic acid bacteria in the food industry[J].Science and Technology of Food Industry,2007,28(2):242-244.(in Chinese)
[7]王炜宏,杜晓华,张家超,等.内蒙古鄂尔多斯地区酸粥发酵液中乳酸菌的分离鉴定 [J].食品与生物技术学报,2010,29(2):265-270.
WANG Wei-hong,DU Xiao-hua,ZHANG Jia-chao,et al.Isolation and identification of LAB from naturally fermented congee in Ordos area of Inner Mongolia[J].Journal of Food Science and Biotechnology,2010,29(2):265-270.
[8]WANG JG,CHEN X,LIU WJ,et al.Identification of Lactobacillus from koumiss by conventional and molecular methods[J].European Food Research and Technology,2008,227(5):1555-1561.
[9]于洁,孙志宏,张家超,等.16S r DNA-RFLP技术鉴定西藏地区乳制品中的乳杆菌[J].食品与生物技术学报,2009,28(6):804-810.
YU Jie,SUN Zhi-hong,ZHANG Jia-chao,et al.Identification of lactobacillus isolated from home-made fermented milk in Tibet by 16S rDNA-RFLP[J].Journal of Food Science and Biotechnology,2009,28(6):804-810.(in Chinese)
[10]剧柠,张家超,孙志宏,等.制作云南乳扇用酸乳清中乳杆菌的多样性分析 [J].食品与生物技术学报,2010,29(5):735-741.
JU Ning,ZHANG Jia-chao,SUN Zhi-hong,et al.The Polymorphism ofLactobacillifrom acid whey for dairy fan in Yunnan Area[J].Journal of Food Science and Biotechnology,2010,29(5):735-741.
[11]小崎道雄,内村泰,冈田早苗,等.乳酸菌実験マニュアル[M].東京:朝倉書店,1992:29-72.
[12]Torriani S,Felis G E,Dellaglio F.Differentiation ofLactobacillus plantarum,L.pentosusandL.paraplantarumbyrecA gene sequence analysis and multiplex PCR assay withrecA gene-derived primers[J].American Society for Microbiology,2001,67(8):3450-3454.
[13]Goh S H,Facklam R R,Chang M,et al.Identification ofEnterococcusspecies and phenotypically similarLactococcusandVagococcusspecies by reverse checkerboard hybridization to chaperonin 60 gene sequences[J].Journal of Clinical Microbiology,2000,38:3953-3959.
[14]刘长建,权春善,范圣第,等.16S rDNA和rec A-gene对乳酸II32的鉴定 [J].大连民族学院学报,2007,36:50-52.
LIU Chang-jian,QUAN Chun-shan,FAN Sheng-di,et al.Identification of lactic acid bacteria II32 by sequence analysis of 16S r DNA and rec A-gene[J].Journal of Dalian Nationalities University,2007,36:50-52.(in Chinese)
Molecular Identification of Nine Strains Lactic Acid Bacteria Isolated from Yak Milk and Triton in Sichuan
BAO Qiu-hua,LU Qiang,YU Jie,WANG Hong-mei,ZHANG He-ping,SUN Tian-song*
(Key Laboratory of Dairy Biotechnology and Engineering Ministry of Education,Inner Mongolia Agricultural University,Hohhot 010018,China)
In this study,To accurately identify lactic acid bacteria(LAB)that were isolated from fresh yak milk and Qula in Sichuan region of China,nine LAB strains were classified and identified to species level by using 16S r DNA sequencing analysis,multiplex PCR assay of rec A gene,hsp60-PCR-RFLP analysis etc.The results showed that nine LAB strains were initially characterized as Lactobacillus plantarum group (4 strains),Leuconostoc mesenteroides(4 strains)andLactobacillus helveticus(1 strain)by 16S r DNA sequencing analysis.However,16S r DNA sequencing analysis could not distinguish betweenL.plantarumandL.pentosus.For further identification of strains inL.plantarumgroup,multiplex PCR assay ofrecA gene andhsp60-RFLP analysis were conducted.The results revealed that multiplex PCR assay ofrecA gene andhsp60-RFLP could identifyL.plantarumgroup to species level and 4 LAB strains inL.plantarumgroup were identified asL.plantarum.
lactic acid bacteria,identification,16S r DNA,RecA gene,Hsp60,RFLP
*通信作者:孙天松(1967-),女,内蒙古呼和浩特人,工学博士,教授,博士研究生导师,主要从事乳品微生物方面的研究。Email:sts9940@sina.com
Q 789
A
1673-1689(2012)04-0396-06
2011-03-23
国家自然科学基金地区重点项目(30860219);内蒙古自治区自然科学基金重点项目(2010Zd15)。
包秋华(1973-),女,内蒙古赤峰人,乳品生物技术与工程博士研究生,助理研究员,E-mail:nmgbqh@126.com
