棉花NLP(Nin-Like Protein)基因家族的全基因组鉴定及表达分析
2023-10-25丁国华肖光辉竺丽萍
丁国华,肖光辉,竺丽萍
棉花NLP(Nin-Like Protein)基因家族的全基因组鉴定及表达分析
丁国华,肖光辉,竺丽萍
陕西师范大学生命科学学院,西安 710119
【目的】全基因组范围内探究棉花NLP转录因子的结构特点及进化特征,深入了解棉花NLP转录因子表达模式,为后续NLP的功能研究和利用奠定基础。【方法】采用blastp和HMMsearch 2种策略进行搜索,鉴定亚洲棉、雷蒙德氏棉、海岛棉、陆地棉4种棉花全基因组范围内的NLP转录因子家族成员。对确认后的棉花NLP家族成员进一步展开生物信息学分析,利用在线软件Expasy对分子量、理论等电点等理化性质进行预测;使用MEGA 7软件构建系统进化树;通过网站MEME进行蛋白保守基序分析;运用在线软件GSDS 2.0分析基因结构;TBtools进行染色体定位绘制;McscanX进行棉花NLP家族复制基因分析;利用PlantCARE网站预测棉花NLP基因家族启动子元件;通过TBtools绘制不同组织及非生物胁迫下棉花NLP基因表达热图,分析组织表达特性和响应非生物胁迫的特征。通过RT-qPCR分析缺氮和复氮处理后棉花NLP基因的表达情况。【结果】从亚洲棉、雷蒙德氏棉、海岛棉、陆地棉蛋白数据库中分别筛选出11、11、21和22个NLP转录因子成员,家族蛋白序列长度为693—996个氨基酸,相对分子质量为76.92—110.02 kDa,理论等电点为5.13—7.77,亚细胞定位几乎均定位于细胞核中,NLP基因启动子区发现大量激素响应和逆境响应顺式作用元件。系统进化分析将棉花NLP蛋白分为Ⅰ、Ⅱ和Ⅲ组,基因复制分析发现片段复制是NLP基因在棉花中扩张的主要方式。Ka/Ks均小于1显示棉花中NLP基因进化主要经历纯化选择。表达分析结果也证实棉花响应氮饥饿和复氮过程。【结论】从亚洲棉、雷蒙德氏棉、海岛棉、陆地棉中分别鉴定获得11、11、21和22个NLP转录因子成员,它们之间具有较高的保守性,又有一定程度的差异。陆地棉在缺氮及复氮处理过程中表达量发生显著变化,可能在棉花响应硝酸盐过程中具有一定作用。
棉花;NLP转录因子;氮信号;生物信息学
0 引言
【研究意义】棉花是世界上重要的天然纤维作物及油籽作物,作为世界纺织工业的重要支柱,在全球80多个国家均有种植[1]。随着全球气候和生态环境的变化,棉花的生长受到低温、高温、干旱、盐碱胁迫等多种环境压力的影响,这些逆境胁迫影响作物生理和代谢过程,从而影响其生长发育,最终影响作物产量及质量,降低经济效益[2-3]。干旱胁迫影响植物对氮素的吸收和利用[4],而氮素作为一种大量元素,在维持植物的发芽、生长发育、开花结果、抗逆性及产量等方面均有着必不可少的作用[5]。硝酸盐是土壤中氮元素存在的主要形态之一,对植物的生长发育具有关键的生理及营养作用[5]。氮信号通路基因和转录因子(包括硝酸盐转运蛋白基因(nitrate transporter,NRT)、硝酸还原酶基因(nitrate reductase,NR)、亚硝酸还原酶基因(nitrite reductase,NiR)等)在硝酸盐吸收和同化过程中发挥关键作用[6-10]。NLP(NIN-like protein)转录因子在植物氮信号中有着重要的作用[11],通过研究棉花NLP家族可以进一步了解棉花氮素调节相关基因,对探究棉花的氮素吸收与同化的调控机制具有重要意义,为进一步提高棉花氮肥利用效率提供参考。【前人研究进展】拟南芥中鉴定的第一个感应硝酸盐转运蛋白NRT1.1,可以被CIPK23和CIPK8磷酸化,以调节高亲和力或低亲和力的硝酸盐反应[6]。NRT1.1是细胞外质膜硝酸盐转运传感器,而NLP7被证实是细胞内硝酸盐传感器[11]。NLP是一类植物特异性转录因子家族,在硝酸盐信号转导和同化过程中发挥重要作用[12]。NIN(nodule inception)蛋白最早在豆科模式植物百脉根中被鉴定,起初作为调控共生根瘤早期发育的调节因子[13]。之后在非豆科植物(包括拟南芥[14]、水稻[14]、小麦[15]、玉米[12]、毛果杨[9]、枳[10]、甘蓝型油菜[16]、番茄[17]等)中鉴定到Nin同源蛋白Nin-like蛋白。研究发现NLP转录因子均具有2个典型的特征结构域,氨基端具有高度保守的RWP-RK结构域,可以与启动子上响应硝酸盐的顺式作用元件(nitrate-responsive-element,NRE)结合,激活和调控下游基因的表达[18-20]。羧基端具有PB1结构域,能与其他含有该结构域的蛋白形成二聚体[14]。此外,在其序列的氨基端还有一个GAF-like结构域,在维管植物中,GAF结构域存在于植物色素和与乙烯信号相关的蛋白质中[21-23],但在NLP蛋白中,其功能尚不明确[18]。在模式植物拟南芥中,已对NLP转录因子功能进行了大量研究,拟南芥NLPs可以结合硝酸盐响应顺式元件在初级氮响应调节中起重要作用[20, 24-25]。AtNLP7可与一些氮通路关键基因(包括、、、、等)结合,激活或抑制下游基因来调控氮同化和氮代谢[26-27]。过表达拟南芥可增强氮和碳的同化,在缺氮和氮充足的条件下均能够促进植物生长[28]。AtNLP8被证实可以直接与脱落酸分解代谢酶基因()的启动子结合,是硝酸盐促进种子萌发的主要调节因子[29]。研究发现,在持续的硝酸盐和氮饥饿条件下,AtNLP6和AtNLP7可与另一个关键转录调控因子TCP20相互作用,在调控硝酸盐响应基因的表达过程中发挥重要作用[30]。在硝酸盐环境中,硝酸盐触发Ca2+信号偶联钙依赖性蛋白激酶CPK10/CPK30/CPK32,并在S205位点磷酸化NLP7以确保其定位在核中,并激活初级硝酸盐响应,该信号在营养生长网络中对形成组织器官结构和生物量至关重要[31]。过量表达促进水稻生长,提高水稻氮素利用率和产量[32]。在中等氮水平条件下,过量表达株系的氮利用效率显著提高47%,产量较野生型显著提高30%[33]。另外,研究发现,敲除拟南芥比野生型表现出较强的抗旱能力[24],可见NLP转录因子不仅在调控氮素信号中具有非常重要的作用[18],而且对调节植物抗旱性有一定作用。【本研究切入点】虽然在一些植物中对NLP转录因子家族进行了鉴定,但有关棉花NLP的相关研究尚未见报道。四倍体陆地棉(,(AD)1)和海岛棉(,(AD)2)起源于100万—150万年前[34]。棉花基因组学的发展为棉花基因家族的鉴定提供了必需的资源,多个高质量的基因组数据库(包括2个二倍体棉花——雷蒙德氏棉(,(D5))[35]和亚洲棉(,(A2))[36],以及2个异源四倍体棉花——陆地棉(,(AD1))[37-39]和海岛棉(,(AD2))[38])为鉴定棉花NLP基因及其潜在功能奠定基础。【拟解决的关键问题】本研究基于已发表的4种棉花基因组,采用生物信息学手段,对亚洲棉、陆地棉、海岛棉及雷蒙德氏棉全基因组范围内NLP转录因子成员进行鉴定,并从基因和蛋白水平上对其进行系统地分析,此外,还分析了陆地棉NLP基因组织表达、纤维发育过程、非生物胁迫下的表达情况,以及通过实时荧光定量PCR对氮饥饿和复氮过程的表达进行分析,为棉花NLP转录因子特点与功能的进一步探究提供依据。
1 材料与方法
1.1 植物材料与处理
将饱满、均一的陆地棉种子(徐州142)播种于蛭石,置于30 ℃ 16 h光照/28 ℃ 8 h黑暗条件下。待种子萌发后,挑选长势一致的幼苗,用去离子水洗净根后置于营养液(2.14 mmol·L-1KH2PO4、3 mmol·L-1MgSO4、1.3 mmol·L-1Ca(NO3)2、1.5 mmol·L-1KNO3、25 μmol·L-1H3BO3、2 μmol·L-1ZnSO4、2 μmol·L-1MnCl2、0.5 μmol·L-1CuSO4、0.5 μmol·L-1Na2MoO4和20 μmol·L-1Fe-EDTA)中培养3周后,将营养液替换为缺氮营养液[17]进行缺氮处理,分别于0、6、12、24和48 h取根系。缺氮处理72 h后,用5 mmol·L-1硝酸盐(KNO3)进行复氮处理,于0、0.5、1、2和4 h取植株根系,液氮速冻后,-80 ℃保存,用于RNA提取及RT-qPCR试验。
1.2 棉花NLP转录因子家族成员的鉴定
从Tair网站(http://www.arabidopsis.org/)下载拟南芥()9个NLP成员蛋白序列,亚洲棉基因组数据来源于CottonMD网站(http:// yanglab.hzau.edu.cn/CottonMD)[40],海岛棉数据库来源于MagenDB网站(http://magen.whu.edu.cn)[41],陆地棉下载于CottonFGD(https://cottonfgd.org/),雷蒙德氏棉数据下载于Phytozome(https://phytozome- next.jgi.doe.gov/)数据库,以拟南芥NLP蛋白序列为查询序列,在4种棉花全基因组蛋白数据库中进行本地blastp搜索,e值小于1×e-15;同时,从Pfam数据库[42](http://pfam.xfam.org/)下载保守结构域RWP-RK(PF02042)及PB1(PF00564)的HMM文件,基于隐马尔可夫模型策略,进行HMMsearch搜索,e值小于1×e-15。2种方法得到的NLP蛋白整合后删除重复及冗余转录本,进一步通过EMBL的SMART(http://smart.embl-heidelberg.de/)及NCBI的Batch CD-search(https://www.ncbi.nlm.nih.gov/Structure/ bwrpsb/bwrpsb.cgi)确认结构域,获得4种棉花NLP转录因子家族成员。
1.3 棉花NLP蛋白理化特征及亚细胞定位预测
使用EXPASY网站中的在线程序ProtParam(http://web.expasy.org/protparam/)计算棉花NLP蛋白氨基酸长度、分子量大小、等电点、总平均亲水性(grand average of hydropathicity,GRAVY)等信息。使用网站WoLF PSORT[43](https://www.genscript.com/ wolf-psort.html)进行亚细胞定位预测,生物类型选择植物。
1.4 棉花NLP的系统进化分析
从Tair数据库下载拟南芥()NLP成员蛋白序列,从Phytozome数据库下载甘蓝型油菜()、水稻()、玉米()及二穗短柄草()NLP成员蛋白序列,用于构建系统发生树。首先使用Clustalx软件对9个物种NLP蛋白序列进行多序列比对,之后使用软件MEGA7[44],选择Neighbor-joining方法进行建树,检验方法选择bootstrap method,重复1 000次,置换模型选择Jones-Taylor-Thornton (JTT) Model。
1.5 棉花NLP染色体定位分析
根据棉花NLP基因ID在陆地棉、海岛棉、亚洲棉和雷蒙德氏棉基因组的注释,提取NLP家族成员位置信息及所在染色体的全长信息,利用TBtools[45](https://github.com/CJ-Chen/TBtools)进行染色体定位分析。
1.6 棉花NLP保守基序,基因结构分析及蛋白质结构分析
调取陆地棉、海岛棉、亚洲棉和雷蒙德氏棉的NLP蛋白序列,提交至在线网站MEME(http://meme- suite.org/tools/meme)进行保守基序分析[46]。调取棉花NLP家族成员的基因组注释信息,在基因结构可视化网站GSDS2.0[47](http://gsds.cbi.pku.edu.cn/)中进行棉花NLP家族成员基因结构特征分析。利用SOPMA网站(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page=npsa_sopma. Html)进行NLP蛋白质二级结构预测。
1.7 棉花NLP共线性分析
使用MCScanX进行棉花NLP家族复制基因分析。使用Dual Systeny Plotter程序进行共线性分析(https://github.com/CJ-Chen/TBtools)[45],e值设为1.0×10-5。使用KaKs_Calculator计算每对NLP复制基因的非同义替换率(Ka)和同义替换率(Ks)[48]。
1.8 基因的表达分析
从CottonFGD网站调取已拼接完成的陆地棉不同组织表达转录组数据及非生物胁迫表达转录组数据。以Log2FPKM对转录组数据进行均一化,利用TBtools软件中的heatmap程序绘制基因表达模式热图。通过RT-qPCR进行缺氮及复氮处理过程的表达量分析,使用天根多糖多酚总RNA提取试剂盒提取RNA,使用Thermo Scientific RevertAid第一链cDNA合成试剂盒进行反转录。利用Primer Premier 5.0软件设计定量特异性引物(电子附表1),利用ABI StepOnePlus实时荧光定量PCR仪,诺唯赞ChamQ SYBR qPCR Master Mix,采用两步法进行定量PCR反应。程序设置为95 ℃ 30 s;95 ℃ 10 s,60 ℃ 30 s,40个循环。陆地棉为内参基因,每个反应3次重复,根据2-Ct法分析RT-qPCR数据。
1.9 棉花NLP家族蛋白相互作用网络预测及互作蛋白表达相关性分析
通过蛋白互作在线网站STRING(https://string. db.org)预测棉花NLP家族成员相关的互作关系,选择模式物种拟南芥作为同源蛋白物种。以预测得到的互作蛋白的序列调取陆地棉中同源蛋白,从cottonFGD网站调取其在不同组织中表达量的转录组数据,进行表达特征相关性分析。
2 结果
2.1 棉花NLP转录因子家族成员鉴定及基本信息
通过鉴定和分析4种棉花NLP基因家族,在陆地棉、海岛棉、亚洲棉和雷蒙德氏棉中共鉴定出65个NLP蛋白,分别是22个GHNLP蛋白、21个GbNLP蛋白、11个GaNLP蛋白和11个GrNLP蛋白(表1)。对棉花NLP家族成员进行理化性质分析,发现该家族蛋白长度为693—996个氨基酸,相对分子质量为76.92—110.02 kDa,理论等电点为5.13—7.77,平均等电点值为5.92。亚细胞定位预测显示,除GrNLP8-1位于叶绿体及细胞核,其余成员均位于细胞核。总平均亲水性(GRAVY)为-0.521—-0.330,即棉花NLP家族成员均为低亲水性蛋白。棉花NLP蛋白均各包含1个RWP-RK(PF02042)和1个PB1(PF00564)结构域(表1)。
2.2 棉花NLP蛋白系统发育树,保守基序及基因结构分析
为了分析棉花NLP蛋白的进化关系,将4种棉花NLP氨基酸序列与其他5种植物(包括2种双子叶植物(拟南芥和甘蓝型油菜)和3种单子叶植物(水稻、玉米和二穗短柄草))的NLP氨基酸序列构建邻接进化树(图1)。可以看出棉花NLP家族蛋白明显被分为3组。同时,根据进化树分支及拟南芥NLP命名情况,对棉花NLP蛋白进行重新命名。在棉花NLP家族中,GaNLP1-1/2/3、GaNLP4-1/2、GrNLP1-1/2/3、GrNLP4-1/2、GbNLP1A/D-1/2/3、GbNLP4A/D-1/2、GHNLP1A/D-1/2/3和GHNLP4A/D-1/2为第I组;GaNLP7-1/2/3、GrNLP7-1/2/3、GHNLP7A/D-1/2/3、GbNLP7A/D-1/3和GbNLP7D-2蛋白属于第Ⅱ组;第Ⅲ组包含GaNLP8-1/2/3、GbNLP8A/D-1/2/3、GrNLP8-1/2/3和GHNLP8A/D-1/2/3。每个分支中均存在双子叶植物和单子叶植物,表明NLP家族的基因扩增发生在单子叶植物和双子叶植物分化之前。还发现,NLP蛋白在4种棉花之间的进化关系更紧密,而单子叶植物水稻、玉米和二穗短柄草NLP蛋白和4种棉花NLP蛋白的亲缘关系较远。

表1 棉花NLP基因家族成员信息
a、b:保守结构域RWP-RK(PF02042)和保守结构域PB1(PF00564)的个数
a,b: the numbers of conserved RWP-RK domain (PF02042) and PB1 domain (PF00564)

GH:陆地棉;Gb:海岛棉;Ga:亚洲棉;Gr:雷蒙德氏棉;At:拟南芥;Os:水稻;Zm:玉米;Bna:甘蓝型油菜;Bd:二穗短柄草
通过对4种棉花NLP蛋白序列构建系统发育树,将棉花NLP家族成员分为3组(图2-A)。经MEME分析,在4种棉花NLP蛋白中识别10种保守基序(图2-B),其中,Motif1组成RWP-RK保守结构域,由大约50个氨基酸组成。Motif4和Motif8组成PB1保守结构域(图2-D)[18],由大约80个氨基酸组成。此外,Motif7、Motif3和Motif9组成GAF-like保守结构域[18]。GrNLP8-1、GbNLP7A/D-3缺少Motif7、Motif3和Motif9,GHNLP7A-2缺少Motif7,其余NLP序列中均包含这10种保守基序。为了分析4种棉花NLP基因序列的结构特征,利用GSDS网站对NLP家族基因的外显子/内含子结构进行分析,结果显示,同一组内基因具有相似的基因结构,外显子长度及分布相近(图2-C)。第I组中,除和包含5个外显子外,多数基因包含4个外显子。第Ⅱ、Ⅲ组多数基因包含5个外显子。第Ⅱ组中,包含3个外显子,、和包含6个外显子。第Ⅲ组中,包含6个外显子。
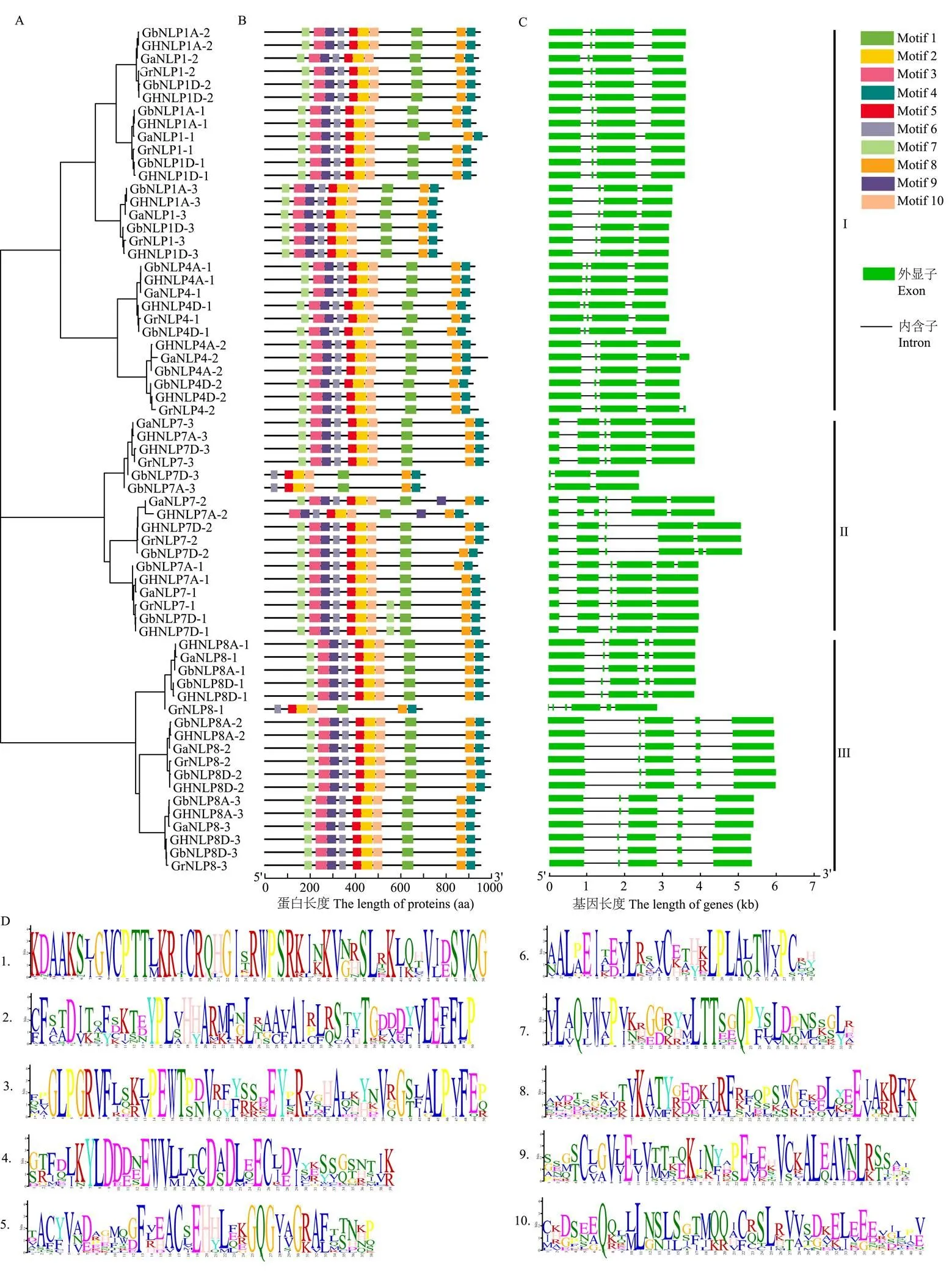
图2 4种棉花NLP蛋白系统进化树(A)、蛋白保守基序(B)、基因结构(C)的分析及保守基序的序列(D)
2.3 棉花NLP蛋白二级结构预测
预测棉花NLP蛋白中的二级结构(表2),65个棉花NLP蛋白质二级结构主要包含无规则卷曲和α-螺旋,其中,无规卷曲所占比例最大,此外还包含延长链和β-转角,β-转角在所有NLP成员中均占最小比例,而且在这些棉花NLP成员蛋白质中各类二级结构的占比相近。
2.4 棉花NLP染色体的定位分析
从基因组注释文件中提取4种棉花NLP基因家族所在染色体位置的信息,分析NLP基因家族成员在染色体上的分布情况(图3)。在亚洲棉中,11个GaNLP主要分布在9条染色体上,分别是Chr.2、Chr.3、Chr.5、Chr.6、Chr.7、Chr.9、Chr.10、Chr.11和Chr.13,其中,Chr.5染色体含有2个GaNLP基因(和),Chr.13染色体含有2个GaNLP基因(和),Chr.2、Chr.3、Chr.6、Chr.7、Chr.9、Chr.10和Chr.11染色体分别含有1个GaNLP基因(分别为、、、、、和)。

表2 棉花NLP蛋白质二级结构

续表2 Continued table 2

图3 NLP家族成员在4种棉花中的染色体定位分析
雷蒙德氏棉NLP成员分布在8条染色体上,分别为Chr.2、Chr.3、Chr.4、Chr.5、Chr.6、Chr.8、Chr.9和Chr.11,其中,Chr.5染色体含有3个GrNLP基因(、和),Chr.2染色体含有2个GrNLP基因(和),Chr.3、Chr.4、Chr.6、Chr.8、Chr.9和Chr.11染色体各含有1个GrNLP基因(分别为、、、、和)。
在陆地棉中,共含有22个GHNLP基因,分布在16条染色体上,在A亚基因组的8条染色体上分布着11个GHNLP基因,A02染色体上含有、和,A01染色体上含有2个成员(和),A03、A05、A08、A09、A10和A12染色体上分别含有1个NLP成员(分别为、、、、和)。在D亚基因组的8条染色体上分布着11个GHNLP成员。其中,D01染色体上含有2个成员(分别为和),D02染色体上含有3个成员(、和),D03、D05、D08、D09、D10、D12染色体上分别含有1个GHNLP成员,分别为、、、、和。
在海岛棉中,21个GbNLP成员分布在15条染色体上。10个GbNLP成员分布在7条A亚基因组染色体上,其中,3个成员分布在A02染色体上(、和),A01染色体上含有2个GbNLP成员(和),A03、A05、A09、A10和A12染色体上分别有1个GbNLP成员,分别为、、、和。11个GbNLP成员分布在8条D亚基因组染色体上,其中,3个成员分布在D02染色体上(、和),D01染色体上含有2个成员(和),D03、D05、D08、D09、D10和D12染色体分别包含1个GbNLP成员,分别为、、、、和。
2.5 棉花NLP家族基因的复制及共线性分析
串联重复和片段复制是基因家族扩张的主要驱动力,促进基因家族的形成与基因组的进化。为了探究棉花NLP基因扩张可能的驱动力,分析4种棉花中NLP成员的复制事件,其中,在亚洲棉的5个GaNLP成员中发现3对片段复制事件,在雷蒙德氏棉的7个GrNLP成员中发现6对片段复制事件,在海岛棉的13个GbNLP成员中发现16对片段复制事件,在陆地棉的22个GHNLP成员中发现51对片段复制事件,表明片段复制可能是NLP基因在棉花中扩张的主要驱动力。进一步分析棉花NLP复制基因对的Ka、Ks和Ka/Ks值(表3)。结果表明,4种棉花的NLP复制基因对的Ka/Ks值均小于1(0.118—0.530),推断4种棉花NLP基因在进化过程中受到了纯化选择,表明棉花NLP功能较为保守。
为了探究四倍体与二倍体棉花NLP成员间的进化关系,对四倍体与二倍体棉花间的共线性进行分析,结果表明,22个陆地棉GHNLP基因与11个亚洲棉GaNLP基因存在共线关系,与11个雷蒙德氏棉GrNLP基因存在共线关系(图4-A)。海岛棉21个GbNLP基因与11个亚洲棉GaNLP基因存在共线关系,与11个雷蒙德氏棉GrNLP基因存在共线关系(图4-B)。

A:陆地棉、亚洲棉和雷蒙德氏棉NLP基因的共线性分析;B:海岛棉、亚洲棉和雷蒙德氏棉NLP基因的共线性分析

表3 棉花NLP复制基因对的非同义替换率(Ka)与同义替换率(Ks)比值
2.6 棉花NLP基因启动子区顺式作用元件分析
通过对棉花NLP家族基因启动子区顺式作用元件进行分析(图5),鉴定到大量和激素相关及胁迫响应相关的顺式作用元件,其中,激素响应元件数量较多的为响应乙烯的ERE,响应生长素的TGA- element和AuxRR-core,此外,还有响应脱落酸的AAGAA-motif和ABRE,响应赤霉素的GARE-motif、P-box和TATC-box,响应水杨酸的TCA-element等元件,响应茉莉酸甲酯的CGTCA-motif和TGACG- motif。与胁迫相应相关的元件中数量较多的为MYB、MYC及厌氧响应元件ARE,损伤诱导元件WUN- motif,此外,还有as-1、STRE、AP-1、DRE core、MYB、MYC、TC-rich repeats和box S,干旱响应元件MBS和CCAAT-box,低温响应元件LTR等。另外,棉花NLP基因启动子区还有与氮素响应相关的GCN4元件[49]。
2.7 棉花NLP基因家族不同组织表达模式及响应非生物胁迫表达模式分析
为了探究陆地棉GHNLP成员在棉花生长发育中潜在的作用,对GHNLP家族成员的表达模式进行了分析(图6),陆地棉NLP成员的组织表达谱显示,在根、茎、叶、花瓣、花药、雌蕊、副萼、萼片和花托各组织中表达量都很高,、在各组织中也有较高表达量,其中,在根中表达最高。同时,和在纤维起始时胚珠中表达量较高,随后在胚珠中表达量降低,而在20和25 d时在纤维中表达量升高。在20 d纤维中表达较高,和在纤维起始时胚珠中表达量较高。
为了研究棉花NLP家族基因在非生物胁迫中的潜在作用,对陆地棉GHNLP基因在低温、高温、盐、干旱胁迫下的表达模式进行了分析(图7)。结果显示,、和在高温处理1 h后表达升高,在高温处理3 h后表达升高,在高温处理1、12和24 h时表达升高。在低温处理后表达升高,低温处理3 h,表达升高,在低温处理6、12和24 h时表达量升高,低温处理24 h后,、和表达量升高。NaCl处理1 h后,和表达量升高,在NaCl处理3 h维持较高表达量,随后逐渐降低,NaCl处理24 h后,、和表达量升高。干旱处理3 h后,和表达量升高,随后逐渐下降。在干旱处理6 h后表达上升。

每一列代表一种顺式作用元件,每行代表一个棉花NLP家族基因,方格中数字代表顺式作用元件数量,右上角图例代表顺式作用元件数量范围,红色越深代表数量越多,蓝色越深代表数量越少

A:GHNLP基因在不同组织中的表达水平热图;B:GHNLP基因在胚珠以及纤维发育不同时期的表达水平热图

图7 陆地棉GHNLP基因在非生物胁迫下的表达分析
2.8 棉花NLP蛋白相互作用网络及互作蛋白表达相关性分析
为进一步了解陆地棉中NLP转录因子潜在的功能,在STRING网站中,利用陆地棉NLP蛋白在模式物种拟南芥中的同源序列,构建陆地棉NLP蛋白相互作用网络(图8-A),其中,陆地棉中GHNLP1A/D-1/2/3在拟南芥中同源性最高蛋白均对应AT4G35270,GHNLP4A/D-1/2均对应AT1G20640,GHNLP7A/D-1/ 2/3对应NLP7,GHNLP8A/D-1/2/3对应AT2G43500。网络图中可看出,NLP7蛋白可与硝酸盐转运蛋白NRT1.1、NRT2.1,胞质硝酸还原酶NIA1及转录因子HRS1、LBD37、AGL44等相互作用,可以看出,NLP蛋白和氮素转运、同化等蛋白相互作用,且NLP7位于核心位置。调取陆地棉中同源的NRT1.1、NRT2.1、NIA1、NIR1及HRS1,LBD37、AGL44蛋白对应基因在不同组织转录组的表达量,进行表达特征相关性分析(图8-B),结果表明,陆地棉中与氮素响应相关的互作蛋白对应基因的表达特征均与、和具有很高的相关性,且这些互作蛋白对应基因之间表达也具有很高的相关性。
2.9 氮饥饿及复氮过程中GHNLPs的表达模式
为了研究氮素缺乏条件下棉花的响应情况,对陆地棉幼苗进行氮饥饿和复氮处理后进行RT-qPCR分析,结果发现,在氮饥饿处理过程中,的表达表现出不同程度的变化,整体而言,在氮饥饿处理的几个时间点,几乎所有的表达量较0 h均有不同程度的提高,呈现出不同的变化趋势。表明对氮饥饿的响应随时间变化,各个成员之间也存在一定的差异(图9)。、、和表达量在处理48 h时最高,、、和表达量在氮饥饿处理24 h时最高。
对氮饥饿72 h后的棉花幼苗进行复氮处理,复氮过程中,的表达量在短时间内均显著上调,表明复氮促进了的表达,对氮的响应迅速,复氮4 h后表达量逐渐下降(图10)。、、、、、、、和在复氮0.5 h时即具有迅速响应,表达量达到最高。复氮1 h后表达量最高,、、和复氮2 h后表达量最高。
3 讨论
3.1 棉花NLP基因家族的特征
NLP转录因子在氮素响应过程中起着重要的作用[18, 20],NLP广泛存在于豆科植物以及非豆科植物中,受硝酸盐信号调节,该家族蛋白能够与具有氮响应顺式作用元件(NRE)的启动子结合激活下游相关基因表达[10, 18, 20]。除了响应硝酸盐信号,也有研究表明NLP转录因子可在调节干旱胁迫抗性中发挥一定作用[24]。但棉花NLP基因功能和家族分析并未见报道。本研究以4种棉花为研究对象,系统分析了亚洲棉、雷蒙德氏棉、陆地棉和海岛棉NLP家族的序列特征和进化关系以及陆地棉GHNLP基因的组织表达情况及非生物胁迫处理后表达水平变化情况。在这4种棉花中分别识别到了11、11、22和21个NLP家族成员。在不同物种中NLP蛋白家族成员的数量差异较大,在拟南芥中含有9个NLP[14],水稻中含有6个[14],玉米中[12]鉴定了9个NLP,在枳基因组数据中筛选到4个NLP[10],二穗短柄草中鉴定了7个NLP[15],甘蓝型油菜基因组包含31个NLP[16],毛竹基因组中得到10个NLP基因[50],毛果杨中共鉴定出14个NLP基因家族成员[9],而四倍体棉花和甘蓝型油菜中NLP家族成员较多可能是由于其在进化过程中经历的基因组加倍事件[51-52]。海岛棉中鉴定到21个NLP基因,推测海岛棉在二倍体杂交形成四倍体的进化过程中可能存在基因丢失[53]。棉花NLP蛋白均各包含1个RWP-RK和1个PB1结构域,大部分棉花NLP蛋白在N端还包含1个GAF-like结构域。RWP-RK结构域包含1个螺旋-转角-螺旋基序和1个两亲性亮氨酸拉链,可能参与DNA结合[13-14],PB1结构域包含2个α-螺旋和1个混合的五链β-折叠,可能在其蛋白结合能力中发挥关键作用[54]。这3个结构域(GAF、RWP-RK和PB1)使NLP在非固氮植物氮响应和适应氮缺乏中发挥重要作用[12]。亚细胞定位预测显示65个棉花NLP有64个定位在细胞核中,这与其转录因子的特性相符,而只有GrNLP8-1定位于叶绿体及细胞核,这可能是由于NLP家族的核穿梭现象[27],拟南芥、水稻、毛竹的NLP家族中也有类似现象[33, 50]。总平均亲水性(GRAVY)值表明4种棉花NLP均为低亲水性蛋白(表1)。一些物种中NLP家族蛋白具有较长的氨基酸和较低的等电点值[12,14,18],在棉花NLP家族蛋白中也得到类似的结果(表1),表明NLP转录因子在酸性条件下具有活性。在进化树第I组除和包含5个外显子,其他均包含4个外显子,第Ⅱ组基因包含3—6个外显子,第Ⅲ组基因包含5—6个外显子,总之,较第I、Ⅱ组基因外显子数目多,表明棉花NLP基因结构多样性,该结果与甘蓝型油菜中NLP基因相似[16]。在棉花NLP启动子区识别到大量乙烯响应元件ERE(图5),据文献报道NLP蛋白中的GAF结构域存在于乙烯信号相关的蛋白质中[21-23],由此推测部分棉花NLP蛋白可能与乙烯信号相关,且与GAF结构域有关联。此外在棉花NLP蛋白启动子区识别到大量MYB元件,推测部分棉花NLP可能与MYB转录因子结合。

A:基于拟南芥同源蛋白的陆地棉GHNLP蛋白相互作用网络;B:NLP互作网络蛋白基因表达特征相关性分析。红色字体为棉花NLP蛋白编号
A: PPI network of GHNLPs from upland cotton based on the homologous proteins in; B: Correlation analysis of the expression characteristics of proteins interacted with NLPs in the PPI network. The name of cotton NLP proteins are highlighted in red color
图8 GHNLPs蛋白相互作用网络及互作蛋白表达相关性分析
Fig. 8 PPI network of GHNLPs proteins and the expression correlation analysis of proteins interacted with NLPs
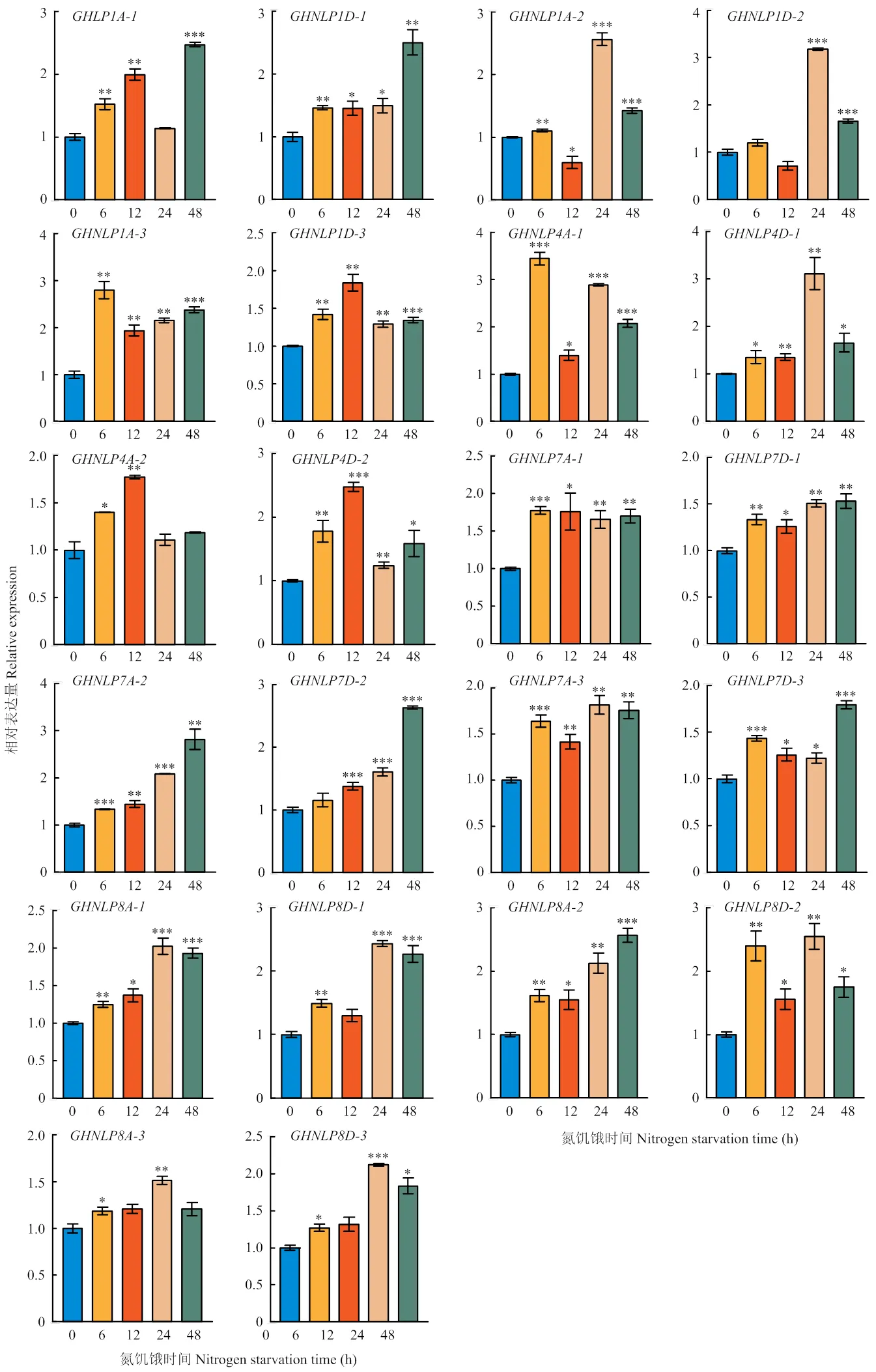
***:<0.001;**:<0.01;*:<0.05
图9 氮饥饿过程中的表达分析
Fig. 9 Expression analysis ofduring nitrogen starvation

图10 复氮过程中GHNLPs的表达分析
3.2 棉花NLPs的系统发育及复制关系
进化分析表明各物种NLP家族均可分为3组(图1),包括拟南芥、甘蓝型油菜、水稻、玉米、二穗短柄草及4种棉花。本研究中棉花和其他物种的分组一致,说明棉花中没有特异进化的NLP成员。而4种棉花NLP成员的基因结构分析和MEME保守基序分析发现,位于相同亚组的NLP家族成员外显子数量及分布相似(图2-C),保守基序分布相似(图2-B)。说明同一亚组的NLP成员可能具有相似的生物学功能。4种棉花NLP家族的复制基因分析发现片段复制是NLP基因在棉花中扩张的主要驱动力。进一步分析了4种棉花中存在片段复制的NLP复制基因对的非同义替换率(Ka)与同义替换率(Ks)及Ka/Ks值,结果表明,棉花中NLP复制基因对的Ka/Ks值均小于1(表3),由此推断4种棉花中NLP基因在进化过程中受到了纯化选择。共线性分析表明,在22个陆地棉GHNLP基因和11个亚洲棉GaNLP基因间存在42对共线关系,在22个陆地棉GHNLP基因和11个雷蒙德氏棉GrNLP基因间存在46对共线关系,在21个海岛棉GbNLP基因和11个亚洲棉GaNLP基因间存在42对共线关系,在21个海岛棉GbNLP基因和11个雷蒙德氏棉GrNLP基因间存在44对共线关系。其中在陆地棉中有2个共线基因和,而在海岛棉中只有1个共线基因。
3.3 棉花NLP基因表达谱及其对缺氮和复氮条件的响应
陆地棉GHNLP基因在根、茎、叶、花瓣、花药、雌蕊、副萼、萼片和花托不同组织的特异性的表达,能够揭示GHNLP基因在棉花生长发育过程中的潜在作用。在所有组织中均有较高表达,说明可能在棉花生长发育过程中发挥重要作用。在根中的表达量最高,拟南芥中是硝酸盐信号通路的关键基因[31],推测棉花中也可能在棉花根中响应硝酸盐过程中发挥重要作用。在花瓣、花药、雌蕊、副萼、萼片和花托中具有较高表达,即在开花期间上调,表明可能调节氮转运和同化以支持开花。陆地棉NLP蛋白相互作用网络图中,NLP7蛋白可与硝酸盐转运蛋白NRT1.1、NRT2.1,胞质硝酸还原酶NIA1等氮素转运、同化相关蛋白相互作用,表明棉花中与NLP7同源的GHNLP7A/D-1/3也可能参与硝酸盐响应过程。
此外,棉纤维发育可以分为起始、伸长、次级细胞壁加厚和成熟阶段4个相互重叠的阶段,开花后0 d(0 DPA)为纤维起始阶段,0—26 DPA纤维长度增长并逐步达到最大值,该时期与纤维的长度紧密相关[55]。次级细胞壁加厚阶段合成大量纤维素,此阶段主要集中在15—40 DPA,对纤维强度至关重要[56]。进一步分析了陆地棉GHNLP基因在棉纤维发育过程中的潜在作用,在纤维起始阶段具有较高表达,表明其可能在纤维起始过程中发挥作用。在棉纤维发育过程中的细胞次级细胞壁加厚期表达量最高。
NLP转录因子还参与逆境响应[27],研究表明NLP转录因子还在调节干旱胁迫抗性中起一定作用[24],通过分析陆地棉GHNLP基因在盐、干旱、低温、高温胁迫下的表达模式(图7),探究陆地棉GHNLP家族成员在非生物胁迫中可能的作用。结果显示,、响应低温和高温胁迫,同时响应高温、低温和干旱胁迫,对低温、高温和盐胁迫均有响应,对干旱和盐胁迫均有响应,响应低温和盐胁迫,和响应盐胁迫,对高温胁迫响应。在干旱胁迫时表达量升高,6 h时表达量最高,之后逐渐下降,与苹果、枳等NLP在干旱胁迫下表达相似[10, 57]。这些基因可能在棉花对非生物胁迫的调节过程中具有一定作用。氮饥饿过程中,几乎均不同程度上调表达,表明在陆地棉响应缺氮的过程中发挥重要作用,且不同成员的表达随着处理时间变化具有不同表达趋势。这与苹果、毛竹等在缺氮处理时表达类似[10, 50]。在氮饥饿后复氮处理过程中,均能短时间内作出快速反应,所有的表达均上调,大部分成员在0.5 h内显著上调,说明硝酸盐诱导的表达。该结果与番茄中表达相似[17]。结合互作网络预测,以及表达特征相关性分析的结果,与拟南芥同源性较高的基因中,和可能具有与类似的调控关系。本研究结果为进一步研究GHNLP基因家族在硝酸盐吸收同化及抵御非生物胁迫方面提供了理论基础。
4 结论
对亚洲棉、雷蒙德氏棉、海岛棉、陆地棉4种棉花NLP转录因子进行全基因组分析,分别识别到11、11、21和22个NLP转录因子成员,均包含RWP-RK和PB1保守结构域,进化分析可分为3组。启动子区识别到大量激素和胁迫响应的顺式作用元件,数量最多的为乙烯响应元件ERE和MYB,与AtNLP7同源性较高的分支中,在根中及纤维起始表达量高,同时,响应盐胁迫,响应高温、低温和盐胁迫,响应高温、低温和干旱胁迫。对缺氮及复氮过程有响应。
[1] CHEN Z J, SCHEFFLER B E, DENNIS E, TRIPLETT B A, ZHANG T Z, GUO W Z, CHEN X Y, STELLY D M, RABINOWICZ P D, TOWN C D, ARIOLI T, BRUBAKER C, CANTRELL R G, LACAPE J M, ULLOA M, PENG C E, GINGLE A R, HAIGLER C H, PERCY R, SAHA S, WILKINS T, WRIGHT R J, VAN DEYNZE A, ZHU Y X, YU S X, ABDURAKHMONOV I, KATAGERI I, KUMAR P A, RAHMAN M U, ZAFAR Y, YU J Z, KOHEL R J, WENDEL J F, PATERSON A H. Toward sequencing cotton () genomes. Plant Physiology, 2007, 145(4): 1303-1310.
[2] YANG Z R, QANMBER G, WANG Z, YANG Z E, LI F G.genomics: trends, scope, and utilization for cotton improvement. Trends in Plant Science, 2020, 25(5): 488-500.
[3] WANG R, LIU L, KONG Z S, LI S D, LU L L, KABIR N, CHEN G Q, ZHANG J X, QANMBER G, LIU Z. Identification ofgene family revealed thatis involved in regulating salinity tolerance in cotton (L.). Plant Physiology and Biochemistry, 2021, 166: 328-340.
[4] XIE S X, CAO S Y, LIU Q, XIONG X Y, LU X P. Effect of water deficit stress on isotope15N uptake and nitrogen metabolism of Newhall orange and Yamasitaka mandarin seedling. Journal of Life Sciences, 2013, 7(11): 1170-1178.
[5] CRAWFORD N M. Nitrate: Nutrient and signal for plant growth. The Plant Cell, 1995, 7(7): 859-868.
[6] HACHIYA T, MIZOKAMI Y, MIYATA K, THOLEN D, WATANABE C K, NOGUCHI K. Evidence for a nitrate-independent function of the nitrate sensor NRT1.1 in. Journal of Plant Research, 2011, 124(3): 425-430.
[7] Forde B G. Local and long-range signaling pathways regulating plant responses to nitrate. Annual Review of Plant Biology, 2002, 53: 203-224.
[8] Bi Y M, Wang R L, Zhu T, Rothstein S J. Global transcription profiling reveals differential responses to chronic nitrogen stress and putative nitrogen regulatory components in. BMC Genomics, 2007, 8: 281.
[9] 吴翔宇, 许志茹, 曲春浦, 李蔚, 孙琦, 刘关君. 毛果杨基因家族生物信息学分析与鉴定. 植物研究, 2014, 34(1): 37-43, 61.
WU X Y, XU Z R, QU C P, LI W, SUN Q, LIU G J. Genome-wide identification and characterization ofgene family in. Bulletin of Botanical Research, 2014, 34(1): 37-43, 61. (in Chinese)
[10] 曹雄军, 卢晓鹏, 熊江, 李静, 吴倩, 周芳芳, 谢深喜. 枳NLP转录因子克隆及其在不同水分条件下的表达. 中国农业科学, 2016, 49(2): 381-390.
CAO X J, LU X P, XIONG J, LI J, WU Q, ZHOU F F, XIE S X. Cloning and expression of(L.) raf. NIN-like transcription factors under different water conditions. Scientia Agricultura Sinica, 2016, 49(2): 381-390. (in Chinese)
[11] LIU K H, LIU M H, LIN Z W, WANG Z F, CHEN B Q, LIU C, GUO A P, KONISHI M, YANAGISAWA S, WAGNER G, Sheen J. NIN-like protein 7 transcription factor is a plant nitrate sensor. Science, 2022, 377(6613): 1419-1425.
[12] GE M, LIU Y H, JIANG L, WANG Y C, LV Y D, ZHOU L, LIANG S Q, BAO H B, ZHAO H. Genome-wide analysis of maize NLP transcription factor family revealed the roles in nitrogen response.Plant Growth Regulation, 2018, 84(1): 95-105.
[13] SCHAUSER L, ROUSSIS A, STILLER J, STOUGAARD J. A plant regulator controlling development of symbiotic root nodules. Nature, 1999, 402(6758): 191-195.
[14] SCHAUSER L, WIELOCH W, STOUGAARD J. Evolution of NIN-like proteins in, rice, and. Journal of Molecular Evolution, 2005, 60(2): 229-237.
[15] KUMAR A, BATRA R, GAHLAUT V, GAUTAM T, KUMAR S, SHARMA M, TYAGI S, SINGH K P, BALYAN H S, PANDEY R, GUPTA P K. Genome-wide identification and characterization of gene family for RWP-RK transcription factors in wheat (L.). PLoS ONE, 2018, 13(12): e0208409.
[16] LIU M, CHANG W, FAN Y H, SUN W, QU C M, ZHANG K, LIU L Z, XU X F, TANG Z L, LI J N, LU K. Genome-wide identification and characterization of NODULE-INCEPTION-LIKE protein (NLP) family genes in. International Journal of Molecular Sciences, 2018, 19(8): 2270.
[17] LIU M Y, ZHI X N, WANG Y, WANG Y. Genome-wide survey and expression analysis of NIN‑like Protein (NLP) genes reveals its potential roles in the response to nitrate signaling in tomato. BMC Plant Biology, 2021, 21: 347.
[18] CHARDIN C, GIRIN T, ROUDIER F, MEYER C, KRAPP A. The plant RWP-RK transcription factors: key regulators of nitrogen responses and of gametophyte development. Journal of Experimental Botany, 2014, 65(19): 5577-5587.
[19] KONISHI M, YANAGISAWA S. Identification of a nitrate-responsive-element in thepromoter defines the presence of multiple-regulatory elements for nitrogen response. The Plant Journal, 2010, 63(2): 269-282.
[20] Konishi M, Yanagisawa S.NIN-like transcription factors have a central role in nitrate signalling. Nature Communications, 2013, 4: 1617.
[21] SU Y S, LAGARIAS J C. Light-independent phytochrome signaling mediated by dominant GAF domain tyrosine mutants ofphytochromes in transgenic plants. The Plant Cell, 2007, 19(7): 2124-2139.
[22] GAO Z Y, WEN C K, BINDER B M, CHEN Y F, CHANG J H, CHIANG Y H, KERRIS R J, CHANG C R, SCHALLER G E. Heteromeric interactions among ethylene receptors mediate signaling in. Journal of Biological Chemistry, 2008, 283(35): 23801-23810.
[23] GREFEN C, STÄDELE K, RŮZICKA K, OBRDLIK P, HARTER K, HORÁK J. Subcellular localization andinteractions of theethylene receptor family members. Molecular Plant, 2008, 1(2): 308-320.
[24] CASTAINGS L, CAMARGO A, POCHOLLE D, GAUDON V, TEXIER Y, BOUTET-MERCEY S, TACONNAT L, RENOU J P, DANIEL-VEDELE F, FERNANDEZ E, MEYER C, KRAPP A. The nodule inception-like protein 7 modulates nitrate sensing and metabolism in.The Plant Journal, 2009, 57(3): 426-435.
[25] KONISHI M, YANAGISAWA S. The regulatory region controlling the nitrate-responsive expression of a nitrate reductase gene,, in. Plant and Cell Physiology, 2011, 52(5): 824-836.
[26] JIAN W, ZHANG D W, ZHU F, WANG S X, ZHU T, PU X J, ZHENG T, FENG H, LIN H H. Nitrate reductase-dependent nitric oxide production is required for regulation alternative oxidase pathway involved in the resistance to Cucumber mosaic virus infection in. Plant Growth Regulation, 2015, 77(1): 99-107.
[27] MARCHIVE C, ROUDIER F, CASTAINGS L, BRÉHAUT V, BLONDET E, COLOT V, MEYER C, KRAPP A. Nuclear retention of the transcription factor NLP7 orchestrates the early response to nitrate in plants. Nature Communications, 2013, 4:1713.
[28] YU L H, WU J, TANG H, YUAN Y, WANG S M, WANG Y P, ZHU Q S, LI S G, XIANG C B. Overexpression ofimproves plant growth under both nitrogen-limiting and-sufficient conditions by enhancing nitrogen and carbon assimilation. Scientific Reports, 2016, 6: 27795.
[29] YAN D W, EASWARAN V, CHAU V, OKAMOTO M, IERULLO M, KIMURA M, ENDO A, YANO R, PASHA A, GONG Y C, Bi Y M, PROVART N, GUTTMAN D, KRAPP A, ROTHSTEIN S J, NAMBARA E. NIN-like protein 8 is a master regulator of nitrate- promoted seed germination in. Nature Communications, 2016, 7: 13179.
[30] GUAN P Z, RIPOLL J J, WANG R H, VUONG L, BAILEY- STEINITZ L J, Ye D N, Crawford N M. Interacting TCP and NLP transcription factors control plant responses to nitrate availability. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(9): 2419-2424.
[31] LIU K H, NIU Y J, KONISHI M, WU Y, DU H, SUN CHUNG H, LI L, BOUDSOCQ M, MCCORMACK M, MAEKAWA S, ISHIDA T, ZHANG C, SHOKAT K, YANAGISAWA S, SHEEN J. Discovery of nitrate-CPK-NLP signalling in central nutrient-growth networks. Nature, 2017, 545(7654): 311-316.
[32] ALFATIH A, WU J, ZHANG Z S, XIA J Q, JAN S U, YU L H, XIANG C B. Rice NIN-LikeProtein 1 rapidly responds to nitrogen deficiency and improves yield and nitrogen use efficiency. Journal of Experimental Botany, 2020, 71(19): 6032-6042.
[33] WU J, ZHANG Z S, Xia J Q, ALFATIH A, SONG Y, HUANG Y J, WAN G Y, Sun L Q, TANG H, LIU Y, WANG S M, ZHU Q S, QIN P, WANG Y P, LI S G, MAO C Z, ZHANG G Q, CHU C C, YU L H, XIANG C B. Rice NIN-LIKE PROTEIN 4 plays a pivotal role in nitrogen use efficiency. Plant Biotechnology Journal, 2021, 19(3): 448-461.
[34] ENDRIZZI J E, TURCOTTE E L, KOHEL R J. Genetics, cytology, and evolution of. Advances in Genetics, 1985, 23: 271-375.
[35] PATERSON A H, WENDEL J F, GUNDLACH H, GUO H, JENKINS J, JIN D C, LLEWELLYN D, SHOWMAKER K C, SHU S Q, UDALL J, YOO M J, BYERS R, CHEN W, DORON-FAIGENBOIM A, DUKE M V, GONG L, GRIMWOOD J, GROVER C, GRUPP K, HU G J, Lee T H, LI J P, LIN L F, LIU T, MARLER B S, PAGE J T, ROBERTS A W, ROMANEL E, SANDERS W S, SZADKOWSKI E, TAN X, TANG H B, XU C M, WANG J P, WANG Z N, ZHANG D, ZHANG L, ASHRAFI H, BEDON F, BOWERS J E, BRUBAKER C L, CHEE P W, DAS S, GINGLE A R, HAIGLER C H, HARKER D, HOFFMANN L V, HOVAV R, JONES D C, LEMKE C, MANSOOR S, RAHMAN M U, RAINVILLE L N, RAMBANI A, REDDY U K, RONG J K, SARANGA Y, SCHEFFLER B E, SCHEFFLER J A, STELLY D M, TRIPLETT B A, VAN DEYNZE A V, VASLIN M F S, WAGHMARE V N, WALFORD S A, WRIGHT R J, ZAKI E A, ZHANG T Z, DENNIS E S, MAYER K F X, PETERSON D G, ROKHSAR D S, WANG X Y, SCHMUTZ J. Repeated polyploidization ofgenomes and the evolution of spinnable cotton fibres. Nature, 2012, 492(7429): 423-427.
[36] DU X M, HUANG G, HE S P, YANG Z E, SUN G F, MA X F, LI N, ZHANG X Y, SUN J L, LIU M, JIA Y H, PAN Z E, GONG W F, LIU Z H, ZHU H Q, MA L, LIU F Y, YANG D G, WANG F, FAN W, GONG Q, PENG Z, WANG L R, WANG X Y, XU S J, SHANG H H, LU C R, ZHENG H K, HUANG S W, LIN T, ZHU Y X, LI F G. Resequencing of 243 diploid cotton accessions based on an updated a genome identifies the genetic basis of key agronomic traits. Nature Genetics, 2018, 50(6): 796-802.
[37] LI F G, FAN G Y, LU C R, XIAO G H, ZOU C S, KOHEL R J, MA Z Y, SHANG H H, Ma X F, Wu J Y, LIANG X M, HUANG G, PERCY R G, LIU K, YANG W H, CHEN W B, DU X M, SHI C C, YUAN Y L, YE W W, LIU X, ZHANG X Y, LIU W Q, WEI H L, Wei S J, HUANG G D, ZHANG X L, ZHU S J, ZHANG H, SUN F M, WANG X F, LIANG J, WANG J H, HE Q, HUANG L H, WANG J, CUI J J, SONG G L, WANG K B, XU X, YU J Z, ZHU Y X, YU S X. Genome sequence of cultivated upland cotton (TM-1) provides insights into genome evolution. Nature Biotechnology, 2015, 33(5): 524-530.
[38] WANG M J, TU L L, YUAN D J, ZHU D, SHEN C, LI J Y, LIU F Y, PEI L L, WANG P C, ZHAO G N, YE Z X, HUANG H, YAN F L, MA Y Z, ZHANG L, LIU M, YOU J Q, YANG Y C, LIU Z P, HUANG F, LI B Q, QIU P, ZHANG Q H, ZHU L F, Jin S X, YANG X Y, MIN L, LI G L, CHEN L L, ZHENG H K, LINDSEY K, LIN Z X , UDALL J A, ZHANG X L. Reference genome sequences of two cultivated allotetraploid cottons,and. Nature Genetics, 2019, 51(2): 224-229.
[39] HU Y, CHEN J D, FANG L, ZHANG Z Y, MA W, NIU Y C, JU L Z, DENG J Q, ZHAO T, LIAN J M, BARUCH K, FANG D, LIU X, RUAN Y L, RAHMAN M U, HAN J L, WANG K, WANG Q, WU H T, MEI G F, ZANG Y H, HAN Z G, XU C Y, SHEN W J, YANG D F, SI Z F, DAI F, ZOU L F, HUANG F, BAI Y L, ZHANG Y G, BRODT A, HAMO H B, ZHU X F, ZHOU B L, GUAN X Y, ZHU S J, CHEN X Y, ZHANG T Z.andgenomes provide insights into the origin and evolution of allotetraploid cotton. Nature Genetics, 2019, 51(4): 739-748.
[40] YANG Z Q, WANG J, HUANG Y M, WANG S B, Wei L L, LIU D X, WENG Y L, XIANG J H, ZHU Q, YANG Z E, NIE X H, YU Y, YANG Z R, YANG Q Y. CottonMD: a multi-omics database for cotton biological study. Nucleic Acids Research, 2022, gkac863: 1-11.
[41] WANG D H, FAN W L, GUO X L, WU K, ZHOU S Y, CHEN Z G, LI D Y, WANG K, ZHU Y X, ZHOU Y. MaGenDB: a functional genomics hub for Malvaceae plants. Nucleic Acids Research, 2020, 48(D1): D1076-D1084.
[42] FINN R D, BATEMAN A, CLEMENTS J, COGGILL P, EBERHARDT R Y, EDDY S R, HEGER A, HETHERINGTON K, HOLM L, MISTRY J, SONNHAMMER E L L, TATE J, PUNTA M. Pfam: The protein families database. Nucleic Acids Research, 2014, 42(D1): D222-D230.
[43] HORTON P, PARK K J, OBAYASHI T, FUJITA N, HARADA H, ADAMS-COLLIER C J, NAKAI K T. WoLF PSORT: Protein localization predictor. Nucleic Acids Research, 2007, 35(suppl_2): W585-W587.
[44] KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[45] CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecualr Plant, 2020, 13(8): 1194-1202.
[46] BAILEY T L, WILLIAMS N, MISLEH C, LI W W. MEME: Discovering and analyzing DNA and protein sequence motifs. Nucleic Acids Research, 2006, 34(suppl_2): W369-W373.
[47] HU B, JIN J P, GUO A Y, ZHANG H, LUO J C, GAO G. GSDS 2.0: An upgraded gene feature visualization server. Bioinformatics, 2014, 31(8): 1296-1297.
[48] WANG D P, ZHANG Y B, ZHANG Z, ZHU J, YU J. KaKs_ Calculator 2.0: a toolkit incorporating gamma-series methods and sliding window strategies.Genomics, Proteomics & Bioinformatics, 2010, 8(1): 77-80.
[49] MÜLLER M, KNUDSEN S. The nitrogen response of a barley C-hordein promoter is controlled by positive and negative regulation of the GCN4 and endosperm box. The Plant Journal, 1993, 4(2): 343-355.
[50] 袁婷婷, 朱成磊, 李紫阳, 宋新章, 高志民. 毛竹NLP转录因子鉴定及其响应氮素的表达模式. 林业科学研究, 2021, 34(5): 39-48.
YUAN T T, ZHU C L, LI Z Y, SONG X Z, GAO Z M. Identification of NLP transcription factors ofand their expression patterns in response to nitrogen. Forest Research, 2021, 34(5): 39-48. (in Chinese)
[51] CHALHOUB B, DENOEUDF, LIU S Y, PARKIN I A P, TANG H B, WANG X Y, CHIQUET J, BELCRAM H, TONG C B, SAMANS B,. Early allopolyploid evolution in the post-Neolithicoilseed genome. Science, 2014, 345: 950-953.
[52] HUANG G, WU Z G, PERCY R G, BAI M Z, LI Y, FRELICHOWSKI J E, HU J, WANG K, YU J Z, ZHU Y X. Genome sequence ofand genome updates ofandprovide insights into cotton A-genome evolution.Nature Genetics, 2020, 52(5): 516-524.
[53] LYNCH M, CONERY J S. The evolutionary fate and consequences of duplicate genes. Science, 2000, 290(5494): 1151-1155.
[54] SUMIMOTO H, KAMAKURA S, Ito T. Structure and function of the PB1 domain, a protein interaction module conserved in animals, fungi, amoebas, and plants. Sciemce Stke, 2007, 2077(401): re6.
[55] ZHAO W Q, DONG H R, ZAHOORA R, ZHOU Z G, SNIDER J L, CHEN Y L, SIDDIQUE K H M, WANG Y H. Ameliorative effects of potassium on drought-induced decreases in fiber length of cotton (L.) are associated with osmolyte dynamics during fiber development. The Crop Journal, 2019, 7(5): 619-634.
[56] SONG Z Q, CHEN Y, ZHANG C Y, ZHANG J X, Huo X H, GAO Y, PAN A, DU Z H, ZHOU J, ZHAO Y X, LIU Z, WANG F R, ZHANG J. RNA-seq reveals hormone-regulated synthesis of non-cellulose polysaccharides associated with fiber strength in a single- chromosomal-fragment-substituted upland cotton line. The Crop Journal, 2020, 8(2): 273-286.
[57] 王寻, 陈西霞, 李宏亮, 张富军, 赵先炎, 韩月彭, 王小非, 郝玉金. 苹果NLP(Nin-Like Protein)转录因子基因家族全基因组鉴定及表达模式分析. 中国农业科学, 2019, 52(23): 4333-4349.
WANG X, CHEN X X, LI H L, ZHANG F J, ZHAO X Y, HAN Y P, WANG X F, HAO Y J. Genome-wide identification and expression pattern analysis of NLP (Nin-Like Protein) transcription factor gene family in apple. Scientia Agricultura Sinica, 2019, 52(23): 4333-4349. (in Chinese)
Genome-Wide Identification and Expression Analysis of NLP (Nin- Like Protein) Transcription Factor Gene Family in cotton
DING GuoHua, XIAO GuangHui, ZHU LiPing
College of Life Sciences, Shaanxi Normal University, Xi’an 710119
【Objective】To explore the structure and evolution characteristics of cotton NLP transcription factors in the whole genome, and further understand their expressions patterns, so as to lay a foundation for the further function research and utilization of NLP genes. 【Method】The NLP transcription factor family members in the whole genomes of four cotton species,(, Ga),(, Gr),(, Gb) and(, GH), were identified using two strategies, BLASTP and HMM search. Further bioinformatics analysis was carried out on the confirmed cotton NLP family members. The molecular weights, theoretical isoelectric points and other physical and chemical properties were predicted using online software Expasy; the MEGA 7 software was used to build the phylogenetic tree; protein conservative motifs were analyzed through MEME website; online software GSDS 2.0 was used to analyze gene structures; TBtools was used to view the chromosome localizations; McscanX was used to analyze the replication genes of cotton NLP family members; the PlantCARE website was used to predict the-acting elements in the promoters of cotton NLP family genes. The heat maps of cotton NLP genes expression levels of different tissues and under abiotic stresses were drawn through TBtools to analyze the tissue expression characteristics and abiotic stresses response characteristics. The expressions ofin cotton under nitrogen starvation and nitrogen resupply treatments were analyzed by RT-qPCR. 【Result】A total of 11, 11, 21 and 22 NLP members were screened from the four cotton protein databases of,,and, respectively. These NLP family genes encoded 693-996 amino acids. The relative molecular masses ranged from 76.92-110.02 kDa and the theoretical isoelectric points were 5.13-7.77. The subcellular localization prediction results showed that almost all the NLP members located in the nucleus. Promoter analysis found a large number of-acting elements related to phytohormone and stress response. Phylogenetic analysis showed cotton NLPs were divided into three groups, I, II and III. Gene replication analysis showed that fragment replication was the main force formembers expansion in cotton. All the Ka/Ks values were less than 1, indicating that evolution of NLP family in cotton mainly underwent purification selection. The results of expression analysis also confirmed thatresponded to nitrogen starvation and nitrogen resupply. 【Conclusion】From the whole genome of,,, and, 11, 11, 21 and 22 NLP transcription factor members were identified respectively. They had high conservatism and some degree of differences. The expression levels ofchanged significantly during nitrogen starvation and nitrogen resupply processes, which may play a role in the response of cotton to nitrate.
cotton; NLP transcription factor; nitrogen signal; bioinformatics
10.3864/j.issn.0578-1752.2023.19.003
2022-11-07;
2023-01-18
国家自然科学基金(32070549、32270578和32200444)、中国博士后科学基金(2022M712005)、陕西省自然科学基础研究计划(2022JQ-197)、安徽省自然科学基础研究计划(208085QC85)、中央高校基本科研业务费专项资金(GK202002005)、中央高校基本科研业务费(GK202304018,GK202304016,GK202304015)、棉花生物学国家重点实验室开放课题(CB2022A01、CB2021A05和CB2021A21)
丁国华,E-mail:dingguohuasw@163.com。通信作者竺丽萍,E-mail:zhuliping0903@163.com。通信作者肖光辉,E-mail:guanghuix@snnu.edu.cn
(责任编辑 李莉)
