球等鞭金藻MYB基因家族成员全基因组鉴定和特性分析
2023-07-13韩玉莹陈璐瑶陈由强陈多陈建楠
韩玉莹 陈璐瑶 陈由强 陈多 陈建楠
摘 要:为了深入理解球等鞭金藻MYB基因家族在基因调控网络中的作用机制,使用MYB_DNA-binding保守结构域在球等鞭金藻蛋白序列中筛选出60个MYB基因家族成员,并对这些基因家族成员的蛋白(一、二、三级)结构预测和分析、亚细胞结构定位、基因结构及保守结构域等方面进行了分析。结果表明:这些MYB基因家族成员可分为3个cluster,其中R2-MYB和R3-MYB主要集中在cluster 3中,R2R3-MYB则含有大部分家族成员,表明这些基因在球等鞭金藻中起着重要的生物学功能。不同分支的亲缘关系较远,同一分支的關系较近,这表明球等鞭金藻MYB基因家族在演化过程中形成了一些适应性的变化。通过基因组内共线性分析,研究发现球等鞭金藻全基因组内有177对片段复制基因和46对串联复制基因,其中MYB基因家族有2对复制基因,包括串联重复基因对IZ004267和IZ007345以及片段重复基因对IZ006462和IZ006102。结合染色体定位分析,IZ006462和IZ006102位于9号染色体,这也支持上述片段重复的判断。这些重复基因对的Ka/Ks值小于1,表明这些基因在扩张过程中受到了基因纯化选择,功能相当保守。
关键词:球等鞭金藻;MYB基因家族;全基因组;光质;表达分析
中图分类号:Q 943.2 文献标志码:A 文章编号:0253-2301(2023)04-0022-14
DOI: 10.13651/j.cnki.fjnykj.2023.04.004
Genome-wide Identification and Characteristic Analysis ofMYB Gene Family Members in Isochrysis galbana
HAN Yu-ying1,2,3, CHEN Lu-yao1,3, CHEN You-qiang1,2,3, CHEN Duo1,2,3, CHEN Jian-nan1,2,3*
(1. Public Service Platform for Industrialization Development Technology of Marine Biological Medicine and Products,
Fuzhou, Fujian 350117, China; 2. Fujian Key Laboratory of Special Marine Bioresource Sustainable Utilization, Fuzhou,
Fujian 350117, China; 3. College of Life Science, Fujian Normal University, Fuzhou, Fujian 350117, China)
Abstract: In order to further understand the mechanism of the MYB gene family in the gene regulatory network, the MYB_DNA-binding conserved domain was used to screen 60 MYB gene family members from the protein sequences of Isochrysis galbana. Then, the protein (primary, secondary and tertiary) structure prediction and analysis, subcellular structure localization, gene structure, and conserved domains of these gene family members were analyzed. The results showed that: these MYB gene family members could be divided into three clusters, among which R2-MYB and R3-MYB were mainly concentrated in cluster 3, and R2R3-MYB contained most of the family members, indicating that these genes played an important biological function in Isochrysis galbana. The distant relationship between different branches and the close relationship within the same branch indicated that the MYB gene family in Isochrysis galbana had undergone some adaptive changes during the evolution process. Through the analysis of collinearity in the genome, the study found that there were 177 pairs of segmental duplication genes and 46 pairs of tandem duplication genes in the whole genome of Isochrysis galbana, among which there were 2 pairs of duplication genes in the MYB gene family, including the tandem duplicated gene pair IZ004267 and IZ007345, and the segmental duplicated gene pair IZ006462 and IZ006102. By combining with the analysis of chromosomal localization, IZ006462 and IZ006102 were located on chromosome 9, which also supported the above judgment of segmental duplication. The Ka/Ks values of these duplicated gene pairs were less than 1, indicating that these genes were subjected to gene purification selection during expansion and their functions were quite conserved.
Key words: Isochrysis galbana;MYB gene family; Whole genome; Light quality; Expression analysis
球等鞭金藻Isochrysis galbana是一种单细胞海藻,大小约5 μm,呈金褐色,通常呈椭圆形或球形,有两条等长光滑的鞭毛,在水体中能缓慢转动或旋涡状运动[1]。球等鞭金藻具有完整的生物代谢通路和细胞器,具有较高的研究价值。作为一种模式生物,球等鞭金藻在光合作用、细胞周期调控、信号转导、膜转运、细胞骨架和药物发现等领域被广泛研究。球等鞭金藻富含多种不饱和脂肪酸,如岩藻黄素和DHA,是一种珍贵的藻类资源[2-5]。MYB基因家族是一类重要的转录因子家族,广泛参与植物生长发育、代谢物合成、激素信号传导等生理过程。MYB转录因子在植物中数量和功能最多[6-7]。对于该转录因子在类胡萝卜素合成过程中调控作用的研究也比较多。王拓璋等[8]通过对鳄梨全基因组的MYB转录因子基因序列分析,得到92个PaMYB基因序列,转录组表达分析显示有68个PaMYB基因在干旱胁迫下表达,72个PaMYB基因在低温胁迫下表达。为深入研究PaMYB转录因子非生物胁迫提供科学理论依据。阙秋霞等[9]从刺葡萄愈伤组织转录组数据鉴定并克隆出9个VdMYB家族基因,并利用生物信息学方法分析和预测其结构和功能,同时分析其在不同光质诱导下的表达模式,9个VdMYB启动子序列中均有大量光响应等多种生物/非生物响应元件。qRT-PCR分析表明,光质显著影响VdMYBs基因的表达,不同光质作用下,VdMYBs基因响应模式与结构特征相对应;VdMYB31/VdMYBB1和VdMYB4A基因分别作为正负调控因子参与花青素生物合成。本研究为进一步解析MYB基因通过响应光信号参与调控刺葡萄愈伤组织花青素生物合成的分子机制提供理论参考。
本课题组前期在球等鞭金藻研究中,已经发现了一些MYB基因的存在,MYB基因在不同光质培养下的表达模式也存在差异,蓝光抑制球等鞭金藻岩藻黄素含量的积累,不利于脂肪酸积累,转录组学分析揭示了R2-MYB转录因子IZ014151可能是球等鞭金藻光合色素岩藻黄素合成的关键调控基因。本研究对球等鞭金藻MYB基因家族进行了全面鉴定,包括基因家族成员鉴定和蛋白质理化特性分析、亚细胞结构定位分析、二级结构和三级结构预测分析、基因家族结构和保守结构域分析、基因家族特征motif分析、染色体定位分析、系统发育进化树构建、共线性分析及基因复制对中的Ka/Ks分析和基因家族启动子分析,并揭示了其在不同光质下的表达模式和进化特征,可为后续深入研究球等鞭金藻中MYB基因家族的功能打下基础。
1 材料与方法
1.1 试验材料
球等鞭金藻Isochrysis galbana藻株为LG007,系本课题组林岗教授分离,保藏于本实验室。将球等鞭金藻以1×106个·mL-1密度接种于锥形瓶内,均放置于白光培养箱培养。所用培养基为f/2培养基。培养条件:光照强度70 μmol·m-2·s-1,温度(22±1)℃,光照周期24 h,每天定时摇瓶3次。
1.2 基因家族成员鉴定和蛋白质理化特性分析
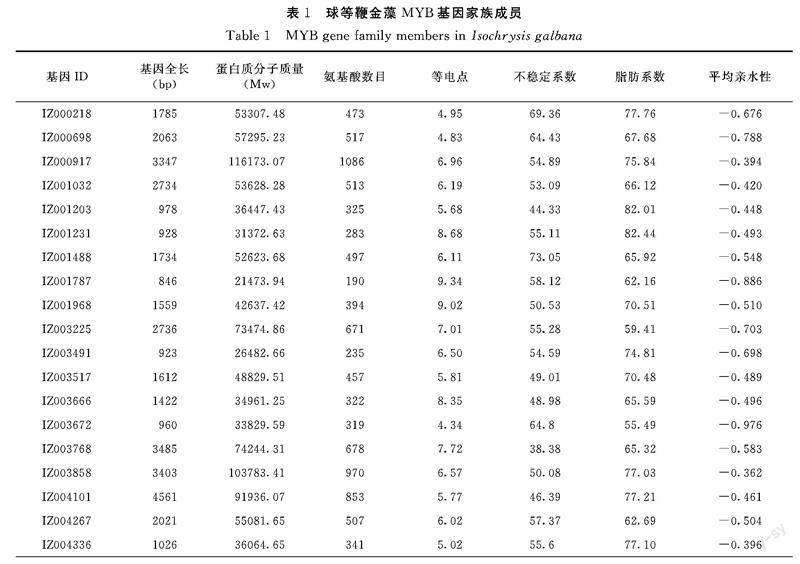
从Pfam(http://pfam.xfam.org)数据库中下载MYB_DNA-binding的隐马尔科夫模型的HMM文件,Pfam编号PF00249。在HMMER 3.0软件通过hummerseach命令(hmmsearch.exe./ MYB_DNA-binding.hmm./ IZ.pep>IZ-MYB.txt)提取出含有MYB保守结构域的蛋白序列ID(E-value为1e-05),利用TBtools软件和球等鞭金藻蛋白fasta文件提取候选ID的序列。通过Batch CD-Searc(CDD)(Batch CD-Search:https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi),将结构域不完整以及重复冗长的序列删除,剩余的即为基因家族成员。将鉴定的基因家族成员的蛋白序列上传至ProtParam(ProtParam:https://web.expasy.org/protparam),分析其等电点、分子量等理化性质,从基因组的gff文件中提取MYB家族成员的全长和CDS长度信息。
1.3 亚细胞结构定位分析
1.3.1 亚细胞结构定位分析 利用Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi)、ProtComp 9.0(http://linux1.softberry.com/all.htm)和WolfPSORT(https://wolfpsort.hgc.jp)在線软件分别对基因家族成员蛋白进行亚细胞结构定位预测。综合3种预测结果,评估预测结果。
1.3.2 跨膜结构域预测 利用在线软件TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM)进行跨膜结构域预测。
1.3.3 信号肽预测 利用在线软件SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP-4.1)对基因家族蛋白成员进行信号肽预测,输出结果mean D显示“YES”则表示有信号肽,“NO”表示没有信号肽。
1.4 二级结构和三级结构预测分析
通利用在线软件GOR IV(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)预测基因家族成员的二级结构。
利用SWISS-MODEL(https://www.swissmodel.expasy.org)在线软件预测蛋白的三级结构。利用GMQE值和QMEAN值进行评估,GMQE值为0~1,QMEAN值为-4~0,其中GMQE值越大表示模型越可靠,QMEAN值越大表示预测蛋白与模板蛋白的匹配度越高。
1.5 基因家族结构和保守结构域分析
首先利用Batch WB CD search tool(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)在线工具将最终确定的基因家族进行功能结构域分析,将分析结果下载后在Excel中打开,只保留Query、From、To、short name这四列,将这四列的信息复制到TBtools软件中,并将gff3文件也拖入该软件中,绘制基因结构图。将基因家族成员的蛋白序列上传至Pfam数据库,分析其保守结构域,保存结果,然后利用TBtools的Visualize Domain Pattern程序对Pfam的分析结果进行可视化分析。
1.6 基因家族特征motif分析
通过在线工具MEME(http://meme-suite.org)分析保守的蛋白结构域,将已确定的蛋白序列上传到MEME中,将得到的mast.xml文件结果保存,利用蛋白ID和mast.xml文件在TBtools软件中绘制并优化图片。
1.7 染色体定位分析
用TBtools在gff3文件中提取出球等鞭金藻的基因所在染色体的位置文件以及染色体的长度文件,在这两个文件中提取出基因家族的信息和基因所在染色体的长度。之后用MapGene2Chromosome v2(http://mg2c.iask.in/mg2c_v2.0)在线工具进行基因染色体定位的绘制和编辑。
1.8 系统发育进化树构建
从NCBI数据库中下载莱茵衣藻(Chlamydomonasreinhardtii)和三角褐指藻(Phaeodactylumtricornutum)蛋白质序列fasta文件,用ClustalW软件对Phaeodactylumtricornutum、Chlamydomonasreinhardtii以及球等鞭金藻基因家族成员蛋白序列进行氨基酸序列比对,分别构建球等鞭金藻、莱茵衣藻和三角褐指藻的MYB家族的系统发育进化树。利用iTOL在线软件进行进化树优化。
1.9 共线性分析及基因复制对中的Ka/Ks分析
首先利用MCScanX(http://chibba.pgml.uga.edu/mcscan2)软件进行基因组内共线性分析,利用TBtools软件对共线性分析结果进行可视化。利用gff文件和blast文件(基因组内自身blast文件),运行MCScanX软件得到collinearity 共线性文件和tandem串联基因对文件。
TBtools软件可视化步骤如下:先将collinearity文件转换成基因对txt文件(用Text Merge for MCScanX功能),然后利用TBtools软件从gff文件中提取染色体长度和基因位置的信息,整理成txt文件,在Text Transformat for Micro-Synteny View功能中输入染色体长度和位置的txt文件,得到基因对inks文件。最后在Advanced Circos程序中输入染色体长度txt文件和基因对inks文件进行可视化分析。
利用TBtools程序中的Simple Ka/Ks Calculator9(NG)计算重复基因对的Ka/Ks(非同义替换率与同义替换率的比值)。当Ka/Ks>> 1,基因受正向选择(Positive selection);当Ka/Ks=1,基因中性进化(Neutral evolution);当Ka/Ks<< 1,基因受纯化选择(Purify selection)。
1.10 基因家族启动子分析
利用TBtools程序中的Gtf/Gff3 Sequences Extractor,提取球等鞭金藻CDS上游2000 bp的fasta文件,然后再利用启动子序列在Quick Fasta Extractor or Filter 中提取目标基因的启动子序列,检查文件信息是否正确(Fasta Stater),最后将其序列全部转化成大写,提交到PlantCARE(http: //bioinformatics. psb. ugent.be/webtools/plantcare/html)网站进行顺势作用元件预测。将得到的结果进行整理和简化,上传至TBtools进行启动子可视化分析。
2 结果与分析
2.1 MYB基因家族成员鉴定及蛋白特性分析
由表1可知,这60个MYB基因序列长度差异较大,长度在752~4561 bp;蛋白质相对分子质量在17517.93~116173.07 Da;氨基酸数目差异较大,最少的有155个氨基酸,最多的有1463个;等电点在4.34~10.18,其中只有少数为碱性蛋白;不稳定系数在22.02~77.91(>40时认为是不稳定蛋白),脂肪系数在52.48~84.96,平均亲水性均小于0,所以该家族成员多为不稳定亲水性蛋白。以上结果表明,同一家族成员的理化特性可能存在较大的差异,因此,它们的结构功能也会有所不同。
2.2 MYB基因家族亚细胞结构定位
利用3种在线软件对球等鞭金藻MYB蛋白进行亚细胞定位预测分析。Cell-PLoc的预测结果显示,有59个MYB蛋白定位于细胞核,只有1个蛋白(IZ003768)定位于细胞膜。ProtComp9.0的预测结果显示44个MYB蛋白定位于细胞核。WolfPSORT的预测结果显示有56个MYB蛋白定位于细胞核,其中IZ001203蛋白定位于液泡,IZ007734和IZ008056蛋白定位于线粒体,IZ010753蛋白定位于叶绿体。在WolfPSORT和ProtComp 9.0软件均预测IZ008056蛋白定位于线粒体。对于IZ001203、IZ007734、IZ010753、IZ008056蛋白,3个软件预测结果不一致,还有待进一步确定。由表2可知,3个软件预测均定位于细胞核的蛋白有34个(IZ000218、IZ001231、IZ001488、IZ001787、IZ001968、IZ003225、IZ003491、IZ003666、IZ003672、IZ004101、IZ004267、IZ004469、IZ004656、IZ004905、IZ005555、IZ005588、IZ006315、IZ007092、IZ007542、IZ007617、IZ010306、IZ010353、IZ010554、IZ010827、IZ011696、IZ011780、IZ011921、IZ012323、IZ012697、IZ012811、IZ013719、IZ014349、IZ014568、IZ014686),故推測这些蛋白有着相似的结构和表型理化特征。
对目的蛋白进行跨膜结构域预测,结果表明除了IZ001203、IZ003517蛋白(图1)外,其他均无跨膜结构域,说明他们都不属于跨膜类蛋白,因此也不会产生跨膜运动。而IZ001203蛋白在前端约25个氨基酸残基的位置,IZ003517蛋白在末端约25个氨基酸残基的位置有跨膜结构域,故推测IZ001203和IZ003517蛋白具跨膜功能。对目的蛋白进行信号肽预测,结果表明60个蛋白中只有IZ001203蛋白(图2)有信号肽。结合蛋白跨膜结构域预测结果,IZ001203蛋白具有跨膜功能,与信号肽预测结果相符。综上,IZ001203属稳定亲水性蛋白,有信号肽而且可以进行跨膜运动。
2.3 MYB基因家族二级结构、三级结构分析
由表3可知,对获得的60个MYB基因编码的蛋白进行二级结构预测,这60个蛋白均含有α-螺旋,所占比例在18.61%~58.41%;无规则卷曲是最主要的结构形式,所占比例高达35.2%~68.61%;延伸链结构所占比例最少,在2.27%~22.93%。
由图3可知,对球等鞭金藻MYB转录因子进行三级结构分析预测,可知每个MYB_DNA-binding结构域都有3个α-螺旋,这3个螺旋之间互相形成一定的角度,且第2个与第3螺旋之间形成1个β-转角,与二级结构预测结果基本相符。
2.4 MYB基因家族结构和保守结构域分析
根据MYB_DNA-binding结构域在每个MYB蛋白上重复出现次数,将60个MYB家族成员按照R1-MYB、R2-MYB(R2R3-MYB)、R3-MYB、R4-MYB这4种类型进行归类,详细信息见表4。结果表明球等鞭金藻R1-MYB有19个,R2-MYB有22个,R3-MYB有6个,R4-MYB有3个。
由图4可知,60个球等鞭金藻MYB基因家族成员的结构存在较大差异,其中有21个基因无内含子,其他基因内含子数目为1~10个,编码区数量为1~10个。综合来看,MYB基因家族成员中IZ004101基因长度最长,为4561 bp,且内含子和编码区数量最多。保守结构域大多数分布在基因的5′端,且保守结构域的长度相似。
此外,还发现有10个MYB成员有MYB_DNA-bind_6结构域(IZ00218、IZ012697、IZ012811、IZ001231、IZ003225、IZ004469、IZ007941、IZ011921、IZ003491、IZ004905)。MYB_DNA-binding和MYB_DNA-bind_6家族均包含来自MYB蛋白的DNA结合结构域,所以推测MYB_DNA-binding、MYB_DNA-bind_6的保守结构域相似。
2.5 MYB基因家族特征motif分析
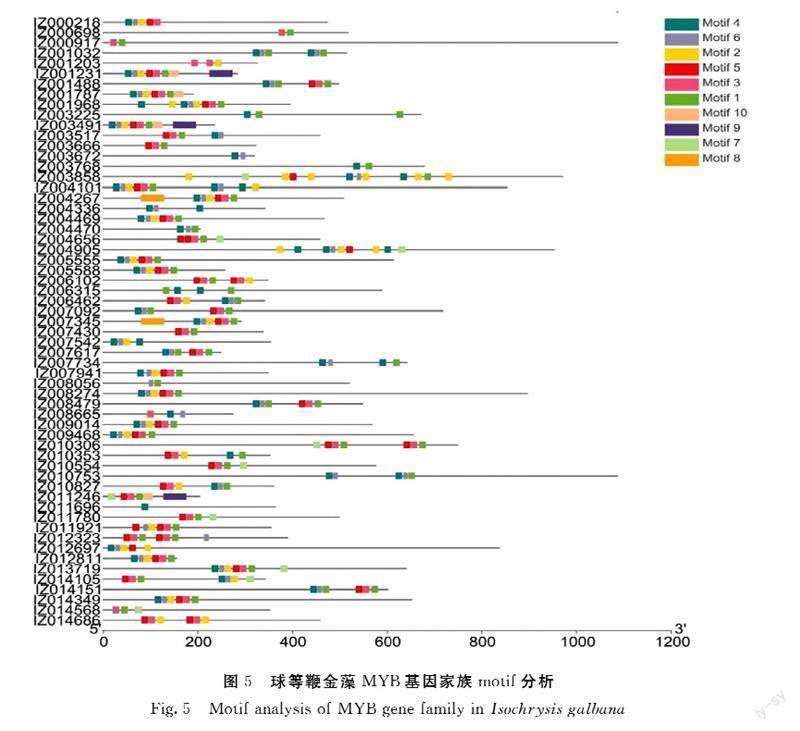
由图5可知,MYB基因家族motif分析,该基因家族共有10个motif,motif 4、6、2多位于MYB蛋白的5′端,motif 1位于MYB功能结构域中间位置,motif 7位于MYB功能结构域3′端。Motif数量最少的仅有1个,最多有12个。大部分球等鞭金藻MYB蛋白均不含motif 9和motif 8,只有IZ001231、IZ011246、IZ003491含有motif 9,IZ004267、IZ007345含有motif 8。
2.6 MYB基因家族染色體定位
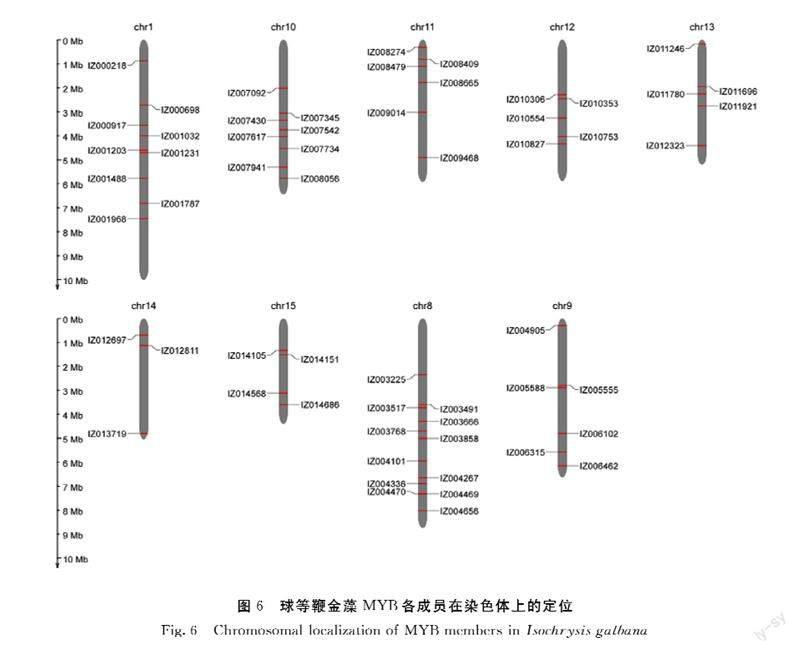
由图6可知,球等鞭金藻MYB基因家族各成员分布在9条染色体上,每条染色体上基因数目存在差异,数量在3~12个,其中8号染色体上数量最多,共有12个基因,且各基因在染色体上的距离较近,其中IZ003517和IZ003491基因在染色体上的位置相近。其次是1号染色体,包含9个基因,其中IZ001203和IZ001231这两个基因位置相近。12号和9号染色体上,IZ010306和IZ010353、IZ005588和IZ005555基因位置也相邻。从11号、12号、8号染色体中可以看到,在染色体上的某个区域家族成员比较集中。IZ004469和IZ004470基因长度分别为1404 bp、1469 bp,且均定位于细胞核。结合染色体定位分析发现IZ004469和IZ004470这两个基因位置紧密相连,形成明显的基因簇。
2.7 MYB基因家族系统发育进化树
由图7可知,球等鞭金藻、莱茵衣藻和三角褐指藻的MYB基因家族系统发育进化树划分为3个cluster,cluster 1包含17个成员,8个为球等鞭金藻的MYB家族成员;cluster 2仅包含5个球等鞭金藻的MYB家族成员;cluster 3包含成员最多,83个成员中包含47球等鞭金藻的MYB家族成员。根据进化树分析可知,4R-MYB主要在cluster 2中,包括IZ001032和IZ001488。此外,第9 d白光组与蓝光组的差异表达基因IZ014151、IZ006462(R2-MYB转录因子)也被划分到cluster 2中。R1-MYB 在cluster1中有分布,但主要和R2-MYB、R3-MYB集中在cluster 3中。
2.8 基因组内共线性分析及基因复制对分析
由图8可知,为球等鞭金藻基因组内共线性分析,发掘MYB家族的基因复制事件。黄色的一圈每个模块表示球等鞭金藻基因组的染色体,红色的一圈代表染色体相对应的基因密度。共线性基因(片段复制基因)用线条连接,灰色连线为球等鞭金藻基因中全部片段复制基因对的连线,最外圈标注为MYB基因家族的片段复制基因ID,其共线性关系用红色连线展示。通过共线性分析发现,球等鞭金藻全基因组内有177对片段复制基因和46对串联复制基因。其中MYB有2对复制基因,表明MYB基因家族的扩张主要来自串联复制和片段复制。
由表5可知,显示这些重复基因Ka/Ks<1,表明MYB基因家族扩张过程中,基因复制对受到基因纯化选择,所以它们的功能也相当保守。
3 结论与讨论
MYB_DNA-binding保守结构域是一种广泛存在于不同生物界中的转录因子保守结构域,通常由3个串联的结构域组成:R1-MYB、R2-MYB和R3-MYB。这3个结构域中的R2-MYB结构域通常是DNA结合的主要部位,包含了一个双顶花环结构,该结构通过与DNA中的核苷酸相互作用,实现MYB蛋白与DNA序列的结合[10]。MYB_DNA-binding保守结构域在植物、动物和真菌中均有发现,它对于调控生长发育、代谢物合成以及激素信号传导等生理过程具有重要作用[11]。本研究利用Myb_DNA-binding保守结构域在球等鞭金藻蛋白序列、利用Pfam数据库以及hummer程序中筛选鉴定到60个MYB基因家族成员。对这些基因家族成员蛋白一、二、三级结构预测及分析、亚细胞结构定位、基因结构及保守结构域等方面进行分析。经分析球等鞭金藻MYB家族基因序列长度差异较大,在752~4561 bp,蛋白质相对分子质量在17517.93~116173.07 Da,氨基酸數目差异较大,155~1463个,多为不稳定亲水性蛋白,其中只有IZ001203和IZ003517蛋白为跨膜类蛋白;分布在9条染色体上,IZ004469和IZ004470这两个基因在8号染色体上紧密相连,形成明显的基因簇。
球等鞭金藻MYB基因家族可以划分为3个cluster,其中R2-MYB和R3-MYB主要集中在cluster 3中,不同分支的亲缘关系较远,同一分支的关系较近,可以推测,球等鞭金藻MYB基因家族在不断的演化过程中形成一些适应性的变化。
基因共线性是指在具有同源性关系的2个物种中,其基因组中有共同的连锁基因,且同源基因的相对顺序具有较高保守性的现象[12]。在生物进化中,基因组会在全基因组复制、染色体重组、染色体倒位和易位等过程中发生结构和数量的变化[13-14]。因此,基因组的共线性分析在非编码序列的确认、新测序物种的注释和全基因组复制事件的估计等过程中具有重要作用[15-16]。
通过基因组内共线性分析,发现球等鞭金藻全基因组内有177对片段复制基因和46对串联复制基因。其中MYB基因家族有2对复制基因,串联重复基因对:IZ004267与IZ007345;片段重复基因对:IZ006462与IZ006102。结合染色体定位分析,IZ006462与IZ006102位于同一条染色体(9号染色体),且在染色体上的位置相近,这也支持上述片段重复的判断。这些重复基因对Ka/Ks<1,表明MYB基因家族扩张过程中,这些重复基因对都受到基因纯化选择,所以它们的功能也相当保守。
球等鞭金藻MYB基因家族成员均含有多个光响应元件,且光响应元件是基因家族成员所有启动子顺式作用元件中最多的作用元件,可能与光的接收、生物钟的调控有关。光会影响植物在整个生命周期的发展过程,在植物中凡是能感受到不同光现象的物质均被称为光受体。光周期途径是光受体对不同的光信号感知后做出一系列的响应活动,进而达到调控植物开花,影响根、茎的形成以及调节叶落和芽休眠时间的作用[17-19]。此外,特定基因组的转录调控是光调节植物生长发育的关键机制之一。也有许多与植物生长激素相关的响应元件,如脱落酸响应元件、生长素响应元件、茉莉酸响应元件及水杨酸响应元件等,说明这3个家族与植物生长发育密切相关;还有其他非生物胁迫响应元件,说明它们可能参与逆境胁迫响应等其他功能。
参考文献:
[1]王珺,王永强,陈雪芬,等.金藻0898培养的生态条件研究[J].海南大学学报(自然科学版),2008(1):93-98.
[2]JIA Y C,SIMX,YING Y W,et al.Integrated Analyses of miRNome and Transcriptome Reveal the Critical Role of miRNAs Toward Heat Stress Response in Isochrysisgalbana[J].Marine biotechnology,2022,24(4):753-762.
[3]AGUILERASAEZ L M,ABREU A C,CAMACHO R J,et al.NMR Metabolomics as an Effective Tool To Unravel the Effect of Light Intensity and Temperature on the Composition of the Marine Microalgae Isochrysisgalbana[J].Journal of agricultural and food chemistry,2019,67(14):3879-3889.
[4]HONG L Z,FENG R G,KUN Y S,et al.Docosahexaenoic acid production of the marine microalga Isochrysisgalbana cultivated on renewable substrates from food processing waste under CO2 enrichment[J].The Science of the total environment,2022,848:157654.
[5]ZHANGY,ZHANG X,GUO R,et al.Effects of florfenicol on growth,photosynthesis and antioxidant system of the non-target organism Isochrysisgalbana[J].Comparative Biochemistry and Physiology Part C Toxicology & Pharmacology,2020,233,108764.
[6]LIX,DHAUBHADEL S.1433 proteins act as scaffolds for GmMYB62 and GmMYB176 and regulate their intracellular localization in soybean[J].Plant Signal Behav,2012,7(8):965-968.
[7]刘蕾,杜海,唐晓凤,等.MYB转录因子在植物抗逆胁迫中的作用及其分子机理[J].遗传,2008,30(10):7.
[8]王拓璋,王毅,陈娇娇,等.鳄梨干旱与低温胁迫相关MYB转录因子的鉴定及表达分析[J].分子植物育种,2023:https://kns.cnki.net/kcms/detail/46.1068.S.20230506.0944.004.html.
[9]阙秋霞,赖恭梯,潘若,等.刺葡萄MYB基因家族鉴定及其在不同光质下的表达模式分析[J].农业生物技术学报,2023,30(2):298-310.
[10]MILLARD P S,KRAGELUND B B,BUROW M.R2R3 MYB Transcription Factors-Functions outside the DNA-Binding Domain[J].Trends in Plant Science,2019,24(10):934-946.
[11]OGATA K,MORIKAWA S,NAKAMURA H,et al.Solution structure of a specific DNA complex of the Myb DNA-binding domain with cooperative recognition helices[J].Cell,1994,79(4):639-648.
[12]HAI B T,JOHN E B,XI Y W,et al.Paterson.Synteny and Collinearity in Plant Genomes[J].Science,2008,320(5875):486-488.
[13]WOLFE KH.Comparative genomics and genome evolution in yeasts[J].Philosophical Transactions of the Royal Society B: Biological Sciences,2006,361(1467):403-412.
[14]NAKATANI Y,TAKEDAH,KOHARA Y J,et al.Reconstruction of the vertebrate ancestral genome reveals dynamic genome reorganization in early vertebrates[J].Genome research,2007,17(9):1254-1265.
[15]LYONS E,PEDERSEN B,KANE J,et al.Finding and comparing syntenic regions among Arabidopsis and the outgroups papaya, poplar, and grape: CoGe with rosids[J].Plant physiology,2008,148(4):1772-1781.
[16]SODERLUNDC,BOMHOFFM,NELSONWM.SyMAP v3.4:a turnkey synteny system with application to plant genomes[J].Nucleic acids research,2011,39(10):e68.
[17]OIDE M,NAKASAKOM.Red light-induced structure changes in phytochrome A from Pisumsativum[J].Scientific Reports,2021,11(1):2827.
[18]CHRISTIE JM.Phototropin Blue-Light Receptors[J].Annual Review of Plant Biology,2007,58(1):21-45.
[19]INES C,RICHARD P,MARTINB,et al.The Cryptochromes: Blue Light Photoreceptors in Plants and Animals[J].Annual Review of Plant Biology,2011,62(1):335-364.
(責任编辑:柯文辉)
收稿日期:2023-03-17
作者简介:韩玉莹,女,1994年生,硕士,主要从事分子生物学研究。
*通信作者:陈建楠,男,1991年生,实验师,主要从事球等鞭金藻培育研究(E-mail:381220681@qq.com)。
基金项目:国家现代农业产业技术体系项目 (CARA-170501)。
