基于55K SNP芯片分析的优质小麦的遗传多样性
2022-05-20彭义峰李亚青张士昌李孟军何明琦
张 楠,彭义峰,李亚青,张士昌,李孟军,何明琦
(石家庄市农林科学研究院,河北省小麦工程技术研究中心,河北石家庄 050041)
随着人民生活水平的不断提高,市场对优质小麦的需求量不断增长。当前,优质小麦生产面临着大面积推广应用品种少、品种抗逆性不佳、品质不稳定等问题。在优质小麦育种实践中,可选用的育种亲本数目有限,仅师栾02-1、藁优2018、济南17、新麦26、西农979等优质强筋小麦可作为育种的亲本。利用分子标记(如SSR、SNP芯片)分析优质小麦品种的亲缘关系和遗传相似度,对优质小麦种质创新和育种具有重要意义,但目前有关优质小麦遗传多样性的研究报道较少。利用SNP芯片对中国主要优质小麦品种的遗传多样性进行系统研究尚未见报道。
小麦基因组测序工作的不断深入,极大促进了SNP标记的研究与开发。SNP芯片具有标记数量多、通量高等优点。目前,已开发的小麦SNP芯片主要有9K iSelect、90K iSelect、820K Axiom array、35K Axiom array、50K TraitBreed、15K、660K Axiom array、55K等。研究表明,小麦SNP芯片是研究小麦遗传多样性、群体结构和QTL定位的有效技术手段。与小麦其他SNP芯片比较,55K SNP芯片的标记有效性更高,已用于QTL定位、农艺性状关联分析、遗传多样性分析等研究。本研究选取中国主要优质小麦品种,利用55K SNP芯片揭示其遗传多样性和亲缘关系,以期为优质小麦育种亲本选配、种质资源创制和分子标记辅助选择提供参考依据。
1 材料与方法
1.1 供试材料
45个优质小麦品种来自国家产业体系历年种质资源征集品种;5个优质高代品系(品鉴01、品鉴02、品鉴03、品鉴04和品鉴05)由石家庄市农林科学研究院小麦研究所提供。所有材料现保存于石家庄市农林科学研究院分子育种实验室,品种(系)的具体信息见表1。
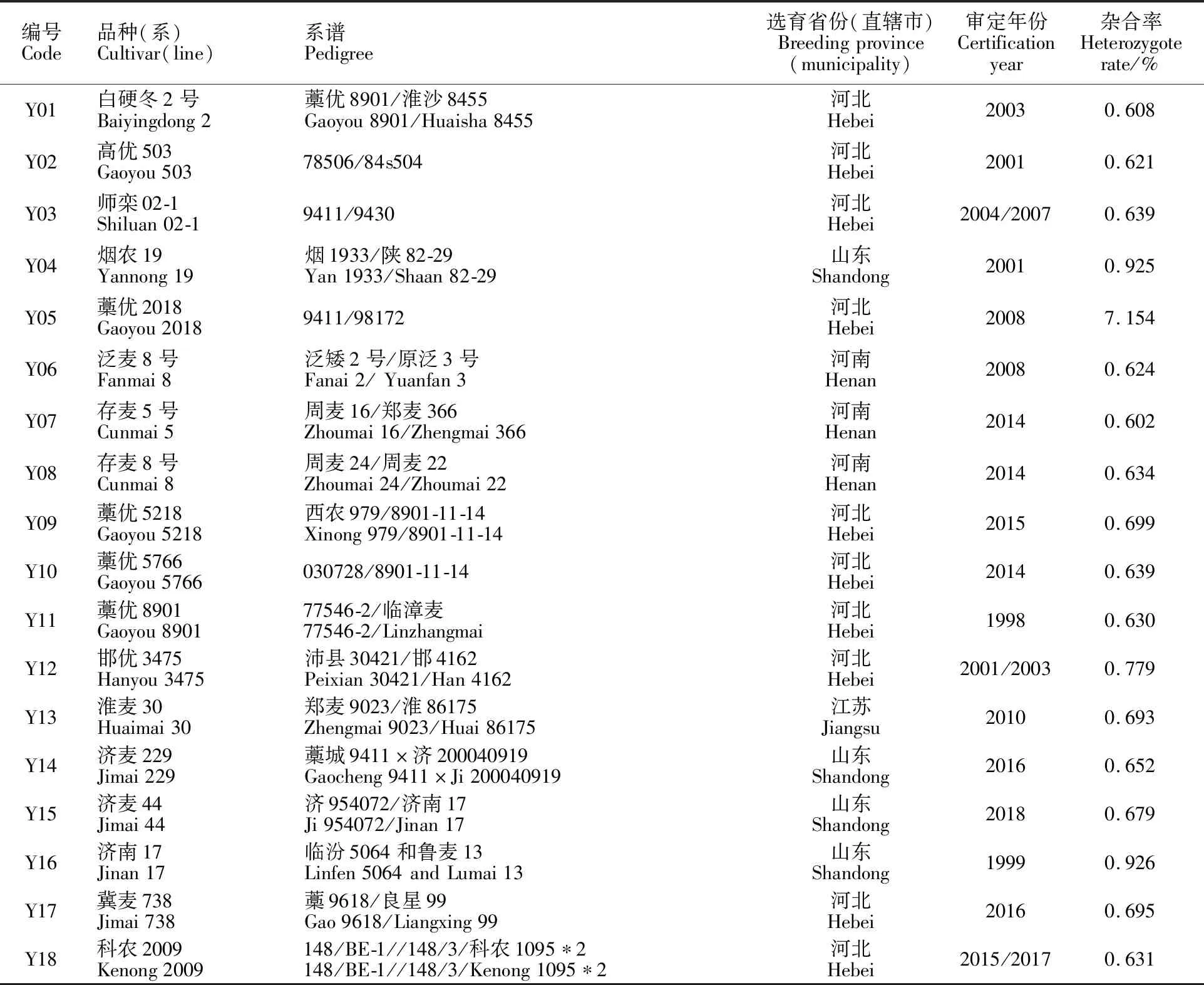
表1 供试小麦品种(系)的来源及其杂合率Table 1 Origin and heterozygote rate of wheat cultivars(lines)used in the study

(续表1 Continued table 1)
1.2 基因组DNA提取
用于SNP芯片分析的50个小麦品种(品系)为经过连续2代田间选择的单株。试验材料在植物生长室中培养,待长至二叶一心时,取小麦叶片,用CTAB法提取基因组DNA。
1.3 SNP标记分析
1.4 统计分析
对Dish QC(DQC)>0.82和标记检出率(CR)>95% 的样品进行SNP位点质控。以缺失率(MISS)<0.1和最小等位基因频率(MAF)>0.05为标准对SNP标记进行过滤。以杂合标记数除以总标记数计算品种的杂合率。以基因型一致的标记数除以成功分型的标记数计算品种的遗传相似度。使用Structure软件进行群体结构分析。使用TreeBest软件的NJ-tree模型构建进化树。
2 结果与分析
2.1 染色体低多态性区段
染色体低多态性区段是指SNP多态性低的染色体区段,由MAF值较低的一段SNP位点组成。55K SNP芯片共有53 007个探针位点,通过对DQC>0.82 和CR>95%的样本进行SNP位点质控,共获得36 691个有效标记,去除无染色体和物理位置的标记,最终获得33 876个有效标记,可用于染色体低多态性区段分析。其中,多态性标记有33 202个,占有效标记数的98.01%。B基因组多态性位点最多,为13 207个,占比 39.78%,其中4B染色体最多;A基因组上多态性位点有12 873个,占比38.77%,其中5A染色体上最多;D基因组上多态性位点最少,为7 122个,占比21.45%,4D和5D染色体上分别仅有648和674个。多态性SNP位点在部分同源群上的分布频率大小依次为7号染色体>4号染色体>3号染色体>2号染色体>5号染色体>1号染色体>6号染色体。
在33 876个有效标记中,MAF在0.00~ 0.20之间的SNP标记占比31.45%(图1A),因此,进一步分析阈值为0.10和0.05两种情况(图1B和1C)。低多态性区段在优质小麦品种(系)染色体上的分布表明,A基因组和D基因组染色体上的低多态性区段均多于B基因组。6A、4D和6D染色体上的低多态性区段多于其他染色体 (图1)。
2.2 品种的杂合率
杂合率反映优质小麦品种(系)在基因组水平上的杂合程度。一般而言,自交代数越高的材料,其杂合率越低。利用55K SNP芯片对50个小麦品种(系)进行分析,结果(表1)发现,在45个优质小麦品种中,除藁优2018的杂合率为7.154%外,其余44个优质小麦品种的杂合率变化范围为0.496%~1.457%,平均值为0.849%,表明这些优质小麦品种的纯合程度整体较高。5个优质高代品系中,品鉴2为F代株系,杂合率高达 8.399%,但其群体农艺性状表现一致。
2.3 基于SNP标记的品种间遗传相似度
遗传相似度分析能够为优质小麦亲本组配(相似度低的材料)和群体改良(相似度高的材料)提供数据支撑。45个优质小麦品种间的遗传相似度系数(genetic similarity,GS)变化范围为 0.488~0.928,平均值为0.603;有1 944个GS变化范围为0.500~0.800,占98.18%,其中 0.500~0.600、0.600~0.700和0.700~0.800分别占54.34%、38.18%和5.66%。GS小于 0.500的有20个(1.01%),大于0.800的有16个(0.81%)。因此,中国优质小麦品种间具有相对较低的遗传多样性。
河北省15个优质小麦品种间的GS变化范围为0.467~0.928,平均值为0.618;山东省8个优质品种间的GS变化范围为0.557~0.740,平均值为0.638;河南省9个优质品种间的GS变化范围为0.564~0.736,平均值为0.630。可以看出,河北省优质小麦品种间的GS低于山东省和河南省。系谱分析显示,这种现象与河北省优质小麦的育种亲本较为广泛有关。
品种间GS低于0.500的优质小麦品种为:(1)师栾02-1和藁优2018;(2)藁优2018和藁优5818;(3)高优503和龙麦40;(4)藁优8901和西农889;(5)藁优8901和藁优2018;(6)白硬冬和龙麦40;(7)邯优3475和镇麦168;(8)泛麦8号和藁优8901;(9)藁优2018和科农2009;(10)冀麦738和运旱618,这些优质小麦品种分别来自河北、河南、黑龙江、江苏、陕西和山西,亲缘关系较远。品种间GS高于0.800的优质小麦品种为:(1)西农20和存麦5号;(2)石4366和石优17;(3)石优20和济南17;(4)中麦578和济麦44;(5)西农979、西农9718、藁优5218和藁优5818;(6)龙麦26和龙麦40;(7)藁优5766和藁优5218。其中龙麦26和龙麦40、藁优5766和藁优5218的GS均高于0.900。这些品种属于近似品种,在优质小麦种质创新和育种中应予以注意。

a、b和c分别表示染色体上0.00≤MAF≤0.20、0.00≤MAF≤0.10和0.00≤MAF≤0.05的低多态性区段。蓝色表示小于等于MAF阈值的多态性区段,红色表示大于阈值的多态性区段。a,b and c indicate low-diversity chromosomal regions with 0.00≤MAF≤0.20,0.00≤MAF≤0.10 and 0.00≤MAF≤0.05,respectively.Red indicates the MAFs of chromosomal regions are higher than threshold value,and blue indicates the other regions.图1 低多态性区段在优质小麦品种(系)染色体上的分布Fig.1 Distribution of low-diversity regions on high-quality wheat chromosomes
2.4 群体结构
群体结构分析是按照一定标准将一个群体分为若干亚群,处于同一亚群内的个体亲缘关系较高。群体结构分析可了解自然群体的遗传多样性和遗传分化情况。在群体结构分析中,最低的交叉验证误差(cross-validation error,CV error)对应的值(群数)是最理想的选择。在50个小麦品种(系)中,=2 时,CV errors最低(1.15),因此,选择=2进行进一步分析。当=2时,有12个优质小麦品种(系)血统混杂,在不同的群结构分析中可能发生变化,但不影响整体分群评估(图2A)。
2.5 进化树
使用TreeBest软件的NJ-tree模型将50个小麦品种(系)分为2个类群,这与群体结构分析结果一致。其中,类群1包含6个亚群(亚群Ⅰ~Ⅵ),有39个品种(系);类群2仅包含亚群Ⅶ,有11个品种(系)。类群1中,亚群I包含6个品种(系),其中品鉴01与中麦578亲缘关系较近,聚类在一个分支上。亚群Ⅱ包含5个品种,其中,石优17是石4366的父本,聚类在一个分支上。亚群Ⅲ包含3个品种,济南17是石优20和新麦26的父本。亚群Ⅳ包含6个品种(系),其中,郑麦7698是品鉴03、品鉴04和品鉴05的父本;郑麦366是西农20和存麦5号的共同亲本。亚群Ⅴ包含10个品种,其中郑麦98、烟农21、西农889、泛麦8号、郑麦366血统混杂,该亚群聚类结果与系谱关系不明;亚群Ⅵ包含9个品种,有2个分支,其中,1个分支是西农2611或西农979的后代;另1个分支包含4个品种,其中3个为龙麦系列品种,龙麦26是龙麦35的父本。类群2中,济麦229、邯麦18、冀麦738、龙麦20和邯优3475血统混杂。类群2有2个分支,其中1个分支为临漳麦或藁优8901的后代,另1个分支中,邯麦18、师栾02-1和冀麦738均具有藁优8901血统,龙麦20和邯优3475的聚类结果与系谱关系不明(图2B)。
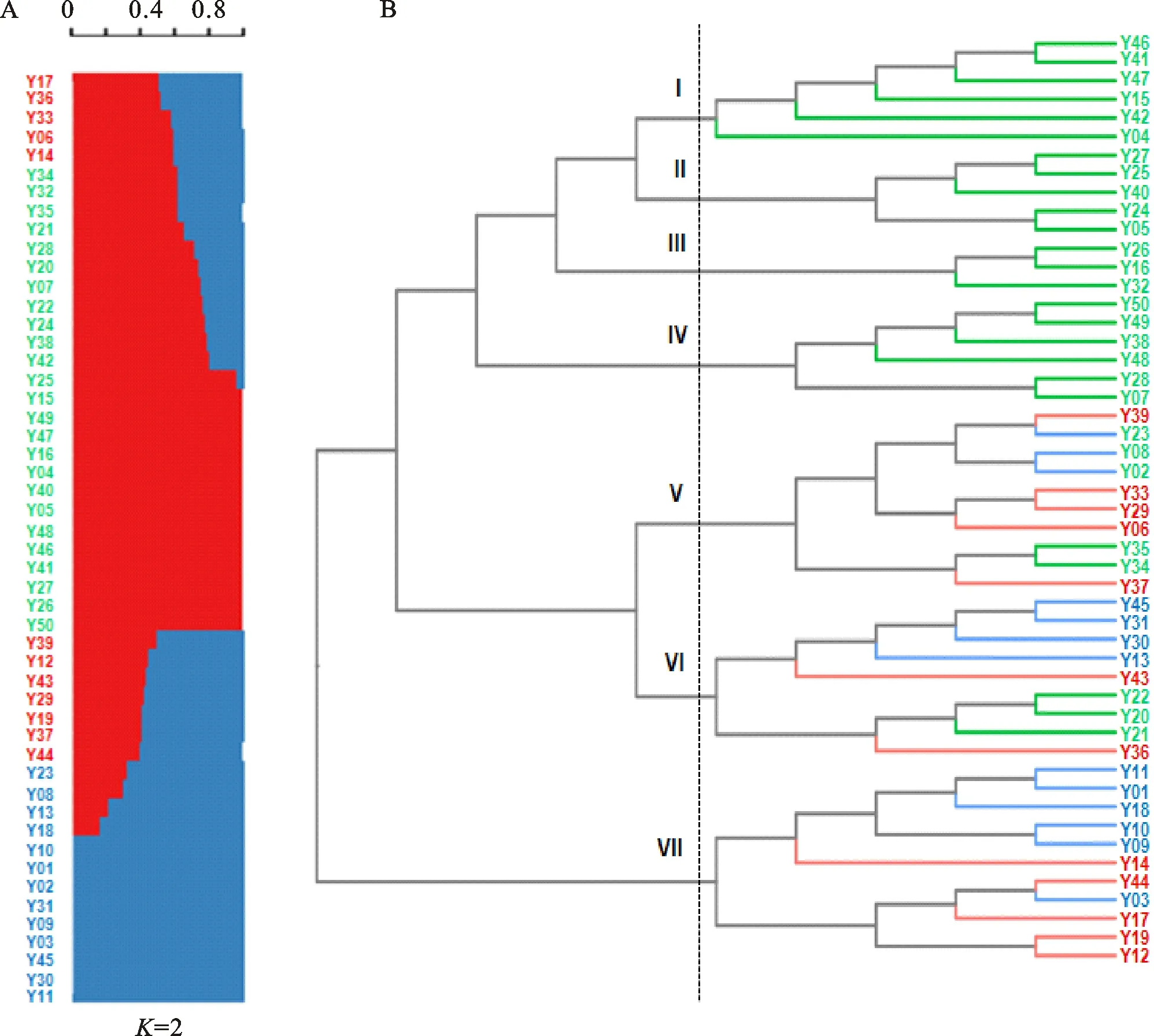
A:群体结构分析;B:进化树分析。品种(系)编号对应的品种(系)见表1。绿色和蓝色字体表示不同类群,图B中分枝线条颜色与图A不同类群对应;红色字体表示血统混杂品种(系)。A:Population structure analysis;B:Phylogeny tree analysis.Cultivars(lines)correspond to numbers showed in table 1.Green and red fonts represent different population,and the colors of branch lines in figure B correspond to different groups in figure A;Red font represents mixed ancestry cultivars(lines).图2 供试小麦品种(系)的群体遗传结构和进化树分析Fig.2 Population structure and phylogeny tree analysis of the tested wheat cultivars(lines)
3 讨 论
本研究发现,55K SNP芯片中有效标记和多态性标记数量分别占总标记数量的63.91%和62.64%。有效标记中多态性标记占比高达 98.01%。D基因组中有效标记和多态性标记数量显著低于A基因组和B 基因组,但多态性比例无显著差异。7个部分同源群上的有效标记数量为3 686~5 051个,相应多态性标记占比 97.38%~98.90%。且55K SNP芯片中有效标记的多态性以及在不同基因组和7个部分同源群上的分布均优于90K SNP芯片。因此,55K SNP 芯片适用于对中国优质小麦开展遗传多样性研究。
GS反映不同小麦品种(系)间在基因组水平上的相似程度。郑永胜等基于SSR标记,建议在DUS测试中与申请品种遗传相似度系数高于0.800的已知品种作为近似品种进行田间种植和特异性评价。王立新等利用Genomic-SSR、EST-SSR、AFLP-SCAR标记筛查547个国家冬小麦区域试验品系及134个国审品种,发现绝大多数相似品种(系)间的GS 均大于0.900。曹廷杰等利用90K SNP芯片对96个河南省小麦品种进行遗传多样性分析,发现品种间GS变化范围为0.552~0.998,平均值为0.719,94.3%的品种GS分布在0.652~0.812 之间;李珊珊等利用90K SNP芯片对143份河北省推广小麦品种进行遗传多样性分析,发现品种间GS变化范围为 0.438~0.997,平均值为0.602,89.23%的品种间GS分布在0.520~0.730之间。本研究表明,中国优质小麦GS变化范围为0.488~0.928,平均值为0.603,98.18%的品种间GS分布在 0.500~0.800之间。这表明,中国优质小麦品种遗传多样性总体不高。随着中国优质小麦品种审定标准的提高,优质小麦的遗传多样性有进一步降低的趋势。在45个优质小麦品种中,0.81%的优质小麦品种间GS高于0.800,但其农艺性状存在显著差异,因此,这些品种均具有特异性,符合现行国家小麦品种审定标准。SNP芯片探针根据基因组SNP设计,很多标记与重要农艺性状基因无关,可能是导致上述现象的原因之一。
本研究群体结构和进化树分析结果表明,50个供试小麦品种(系)仅分成 2个类群7个亚群,同时,2个类群间存在较严重的混血现象,这种现象是遗传多样性较低的一种表现。50个小麦品种(系)有12个血统混杂,这些血统混杂品种在不同群结构分析中变化较大,但不影响整体分群评估。聚类结果与系谱比较分析表明,除亚群Ⅴ外,供试小麦品种(系)聚类分析与系谱分析结果基本吻合。因此,利用55K SNP芯片能够较好地反映中国小麦品种(系)的遗传多样性和亲缘关系。
在50个供试小麦品种(系)中,多态性SNP标记数量在B基因组中最多,A基因组次之,D基因组最少,与利用RFLP标记、SSR标记和90K SNP芯片对小麦遗传多样性的研究结果一致。A基因组和D基因组的染色体低多态性区段均多于B基因组,这与多态性标记的分布规律相符合。6A、4D和6D染色体上的低多态性区段多于其他染色体,这表明,在优质小麦育种过程中,6A和6D染色体这些区段受到高强度选择。一般认为,六倍体小麦D基因组的遗传多样性较低,4D染色体上的多态性标记最少。本研究在50个供试优质小麦品种(系)中,4D染色体上的多态性标记也最少(674个),多态性标记在有效标记中的比例也最低 (88.77%)。供试小麦品种(系)染色体上的SNP标记MAF在 0.00~0.20之间的占比31.45%,且4D染色体上占比39.04%,这表明,4D染色体在优质小麦育种过程中也受到了高强度选择。
