生物信息学在筛选影响结直肠癌预后基因中的应用*
2021-05-26伏小红冯宦翔屈春燕
伏小红,冯宦翔,苟 伟,杨 敏,郭 靓,屈春燕
四川省成都市第七人民医院:1.检验科;2.神经内科,四川成都 610000
结直肠癌(CRC)的发病率和病死率在消化系统恶性肿瘤中仅次于胃癌、食管癌和原发性肝癌[1],近年来其发病率在我国不少地区有不断上升的趋势[2]。有研究发现,普遍的筛查可有效提升CRC患者的生存率,降低其病死率[3]。鉴于筛查的现实需要和目前筛查方式的不足,急需寻找更方便快捷的筛查方法。基因芯片和基因图谱是一种很成熟的检测技术,特别适用于筛选差异表达基因(DEGs)[4]。运用生物信息学方法整合和分析基因芯片应用过程中积累下来的大量CRC数据,并与表达谱技术相结合,非常适用于研究CRC可能存在的生物标记物基因。本研究结合多种数据库开展CRC的研究。
1 资料与方法
1.1筛查出有差异的上调和下调基因 从基因芯片数据库(NCBI-GEO)中获取GSE110224、GSE41328和GSE15960基因在CRC组织及正常结直肠组织中的表达谱,均是GPL570[G-U133_Plus_2]Affymetrix Human Genome U133 Plus 2.0 Array,包括28个正常结直肠组织标本和38个CRC组织标本;采用GEO2R在线工具对CRC组织标本和正常结直肠组织标本的DEGs数据进行分析(|logFC|>2和调整P<0.05);然后再用Venn软件对原始数据进行分析,检测3个基因芯片之间的差异,logFC<0的DEGs为下调基因,logFC>0的DEGs为上调基因。
1.2分析筛选出的DEGs的生物学特征 在DAVID数据库里,用基因本体论(GO)分析筛选出的DEGs的生物学特征,分成生物过程(BP)组、分子功能(MF)组和细胞成分(CC)组3组。用京都基因与基因组百科全书(KEGG)分析信号通路情况。
1.3分析蛋白质相互作用 通过检索相互作用基因的搜索工具(STRING)分析蛋白-蛋白相互作用(PPI)网络,再用MCODE插件进一步分析。
1.4分析筛选出的DEGs对生存率的影响及对RNA的表达 利用UALCAN数据库评估筛选出的基因对生存率的影响,计算出有95%置信区间的logrankP值和风险比(HR)。利用基因表达数据交互分析(GEPIA)网站对标本的RNA表达数据进行分析。
2 结 果
2.1CRC的DEGs分析 用于鉴别的DEGs共包括38份CRC组织标本和28份正常结直肠组织标本。通过GEO2R在线工具,分别从GSE110224、GSE41328和GSE15960中提取出177、290和3 235个DEGs。用Venn软件对3个数据集中的常用数据集进行识别,共检测到62个常见DEGs(21个上调基因,41个下调基因)。见表1、图1。
2.2DEGs的GO和KEGG通路分析 在DAVID数据库里采用GO分析筛选出的62个DEGs,KEGG通路分析表明,下调的DEGs在肾素分泌和胰腺分泌调节信号通路富集,主要相关的基因是CLCA1和CLCA4,而上调的DEGs未见明显的信号通路(P<0.05),见表2、3。
2.3PPI和MCODE插件分析 PPI分析36个DEGs被导入到含有59个节点和60条变的DEGs PPI网络复合体中,62个DEGs中有36个被纳入到DEGs PPI网络,其中包括10个上调基因和26个下调基因。采用MCODE插件进一步分析结果显示,6个中心节点均为下调基因,见图2。
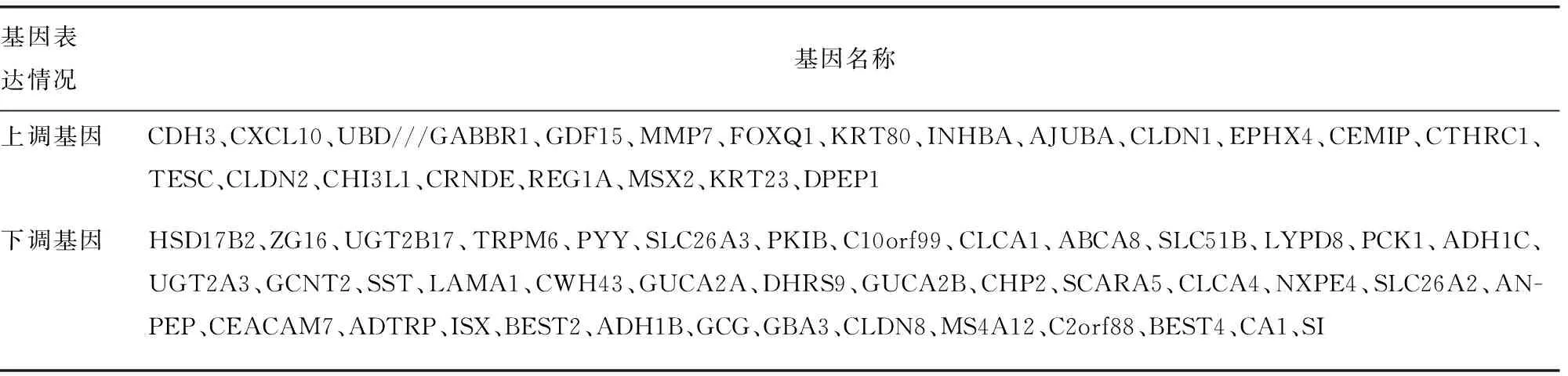
表1 CRC组织与正常结直肠组织相比得出的3个基因芯片都包含的21个上调基因和41个下调基因
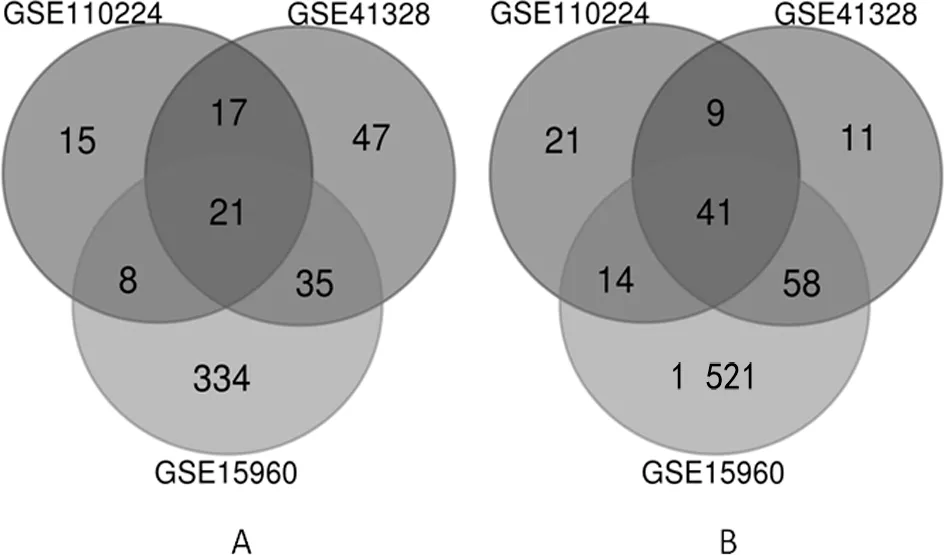
注:A为上调基因;B为下调基因;数据为DEGs。
2.4UALCAN和GEPIA的核心基因分析 CRC基因表达数据来源于数据库中的结肠腺癌(COAD)和直肠腺癌(READ)两种癌症所表达的数据。利用UALCAN数据库对6个核心基因对CRC的预后影响进行分析,发现CLCA1和CLCA4基因低表达时对COAD的预后有明显影响(P<0.05),对READ无明显影响,且剩余4个基因差异无统计学意义(P>0.05)。用GEPIA对核心基因再进行分析发现,CRC组织与正常结直肠组织相比,CLCA1和CLCA4基因在CRC中表达明显降低(P<0.05)。见图3、4。
2.5KEGG通路富集分析 通过DAVID数据库重新对这3个筛选出的基因进行KEGG通路富集分析,结果显示只有CLCA4在肾素分泌的通路中明显表达降低(P<0.05)。
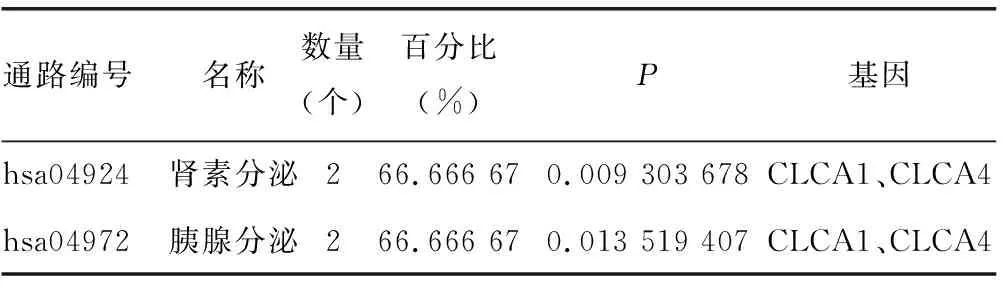
表2 CRC的DEGs的KEGG通路分析
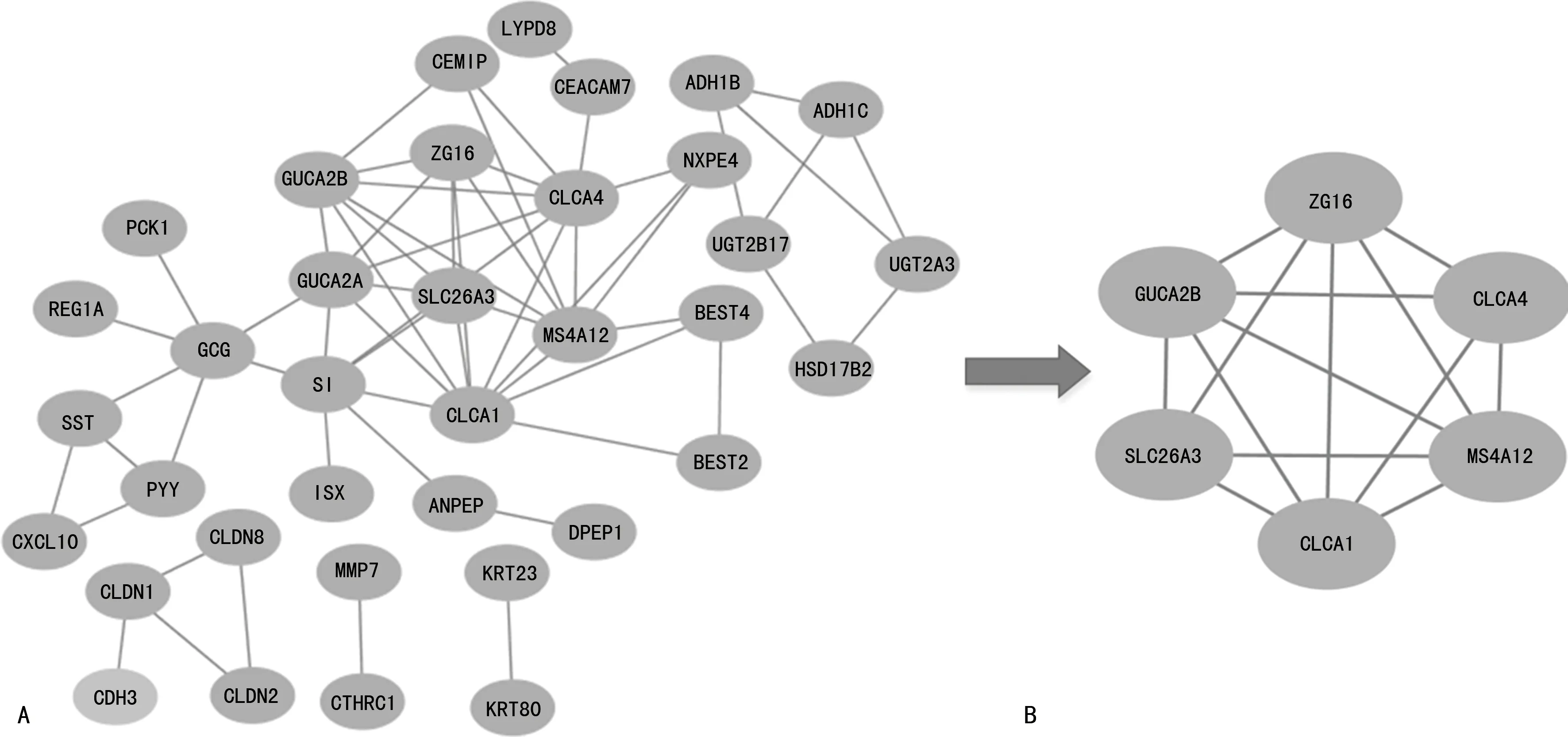
注: A为STRING分析PPI结果;B为MCODE分析所得中心节点。
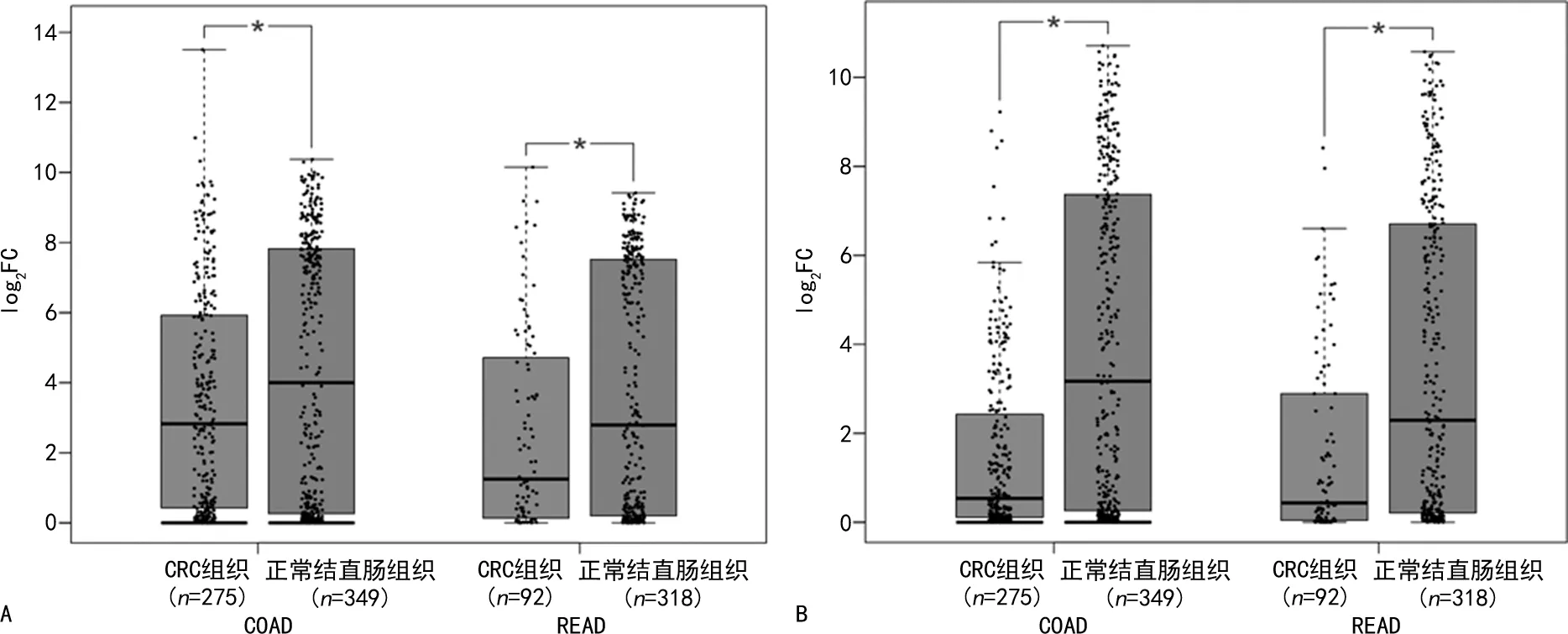
注:A为CLCA1基因;B为CLCA4基因;*P<0.05。
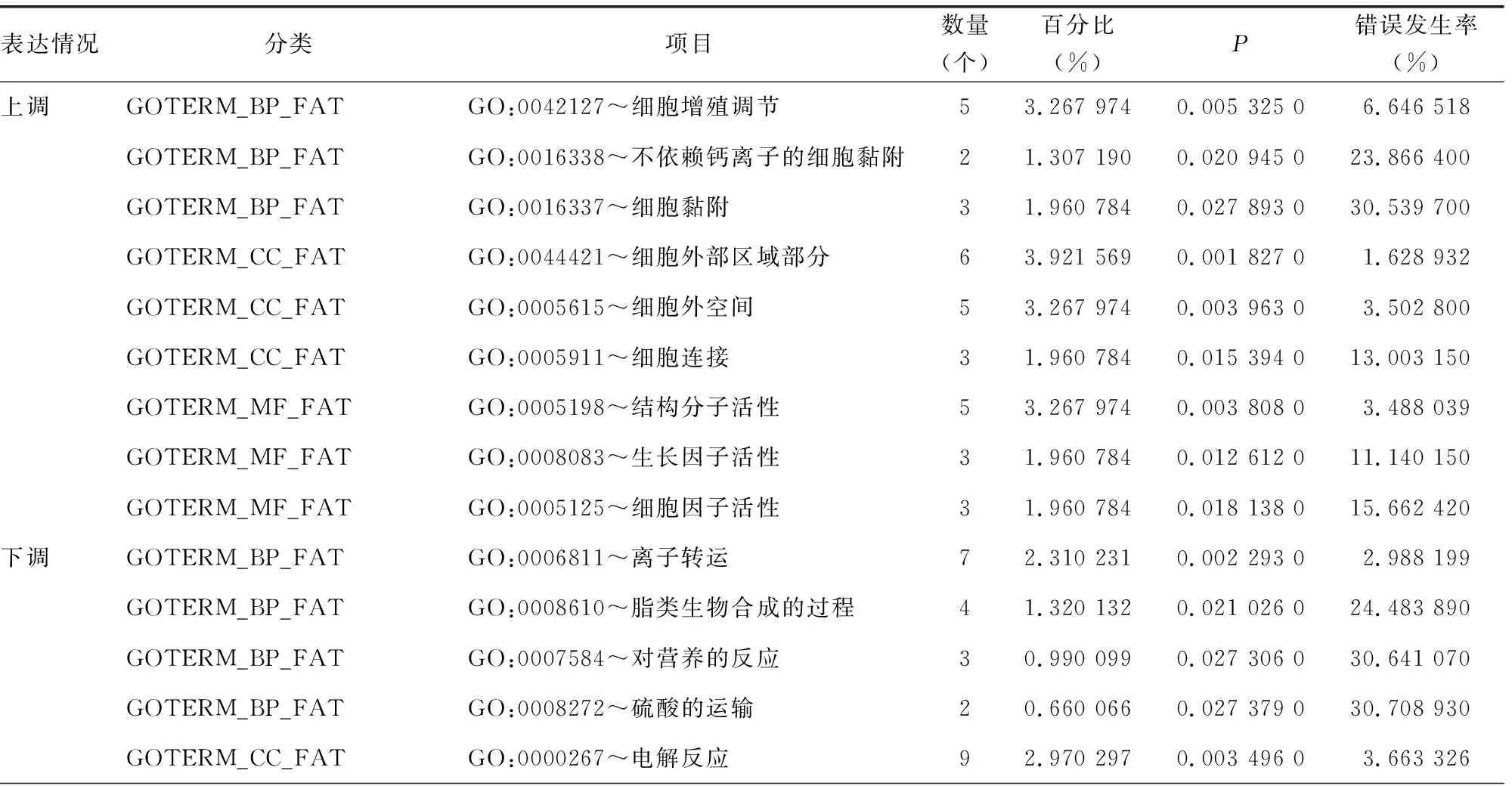
表3 CRC的DEGs的GO分析
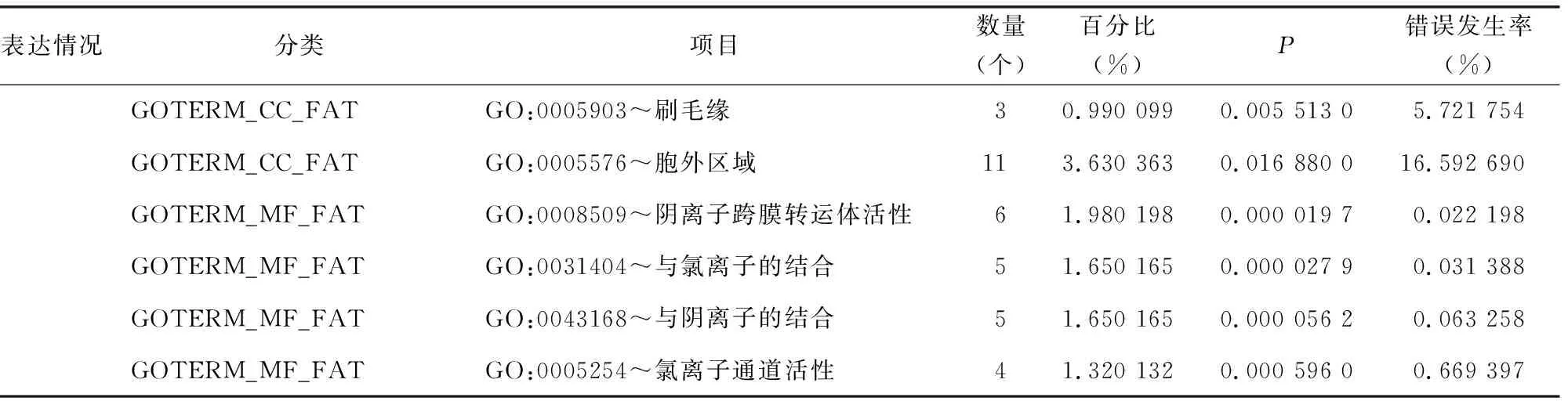
续表3 CRC的DEGs的GO分析
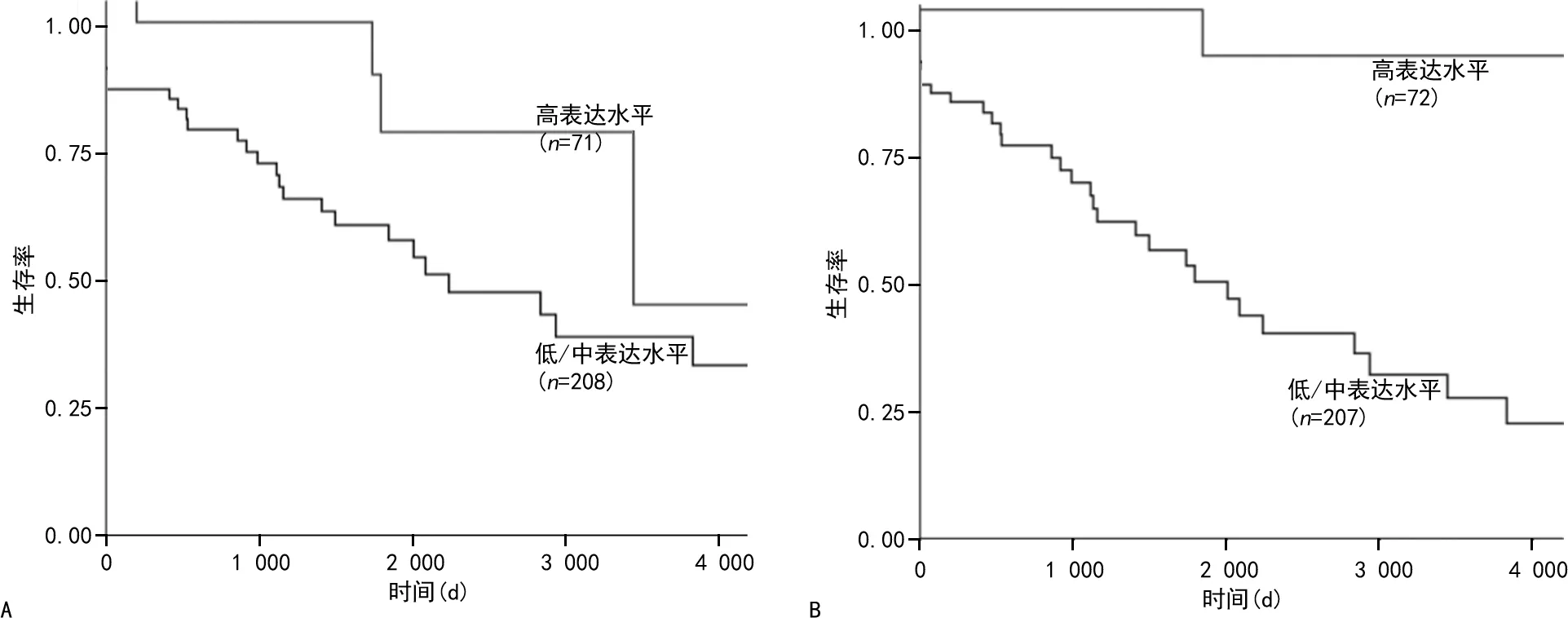
注:A为CLCA1的表达水平对COAD患者生存率的影响;B为CLCA4的表达水平对COAD患者生存率的影响。
3 讨 论
CRC作为全球癌症相关死亡的第4大原因,在CRC不同发展阶段诊断的患者5年生存率差异很大,早期诊断的患者生存率超过70%,而晚期诊断的患者生存率不到10%[5]。超过一半的CRC患者在诊断时已有局部和远处转移,尤其是淋巴结转移严重影响患者的预后[6]。并且有研究发现越年轻的CRC患者预后也越好[7],因此如果有便捷的早期筛查方法在常规体检或者是转移发生前及早地发现CRC,能够有效提高CRC的生存率。
本研究利用生物信息学方法对3个芯片进行了深入分析,集中研究了多个基因,与正常结直肠组织相比,CRC组织中的CLCA1和 CLCA4存在明显差异,同时发现这两个基因的低表达能够对COAD患者的生存率产生影响,这一发现提示CLCA1和 CLCA4可能是诊断和治疗CRC的潜在生物标志物。但CLCA1和 CLCA4的低表达通过GEPIA分析只对COAD的预后有明显影响,而在READ组织中的明显低表达却未对其预后有明显影响,这可能与数据库的标本收入量等有关,还需进一步做更深入的研究。
CLCA1和CLCA4是钙活化氯离子通道家族中的成员,与CLCA2一起具有相似的一级结构。钙活化氯离子通道调控因子作为一种在氨基酸末端中具有对称多半胱氨酸基序的蛋白质[8],在多种癌症中都出现表达失调[9-10]。同时,对钙活化氯离子通道蛋白的研究发现,其不仅可以调节炎性反应,还参与致癌[11],由于其相似的一级结构,使其可能在作用效能上也存在类似情况。
有研究发现,CRC患者的CLCA1血清水平较健康对照者明显降低,并且CLCA1血清水平和CLCA1mRNA表达水平与CRC的转移和分期呈负相关,还发现CLCA1可能通过下调Wnt/β-catenin信号通路和上皮细胞-间质转化(EMT)发挥抑癌作用[12]。异常的Wnt信号可以触发EMT过程,这在癌症转移中至关重要。CLCA1表达的增加降低了β-catenin的表达水平,抑制了EMT从而抑制肿瘤的活性,这与一些文章发现的Wnt/β-catenin激活和炎症性肠病是导致CRC的两个主要因素的结论不谋而合,这两个因素能导致肠道内肠上皮内环境稳态的破坏,如细胞增殖增加、细胞分化和凋亡减少等[13-15]。同时,EMT过程使上皮细胞转化为间质细胞,使细胞失去黏附和极性,从而发生迁移和侵袭[16]。
对于CLCA4,近期也有研究指出,CLCA4在细胞中发挥生物学功能是通过使PI3K/AKT信号失活和调节EMT介导的,并且CLCA4 的降低与CRC患者的淋巴结转移有关[17],这与LIU等[18]发现的CLCA4抑制肝癌细胞的增殖和侵袭的机制相似。
综上所述,应用生物信息学分析方法,本研究筛选出了CLCA1和CLCA4两个基因,对其进行更进一步的作用机制讨论,发现它们严重影响CRC的转移,且最终影响CRC患者的预后。本研究丰富了CLCA1和CLCA4在CRC中的认识,二者有可能可以作为CRC的诊断依据和治疗靶点。
