结合多头自注意力机制与BiLSTM-CRF的中文临床实体识别
2021-05-06罗熹夏先运安莹陈先来
罗熹 夏先运 安莹 陈先来
摘 要:命名实体是电子病历中相关医学知识的主要载体,因此,临床命名实体识别(Clinical Named Entity Recognition,CNER)也就成为了临床文本分析处理的基础性任务之一. 由于文本结构和语言等方面的特殊性,面向中文电子病历(Electronic Medical Records,EMRs)的临床命名实体识别依然存在着巨大的挑战. 本文提出了一种基于多头自注意力神经网络的中文临床命名实体识别方法. 该方法使用了一种新颖的融合领域词典的字符级特征表示方法,并在BiLSTM-CRF模型的基础上,结合多头自注意力机制来准确地捕获字符间潜在的依赖权重、语境和语义关联等多方面的特征,从而有效地提升了中文临床命名实体的识别能力. 实验结果表明本文方法超过现有的其他方法获得了较优的识别性能.
关键词:中文电子病历;命名实体识别;长短期记忆;多头自注意力
中图分类号:TP391 文献标志码:A
Chinese CNER Combined with Multi-head
Self-attention and BiLSTM-CRF
LUO Xi1,2,XIA Xianyun2,AN Ying1,CHEN Xianlai1
(1. Big Data Institute,Central South University,Changsha 410083,China;
2. Key Laboratory of Network Crime Investigation of Hunan Provincial Colleges,
Hunan Police Academy,Changsha 410138,China)
Abstract:Named entity is the main carrier of relevant medical knowledge in Electronic Medical Records (EMRs),so clinical named entity recognition(CNER) has become one of the basic and crucial tasks of clinical text analysis and processing. Due to the particularity of medical text structure and Chinese language,the recognition of clinical named entities for Chinese EMRs still faces great challenges. In this paper, a Chinese clinical named entity recognition method based on multi-head self-attention neural network is proposed . In this method, a character-level feature representation method combined with a domain dictionary is presented. Moreover, based on the BiLSTM-CRF model, a multi-head self-attention mechanism is incorporated to accurately capture the multiple features from different aspects, such as dependency weights between characters and contextual semantic relationships, thereby effectively improving the ability of Chinese clinical named entity recognition. Experimental results demonstrate that the proposed method outperforms other existing methods and has the best recognition performance.
Key words:Chinese electronic medical record;named entity recognition;long short-term memory;multi-head self-attention
隨着医疗信息化的快速发展,医疗机构中积累了大量的电子病历数据. 这些电子病历是病人在医院就诊及治疗过程中所产生的重要记录,包含了临床文本、医学图表、医学影像等多种类型的临床记录数据. 其中,诸如主诉、诊断结果、入院/出院记录和治疗过程等临床文本中蕴含着极为丰富的临床经验知识以及与病人健康状态紧密相关的临床信息,但是,这些以非结构化自由文本形式存储的信息很难直接加以分析和利用,需要首先通过自然语言处理技术进行必要的信息抽取,准确地识别出文本中的相关概念、属性和语义关系等重要信息. 命名实体(Named Entity,NE)是电子病历中相关医学知识的主要载体,因此,临床命名实体识别也就成为了临床文本分析处理极为关键的基础性任务之一.
近年来,关于临床命名实体识别的研究得到了研究人员大量的关注,并在英文临床文本领域产生了一系列的研究成果,其中基于统计机器学习方法的条件随机场(CRF)[1]和基于深度学习方法的长短时记忆网络与条件随机场(BiLSTM-CRF)[2]应用最为广泛. 然而,由于在语言结构和表达形式等方面的特殊性,面向中文电子病历的临床命名实体识别依然存在着巨大的挑战.
随着深度学习技术的发展,很多研究人员尝试使用基于循环神经网络(RNN)的深度学习模型解决中文临床命名实体识别任务,该模型将中文文本中的一个句子看作是一条由汉字词语或者字符组成的序列,然后执行循环遍历,利用RNN中的隐藏单元来学习文本中的上下文信息. 但是,已有的研究方法仍然存在着很多的欠缺. 首先,传统的RNN学习长序列中的依赖关系的能力不足,当文本序列较长时容易损失大量有用信息. 其次,现有的方法大多仅将一个文本序列映射为单一的表示,缺乏获取多角度文本序列特征的能力. 此外,这些基于深度学习模型的方法的识别性能很大程度上依赖于大量的标注训练数据集,而且未能对已有的领域特征加以有效的利用. 再加上中文临床文本中存在的很多语法及表述方面的问题,如大量的医学专有名词、非标准化的名词缩写以及由于书写或表达错误导致的噪声,都严重地影响了中文临床文本命名实体识别的性能.
为了解决以上的这些挑战,本文提出了一种结合多头自注意力机制与BiLSTM-CRF的深度网络模型(Multi-Head self-Attention BiLSTM-CRF,MHA-BiLSTM-CRF). 该模型将中文文本字符特征和临床领域知识特征进行融合,以获得更全面的字符级嵌入表示. 然后使用BiLSTM网络从输入序列中捕获相关的时序特征和上下文关系,同时引入多头自注意力机制,从多个角度获得中文临床文本中字符间关联权重表示来更准确地关注句子中的重要字符或单词,从而有效地提升中文临床命名实体的识别能力.
1 相关工作
1.1 中文临床命名实体识别
命名实体识别(Named Entity Recognition,NER)本质上可以看成是一个多标签序列分类问题. 它以由多个字符或词语构成的文本序列为输入,旨在识别文本中的专有名词、数字信息和其他重要名词并将其分类为预先定义的类别,例如人员名称、组织、位置、时间表达等. 多年来,研究人员针对该问题开展了广泛的研究并提出了许多命名实体识别的相关方法[3]. 这些方法大致可以分为三种类型:基于规则和词典匹配的方法、基于传统特征工程的机器学习的方法[4-6]以及目前比较流行的基于深度学习的方法[2,7-9].
在临床领域中,命名实体识别的目标是从给定的临床文本中提取出那些与医疗过程密切相关的实体指代,并将它们准确地划分为疾病、症状、检查、身体部位以及治疗等特定的实体类别. 目前,研究人员针对英文临床文本的命名实体识别已经开展了大量的研究[3]. 而且,得益于英文语言所具有的以空格作为分割符、专业术语的字母大写特征等天然优势,使得许多命名实体识别方法在英文临床文本中得到了较为成功的应用. 然而,由于不同语言在句法结构、表达方式等方面的特殊性,针对其他语言的临床文本,特别是中文临床文本的命名实体识别依然是一项极具挑战性的任务.
为了推动中文临床命名实体识别技术的发展,2017年由中文信息学会组织的全国知识图谱与语义计算大会(CCKS2017)首次设立了中文临床文本命名实体识别相关的测评任务,吸引了大量研究人员的积极参与并产生了一系列较为有效的中文命名实体识别方法. 例如,Li等人[10]将中文临床命名实体识别看作一个词级别的序列标注任务,提出了一个基于BiLSTM-CRF的深度模型,并通过使用额外的医疗词典及数据集作为补充来得到更加丰富的、具有领域特征的词向量,从而有效地提高识别的准确率. Ouyang等人[11]结合分词特征、词性特征以及医疗词典特征,提出了一种基于n-gram字符表示策略的BiLSTM-CRF模型. Xia等人[12]则在BiLSTM-CRF模型的基础上引入自学习和主动学习策略,充分利用未标注数据来增强模型的识别能力. 此外,Hu等人[13]還提出了一种基于投票的混合模型,将基于规则的方法、基于CRF的方法以及融合特征的RNN方法进行结合,以有效地实现中文临床文本中的实体识别.
近年来,研究人员在之前工作的基础上对CNER方法进行了大量的改进. Wang等人[14]将数据驱动的深度学习方法与知识驱动的词典方法结合起来,提出了一种融合领域词典的深度神经网络模型,并利用一种改进的词典特征表示方法,在中文临床文本上获得了较高的命名实体识别性能. Qiu等人[15]采用了一个带条件随机场的残差卷积神经网络模型(RD-CNN-CRF)来解决中文临床命名实体识别的问题. 该方法首先将汉字和字典特征映射为对应的向量表示,然后将其输入到RD-CNN-CRF模型中以捕获相关的上下文特征. 由于CNN出色的并行执行能力,该方法在识别能力和训练时间等方面获得了与现有其他基于RNN的方法相当或更高的性能. Tang等人[16]则提出了一种基于注意力机制的CNN-LSTM-CRF模型. 该模型通过引入CNN层和注意力层来更好地捕获词语的局部上下文信息以及词间关联强度,从而有效地扩展和增强了BiLSTM-CRF模型的学习能力. 尽管上述改进方法在提升中文临床命名实体识别性能方面取得了一定的效果,但它们大多没有充分利用中文文本序列中重要的全局特性,而且往往忽略了数据集中临床实体分布的不均衡性,从而严重影响了其实体识别的准确性.
因此,为了弥补现有方法的上述缺陷,本文提出了一种基于多头自注意力机制的BiLSTM-CRF模型,通过有效地捕获和融合临床文本中字符自身特征、字符间的依赖关系、文本序列中的语义和上下文信息以及词典特征等多层面的文本特征来提升中文临床命名实体的识别能力.
1.2 多头自注意力机制
Google机器翻译团队在2017年发表的论文中提出了一种包含自注意力以及多头自注意力机制的神经网络架构,并在机器翻译任务中取得了较为出色的效果[17].自注意力机制是一种特殊的注意力机制,它通过计算单个文本序列中不同位置字符间的关联关系,以便获得序列的交互表示. 而多头自注意力机制则是通过结合多次并行的自注意力计算来捕获同一序列在不同表示子空间上的信息,进而从多角度多层面得到更全面的相关特征. 自注意力机制自其被提出开始就在自然语言处理相关的诸多领域得到了大量应用,例如,自动文本摘要、自然语言推理、机器翻译及语言理解等.
在中文临床命名实体识别任务中,临床文本语料规模通常较小且其中存在大量不规则的文本表达. 而与RNN和CNN相比,多头自注意力机制具有很多优势. 首先,多头自注意力机制可以捕获句子中任意位置的字符之间的关联关系,使得模型更加方便地学习到长句子的前后文依赖信息. 其次,注意力机制使用权重求和的方式产生输出向量,使其梯度在网络模型中的传播比RNN和CNN更加容易. 此外,多头自注意力机制的并行执行能力更强,具有更快的训练速度. 因此,多头自注意力机制将成为进一步提升现有方法中文临床命名实体识别能力的一种有效手段. 该机制将会选择性地关注某些重要的信息,同时相应地忽略其他次要信息,并且根据文本中文字的重要性将较高的权重分配给相对更重要的文字,获取更多的临床文本字符之间的关联权重特征.
2 MHA-BiLSTM-CRF模型设计
中文临床命名实体识别任务通常被当作序列标注任务来处理. 由于中文文本中词语边界的模糊性,本文对文本序列采用了字符级别的标注方式以避免分词错误对实体识别性能带来的影响. 对于给定的一段文本字符序列S = [w1,w2,w3,…,wn],其中wt表示其中的第t个字符,我们的目标是训练出一个模型准确地为文本中的每一个字符给出包含实体类别和位置信息的对应标签,得到相应的标签序列Y = [y1,y2,y3,…,yn]. 本节将简要介绍一下本文方法的基本原理及处理流程.
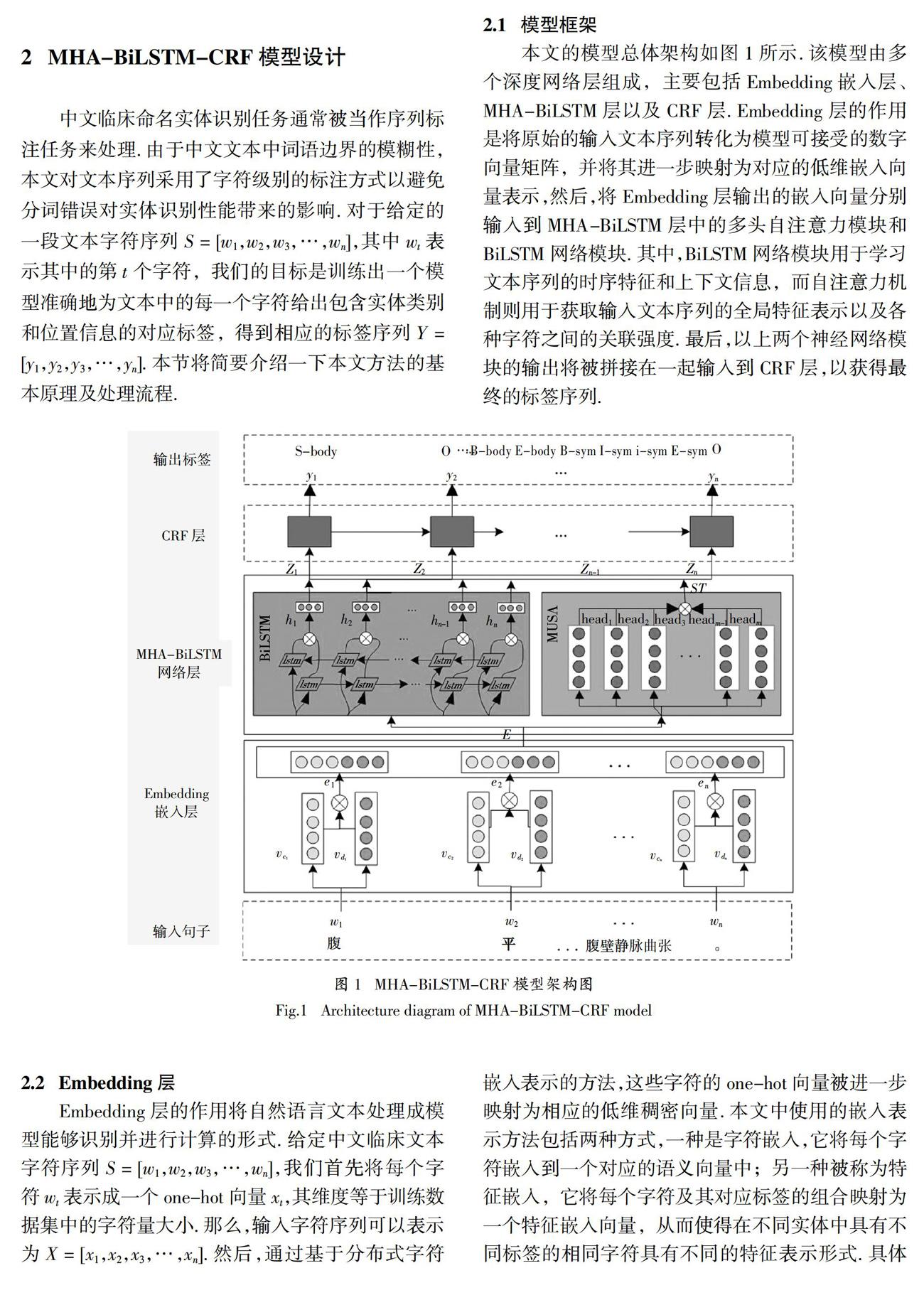
2.1 模型框架
本文的模型總体架构如图1所示. 该模型由多个深度网络层组成,主要包括Embedding嵌入层、MHA-BiLSTM层以及CRF层. Embedding层的作用是将原始的输入文本序列转化为模型可接受的数字向量矩阵,并将其进一步映射为对应的低维嵌入向量表示,然后,将Embedding层输出的嵌入向量分别输入到MHA-BiLSTM层中的多头自注意力模块和BiLSTM网络模块. 其中,BiLSTM网络模块用于学习文本序列的时序特征和上下文信息,而自注意力机制则用于获取输入文本序列的全局特征表示以及各种字符之间的关联强度. 最后,以上两个神经网络模块的输出将被拼接在一起输入到CRF层,以获得最终的标签序列.
2.2 Embedding层
Embedding层的作用将自然语言文本处理成模型能够识别并进行计算的形式. 给定中文临床文本字符序列S = [w1,w2,w3,…,wn],我们首先将每个字符wt表示成一个one-hot向量xt,其维度等于训练数据集中的字符量大小. 那么,输入字符序列可以表示为X = [x1,x2,x3,…,xn]. 然后,通过基于分布式字符嵌入表示的方法,这些字符的one-hot向量被进一步映射为相应的低维稠密向量. 本文中使用的嵌入表示方法包括两种方式,一种是字符嵌入,它将每个字符嵌入到一个对应的语义向量中;另一种被称为特征嵌入,它将每个字符及其对应标签的组合映射为一个特征嵌入向量,从而使得在不同实体中具有不同标签的相同字符具有不同的特征表示形式. 具体过程描述如下.
2.2.1 字符嵌入
字符是中文自然语言处理的最小语义单元,本文采用预训练字符嵌入向量表来获得丰富的字符级文本特征表示. 该查找表通过未标注的中文维基百科数据集经word2vec训练得到,包含了16 691个汉字及其对应的向量表示. 在获取字符嵌入向量的过程中,对于中文临床文本中的任意字符,我们首先在该字符向量表中查找是否存在该字符对应的嵌入向量,若找到匹配项,则将该字符以匹配的字符嵌入向量表示;否则,将其表示为一个随机向量. 这样,输入序列将被转换为字符嵌入向量序列Vc = [v c1,v c2,v c3,…,v cn]. 其中,每个字符嵌入向量的维数d设为100.
2.2.2 特征嵌入
字符嵌入仅实现了字符自身特征的降维表示,然而同一个字符可能出现在不同临床命名实体中的不同位置,这些信息蕴含了与相关实体的依赖关系,对于命名实体的识别具有十分重要的意义. 现有的方法大多采用单独将字符的特征标签(如所属实体的类别以及BIOES位置标注等)转化为对应的特征向量的方式. 但是,这种方式把字符和其对应的标签完全分离开来进行表示,无法捕获字符在对应实体中的依赖关系特征. 因此,在本文的方法中,我们将每个字符与其对应标签作为一个整体,然后通过word2vec将其表示为相应的特征嵌入向量,从而尽可能全面地捕获字符与实体的相关特征以得到更丰富的字符特征表示.
为了获得更全面的字符特征标签,我们使用了一个包含多种临床实体的医学领域词典. 其中的临床实体均由ICD-10、ICD-9-CM以及如搜狗细胞词库、在线医疗咨询网站等其他系统中抽取得来,主要包括五种实体类别:疾病、症状描述、检查项目、身体部位以及治疗. 对于给定的输入文本序列S,我们首先根据该词典利用双向最大化匹配算法(Bi-Direction Maximum Matching,BDMM),对输入文本进行实体划分. 若输入文本序列中的某个子串在词典中发生匹配,则将该子串作为一个临床实体并对其进行相应的类别标注. 没有发生匹配的字符则统一标记为“None”. 然后,我们再利用BIOES标注机制为每个字符加上其所在实体的位置标签. 标注样具体如表1所示. 接着,我们将字符与其对应标签(实体类别标签+位置标签)结合起来转化为相应的联合特征嵌入向量v dt.
最终,对于任意字符的输入xt,我们将上述两个嵌入过程得到的字符嵌入向量v ct和联合特征嵌入向量v dt拼接起来作为它的最终向量表示et,如公式(1)所示.
式中:表示向量拼接运算. 这样,输入字符序列即可表示为E = [e1,e2,e3,…,en].
2.3 MHA-BiLSTM层
为了更好地获取文本的时序特征、字符上下文信息以及文本序列中字符之间的相关权重,本模型在Embedding层之后部署了一个由两个独立模块组成的MHA-BiLSTM层. 一个是BiLSTM模块,另一个则是多头自注意力模块. Embedding层输出的向量矩阵将分别输入到上述两个模块进行处理,然后再将两个模块的输出向量拼接后得到该层的最终输出.
2.3.1 BiLSTM模块
BiLSTM利用来自Embedding层的输出向量矩阵E,通过结合序列中每个字符的上下文信息来获取更全面的特征表示.
对于输入序列中任意位置t上的字符向量et,LSTM将结合et和前一时刻的状态ht-1来计算当前的隐藏状态ht . 具体实现过程如公式(2)~(7)所示:
2.3.2 多头自注意力模块
传统的BiLSTM-CRF模型无法充分表达文本序列的全局信息以及句中各个字符的重要性,而且随着句子长度的增加,BiLSTM在训练过程中有可能会遗失大量对于命名实体识别极为重要的信息. 例如,不同的词语或字符在同一语句中往往具有不同的作用,同一词语或字符在不同语句中的含义也可能存在明显的差异. 这些特征对于准确地理解词语/字符间的关系和上下文含义具有重要意义. 因此,我们引入了多头自注意力机制作为BiLSTM模塊的补充,从字符、单词和句子的层面捕获多种语义特征来进一步提高临床命名实体识别的性能.
该过程将Embedding层的输出向量矩阵E通过三次不同的映射操作分别转换成三个维度均为dk的输入矩阵:查询Q、键K和值V,并传入如公式(9)所示的注意力函数中. 注意力函数将根据Q与K之间的相关性计算V上的权值,进而得到相应的混合向量表示.
在多头自注意力模块中,查询Q、键K和值V将分别使用不同的参数矩阵进行h次独立的线性映射,然后相应地输入到h个并行头中执行上述的注意力函数运算. 这样,每个并行头都可以捕获文本序列中各字符在不同表示子空间上独特的特征信息. 接下来,h个并行头上的计算结果进行合并后再经过一次线性映射得到最终的输出ST = [st1,st2,st3,…,stn],具体过程如公式(10)和(11)所示.
2.4 CRF层
充分考虑相邻字符标签之间的依赖关系和约束条件对于中文临床命名实体的识别和分类具有重要的意义. 因此,我们在模型的最后采用条件随机场(CRF)对MHA-BiLSTM层生成的融合特征信息进行解码来得到文本的字符标签序列.
将上层的输出序列Z = [st1,st2,st3,…,stn]作为输入,CRF层根据上下文前后的字符标签来预测得到可能性最大的标签序列Y = [y1,y2,y3,…,yn].
令θ表示CRF层的参数集合,那么,我们可以通过最大化对数似然函数,如公式(13)所示,来得到所有参数的估计:
式中:Y是文本字符序列对应的标签序列;p表示给定输入特征序列Z和参数集合θ时Y的条件概率.
为了求得式(13)中的条件概率p,我们先根据公式(14)对转移概率矩阵A与MHA-BiLSTM层的输出Z进行求和计算出预测序列Y的得分Sθ(Z,Y),然后,再对Sθ(Z,Y)进行归一化即得到对应的条件概率p.
Sθ(Z,Y) = ∑n t=1(Z yt,t + A yt-1,yt) (14)
这里,Z yt,t表示输入的当前时刻的字符wt被标记为yt标签的概率值,A yt-1,yt则表示t-1时刻的字符被wt标记为标签并且 被标记为 标签的概率. 最终的解码阶段通过CRF中的标准Viterbi算法[18],预测出全局最优的标注序列.
3 实验结果与分析
本节将通过与几种最新的临床命名实体识别方法的对比实验来评估本文方法的有效性.
3.1 数据集
本文实验中的数据均来自CCKS2017-CNER数据集. 该数据集是目前广泛采用的用于中文临床命名实体识别任务公开测评的标准数据集. 其中包含了来自不同科室的400份中文临床记录(300份作为训练集,余下的100份作为测试集),共计1 596个标注实例(10 024个不同的语句),并经多名临床专家的人工标注将其中的实体分为了疾病、症状、检查项目、身体部位以及治疗五种类别. 同时,样本中的句子均根据中文的标点符号(句号或者感叹号等)划分成了子句以方便处理. 数据集中相关临床实体的详细统计信息如表2所示.
3.2 基准模型
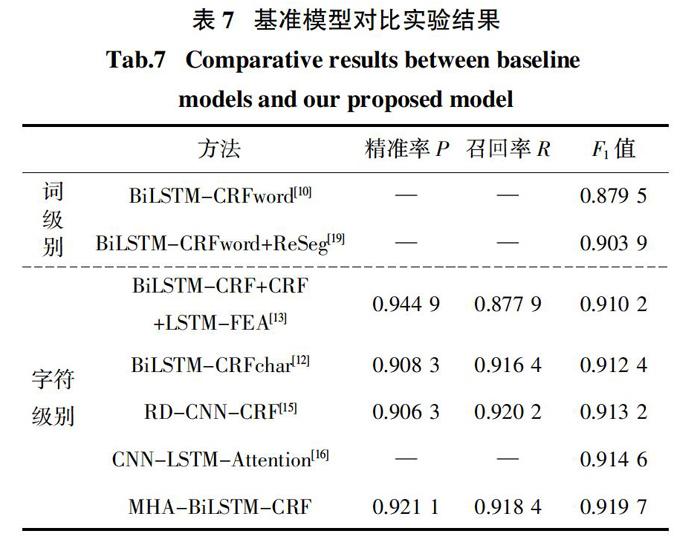
本文选取了几种典型的基于词级特征嵌入和字符级特征嵌入的命名实体识别方法作为基准模型来评估本文方法的有效性,主要包括了词级别的模型BiLSTM-CRFword[10]、BiLSTM-CRFword + ReSeg[19]以及字符级别的模型BiLSTM + CRF + LSTM-FEA[13]、BiLSTM-CRFchar[12]、RD-CNN-CRF[15]、CNN-LSTM-Attention[16].
3.3 实验设置与评价指标
3.3.1 实验设置
本文实验均基于Python 3.6编程语言开发,采用Keras 2.4工具包以及Tensoflow1.13.0进行模型的实现. 训练过程中,所有模型均通过Adam优化算法进行参数优化,初始学习率设为0.01. 同时,采用了early-stop和dropout策略以防止过拟合,并通过梯度裁剪来解决梯度爆炸问题. 主要的实验参数设置如表3所示.
3.3.2 评价指标
本文采用了CCKS2017-CNER挑战赛提供的官方测试数据集和测评标准来对所有模型进行性能评估. 对于模型识别出的实体,仅当实体边界和类别均与标准结果完全一致时才被判定为一次正确的识别. 所用的评价指标主要包括:微平均精准率micro-average precisions(P)、微平均召回率micro-average recall(R)以及微平均F1值(F1).
3.4 结果分析
接下来,我们对本文方法与其他典型的命名实体识别模型的性能进行对比,并从不同的方面验证本文方法的优越性.
3.4.1 多头自注意力机制的优势
本文希望通过引入多头自注意力机制来提升模型的命名实体识别能力,而将注意力机制与BiLSTM-CRF进行结合存在多种可行的方式. 因此,我们首先尝试了4种不同的网络结构,并通过实验性能对比来选择最佳的组合方式. 如图2所示,模型I采用了将多头自注意力模块直接置于BiLSTM网络层之后的方式;模型II将多头自注意力模块置于BiLSTM层与embedding层之间;模型III在模型II的基础上采用残差结构将多头自注意力模块的输出与其经过BiLSTM模块处理后的输出进行融合再输入CRF层的方式;模型IV则将embedding层的输出独立并行地分别送入到BiLSTM与多头自注意力模块中,将这两个模块的输出进行融合后输入CRF层得到最终结果.
从表4中的实验结果可以发现,BiLSTM与多头自注意力机制采用并行组合的两种模型(模型III和模型IV)较之采用串行组合的模型(模型I和模型II)具有更好的效果. 其中,模型IV的精准率、召回率和F1值分别达到了0.905 6、0.909 1和0.907 3,获得了四者中最优的性能. 这说明模型IV能够更好地发挥多头自注意力机制与BiLSTM的互补作用,因此,本文将其作为最终的模型架构.
同时,为了证明多头自注意力机制对于中文临床命名实体识别的好处,我们在使用相同的特征表示方法和字典特征的前提下,选择了两个MHA-BiLSTM-CRF的修改版本来与本文方法进行性能对比. 一个是通过去除MHA-BiLSTM-CRF中的多头自注意力模块得到的BiLSTM-CRF基础模型,另一个则是将MHA-BiLSTM-CRF的多头自注意力模块替换为单头自注意力模块得到的SA-BiLSTM-CRF模型.
从表5所示的实验结果可以看出,由于自注意力機制很大程度上弥补了LSTM模型在捕获字符间关联关系能力方面的不足,因此,不论是否使用字典特征,SA-BiLSTM-CRF的性能均要优于BiLSTM-CRF. 而本文提出的基于多头自注意力机制的MHA-BiLSTM-CRF模型更是超过前两者获得了最佳的性能表现. 这说明多头自注意力机制能充分结合不同层次不同角度的相关特征来增强模型的表示能力,从而进一步提高了实体识别的整体性能. 另外,从表中不难发现,当使用了外部词典特征时,每个模型的性能都出现了明显的提升. 这也验证了领域词典在提高命名实体识别性能方面的重要作用.
为了进一步理解MHA-BiLSTM-CRF模型的优势,我们也做了一些实际的模型识别结果对比分析. 如表6所示,分别给出了BiLSTM-CRF与MHA-BiLSTM-CRF模型的真实识别结果对比. 可以看出,BiLSTM-CRF模型针对中文临床命名实体中实体词较长时具有明显的识别错误,如第一个实例中的治疗实体“腰麻下行阑尾切除术”,BiLSTM-CRF模型将其错误识别为治疗实体“阑尾切除术”,第三个实例中的检查项目实体“肱二、三肌腱反射”,BiLSTM-CRF模型将其错误识别为身体部位实体“三肌腱”,然而MHA-BiLSTM-CRF模型均能准确地识别出这些实体. 这也充分说明了MHA-BiLSTM-CRF能够弥补BiLSTM-CRF的不足,有效学习并捕获到字符序列中的长距离依赖信息,在实体较长时仍然能够正确识别出中文临床命名实体.
3.4.2 与其他基准模型的对比结果
表7展示了几种典型的命名实体识别模型在CCKS2017-CNER数据集上的性能. 从表中我们可以看到两个基于词级特征嵌入的模型(BiLSTM-CRFword和BiLSTM-CRFword + ReSeg)性能相对较差. 其主要原因在于基于词级别特征的方法通常依赖分词工具的准确性,而分词错误必然严重地影响该类方法的实体识别性能. 而与基于词级特征嵌入的模型相比,使用字符级特征的模型摆脱了对分词精度的依赖,因而能够获得相对更高的性能. 例如,同样基于BiLSTM-CRF模型,使用字符级嵌入的BiLSTM-CRFchar的F1值达到了0.912 4,比使用词级嵌入的BiLSTM-CRF高出了0.032 9. RD-CNN-CRF利用残差扩张的卷积神经网络捕获上下文特征,获得了0.913 2的F1值. 然而,CNNs通常主要关注局部特征,没有充分利用文本的长期依赖性,因此其性能在所有对比方法中仅排在第三位. 而CNN-LSTM-Attention则结合了CNN和LSTM各自的优势来获取局部上下文信息和单词的时序依赖关系,并融合了另外两种方法(CRF和LSTM-CRF)来构建集成模型,进一步将F1值提高到了0.914 6. 得益于精确的特征表示以及多头自注意机制的引入,本文方法MHA-BiLSTM-CRF不仅可以全面地挖掘字符级、词级以及句子级的各种语义和结构特征,而且还能捕获句子中不同字符的重要性. 因此,它获得了明显优于其他对比模型性能,F1值达到了0.919 7. 尽管部分模型在精准率或召回率上似乎略高于MHA-BiLSTM-CRF,例如,BiLSTM + CRF + BiLSTM-FEA的精确率达到了0.944 9,而RD-CNN-CRF则获得了0.920 2的召回率. 但是,另一方面,它们的召回率和精确率却分别仅为0.877 9和0.906 3,远远低于我们模型. 作为结合精确率和召回率的综合指标,F1值能更全面有效地评估不同模型的整体性能. 因此,在F1值方面的比较结果更充分地说明了本文方法的优越性.
3.4.3 不同特征表示方法的影响
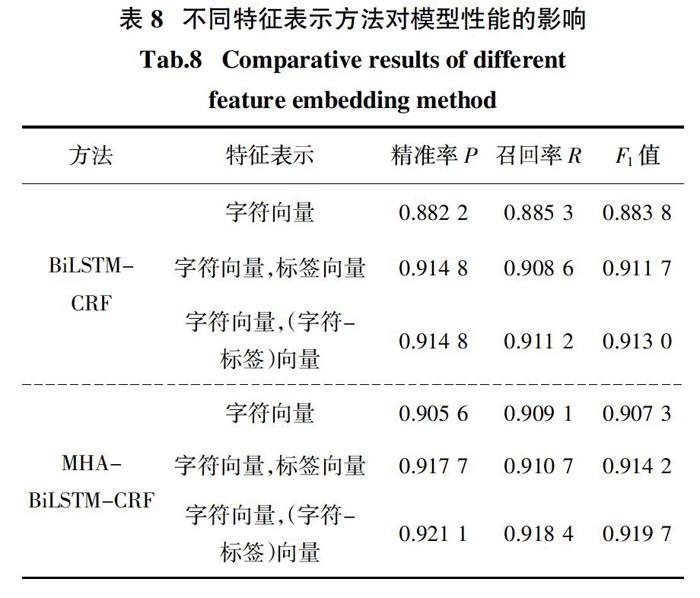
为了分析不同特征嵌入方法对实体识别性能的影响,我们以BiLSTM-CRF和MHA-BiLSTM-CRF模型为例,分别比较采用三种不同的嵌入方法(字符嵌入、字符嵌入+标签嵌入以及字符嵌入+字符-标签嵌入)时的性能. 从表8结果可以看到,本文提出的特征嵌入方法(字符嵌入+字符-标签嵌入)与其他两种嵌入方法相比具有明显的性能优势. 例如,仅采用字符嵌入时,MHA-BiLSTM-CRF模型仅获得了0.907 3的F1值,结合标签嵌入时,其F1值增加到了0.914 2,而采用字符嵌入和字符-标签嵌入的组合时,模型的F1值进一步提升了0.005 5.
这也充分地证明了本文所提出的特征表示方法的有效性. 其主要原因在于,与使用单一标签嵌入的传统方法相比,本文方法将字符和特征标签组合成一个整体来重新训练新的字符标签嵌入向量,该向量可以为出现在不同临床命名实体中的相同字符提供富含领域特性不同表示形式,极大地增强了特征表示的特异性和多样性.
3.4.4 数据不均衡的影响分析
从表2的实体统计数据中可以明显看出,数据集中的实体分布很不均衡,尤其是疾病和治疗实体的数量远远低于其他实体,在训练集中大概只占实体总数的4%左右,然而其他实体的比例超过了25%. 这也导致了模型对于疾病和治疗实体的识别准确率远远低于其他实体,性能结果如图3所示.
因此,为了缓解这一影响,我们对数据集中的样本进行了数据均衡处理,特别针对疾病和治疗实体进行了重采样处理. 实验结果如表9所示,不论是BiLSTM模型还是MHA-BiLSTM-CRF模型,在进行数据不均衡处理之后整体性能明显提升. 此外,还可以从图3中看到,MHA-BiLSTM-CRF识别疾病实体和治疗实体的能力显著增强. 对于治疗实体的识别、精确率、召回率和F1值由0.704 5、0.702 9、0.712 6分别增加至0.841 5、0.802 3、0.821 4. 因此,数据均衡处理提高了整体的识别性能MHA-BiLSTM-CRF的精确率为0.9056,召回率为0.909 1、F1值为0.907 3,比未进行处理的F1-score分别高0.010 8、0.023 7、0.017 2.
4 结 论
临床命名实体识别是许多临床信息抽取任务中最关键也是最基础的环节. 然而,目前很多针对中文临床命名实体识别任务的模型均不能夠很好地捕获文本序列中的全局特征信息以及文本序列内部的字符与字符之间的关联权重信息,且特征表示能力不足. 因此,本文首先设计了一种改进的字符级特征表示方法,将字符嵌入和字符-标签嵌入相结合以增强特征表示的特异性和多样性. 然后,在此基础上提出了一种结合多头自注意力机制和BiLSTM-CRF的中文临床命名实体识别方法. 通过引入多头自注意力机制并结合相关的医学词典,该方法可以更有效地捕获临床文本中的字符间的权重关系和多层次的语义特征信息,从而提高中文临床命名实体的识别能力.
当然,本文方法也存在着一定的局限性. 一方面,本文模型的性能很大程度上依赖于充足的高质量标注数据;另一方面,我们仅在CCKS2017-CNER数据集上对模型进行了性能评估,而在其他数据集上的有效性仍然有待进一步验证. 在未来的工作中,我们将会使用更多其他相关的数据集来测试模型的可扩展性和泛化能力,并测试优化模型在有限标注数据集的情况下的性能.
参考文献
[1] LAFFERTY J,MCCALLUM A,PEREIRA F CN. Conditional random fields:Probabilistic models for segmenting and labeling sequence data[C]// Proceedings of the Eighteenth International Conference on Machine Learning (ICML 2001). San Mateo,CA:Morgan Kaufmann,2001:282—289.
[2] LAMPLE G,BALLESTEROS M,SUBRAMANIAN S,et al. Neural architectures for named entity recognition[C]// Proceedings of the 15th Conference of the North American Chapter of the Association for Computational Linguistics:Human Language Technologies. San Diego,CA:Association for Computational Linguistics,2016:260—270.
[3] DUAN H,ZHENG Y. A study on features of the CRFs-based Chinese named entity recognition[J]. International Journal of Advanced Intelligence Paradigms,2011,3(2):287—294.
[4] ZHOU G D,SU J. Named entity recognition using an HMM-based chunk tagger[C]// Proceedings of the 40th Annual Meeting on Association for Computational Linguistics. Philadelphia,Pennsylvania. Morristown,NJ,USA:Association for Computational Linguistics,2001:473—480.
[5] AMARAPPA S,SATHYANARAYANA S V. Kannada named entity recognition and classification using support vector machine[J]. Transactions on Machine Learning and Artificial Intelligence,2017,5(1):43—43.
[6] MCCALLUM A,LI W. Early results for named entity recognition with conditional random fields,feature induction and web-enhanced lexicons[C]//Proceedings of the Seventh Conference on Natural Language Learning at HLT-NAACL 2003-Volume 4. Stroudsburg,PA:Association for Computational Linguistics,2003:188—191.
[7] HUANG Z,XU W,YU K. Bidirectional LSTM-CRF models for sequence tagging[J]. Computer Science,2015.
[8] 張俊飞,毕志升,王静,等. 基于BLSTM-CRF中文领域命名实体识别框架设计[J].湖南大学学报(自然科学版),2019,46(3):117—121.
ZHANG J F,BI Z S,WANG J,et al. Design of Chinese domain named entity recognition framework based on BLSTM-CRF[J]. Journal of Hunan University(Natural Sciences),2019,46(3):117—121. (In Chinese)
[9] MA X,HOVY E. End-to-end sequence labeling via bi-directional LSTM-CNNs-CRF[C]//Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. San Diego,CA:Association for Computational Linguistics,2016:1064—1074.
[10] LI Z,ZHANG Q,LIU Y,et al. Recurrent neural networks with specialized word embedding for Chinese clinical named entity recognition[C]// Proceedings of the Evaluation Task at the China Conference on Knowledge Graph and Semantic Computing. Berlin,German:Springer,2017:55—60.
[11] OUYANG E,LI Y,JIN L,et al. Exploring n-gram character presentation in bidirectional RNN-CRF for Chinese clinical named entity recognition[C]// Proceedings of the Evaluation Task at the China Conference on Knowledge Graph and Semantic Computing. Berlin,German:Springer,2017:37—42.
[12] XIA Y,WANG Q. Clinical named entity recognition:ECUST in the CCKS-2017 shared task 2[C]// Proceedings of the Evaluation Task at the China Conference on Knowledge Graph and Semantic Computing. Berlin,German:Springer,2017:43—48.
[13] HU J,SHI X,LIU Z,et al. HITSZ_CNER:a hybrid system for entity recognition from Chinese clinical text[C]// Proceedings of the Evaluation Task at the China Conference on Knowledge Graph and Semantic Computing. Berlin,German:Springer,2017:25—30.
[14] WANG Q,ZHOU Y,RUAN T,et al. Incorporating dictionaries into deep neural networks for the Chinese clinical named entity recognition[J]. Journal of Biomedical Informatics,2019,92:103133.
[15] QIU J,ZHOU Y,WANG Q,et al. Chinese clinical named entity recognition using residual dilated convolutional neural network with conditional random field[J]. IEEE Transactions on Nano Bioscience,2019,18(3):306—315.
[16] TANG B,WANG X,YAN J,et al. Entity recognition in Chinese clinical text using attention-based CNN-LSTM-CRF[J]. BMC Medical Informatics and Decision Making,2019,19(3):74.
[17] VASWANI A,SHAZEER N,PARMAR N,et al. Attention is all you need[C]//Proceedings of the 31st Annual Conference on Neural Information Processing Systems. Piscataway,NJ:IEEE,2017:5998—6008.
[18] VITERBI A. Error bounds for convolutional codes and an asymptotically optimum decoding algorithm[J]. IEEE Transactions on Information Theory,1967,13 (2):260—269.
[19] WU J,HU X,ZHAO R,et al. Clinical named entity recognition via bi-directional LSTM-CRF model[C]// Proceedings of the Evaluation Task at the China Conference on Knowledge Graph and Semantic Computing. Berlin,German:Springer,2017:31—36.
收稿日期:2019-10-14
基金項目:湖南省自然科学基金资助项目(2018JJ2534),Natural Science Foundation of Hunan Province(2018JJ2534);网络犯罪侦查湖南省普通高校重点实验室开放基金资助项目(2020WLFZZC003),Open Research Fund of Key Laboratory of Network Crime Investigation of Hunan Provincial Colleges(2020WLFZZC003);国家重点研发计划资助项目(2016YFC0901705),National Key Research and Development Program of China(2016YFC0901705);湖南省重大科技专项(2017SK1040),The Major Science and Technology Special Project of Hunan Province(2017SK1040);高新技术产业科技创新引领计划(2020GK2029),The Science and Technolgy Innovation Leading Program for High and New Technology Industry(2020GK2029)
作者简介:罗熹(1980—),女,湖南长沙人,中南大学副教授
通信联系人,E-mail:anying@csu.edu.com
