基于机器学习方法的拟南芥基因组DNA复制时间预测研究
2021-05-06李东维李昭宏杨若林
李 椰,李东维,李昭宏,杨若林
(西北农林科技大学 生命科学学院,陕西 杨凌 712100)
真核生物基因组的复制在时空上受到严格的调控,细胞内DNA复制发生在细胞周期S期,基因组不同位点DNA在各自开始的时间点精确复制,其间不仅发生DNA复制,也涉及染色质状态、表观信息、染色质空间结构的复制[1-2];同时,该复制过程也伴随着其他生物学过程如核小体重塑[3]。根据DNA复制时间早晚特征,可将基因组分为CTR区(constant timing regions,CTR)和TTR区(timing transition regions, TTR)[4]。CTR区是指由多个相邻复制子构成的一段DNA区域,该区域内的复制子同步激活,出现在S期早期(早期复制区CTR)或晚期(晚期复制区CTR)[5]。TTR区则对应于连接上下游2个具有不同CTR的DNA区域[1]。目前,利用Repli-Seq(replication timing sequencing)测序技术,可以在全基因组水平对DNA的复制时间进行系统研究。该技术通过5-溴-2-脱氧尿苷(bromouridine triphosphate,BrdU)在体内替换胸腺嘧啶,从而对新合成的DNA链进行标记,根据不同时期DNA含量的不同,利用荧光激活细胞分选技术(fluorescence-activated cell sorting,FACS)对不同复制时间的DNA分选,用BrdU抗体免疫沉淀BrdU标记的DNA链,对该DNA进行测序[6]。通过Repli-Seq技术对一些生物的DNA复制时间进行鉴定分析,发现早晚期复制的区域存在着明显差异,例如复制早期的DNA区域的基因密度较高,具有更高的转录活性,表达量更高,且富集激活型的染色质标记[7],复制早晚期也与结构变异类型及突变率存在联系[8]。同时,DNA复制早晚的异常及改变与人类的癌症有关[8]。然而,目前研究人员对DNA复制时间调控机制的了解尚不完善。
DNA复制在不同物种内也存在差异。早期的研究表明,异六聚体起始识别复合物(origin recognition complex, ORC)能特异性地结合到酵母基因组的特定位点[9],这对于保证酵母基因组的准确复制有着重要的作用。然而,在高等真核生物中,仅仅依据结合位点的序列特征并不能完全解释该复合物与DNA的结合,这意味着有DNA序列特征之外的其他因素也参与了DNA复制起始的调控[10]。近年来,针对其他物种的研究发现,基因组局部的染色质状态、表观遗传修饰和染色质三维结构变化会影响DNA复制的起始时间。例如,人类甲基化的RNA基因复制时间较晚[11]。在小鼠中,激活型组蛋白修饰(H3K4me1、H3K4me2、H3K4me3、K3K20me1、H3K36me3、H3K9ac、H3K27ac)与早期复制相关[12];果蝇中也有相似的发现[10]。另外,DNA的乙酰化也会影响复制时间[1,13]。在植物中,玉米的DNA复制与动物存在差异[14]。但有关植物全基因组水平上DNA复制的时空调控机制目前尚少有报道。
随着二代测序技术的普及和发展,植物基因组中尤其是拟南芥基因组中大量的表观遗传修饰被测定[15-25],为探索DNA的复制时间与表观遗传修饰之间的关系提供了有力的数据支撑。与此同时,机器学习方法目前已在图像分类、语音识别等领域取得了重大突破和进展[26-27]。近年来,研究者将机器学习方法与生物组学数据结合,更为全面、系统地揭示了DNA序列与表观遗传修饰的联系[28],以及表观遗传修饰与基因表达之间的生物学机制[29-30]。本研究以模式植物拟南芥为对象,系统地收集和整合染色质开放状态(DNase-Seq)数据以及表观遗传修饰特征(ChIP-Seq)数据,利用机器学习方法,构建拟南芥基因组DNA复制时间的预测模型;在此基础上发现可能参与调控DNA复制时间的表观遗传修饰,以期为后续进一步探究拟南芥DNA复制时间的表观遗传调控机制提供参考。
1 材料与方法
1.1 试验材料
从NCBI数据库收集拟南芥全基因组水平染色质开放状态的DNase-Seq数据和多种表观遗传修饰的ChIP-Seq数据,相关信息如表1所示[15-16,18-25,29,31]。拟南芥基因组从Ensembl plant网站上下载(http://plants.ensembl.org/index.html)。

表1 拟南芥表观遗传修饰特征和染色质状态数据集信息Table 1 Information on datasets of investigated epigenetic features and chromatin status of Arabidopsis thaliana
拟南芥的全基因组复制时间数据来自于PRJNA330547。该研究将基因组划分为长度为1 kb的区间,通过测序将这些区间鉴定和划分为复制早期、中期或晚期,并提供了每个区间的信号值[7]。本研究据此进行DNA复制时间与各种基因组表观遗传修饰信号之间关系的分析。
1.2 方 法
1.2.1 DNase-Seq和ChIP-Seq数据处理 用fastq-dump(v2.8.2)将下载的拟南芥DNase-Seq和ChIP-Seq原始数据(sra格式)转换为fastq格式文件;利用fastqc(v0.11.4)对reads序列的测序质量进行检查,使用Trimmomatic(v0.36)去除不合格reads两端低质量序列和接头序列;使用软件bowtie2将合格的reads序列比对到拟南芥参考基因组上获得bam文件,并将不同组织相同类型的表观修饰数据视为不同的特征。最后共得到31个特征,其中包括29个表观遗传修饰特征和2个染色质开放状态特征。
1.2.2 样本的特征信号值计算 对于每种表观遗传修饰特征,利用其对应的bam文件计算基因组每1 kb区间的表观遗传修饰信号值。以表观修饰特征H3K27ac为例,计算过程为:统计每个区间中的reads数目;为防止某些区域没有reads覆盖,对所有区间的reads数目均加1;对所有区间内的reads数目取对数,利用Z-score方法对压缩后的reads数目标准化,得到每个区间的信号值。具体计算公式如下:


1.2.3 t分布邻域嵌入(t-distributed stochastic neighbor embedding,t-SNE)分析 t-SNE利用非线性降维算法将高维数据投影到低维空间,不仅可以实现很好的可视化效果,而且可以反映这些特征能否将不同类别的数据区分开。t-SNE的核心思想是数据在高维空间内的欧氏距离与降维后低维空间内的欧式距离能够最大程度地保持一致。利用KL散度(Kullback-Leibler divergence)衡量高低维欧式距离的差值,通过最小化KL散度可得到最优降维。该方法既能聚焦于高维数据的局部结构,又能兼顾全局的结构,通过R(v3.6)软件包Rtsen完成计算。
1.2.4 34个特征的相关性 本研究计算了34个特征,包括DNA复制早期、中期、晚期的信号和原本31个表观遗传修饰特征两两之间的线性关系。皮尔逊相关系数(Pearson correlation coefficient,PCC)能够很好地度量两个随机变量的线性关系,其取值范围为[-1,1],其绝对值的大小反映两个变量线性相关性的强弱,而正负方向反映变量之间存在正相关或负相关,通过R(v3.6)软件中的基础包cor.test函数,可计算34个特征两两之间的PCC显著性。
1.2.5 DNA复制期早晚预测模型以及表观遗传修饰特征的重要性评估 用随机森林、多类别逻辑回归模型和支持向量机3种机器学习分类器进行建模分析,分别由R包e1071、nnet和randomForest执行。其中支持向量机的核函数选用径向基函数(radial basis function),其余参数均为默认参数。随机森林选择1 000棵决策树;随机选取80%数据作为训练集,剩余20%数据作为测试集用于评估模型的性能。接受者操作特性曲线(receiver operating characteristic curve,ROC)和ROC曲线下的面积(area under curve,AUC),以及每个模型的表观遗传修饰特征的重要性,均用对应的R函数完成计算。
十折交叉验证:将训练集样本随机分为10份,轮流选取9份构建模型,剩余1份用于评价模型性能。计算每个类别的10次AUC平均值来评估模型的预测性能。
2 结果与分析
2.1 拟南芥基因组表观数据的可预测性及相关性
为了初步检查31个表观遗传修饰特征的联合信号能否预测对应DNA区域的复制时间早晚,首先使用t-SNE变换将所有拟南芥DNA片段的高维表观遗传修饰特征展示到二维空间,然后以3种不同颜色将对应DNA片段的复制时间按早、中、晚类别信号显示,结果见图1。如图1所示,复制晚期的DNA片段明显聚类到一起,与早期和中期片段分开;而早期和中期的DNA片段虽然没有明显分开,但也呈现一定程度的各自聚类模式,说明这些表观遗传修饰特征整体上能够有效地用于预测DNA区域的复制早晚。
31个表观遗传修饰特征与DNA片段复制早、中、晚3个时期信号值之间的相关性结果(图2)显示,一些表观遗传修饰特征之间存在较高的相关性,复制早期的信号与一些表观遗传修饰特征(如H4K16ac)呈较强的正相关(PCC=0.394,P<2.2×10-16),复制晚期的信号则与之相反(PCC=-0.525,P<2.2×10-16),而复制中期的信号与大部分表观遗传修饰特征的相关性较小。而对于表观遗传修饰特征H2AW而言,它们则与复制早期信号呈显著负相关(PCC=-0.508,P<2.2×10-16),而与复制晚期的信号呈正相关(PCC=0.612,P<2.2×10-16)。

图1 拟南芥基因组DNA复制早、中、晚3个时期的样本分群结果Fig.1 Clustering results of Arabidopsis thaliana genome-wide DNA replication timing classification

每个小格的扇形面积表示对应两个特征的相关性大小,×表示对应两个特征之间相关性不显著(P>0.05)The fan-shaped area of each cell represents the degree of correlation between two features,× represents insignificant correlation between two features (P>0.05)图2 拟南芥基因组表观遗传修饰特征与复制时间信号之间的相关性Fig.2 Correlation among all epigenetic features and signal of DNA replication timing in Arabidopsis thaliana
2.2 DNA复制时间的模型预测
拟南芥基因组DNA片段按照复制时间早晚被分为36 671个早期样本、23 113个中期样本和9 052个晚期样本。将其中一类样本作为正样本,其他两类作为负样本进行建模。从中各随机挑选80%样本用于模型构建和性能的评价,剩余20%样本作为独立的测试集。
为了评估随机森林、多类别逻辑回归模型和支持向量机3种分类器的预测性能,对80%数据在3种分类器上进行十折交叉验证。图3展示了3种分类器对复制早期、中期、晚期三个类别的平均AUC以及宏平均(Macro)和微平均(Micro)指数,宏平均和微平均分别表示每个类别的真阳性平均值和所有类别总真阳性。从图3可见,3种模型对DNA复制早期、中期、晚期预测的十折交叉验证平均AUC都在0.8以上,且变异很小;且3种分类器预测性能差异较小,说明复制时间早晚与多种表观遗传修饰的协同作用之间可能存在密切的生物学联系。从图3还可见,DNA复制晚期样本平均AUC最高,分类效果最好,说明表观遗传修饰特征很大程度上与该区域的复制时间有关。这与t-SNE分析结果吻合,较高的Macro和Micro指数表征了3种分类器关于DNA复制时间稳定的综合预测效应。
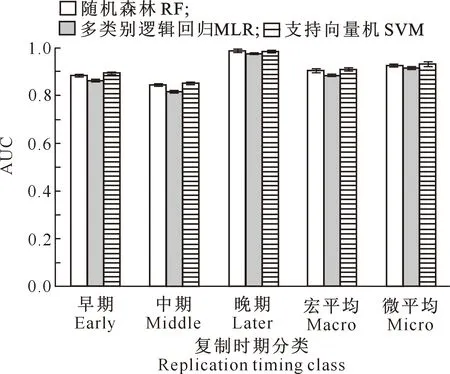
图3 基于3种不同模型的拟南芥DNA复制时间十折交叉验证平均AUCFig.3 Average AUC of 10-fold cross-validation with different classifier of DNA replication timing in Arabidopsis thaliana
为了进一步验证模型性能,本研究利用所选取的80%训练集数据构建了3种模型,再用独立的20%测试集数据验证。3种模型的ROC曲线(图4)和AUC值(表2)均表明,模型对DNA复制时间的预测性能良好,而且与十折交叉验证的结果一致,均表现为复制晚期的分类效果最佳。3种分类器中以支持向量机的预测效果最佳,其次为随机森林和多类别逻辑回归。由于基于独立测试集构建的模型使用了更多样本,因此,它们的AUC值略高于十折交叉验证的AUC值,使得模型训练更加准确。

图4 基于独立测试集的3种拟南芥DNA复制时间预测模型的ROC曲线Fig.4 ROC of three models in predicting DNA replication timing of Arabidopsis thaliana on an independent test set

表2 基于独立测试集的3种拟南芥DNA复制时间预测模型的AUC值Table 2 AUC of three models in predicting DNA replication timing on an independent test set
2.3 拟南芥基因组表观遗传修饰特征的重要性评估
由于在3种分类器中,随机森林(RF)和支持向量机(SVM)2种模型的预测能力更为突出,因此本研究通过随机森林和支持向量机的特征重要性,衡量拟南芥中可能对DNA复制早晚具有影响的表观遗传修饰。图5展示了随机森林和支持向量机2种分类器特征的重要性,其中,随机森林模型的特征重要性通过特征的平均下降精确度大小来衡量。
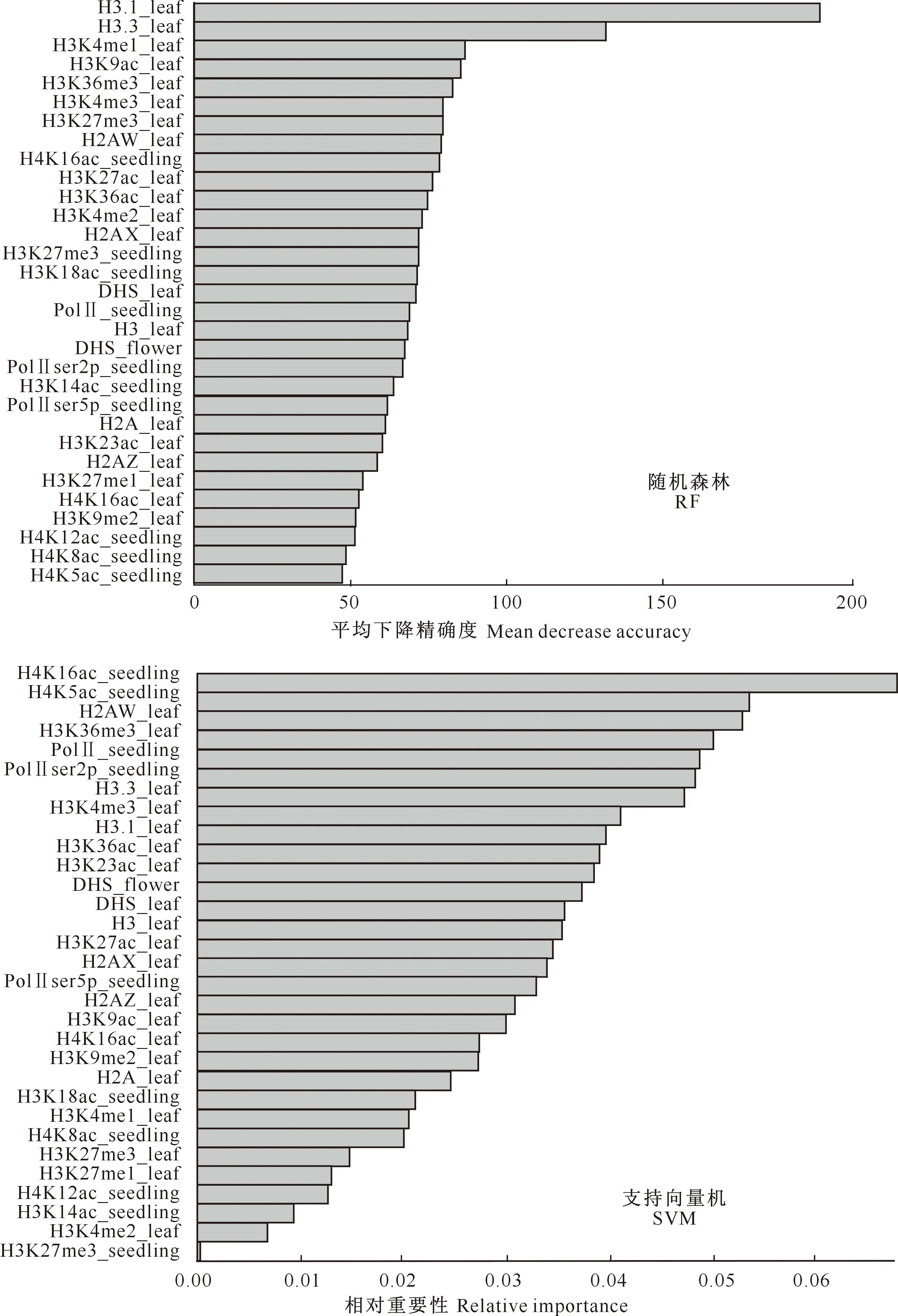
图5 基于随机森林和支持向量机分类器的特征重要性Fig.5 Feature importance of RF and SVM classifiers
图5显示,H3.1_leaf、H3.3_leaf、H2AW_leaf、H3K36me3_leaf、H3K4me3_leaf、H4K16ac_seedling在2个模型特征的重要性排序中都位于前10。H3K4me1_leaf、H3K9ac_leaf、H3K27me3_leaf、H3K4me2_leaf也位于RF模型特征重要性的前10,H4K5ac_seedling、polⅡ_seedling、polⅡser2p_seedling、H3K36ac_leaf也位于SVM模型特征重要性的前10,说明这些表观遗传修饰特征可能影响DNA的复制时间。
3 讨 论
本研究通过整合拟南芥多种表观遗传修饰数据,构建3种分类器(随机森林、多类别逻辑回归、支持向量机),实现对拟南芥基因组DNA复制时间的预测。结果显示,3种预测模型均能够准确地对DNA复制早期、中期、晚期进行预测和分类。
本研究构建的3种分类器均具有较高的预测和分类性能,表明拟南芥基因组中DNA的复制时间可以利用表观遗传修饰进行准确地预测和分类,这与模式生物果蝇中的研究结果[10]有相似之处。通过对各种表观遗传修饰与DNA复制时间的相关性分析发现,一些表观遗传修饰特征与复制时间相关,如大部分的乙酰化修饰、染色质开放程度与复制早期信号呈正相关,与复制晚期信号呈负相关,这些结果与已有研究报道相吻合(如H3K4me1)[10];相反,异染色质的表观遗传修饰特征与复制早期信号呈负相关,与复制晚期信号呈正相关,如H3K9me2(与果蝇上的报道一致)和H2A.W[10,21]。模型的特征重要性分析揭示,在DNA复制时间的表观遗传调控过程中,存在可能扮演重要作用的组蛋白修饰以及组蛋白变体,比如H3.1、H3.3、H3K4me3等。其中H3.1是典型的H3变体,主要在S期表达,并以依赖DNA复制的方式整合到染色质中[17];H3.3位于常染色质的转录活性区域内,包括基因内和启动子区域[32],其是预测DNA的复制时间的重要特征。此外,复制早期信号与RNA聚合酶结合位点、启动子区域相关的特征(H3K4me3)[19],以及激活型表观遗传修饰特征(H3K36me3、H3K4me3、H3K9ac、H4K16ac)呈正相关[19-20]。这可能是因为拟南芥复制早期的DNA位于基因组内高基因密度区域,其他物种中也存在类似现象[33]。
相对于动物和人类的研究,植物的DNA复制时间以及表观遗传修饰的相关研究尚处于起步阶段[7],这不仅表现在相关的调控机制尚不明确,也表现在相关组学数据的匮乏。本研究虽然尽可能全面地收集并整理了拟南芥中已发表和公布的多种表观遗传修饰数据,但是依然无法尽数收集到针对每个特定组织的表观遗传修饰的数据,因此无法构建组织特异性的预测模型。本研究所用的拟南芥复制时间数据是多个组织的平均值[7],但是使用的表观遗传修饰数据却具有组织特异性,这会导致模型的性能下降。但本研究通过分析所构建的机器学习模型对应的表观遗传修饰特征的重要性,获得了一些在DNA复制时间表观遗传调控过程中可能扮演重要作用的组蛋白修饰以及组蛋白变体,但更为具体的DNA复制时间表观遗传修饰机制还需要进一步研究。
4 结 论
本研究构建了一种高精度的基于DNA表观遗传修饰预测基因组DNA复制时间的机器学习模型,结果表明,基因组DNA复制时间可以通过表观遗传修饰进行准确预测,且对DNA复制晚期的预测最为准确。拟南芥基因组DNA复制时间可能受到精细的表观遗传调控,其中H3.1、H3.3、H2AW、H4K16ac、H3K36me3、H3K4me3等可以作为后续验证的重要组蛋白变体及表观遗传修饰。
