SXT/R391元件核心基因的生物信息学分析
2019-12-25张琪周静周江林孔娜胡明达彭小川李北平梁龙靳远任洪广岳俊杰
张琪,周静,周江林,孔娜,胡明达,彭小川,李北平,梁龙,靳远,任洪广,岳俊杰
军事科学院军事医学研究院生物工程研究所,北京100071
原核生物的可移动遗传元件(mobile genetic element,MGEs)可通过接合、转化、转导等方式介导遗传物质在不同种属细菌间的横向(水平)基因转移(lateral/horizontal gene transfer,LGT/HGT)。Burrus 于2002 年首次对那些可以从染色体上剪切并整合进宿主染色体,通过接合的方式在细菌细胞之间进行转移的遗传元件进行了归类,称之为整合性接合元件(integrating conjugative elements,ICEs)[1]。ICEs 兼有质粒和噬菌体的特征,与接合性质粒相似,它可通过接合的方式进行转移;与质粒不同的是,它们不能自主复制,其复制方式与温和噬菌体相似,通过整合进宿主染色体,随宿主染色体一起复制[2]。ICEs 具有吸收和传播包括毒力因子及耐药基因在内的多种特性基因进入细菌宿主细胞的功能。它们常赋予宿主细菌新的性状,如耐药性和致病性等[3]。
迄今,已在多种细菌中发现了ICEs,但目前还没有公认的ICEs 分类方法。欧竑宇提出了根据整合酶序列相似性以及核心结构同线性的特征对ICEs 进行分类的方法[4],按照这种方法可以把发现的大部分ICEs 归为28 个家族。SXT/R391是目前已知的ICEs 中研究最早和最详细的一个家族[5]。
SXT 元件最早发现于1992 年在印度和孟加拉导致霍乱的O139 群霍乱弧菌MO10 的染色体上,该元件能够以整合接合的方式进入宿主菌染色体。SXT 携带多种耐药基因,能够介导霍乱弧菌对磺胺甲恶唑(sulfamethoxazole,Su)、甲氧苄氨嘧啶(trimethoprim,Tm)、链霉素(streptomycin,Sm)和氯霉素(chloramphenicol,Cm)的耐药性。当时根据其携带的基因编码对磺胺甲恶唑和甲氧苄胺嘧啶的抗性而命名为SXT[6]。R391 在上世纪70年代早期被鉴定为赋予抗卡那霉素的质粒,最初被认为是不相容性群质粒“IncJ”的原生代表。20多年后,很多研究表明R391 和其他被称作“R 因子”的遗传元件具有与SXT 高度类似的基因组骨架结构以及固定的染色体插入/重组位点,它们与SXT 实际上属于同一类型的整合接合元件,因此将这一类元件划分为同一个SXT/R391 家族。
目前发现的SXT/R391 元件基因组的大小为70~120 kb。SXT/R391 元件的基因组包括核心保守区和可变区两部分[5,7-8]。SXT/R391 元件的保守区编码的基因介导SXT 元件的整合/切除、接合转移和ICEs 生命周期所需的调控,另外一些保守基因的功能未知。SXT/R391 可变区基因的编码产物主要负责宿主对抗生素的耐药性、重金属离子抗性、生物膜形成和细菌运动能力的调节,另外也编码毒素-抗毒素系统、限制性修饰系统、解旋酶、核酸内切酶等[9]。
作为多药耐药(multi-drug resistance,MDR)基因的载体,SXT 元件在菌株间播散可造成新的优势菌型流行。SXT/R391 元件的进化是造成其扩散能力增强的驱动力。阐明SXT/R391 家族元件的进化特征有助于防止SXT 元件扩散,将对致病菌防控研究起到重要的实践指导意义。
1 材料与方法
1.1 数据搜集
首先以“SXT/R391”及“ICE”为关键词在PubMed 中搜索获得含有报道SXT/R391 元件的文献,通过阅读文献获取SXT/R391 元件的序列信息。接着进行SXT/R391 元件的预测,为了保证获得完整的SXT/R391 元件序列,我们从基因组序列完整度为完整基因组(Complete Genome)和染色体(Chromosome)状态的9632 个细菌基因组序列中搜索SXT/R391 元件。通过BLASTp 软件进行比对,匹配结果中包含整合与剪切模块(integration/excision module)、DNA 分泌模块(DNA secretion module)和调控模块(regulation module)等3 个必需模块的蛋白区域推断为潜在的ICE,如果在一段连续区域内(70~130 kb)出现编码SXT/R391 家族元件的整合酶Int、Ⅳ型分泌系统接合蛋白和调控蛋白SetR 的基因,则预测该基因组存在一个完整的SXT/R391 元件。最终,我们通过文献和预测获得83 个SXT/R391 家族元件。
1.2 研究方法
提取SXT/R391 元件的52 个核心基因序列,对83 个元件做Blast 分析,分析每个基因在83 个元件上的分布和复制情况。
1.2.1 重组分析 应用分子生物学软件包RDP4[10]中的RDP、GENECONV、BootScan、MaxChi、Chimaera 等5 种较为常见的方法,分别探测83 个SXT/R391 元件的52 个核心基因序列的重组事件和重组信号。其中,GENECONV 的参数g-scale 设置为1,允许重组片段内部的错配;其他方法的参数设置为默认值,不作修改。P≤0.01 为具有统计学显著性差异。
1.2.2 正选择分析 选择在线工具Datamonkey(http://datamonkey.org/)[11]进行SXT/R391 元件核心基因的正选择压力分析。首先使用MUSCLE 进行序列比对,提交序列及选择运算模型。我们选择了4 种模型,即随机效应似然法(REL)、固定效应似然法(FEL)、单一祖先计数法(SLAC)及混合效应进化模型方法(MEME)。依据ω 值[ω=dN/dS(非同义置换率/同义置换率)]判断选择的方向。当ω>1 且差异显著时判断为正向选择,0<ω<1 时为净化选择,ω=1 时为中性选择。
2 结果
2.1 SXT/R391 元件的序列整理及菌属分布
通过文献收集和预测共获得83 个SXT/R391元件的基因组序列,包括从文献中提取的54 个和细菌基因组序列中预测的29 个,元件所在宿主基因组的分离时间为1967~2014 年,地理分布包括21 个国家和地区,元件宿主跨越25 个菌属。在预测的29 个SXT/R391ICE 元件中,有12 个曾经被报道,但未提取和报道元件的完整序列。SXT/R391元件的宿主广泛(图1),但分布存在较大偏性,在霍乱弧菌中发现的数量最多,奇异变形杆菌紧随其后。有趣的是,临床发现的SXT/R391 元件也都集中在霍乱弧菌和奇异变形杆菌。在环境中发现的SXT/R391 元件大多来自水环境。SXT/R391的菌种分布不均匀,说明元件能在宿主中存在和保藏可能需要满足一定的遗传背景。
2.2 核心基因分析
Wozniak 等对13 个SXT/R391 元件的基因组进行了比较分析[7],发现该元件具有52 个核心基因。我们提取了SXT/R391 元件的52 个核心基因序列,对获得全基因组信息的83 个SXT/R391 元件的全基因组序列进行同源搜索,分析元件的核心基因的分布情况,结果见图2。
83 个SXT/R391 元件中有一个特殊的成员ICEPmiChn3,该元件缺少20 个核心基因,其中13个发挥接合功能。缺少的20 个核心基因被一个新的由转座子和耐药基因组成的基因簇取代。实验结果表明,该元件的接合功能完全丧失[12],因而ICEPmiChn3 已经不是一个具有完整组件和功能的元件。因此,后续的核心基因数量分析排除了该元件。
经过计算,52 个核心基因中有39 个依然出现在全部元件中。另外有6 个基因仅在1 个元件中缺少,这些基因可称为软核心基因(soft core)[13]。另有7 个基因(rumA、s024、s025、s026、traL/E/K)在2 个或2 个以上元件中缺失,其中的rumA、s024、s025、s026都是SXT/R391 元件扩散所需的最小功能基因集合,缺失这些基因可能会影响元件的传播扩散。以上结果说明从文献报道的13 个元件中推导出的52 个核心基因并不都是严格保守的核心基因,其中有一些基因属于软核心基因。
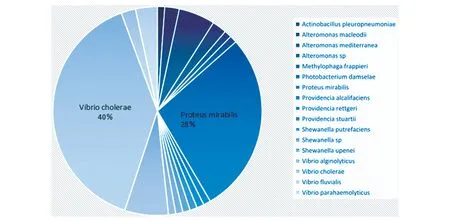
图1 SXT/R391 的菌属分布
值得注意的是,83 个元件的整合酶基因有2类,分别为int-1、int-2,2 类间的序列相似性仅为22%。裂解酶xis基因也有2 类,分别为xis-1、xis-2。我们发现在元件中,int-1与xis-1协同出现,int-2和xis-2协同出现。
另外,从图2 可以看出,traFHG、eex、setC、setD、s082、s083、s084、s086、setR基因在菌株Alteromonas macleodiistr.MED6-ICE 的SXT/R391 元件中出现了整体的基因复制。
2.3 核心基因的融合和裂解
除了基因复制,还有少数核心基因呈现了基因融合/裂解信号。全长rumB基因读框长度为1269 bp,蛋白产物为423 aa,但该基因在46 个元件表现为2 个及更多的片段(图3),说明rumB基因发生了基因融合/裂解,可能与位点附近发生的频繁的插入有关。
此外,trhF与traW在一些元件中为2 个基因。如在元件Proteus mirabilisMD20140901(KX243408)中,trhF读框[54557..55069],长度513 bp,蛋白产物171 aa;traW读框[55080..56204],长度1125 bp,蛋白产物375 aa。再如,在元件Vibrio choleraeO37MZ03(JQ345361)中,trhF读框[26846..27358],长度513 bp,蛋白产物171 aa;traW读框[27369..28493],长度1125 bp,蛋白产物375 aa。但在元件Providencia stuartiiATCC33672(CP008920)中,对应的2 个基因融合为1 个基因,融合基因读框[1904626..1906272](基因组位置),长度1647 bp,融合蛋白产物(549 aa)为type-F conjugative transfer system pilin assembly family protein(AIN64681.1)。
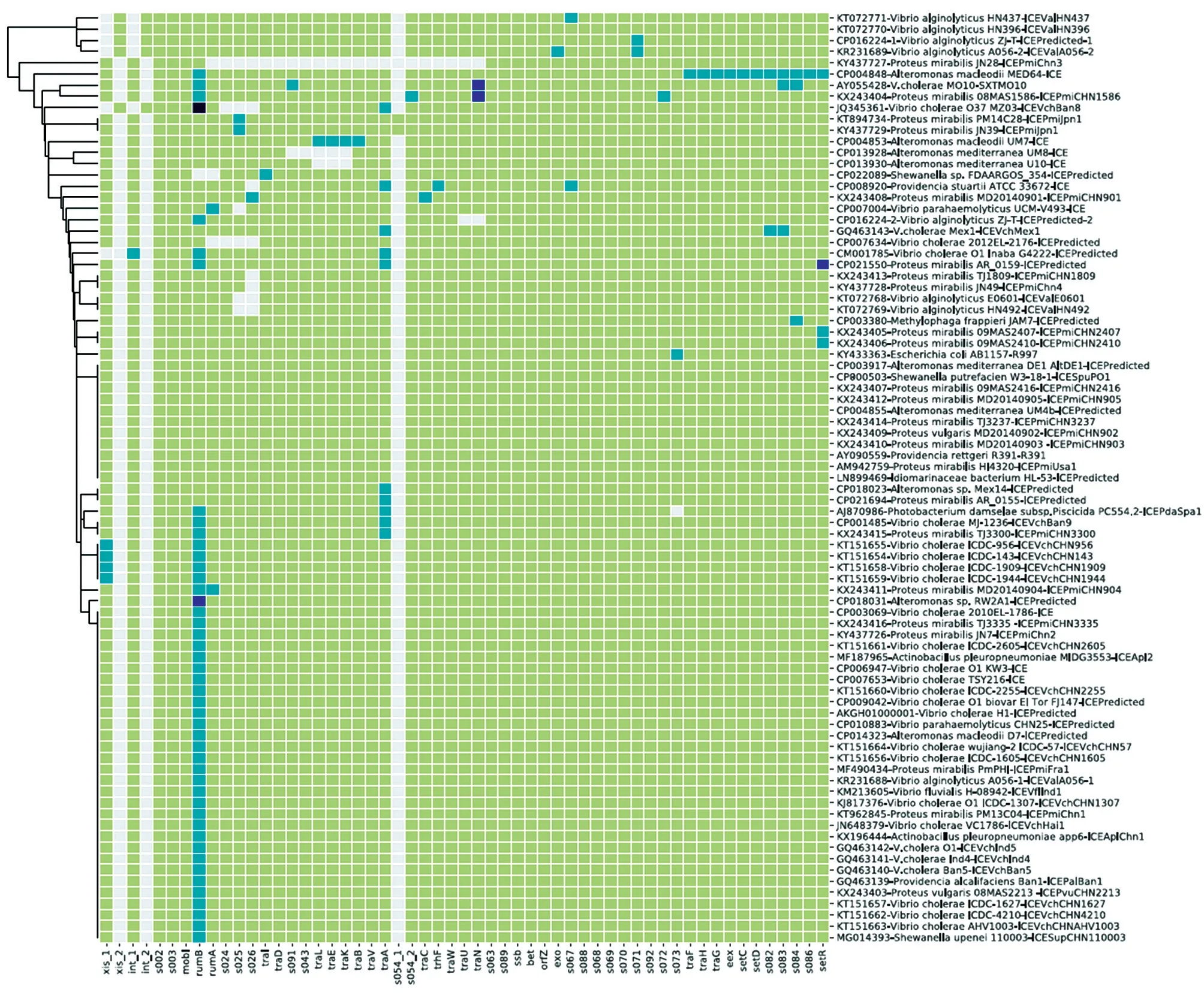
图2 核心基因分布情况
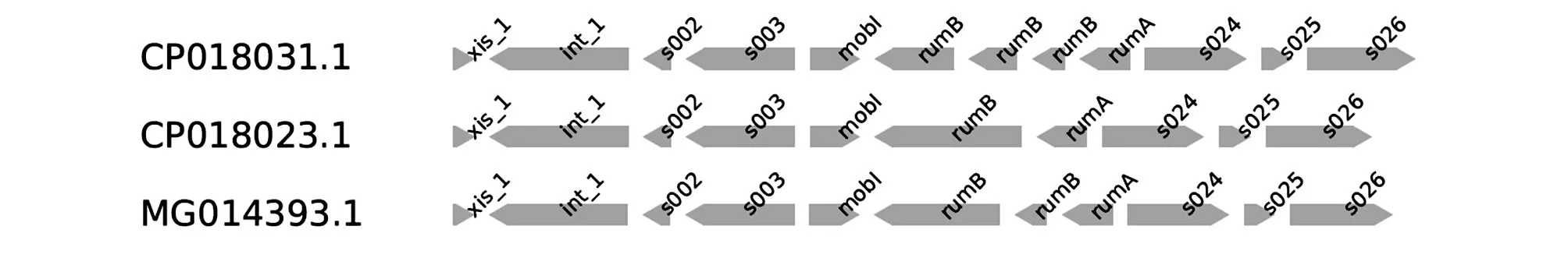
图3 rumB 基因在不同元件中的分布
2.4 核心基因的重组分析
元件之间的重组能导致形成新的杂合元件,重组事件是SXT/R391 元件多样性产生的动力之一。有研究报道,SXT/R391 元件核心区存在基因间重组。重组除了发生在基因及基因以上水平,还可能发生在亚基因水平(sub-gene level)。为了探测SXT/R391 元件的亚基因水平重组事件,我们检测了SXT/R391 元件52 个核心基因的基因内重组。在52 个核心基因中,有22 个核心基因在3种及3 种以上的方法中可以检测到明显的重组信号(表1)。
图4 用红色标出了重组基因在SXT/R391 元件基因组上的位置,这些基因涉及元件核心基因的全部4 个必需功能模块,即整合与剪切模块、DNA 复制及加工模块(replication/DNA processing module)、DNA 分泌模块和调控模块。分泌模块,即Ⅳ型分泌系统(type Ⅳsecretion system,T4SS)有关基因簇的大部分基因都检测到了重组信号。此外,还有一些具有重组信号的核心基因为未知功能基因。
2.5 正选择分析
正选择(positive selection)也是微生物进化过程中独立的遗传变异驱动力,在生物的适应性进化中发挥关键作用。我们检测了SXT/R391 元件基因组中基因的正选择信号。为了避免重组事件对正选择分析造成干扰,我们只在未发生重组事件的核心基因上检测正选择。计算结果显示,除去以上分析的22 个发生重组的核心基因,SXT/R391 元件中没有重组信号的30 个核心基因中有7 个检测到正选择信号(表2)。这7 个基因中的mobI和traJ编码SXT/R391 元件接合转移所需蛋白,其他5 个基因编码未知功能蛋白,且不在SXT/R391 ICE 最小功能基因集合(minimal functional SXT/R391 ICE gene set)中,它们对SXT/R391 元件多样性的贡献有待进一步研究。
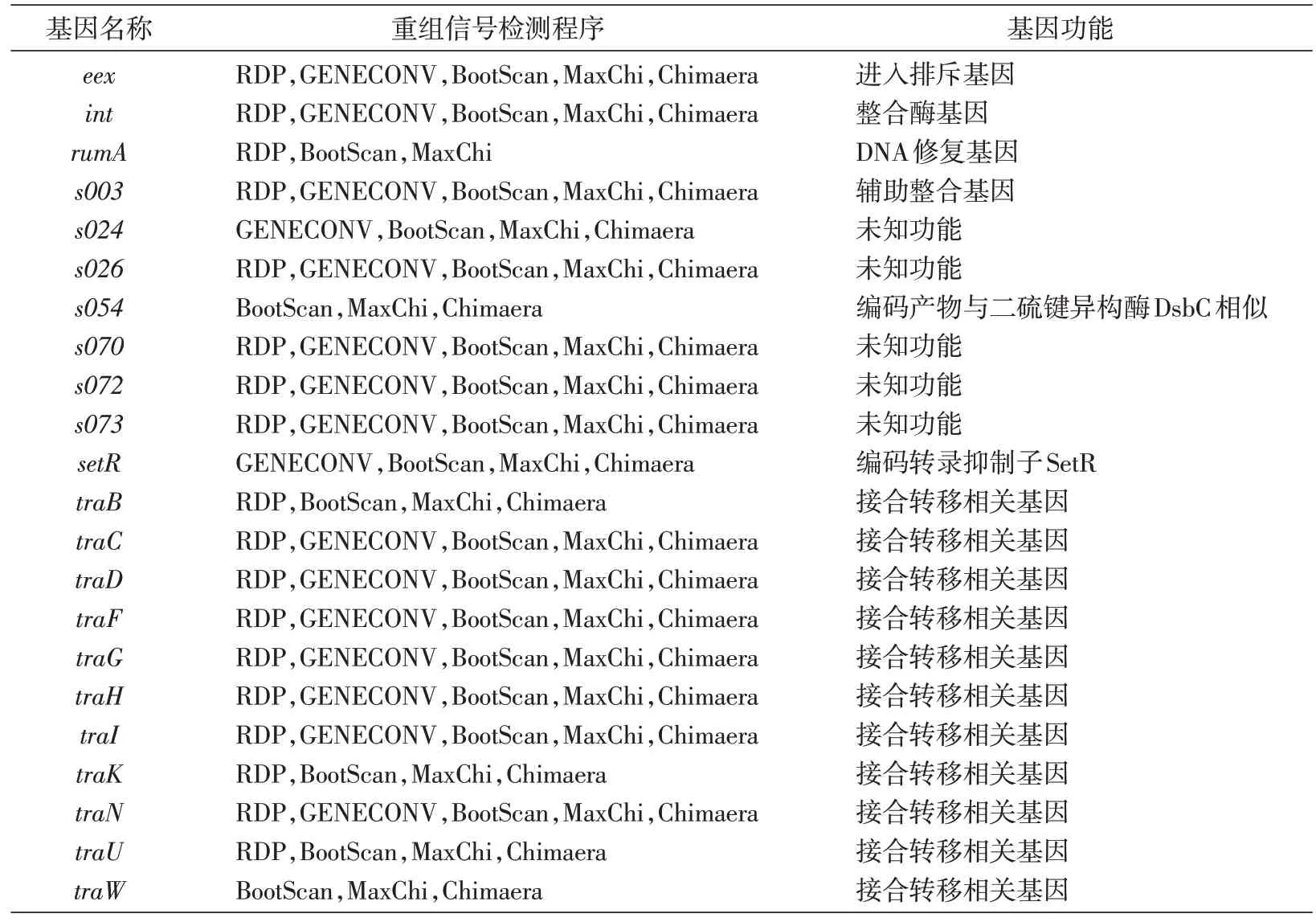
表1 核心基因重组信号检测

图4 22 个发生重组的核心基因在SXT/R391 元件骨架上的位置

表2 核心基因的正选择氨基酸数量与位点
利用I-TASSER 软件对mobI和TraJ基因编码的蛋白进行三维结构预测[14]。从预测的三维结构图(图5)中可以看出,mobI基因编码的蛋白上正选择位点52、104 残基处于α螺旋结构的端头,130残基位于另一α螺旋结构上;TraJ 基因编码的蛋白发生正选择的位点62 残基正处于α螺旋结构的端头位置,195 残基位于α螺旋结构上,根据预测该蛋白的191~210 残基为一个跨膜结构域,正选择位点195 残基处于该结构域内。以上位点所处的蛋白结构域或与转移运输有关,这些位点受到选择压力可能与元件的转移扩散相关。
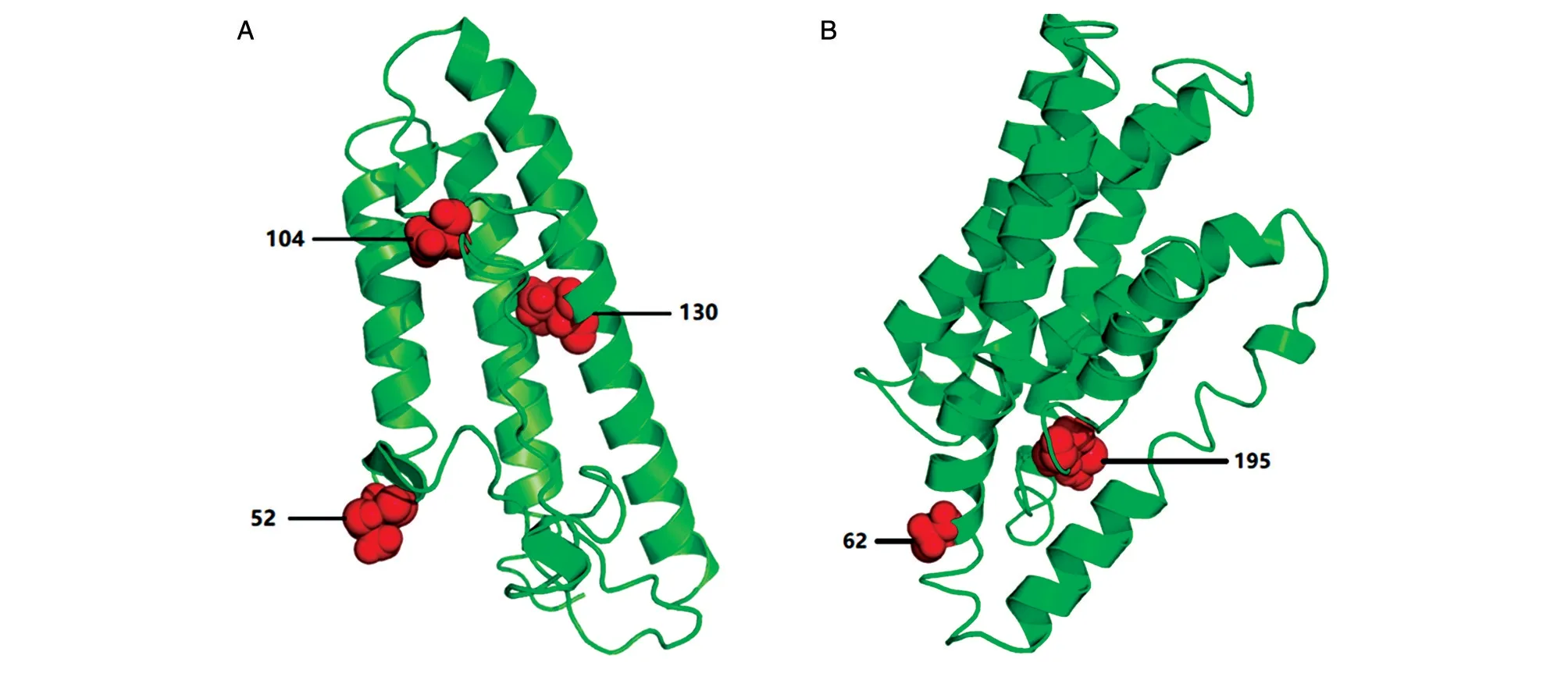
图5 mobI 基因(A)和traJ 基因(B)编码蛋白的三维结构及正选择位点
3 讨论
作为MDR基因的载体,SXT 元件在菌株间播散可造成新的优势菌型流行。SXT/R391 元件的进化是造成其扩散能力增强的驱动力。阐明SXT/R391 家族ICEs 的进化特征、迁移模式及其散播的分子机制,有助于防止SXT 元件扩散,对致病菌防控研究有重要的实践指导意义。SXT/R391 元件的核心基因决定了元件的复制和转移。重组和正选择是微生物进化过程中2 种独立的遗传变异驱动力,在生物的适应性进化中发挥关键作用。本研究收集了目前发现的SXT/R391元件,对核心基因的分布以及核心基因的重组和正选择两大进化特征进行了分析。
本研究表明,同源重组和正选择在SXT/R391元件核心基因的进化过程中发挥了重要作用。我们发现了22 个核心基因在进化中经历了同源重组,导致了元件相当一部分核心基因的遗传变异。另外,有7 个核心基因存在正选择信号,其中2 个基因mobI和traJ与元件的接合转移有关。元件发生重组和正选择变异都是为了SXT/R391 元件适合度的提升,从而能适应生态环境的变化。综上,我们分析了SXT/R391 元件核心基因的特征及适应性进化,为了解SXT/R391 元件的扩散及进化规律奠定了基础。
