斑节对虾α-淀粉酶基因的克隆及其表达分析
2017-11-29杨其彬李运东江世贵黄建华邱丽华朱彩艳周发林
杨其彬 李运东 江世贵 黄建华 姜 松 邱丽华朱彩艳 周发林
(1. 中国水产科学研究院南海水产研究所, 广州 510300; 2. 农业部南海渔业资源开发利用重点实验室, 广州 510300)
斑节对虾α-淀粉酶基因的克隆及其表达分析
杨其彬1,2李运东1,2江世贵1,2黄建华1,2姜 松1,2邱丽华1,2朱彩艳1,2周发林1,2
(1. 中国水产科学研究院南海水产研究所, 广州 510300; 2. 农业部南海渔业资源开发利用重点实验室, 广州 510300)
为了研究斑节对虾α-淀粉酶基因的结构和生物学功能, 根据原实验室构建的斑节对虾(Penaeus monodon) cDNA文库得到的EST序列, 利用RACE技术获得了斑节对虾α-淀粉酶基因(PmAmy)的cDNA全长序列。该基因序列全长2465 bp, 包括2175 bp的开放阅读框, 编码724个氨基酸, 分子总量为78.9 kD, 理论等电点为4.66。PmAmy包含一个α-淀粉酶家族保守的A结构域(Thr34-Ser410)和一个C结构域(Glu420-Ala496)。PmAmy氨基酸序列与其他物种的相似性为47%—99%, 利用PmAmy构建的进化树显示斑节对虾和凡纳滨对虾(Litopenaeus vannamei)的亲缘关系最近。基因表达结果显示PmAmy在肝胰腺组织中的表达量显著高于其他组织(P<0.05)。斑节对虾PmAmy基因在卵巢发育的过程中均有表达, 表达量有所变化, 虽然没有发现显著性的差异(P=0.09)。斑节对虾PmAmy在整个生长阶段的检测中都有表达, 其中幼体发育过程中存在显著性差异,糠虾时期PmAmy表达量显著高于无节幼体、溞状幼体和仔虾时期(P<0.05)。以上实验结果初步说明了PmAmy可能与斑节对虾的幼体发育相关。
斑节对虾; α-淀粉酶; 基因克隆; 基因表达; 幼体发育
α-淀粉酶(α-Amylase)是a-1,4-D-葡聚糖-葡聚糖水解酶(α-1,4-glucan-glucanhydrolase)的简称, 属于糖基水解酶13家族(Glycosyl hydrolase family 13),即a-淀粉酶家族[1]。甲壳动物的生长、发育、繁殖等过程所需的营养素基本来源于消化酶对饵料或储存营养物质的消化及机体的重新合成。甲壳动物肝胰腺中的多种酶类的作用也决定了对营养物质的消化能力, 大量研究表明生物的高生长率、低死亡率和体重等都与消化能力有着紧密的联系[2]。α-淀粉酶是甲壳动物消化腺中一种重要的碳水化合物水解酶类, 它以随机作用的方式切断淀粉、糖原、寡聚或多聚糖分子内的任意α-1,4葡萄糖苷键,生成麦芽糖、葡萄糖和低聚糖等化合物供予机体,与其他类型如β-淀粉酶家族、葡萄糖糖化酶、异淀粉酶等水解淀粉的酶类相比, α-淀粉酶类具有更快的作用[3], 其分泌机能的强弱直接影响生物对食物的消化能力, 从而影响生长繁殖等其他生理过程[4]。
目前α-淀粉酶基因已从多种生物中克隆得到并进行研究, 如黑腹果蝇(Drosophila melanogaster)[5]、斑马鱼(Danio rerio)、草鱼(Ctenopharyngodon idellus)[6]、长牡蛎(Crassostrea gigas)[7]、合浦珠母贝(Pinctada fucata)[8]等。对α-淀粉酶功能的研究也有很多报道, 如Vanwormhoudt等[9]测定了多种从甲壳动物肝脏提取物的α-淀粉酶活性, 发现α-淀粉酶活性在一些虾蟹类(Penaeid, Brachyura, Anomura)中很高, 而在滨蟹属(Carcinus)、龙虾(Palinurus)等品种中非常低。在鱼类上有研究发现, 淀粉酶活性水平与肠道填充度和动物的营养条件相关, 鱼类淀粉酶水平饱腹时较高, 幼鱼比成鱼高[10—12]。Huvet等[13]对太平洋牡蛎(Crassostrea gigas)α-淀粉酶基因多态性与生长性状、消化特性的相关性进行了研究,α-淀粉酶基因对牡蛎的摄食及吸收效率有显著影响, 进而影响牡蛎的生长。此外, Prudence等[14]的研究发现, α-淀粉酶基因的多态性与牡蛎的生长显著相关, 并认为淀粉酶基因的分子标记在育种中具有潜在应用价值。对虾上的现有研究表明α-淀粉酶是对虾的重要消化酶, α-淀粉酶基因对生长发育过程具有重要影响, 被认为是与对虾生长性状相关的重要候选基因之一[15,16]。
斑节对虾(Penaeus monodon)是对虾类中体形最大的一种对虾, 具有生长快、适应性强、营养价值高的优点, 是当前世界上三大虾类养殖品种之一,也是中国和东南亚地区重要的传统优质对虾资源。因此本研究对斑节对虾α-淀粉酶基因cDNA序列克隆分析, 初步探究该基因对斑节对虾生长发育的影响和潜在生物学功能, 为下一步α-淀粉酶基因单核苷酸多态性(SNP)位点筛选及其与生长性状的关联性分析、分子标记辅助育种奠定基础。
1 材料与方法
1.1 实验材料
实验动物: 斑节对虾来源于南海水产研究所(深圳基地)斑节对虾遗传育种中心, 在水温(25±2)℃、盐度为30‰的循环水池中暂养3d后取样, 用于cDNA克隆及组织表达等实验。选取雌性斑节对虾3尾(体质量20—50 g)取组织样品, 包括脑、眼柄、鳃、心、肝胰腺、肠、胃、神经、肌肉、卵巢等组织用于组织表达实验。根据形态学特征[17,18]初步判断卵巢发育期, 取斑节对虾(体质量20—200 g)发育为Ⅰ期至Ⅴ期的卵巢组织用于荧光定量实验。参照江世贵等[19]对斑节对虾生命史的研究, 分别采集斑节对虾的无节幼体、溞状幼体、糠虾和仔虾时期的幼体, 和后期仔虾、幼虾、亚成虾和成虾生长阶段的肝胰腺组织, 用于各生长阶段的基因定量表达实验。以上样品组织都采用RNAlater®Solution保存, 4℃过夜后–80℃保存。
实验试剂: RNAlater Tissue Collection购自普博生物公司; RNA提取试剂盒(RNeasy Plus Mini Kit)购自QIAGEN公司; Trizol Reagent购自Invitrogen公司; 逆转录试剂盒PrimeScript II 1st Strand cDNA Synthesis Kit购自Promega公司; pMT18-T Vector、T4 DNA Ligase和DL5000 DNA Marker, 荧光定量试剂盒TaKaRa SYBR、PrimeScriptTM RTPCR Kit (Perfect Real Time)购自TaKaRa公司; 感受态细胞大肠杆菌菌株由本实验制备。
1.2 试验方法
总RNA提取及cDNA的合成用于cDNA克隆的模板来源于卵巢、精巢、肝胰腺的混合样品(总质量30 mg), 提取斑节对虾上述样品组织的总RNA。提取的RNA经琼脂糖凝胶电泳确定其完整度, 经分光光度计测定纯度。以总RNA (2 μg)为模板采用逆转录试剂盒, 经过42℃ 15min延伸和70℃15min逆转录反应后得到cDNA的第一条链。
斑节对虾α-淀粉酶基因cDNA的克隆对已经构建的cDNA文库中的EST序列进行BLAST分析, 结果显示有一条EST序列与凡纳滨对虾α-淀粉酶基因高度同源。利用Premier5.0软件设计引物,引物由华大基因生物公司合成。利用cDNA末端快速扩增技术(Rapid amplification of cDNA ends,RACE)、降落PCR技术和半巢式PCR (Semi-nest PCR)技术扩增目的基因的3′末端。在3′RACE中,降落PCR使用正向引物AMY-F1和接头通用引物UPM (引物见表1), 所得PCR产物稀释50倍后取2 μL用于巢式PCR, 反应程序及条件参照实验室研究文献设置[20]。反应结束后的产物经1.2%琼脂糖凝胶电泳检测, 胶回收目的片段后与pMT18-T载体进行连接, 然后在大肠杆菌感受态细胞中转化, 将转化后的大肠杆菌均匀涂到培养基上, 培养箱中培养8—10h后挑取平板中的阳性克隆, 经过菌液PCR鉴定后送往华大公司测序。

表1 实验中所用引物序列Tab. 1 Primers sequences used in this experiment
1.3 序列分析
测序结果采用Dnaman软件分析拼接, 运用EdiSeq程序预测开放阅读框及氨基酸序列, 通过GOR4方法(http://www.expasy.org/)进行二级结构分析, 用SMART (Simple Modular Architecture Research Tool)(http://smart.emblheidelberg.de/)软件分析蛋白的结构域, 通过NCBI中BLAST对斑节对虾及其他物种的淀粉酶氨基酸序列进行多重比对,3D结构预测和查看采用phyre2软件(http://www.sbg.bio.ic.ac.uk/phyre2)软件、Swiss-model (http://swissmodel.expasy.org)程序和FirstGlance in Jmol(http://bioinformatics.org/firstglance/)程序, 用Clustalx2.0.11和Bioedit软件进行多序列比对, 采用邻位相接法构建系统进化树, 软件使用MEGA5.0。
1.4 斑节对虾α-淀粉酶基因的表达分析
利用RNA提取试剂盒提取斑节对虾各组织(包括心脏、肝胰腺、淋巴、性腺、肌肉、脑、鳃、肠、胃、眼柄和胸腺神经)的总RNA, 并反转录合成cDNA, 以备荧光定量RT-PCR模板使用。运用同样的方法获得卵巢发育Ⅰ—Ⅴ期的荧光定量RTPCR模板和8个生长时期的荧光定量模板。每个时期设置3个平行, 实验中每个时期样本使用的质量约为30 mg。
根据斑节对虾α-淀粉酶基因的cDNA序列设计特异性引物AMY (RT)-F和AMY (RT)-R, 参照已有研究报道[20]选用β-actin为内参基因, 引物序列为βactin-F和βactin-R (表1)。依照TaKaRa SYBR Premix ExTaqTMKit (Perfect Real Time)试剂盒说明书进行PCR反应。以蒸馏水代替模板作为阴性对照,样品和内参均设3个重复。实验数据采用相对ΔCt法(2–ΔΔCt法)分析PmCatL基因在斑节对虾各组织中的相对表达量。
1.5 统计学分析
所得数据采用SPSS18.0软件的单因素方差分析(One-Way ANOVA)进行处理, 结果值以平均值±标准差(Mean±SD)表示。采用Duncan检验显著性差异(P<0.05)。
2 结果
2.1 斑节对虾α-淀粉酶基因序列分析
利用特异性引物进行扩增, 将扩增产物测序后与EST序列拼接, 获得斑节对虾α-淀粉酶基因的cDNA序列, 命名为PmAmy, 其在NCBI的GenBank登录号为KU308415。PmAmy基因全长2465 bp, 包括73 bp的5′端非编码区(Untranslated region, UTR),217 bp的3′端非编码区和2175 bp的开放阅读框(Open reading frame, ORF), 含有polyA加尾信号AATAAA。经过对其编码氨基酸序列预测分析,PmAmy编码的氨基酸序列含有724个氨基酸, 其中含量最多的是甘氨酸(Gly)和缬氨酸(Val)分别占总氨基酸含量的11.3%和8.6%。分子总量为78.9 kD,理论等电点为4.66。结构域分析表明,PmAmy的N端含有21个氨基酸组成的信号肽;PmAmy还包含有一个α-淀粉酶家族保守的A结构域(Thr34-Ser410)和一个C结构域(Glu420-Ala496)。
2.2 斑节对虾与其他动物α-Amylase同源性分析
PmAmy与凡纳滨对虾的一致性最高为99%。与长牡蛎、合浦珠母贝、人、牛的一致性分别为55%、48%、47%和48%。通过与其他动物α-Amylase的比对, 该基因编码氨基酸中的半胱氨酸位点、活性催化位点、钙结合位点和氯离子结合位点在物种间都非常保守。
通过邻位相连法构建的进化树如图1所示, 斑节对虾与凡纳滨对虾的亲缘关系最近, 其次是罗氏沼虾(Macrobrachium rosenbergii), 三者聚为一小支, 与哺乳动物的亲缘关系较远。这样的进化关系与一致性分析结果一致, 也符合传统形态学上各物种的分类地位。
2.3 斑节对虾PmAmy基因表达特征分析
组织表达结果显示,PmAmymRNA在所检测组织中, 表达量存在显著差异。肝胰腺组织中的表达量显著高于其他各个组织(P<0.05), 在淋巴、鳃、肠中也有一定的表达, 表达量最低的是眼柄神经和心脏组织(图2)。
斑节对虾PmAmy基因在卵巢发育的过程中均有表达, 表达量有所变化, 但是统计学分析显示并没有发现显著性的差异(P=0.09)(图3)。斑节对虾PmAmy在整个生长阶段的检测中都有表达。在幼体发育过程中存在显著性差异, 糠虾时期PmAmy表达量达到峰值, 显著高于无节幼体、糠虾和仔虾三个生长阶段(P<0.05)。随后表达量下降, 此后一直到成虾阶段, 这期间各生长时期的表达量也有变化,但在后期仔虾、幼虾、亚成虾和成虾生长时期并没有发现显著性差异(图4)。
3 讨论

图1 利用Clustal W程序和MEGA 5.0软件构建的PmAmy系统进化树Fig. 1 Phylogenetic tree of the AMY amino acid sequence in different groups by program Clustal W and MEGA 5.0
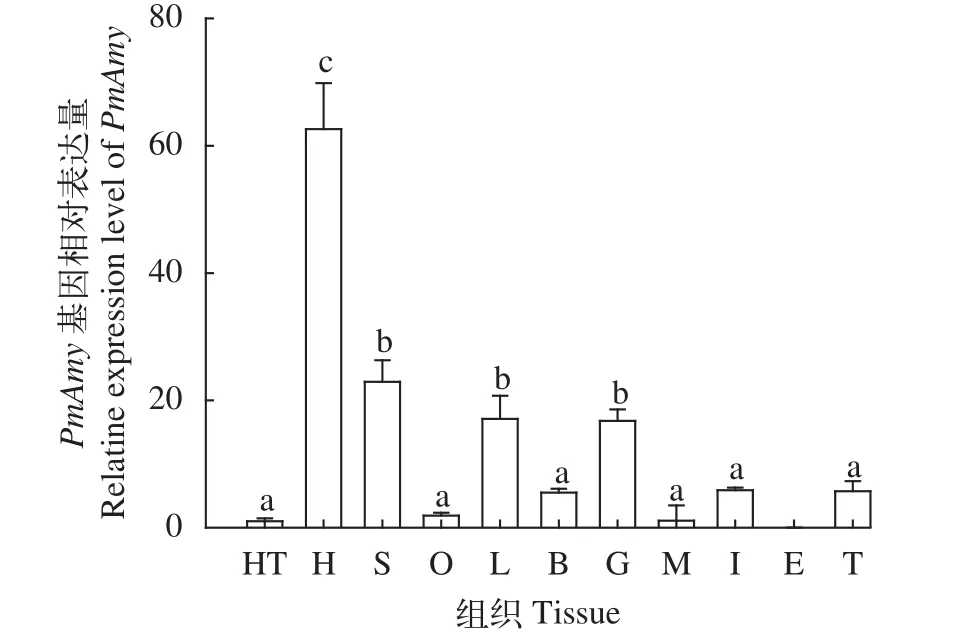
图2 节对虾PmAmy基因在不同组织中的表达分布Fig. 2 Distribution of PmAmy gene expression in different tissues of P. monodon
α-淀粉酶在许多水生动物中控制着碳水化合物的代谢, 并且对生长有着重要的影响。以往较多的研究主要采用测定酶活力的分析方法, 但由于消化酶的活性会受到多种因子的影响, 从酶学水平测定消化酶分布或表达差异存在着方法上的局限性,然而从分子水平检测α-淀粉酶基因的转录水平变化有着很强的特异性, 可以精确地了解α-淀粉酶的合成和分泌情况[8,22]。本研究结果表明斑节对虾α-淀粉酶基因在被检测的多种组织中广泛分布, 且在肝胰腺中的表达量显著高于其他部位。这一结果与鳜(Siniperca chuatsi)、青鱼(Mylopharyngodonpiceus)的组织表达[23]有所不同, 但与合浦珠母贝[8]、草鱼[22]具有相似性, 尤其与甲壳动物中的克氏原螯虾[24](Procambarus clarkii)的研究结果较为一致。本研究中PmAmy基因在肝胰腺中表达量较高的结果可能与肝胰腺是斑节对虾淀粉酶中心合成场所有关[8,20], 而在其他多个组织中的表达同时也说明了肝胰腺并不是PmAmy基因在斑节对虾体内发挥作用的唯一场所。
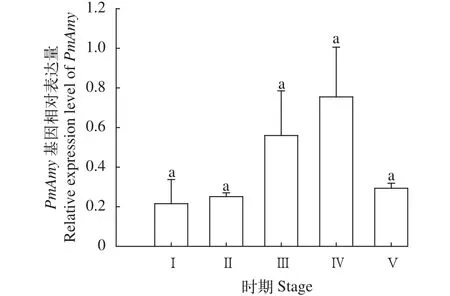
图3 斑节对虾PmAmy基因在卵巢发育各时期的表达特征Fig. 3 Relative expression level of PmAmy mRNA in different development stages of ovary

图4 斑节对虾PmAmy基因在不同生长时期的表达Fig. 4 Expression level of PmAmy in different growth stage of P.monodon
在个体早期发育阶段的功能作用, 一直是α-淀粉酶研究的重要方向之一, 国内外有很多相关报道。阮国良[3]在草鱼和鳡孵化后第二天即检测到α-淀粉酶基因的表达, 并在第7天表现出最大的α-淀粉酶mRNA表达量。其他多种鱼类如日本鳗鲡、太平洋多指马鲅(Polydactylus sexfilis)、湖鲟(Acipenser fulvescens)、河鲈(Perca fluviatilis)、赤鲷(Pagrus pagrus)等[25—28], 也都发现在仔鱼的早期发育阶段有较高的淀粉酶活性, 之后逐渐降低。这些研究表明, 很多鱼类在发育早期缺乏完善的消化腺和稳定消化系统, 这时包括α-淀粉酶在内的多种消化酶在饵料和营养物质的消化过程中发挥着至关重要的作用[23]。本研究在斑节对虾的整个生长阶段均检测到PmAmy基因的表达, 在糠虾时期PmAmy基因表达量达到峰值, 这可能与斑节对虾幼体逐渐摄食大量的轮虫和藻类作为饵料有一定的关系, 这一结果和其他甲壳动物的研究类似但不完全一致。中国明对虾和日本囊对虾在幼体发育过程中, 淀粉酶活性在溞状幼体Ⅲ期时出现一个峰值,以后逐渐减弱[29]。但是也有研究表明淀粉酶基因表达变化与个体自身发育有关, 如在中华绒螯蟹的研究中, 发现整个胚胎发育过程中, 淀粉酶的变化趋势不明显, 但溞状幼体期, 酶的活性显著升高, 这可能与幼体孵化后开口摄食的自身调控机理有关系[30], 影响α-淀粉酶表达量变化的因素还有很多,如蜕皮、温度、盐度等环境因子等[31]。已有研究表明, α-淀粉酶基因在凡纳滨对虾[32,33]、克氏原螯虾[24]等多种甲壳动物的蜕皮诱导应答和生长发育中具有重要作用, 此外还与多种虾蟹类幼体发育过程中食性的变化密切相关, 根据本研究结果推测[22,31], α-淀粉酶基因在斑节对虾生长发育过程中的表达变化特征可能与斑节对虾不同发育阶段的食性变化有关, α-淀粉酶基因可能参与调控斑节对虾的幼体发育过程[33]。
[1]Kuriki T, Imanaka T. The concept of the alpha-amylase family: Structural similarity and common catalytic mechanism [J].Journal of Bioscience and Bioengineering,1999, 87(5): 557—565
[2]Jung H, Lyons R E, Hurwood D A,et al. Genes and growth performance in crustacean species: a review of relevant genomic studies in crustaceans and other taxa [J].Reviews in Aquaculture, 2013, 5(2): 77—110
[3]Ruan G L. Early development of trypsins and amylases of grass carpCtenopharyngodon idellus, yellowcheck carpElopichthys bambusaand topmouth culterCulter alburnus[D]. Thesis for Master of Science, Huazhong Agricultural University. 2010 [阮国良. 草鱼、鳡和翘嘴鲌胰蛋白酶和淀粉酶的早期发育. 硕士学位论文, 华中农业大学. 2010]
[4]Chen L, Liang X F, Wang L,et al. Molecular cloning and sequences analysis of trypsin, amylase and pepsinogen genes from Chinese perch (Siniperca chuatsi) [J].Chinese Journal of Biochemistry and Molecular Biology, 2009,25(12): 1115—1123 [陈亮, 梁旭方, 王琳, 等. 鳜鱼胰蛋白酶和淀粉酶与胃蛋白酶原基因的克隆与序列分析.中国生物化学与分子生物学报, 2009, 25(12):1115—1123]
[5]Boer P H, Hickey D A. The alpha-amylase gene inDrosophila melanogaster: nucleotide sequence, gene structure and expression motifs [J].Nucleic Acids Research,1986, 14(21): 8399—8411
[6]Zhu S L, Li X H, Yang J P,et al. Cloning and analysis of 5' flanking sequence of α-amylase gene fromCtenopharyngodon idellus[J].South China Fisheries Science,2014, 10(5): 87—93 [朱书礼, 李新辉, 杨计平, 等. 草鱼α-淀粉酶基因5′侧翼序列克隆与分析. 南方水产科学,2014, 10(5): 87—93]
[7]Sellos D, Moal J, Degremont L,et al. Structure of amylase genes in populations of pacific cupped oyster(Crassostrea gigas): Tissue expression and allelic polymorphism [J].Marine Biotechnology, 2003, 5(4):360—372
[8]Li L. Genomic structure, SNP screening, growth association and ecological response of α-amylase gene in the pearl oysterPinctada fucata[D]. Thesis for Master of Science, Shanghai Ocean University. 2013 [李璐. 合浦珠母贝α-淀粉酶基因结构、SNP筛选、生长关联与生态响应研究. 硕士学位论文, 上海海洋大学. 2013]
[9]Vanwormhoudt A, Bourreau G, Lemoullac G. Amylase polymorphism in crustacea decapoda-electrophoretic and immunological studies [J].Biochemical Systematics and Ecology, 1995, 23(2): 139—149
[10]Takii K, Shimeno S, Kedeka M. Changes in digestive enzyme activities in eel after feeding [J].Bulletin of the Japanese Society of Scientific Fisheries, 1985, 51(12):2027—2031
[11]Munilla-Morán R, Stark J R. Metabolism in marine flatfish VI. Effect of nutritional state on digestion in turbot,Scophthulmus maximus(L.) [J].Comparative Biochemistry and Physiology Part B, 1990, 95(3): 625—634
[12]Kawai S, Lkeda S. Studies on digestive enzymes of fishes: I. Carbohydrases in digestive organs of several fishes [J].Nsugaf, 1971, 37(4): 333—337
[13]Huvet A, Jeffroy F, Fabioux C,et al. Association among growth, food consumption-related traits and amylase gene polymorphism in the Pacific oysterCrassostrea gigas[J].Animal Genetics, 2008, 39(6): 662—665
[14]Prudence M, Moal J, Boudry P,et al. An amylase gene polymorphism is associated with growth differences in the Pacific cupped oysterCrassostrea gigas[J].Animal Genetics, 2006, 37(4): 348—351
[15]Glenn KL, Grapes L, Suwanasopee T,et al. SNP analysis of AMY2 and CTSL genes inLitopenaeus vannameiandPenaeus monodonshrimp [J].Animal Genetics, 2005,36(3): 235—236
[16]Xin J J, Liu X L, Li X L,et al. PCR-RFLP polymorphism of alpha-amylase gene and its association with growth traits ofLitopenaeus vannamei[J].Acta Oceanologica Sinica, 2011, 33(3): 124—130 [辛静静, 刘小林,李喜莲, 等. 凡纳滨对虾α-淀粉酶基因PCR-RFLP多态性与生长性状的相关分析. 海洋学报(中文版), 2011,33(3): 124—130]
[17]Huang J H, Ma Z M, Zhou F L,et al. Study on anatomy structure and histology of the ovary of wildPenaeus monodon(Fabricius) from the north of South China Sea[J].South China Fisheries Science, 2005, 1(3): 49—53[黄建华, 马之明, 周发林, 等. 南海北部野生斑节对虾卵巢解剖结构及组织学的研究. 南方水产, 2005, 1(3):49—53]
[18]Huang J H, Zhou F L, Ma Z M,et al. Morphological and histological observation on ovary development ofPenaeus monodonfrom northern South China Sea [J].Journal of Tropical Oceanography, 2006, 25(3): 47—52 [黄建华, 周发林, 马之明, 等. 南海北部斑节对虾卵巢发育的形态及组织学观察. 热带海洋学报, 2006, 25(3):47—52]
[19]Jiang S G, Yang C H, Zhou F L,et al. Breeding Technology ofPenaeus monodon[M]. Beijing: Ocean Press.2013, 80—86 [江世贵, 杨丛海, 周发林, 等. 斑节对虾种虾繁育技术. 北京: 海洋出版社. 2013, 80—86]
[20]Li Y D, Zhou F L, Huang J H,et al. Cloning and expression analysis of Cathepsin L cDNA ofPenaeus monodon[J].South China Fisheries Science, 2016, 12(3): 58—66[李运东, 周发林, 黄建华, 等. 斑节对虾组织蛋白酶L基因的克隆及其表达分析. 南方水产科学, 2016, 12(3):58—66]
[21]Zheng L M, Zhou F L, Yang Q B,et al. Molecular cloning and expression analysis of metallothionein fromPenaeus monodon[J].Acta Hydrobiologica Sinica, 2011,35(6): 913—919 [郑丽明, 周发林, 杨其彬, 等. 斑节对虾金属硫蛋白基因cDNA克隆与表达分析. 水生生物学报, 2011, 35(6): 913—919]
[22]Expression analysis of α-amylase gene in various tissues and early development ofCtenopharyngodon idellus[J].Marine Fisheries, 2015, 37(1): 31—37 [唐小红, 樊佳佳,白俊杰. 草鱼α-淀粉酶基因组织表达特征和早期发育的表达谱. 海洋渔业, 2015, 37(1): 31—37]
[23]Wu T T, Zhu X M. Studies on the activity of digestive enzymes in mandarin fish, black carp, grass carp, common carp, crucian carp and silver carp [J].Journal of Fishery Sciences of China, 1994, 1(2): 10—17 [吴婷婷, 朱晓鸣.鳜鱼、青鱼、草鱼、鲤、鲫、鲢消化酶活性的研究.中国水产科学, 1994, 1(2): 10—17]
[24]Peng Tao. Cloning and functional analysis of a-amylase gene inProcambarus clarkia[D]. Anhui Agricultural University, 2015 [彭涛. 克氏原螯虾α-淀粉酶基因的克隆与功能分析. 安徽农业大学, 2015]
[25]Cuvier-Peres A, Kestemont P. Development of some digestive enzymes in Eurasian perch larvaePerca fluviatilis[J].Fish Physiology and Biochemistry, 2001, 24(4):279—285
[26]Darias M J, Murray H M, Gallant J W,et al. Characterization of a partial alpha-amylase clone from red porgy(Pagrus pagrus): Expression during larval development[J].Comparative Biochemistry and Physiology B-Biochemistry amp; Molecular Biology, 2006, 143(2): 209—218
[27]Kim B G, Divakaran S, Brown C L,et al. Comparative digestive enzyme ontogeny in two marine larval fishes:Pacific threadfin (Polydactylus sexfilis) and bluefin trevally (Caranx melampygus) [J].Fish Physiology and Biochemistry, 2001, 24(3): 225—241
[28]Kurokawa T, Suzuki T, Ohta H,et al. Expression of pancreatic enzyme genes during the early larval stage of Japanese eelAnguilla japonica[J].Fisheries Science, 2002,68(4): 736—744
[29]Pan LQ, Liu H Y, Xiao G Q. A review on digestive enzyme of crustacean larvae [J].Journal of Fishery Sciences of China, 2006, 13(3): 492—501 [潘鲁青, 刘泓宇,肖国强. 甲壳动物幼体消化酶研究进展. 中国水产科学,2006, 13(3): 492—501]
[30]Tian H M, Wang Q, Zhao Y L,et al. Digestive enzyme activities and amino acids composition during embryonic development ofEriocheir sinensis[J].Journal of Fishery Sciences of China, 2003, 10(5): 404—408 [田华梅, 王群,赵云龙, 等. 中华绒螯蟹胚胎发育过程中的消化酶活力及氨基酸组成. 中国水产科学, 2003, 10(5): 404—408]
[31]Liu L H, Chen L Q, Zhou Y K,et al. Studies on the digestive enzymes of the chitosan [J].Feed Industry, 2006,27(18): 56—62 [刘立鹤, 陈立侨, 周永奎, 等. 甲壳动物消化酶的研究. 饲料工业, 2006, 27(18): 56—62]
[32]Wei J, Zhang X, Yu Y,et al. Comparative transcriptomic characterization of the early development in pacific white shrimpLitopenaeus vannamei[J].Plos One, 2014, 9(9):e106201
[33]Wei J, Zhang X, Yu Y,et al. RNA-seq reveals the dynamic and diverse features of digestive enzymes during early development of pacific white shrimpLitopenaeus vannamei[J].Comparative Biochemistry amp; Physiology Part D Genomics amp; Proteomics, 2014, 11(5): 37—44
CLONING AND EXPRESSION ANALYSIS OF ALPHA AMYLASE CDNA OF PENAEUS MONODON
YANG Qi-Bin1,2, LI Yun-Dong1,2, JIANG Shi-Gui1,2, HUANG Jian-Hua1,2, JIANG Song1,2, QIU Li-Hua1,2,ZHU Cai-Yan1,2and ZHOU Fa-Lin1,2
(1. South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510300, China;2. Key Laboratory of South China Sea Fishery Resources Exploitation amp;Utilization, Ministry of Agriculture,Guangzhou 510300, China)
In order to study the structure and biological function of the alpha-amylase gene inPenaeus monodon, the full-length cDNA sequence of α-Amylase fromPenaeus monodon(PmAmy) was obtained by high throughput transcriptome sequencing and RACE. ThePmAmycDNA included an open reading frame of 2175 bp encoding a polypeptide of 724 amino acids, and the predicted molecular mass and isoelectric point were 78.9 kD and 4.66, respectively. ThePmAmycontained a conservative A domain (Thr34-Ser410) and a C domain (Glu420-Ala496) of alpha amylase family. Homology analysis revealed that the PmAmy shared 47%—99% identity to other known amylase sequences, and the phylogenetic tree showed that the PmAmy was closely related toLitopenaeus vannamei. The expression levels ofPmAmyin hepatopancreas were significantly higher than those of the other tissues (P<0.05). ThePmAmyexpression was found in five ovarian development stages. The expression level in yolky stage (stageⅣ) was the highest among the five stages, and was the lowest in ovogonium stage (stageⅠ). The expression levels ofPmAmyshowed no significantly difference in ovary development stages. Expression ofPmAmywas detected in all tested growth stages, and the expression level in mysis was significantly higher than that in nauplius, zoea and post larval (P<0.05). These results suggested thatPmAmymight be associated with larval development inP. monodon.
Penaeus monodon; α-Amylase; Gene cloning; Gene expression; Larval development
Q781
A
1000-3207(2017)06-1186-07
2016-07-28;
2017-04-25
现代农业产业技术体系建设专项(CARS-48); 广东省省级科技计划项目(2014B020202003); 广东省海洋与渔业科技推广专项(A201501A06); 深圳市生物产业发展专项资金现代农业生物产业推广扶持计划项目(NYSW201400331010053)资助[Supported by China Agriculture Research System (CARS-48); Guangdong Province Project of China (2014B020202003);Guangdong Oceanic and Fisheries Project of China (A201501A06); Shenzhen Biological Industry Development Project of China(NYSW201400331010053)]
杨其彬(1972—), 男, 广东高州人; 本科; 主要从事水产动物遗传育种与分子生物学研究。E-mail: yangqibin@163.com
周发林(1975—); E-mail: zhoufalin@aliyun.com
10.7541/2017.147
doi: 10.7541/2017.148
