一个可区分银鲫F系和A+系BAC序列的特征、染色体定位和SCAR标记筛选
2017-11-29李熙银桂建芳
洪 玮 李熙银 周 莉 汪 洋 李 志 桂建芳
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
一个可区分银鲫F系和A+系BAC序列的特征、染色体定位和SCAR标记筛选
洪 玮1,2李熙银1周 莉1汪 洋1李 志1桂建芳1
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
为了揭示F系和A+系的遗传差异以及解析F系的遗传特征, 研究基于前期基因组Survey分析发现一个特异于银鲫F系大小为782 bp片段的基础, 首先采用混合池筛选方法从银鲫F系BAC文库中筛选出一个包含该特异片段的BAC克隆(F782-BAC)。鸟枪法测序结果表明, F782-BAC序列全长大小为172625 bp, 富含转座子序列, 包含1个具备完整结构的Tc1-like转座子, 以及不具备完整结构的transposase-like、PIF/Harbinger、target-primed reversed transcription(TPRT) 和transponX-element等6个转座子。用地高辛标记F782-BAC质粒得到的DNA探针进行了银鲫F和A+系有丝分裂中期相的染色体荧光原位杂交, 结果表明不论是在F系还是A+系的有丝分裂中期相中, 3个阳性荧光信号皆清晰地定位于3条大小相同的近端着丝粒染色体短臂的近着丝粒位置。进一步开展了银鲫A+系相应同源的F782-BAC序列的差异鉴定和序列分析, 揭示A+系缺失了大小为3395 bp的一段序列, 且特异于银鲫F系大小为782 bp的片段正好位于其中, 并由此筛选出可作为区分银鲫F系和A+系的SCAR标记。研究为银鲫的多倍化起源提供了进一步的染色体定位证据, 鉴定的SCAR标记可为银鲫分子标记辅助育种提供新的遗传标记。
多倍体银鲫; 转座子;Tc1-like; BAC; SCAR标记
多倍体银鲫通常被视为鲫(Carassius auratusL.)的一个亚种 (Carassius auratus gibelioBloch)[1],然而, 随着研究的深入, 已逐渐被视为一个独立的物种(Carassius gibelioBloch)[2—5]。银鲫不同于其他的单性脊椎动物, 在其自然群体中均发现有雄性个体存在[7—9], 且拥有单性雌核生殖和有性生殖等多重生殖方式[1,2,6]。最近的研究表明, 银鲫D系卵子与异种红鲤雄鱼的精子受精时行单性雌核生殖,当与同一克隆雄鱼的精子受精时行有性生殖, 而当与不同克隆的雄鱼精子受精时行类杂种生殖[2]。银鲫广泛分布于亚欧大陆, 拥有超过156条染色体[10],在经历了连续的两轮多倍化历程后[11], 目前正处在二倍化进程之中, 且生殖方式也正在由单性雌核生殖向两性有性生殖转变[2,3]。在二倍化过程中, 多倍体物种的基因组会发生缺失、复制和重组等巨大变化[12,13], 而在生物进化过程中, 这些染色体重排事件通常是转座子(Transposon)活动的结果[14]。因此在多倍体银鲫中进行转座子的相关研究可理解其基因组进化的内在驱动机制。
我国主养品种异育银鲫“中科3号”(A+系)是银鲫A系的精子在银鲫D系的卵质中通过雄核生殖产生的一个新的银鲫核质杂种克隆[15,16]。银鲫F系是由银鲫E系的卵子与团头鲂(Megaloabrama AmblycephalaYin)的精子经过冷休克处理, 行雌核生殖而来, 并表现出类似团头鲂的隆背性状[17]。染色体荧光原位杂交实验表明, 银鲫F系除了含有母本E系的156条染色体之外, 还含有来自团头鲂的微小染色体片段[17], 是一个具有潜力的育种新品系。为了揭示F系和A+系的遗传差异以及解析F系的遗传特征,在前期的研究中, 我们对F系和A+系分别进行了基因组Survey分析, 并通过基因组Soap分析获得了2个品系之间的差异序列。在本研究中, 我们选取了一个存在于银鲫F系而不存在于A+系、大小为782 bp的差异片段进行分析。我们首先采用混合池筛选方法从银鲫F系BAC文库中筛选了含有该差异片段的一个BAC克隆, 并对该BAC克隆进行了鸟枪法序列测定和生物信息学分析。结果表明, 该BAC克隆富含转座子序列, 其上有一个具有完整结构的转座子Tc1-like (Cg-Tc1-like转座子)。接着以地高辛标记的F782-BAC质粒DNA为探针进行染色体荧光原位杂交, 发现该序列都定位在银鲫F系和A+系中3个相同大小近端部着丝粒染色体的短臂上。最后根据F782-BAC的序列设计引物, 经比较分析筛选获得了2个可用于区分银鲫F系和A+系的SCAR分子标记。
1 材料与方法
1.1 实验材料
银鲫F系[17]和A+系[15]来自中国科学院水生生物研究所官桥实验基地。
1.2 基因组DNA提取及PCR扩增
取银鲫F系和A+系的鱼鳍提取其基因组DNA,使用Wizard®Genomic DNA Purification Kit(Promege公司)试剂盒。基因组提取和PCR扩增方法参照Dan等[18]和Zhu等[19], 引物序列见表1。
1.3 BAC克隆的筛选及提取
采用混合池筛选方法筛选包含特异片段F782的BAC克隆。对于384孔板的筛选, 我们首先构建16个行池和24个列池, 然后利用特异的引物对这16个行池和24个列池进行PCR筛选, 阳性行池和阳性列池的交叉点即为阳性克隆所在[20]。对阳性BAC克隆划线后挑取单克隆, PCR验证后对其进行扩大培养并提取BAC质粒。BAC质粒提取使用PureLink®HiPure Plasmid DNA Purefication Kits(Invitrogen公司)试剂盒, 具体过程参照说明书。
1.4 BAC克隆序列分析
BAC克隆在华大基因公司利用鸟枪法进行完全测序。利用GENESCAN (http://www.eurofins.de/food-analysis/laboratories/eurofins-genescan.aspx)等软件对BAC序列进行基因预测; 通过Repeat-Masker (http://www.repeatmasker.org/)在线利用Repbase重复序列数据库对BAC上的已知重复序列进行对比分析。将通过RepeatMasker预测出的已知重复序列提出后, 剩余的序列与BAC克隆序列本身进行对比, 进一步找出更多的未知重复序列。

表1 实验所用的引物序列Tab. 1 Primer sequences
1.5 转座子序列及结构分析
利用GENESCAN (http://www.eurofins.de/foodanalysis/laboratories/eurofins-genescan.aspx)预测BAC序列上Cg-Tc1-like转座酶的开放阅读框, 随后利用NCBI的CDD数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)确定Cg-Tc1-like的DD(34)E结构域, 之后利用DNAMAN软件对比得到转座子两端的反向重复序列以及TA靶位点重复, 最终确定Cg-Tc1-like转座子的核苷酸全长序列。
1.6 染色体的制备及染色体荧光原位杂交(FISH)
银鲫F系和A+系中期染色体的制备方法参考之前的报道[3,11]。利用DIG-Nick Translation Mix试剂盒(Roche公司)制备BAC质粒DNA荧光探针[3,11]。染色体荧光原位杂交的具体步骤参考文献。
1.7 DNA的回收与测序
PCR扩增产物经1%的琼脂糖凝胶检测后, 使用琼脂糖凝胶DNA回收试剂盒(OMEGA公司)回收特异性目的条带, 回收方法参照说明书。将回收产物连接到pMD18-T Vector (TaKaRa公司)上, 16℃连接3h。转入DH5α感受态大肠杆菌中, 用含氨苄青霉素的固体培养基培养后, PCR筛选阳性克隆送铂尚生物技术(上海)有限公司测序。
2 结果
2.1 银鲫F系特异片段F782的鉴定
根据差异片段F782的序列, 设计特异引物, 分别在银鲫F系和A+系各5尾个体中进行PCR扩增, 结果表明, 与基因组Survery分析结果一致, 该引物只能从银鲫F系5尾个体中扩增出大小为782 bp的特异片段, 而不能从A+系5尾个体中扩增出相应片段(图1)。
2.2 银鲫F系含有F782片段的BAC克隆筛选及其序列特征
采用混合池筛选的方法对银鲫F系BAC文库进行了筛选, 结果表明, 在第16号384孔板的第L行和第23列交叉孔对应的BAC克隆包含了差异片段F782。鸟枪法测序表明, 该F782-BAC克隆全长大小为172625 bp; GENESCAN预测结果显示, F782-BAC克隆序列包含有7个转座子, 其大小和方向如图2A所示, 它们依次排布为target-primed reversed transcription转座子(TPRT)、Cg-Tc1-like转座子、transposase-like转座子、PIF/Harbinger转座子、transpon X-element转座子、PIF/Harbinger转座子和target-primed reversed transcription转座子TPRT。其中Cg-Tc1-like、transposase-like和PIF/Harbinger转座子属于DNA转座子(Ⅱ类转座子), 通过“剪切-粘贴”的方式来进行转座[21,22];TPRT和transpon X-element转座子为反转录转座子(Ⅰ类转座子), 通过“复制-粘贴”的方式来进行转座[21, 22]。

图1 差异片段F782在银鲫A+系和F系中扩增结果的琼脂糖凝胶电泳图Fig. 1 Electrophoretogram of fragment F782 in strain A+ and strain F of gibel carp (M. DL2000 DNA marker)

图2 F782-BAC序列的转座子预测和重复序列分析Fig. 2 Prediction of transposons and repeated sequences in F782-BAC sequence
此外, Repeat Masker软件分析表明, 在F782-BAC序列中有28.9%序列是与转座子有关的重复序列, 其中DNA转座子中的重复序列占F782-BAC序列全长的4.1%, 反转录转座子中的短散在元件(Short interspersed elements, SINEs)、长散在元件(Long interspersed elememts, LINEs)和长末端重复反转座子(Long terminal repeat retrotransposons,LTRs)分别占F782-BAC序列全长的 0.3%、15.1%和 9.4%。除去与转座子相关的重复序列外, 还有1.4%的简单重复序列, 0.2%的低复杂性区段和15.6%的未知重复序列(图2B)。
进一步分析发现, 在所预测出的7个转座子中,transposase-like转座子、PIF/Harbinger转座子和transpon X-element转座子缺失了转座酶开放阅读框(Open reading frame, ORF), 两个TPRT转座子均只含有1个ORF2结构, 而完整的TPRT转座子应同时含有2个ORF结构(ORF1和ORF2), 这2个TPRT转座子也无转录活性; 只有Cg-Tc1-like转座子具有完整的结构, 具有潜在转录活性。Cg-Tc1-like大小为1633 bp, 两端各有一段反向重复序列(Inverted terminal repeats, ITRs)以及一个TA靶位点重复(TA target site duplication, TSD), 其中5′端ITR3长度为225 bp, 3′端ITR3长度为211 bp。Cg-Tc1-like转座子不含内含子, 其ORF长度为996 bp, 编码大小为331个氨基酸的转座酶。转座子序列第768到第1148个碱基编码一个完整的DD (34)E结构域。DD(34)E结构域含有2个天冬氨酸残基(D, D), 在距离第2个天冬氨酸残基的第34个氨基酸处有一个谷氨酸残基(E)[23]。DD (34)E结构域是Tc1-like转座子作用的重要位点, 它在转座子进行“剪切”时催化DNA链在特异位点的裂解与断裂。
2.3 F782-BAC序列在银鲫中染色体的定位
利用地高辛标记的F782-BAC质粒DNA, 分别对银鲫F和A+系有丝分裂中期相的染色体进行荧光原位杂交。如图3所示, 在银鲫F和A+系的有丝分裂中期相中, 3个阳性荧光信号皆清晰地定位于3条相同大小近端着丝粒染色体短臂的近着丝粒位置(图3), 这表明银鲫在其基因组或整套染色体组中的同源染色体有3个, 且F782-BAC序列在银鲫F和A+系存在同源序列。
2.4 银鲫A+系同源的F782-BAC序列的差异鉴定以及区分F系和A+系SCAR标记的筛选
为了揭示银鲫A+系同源的F782-BAC序列的差异, 我们首先根据F系F782-BAC序列设计了10对特异性引物(图4A), 分别在银鲫F和A+系中进行PCR扩增。如图4B所示, 这10对引物均能在银鲫F系中特异地扩增出目标大小的片段; 而在银鲫A+系中, 除F782片段与前述不能扩增出相应片段(图1)外, 在其上下游还各有一对引物不能扩增出片段(第Ⅱ和第Ⅳ片段), 其余7对引物均能扩增出相应的目的片段。接着, 我们在第Ⅰ个和第Ⅱ个片段之间(30122—30144 bp)以及第Ⅳ和第Ⅴ个片段之间(34713—34733 bp)设计了一对引物, 分别在银鲫F系和A+系的5尾个体中进行PCR扩增, 结果表明,在银鲫F系5尾个体中均扩增出一条大小约4600 bp的DNA片段, 而在银鲫A+系的5个个体中均扩增出一条大小约为1200 bp的DNA片段(图5C)。将回收的这2个大小不同的片段分别进行测序表明, 与F系相比, A+系缺失了大小为3395 bp的一段序列(图4D), 特异于银鲫F系大小为782 bp的片段正好位于其中。因此, 这对引物和前面用于扩增F782序列的引物都可作为区分银鲫F系和A+系的SCAR标记。
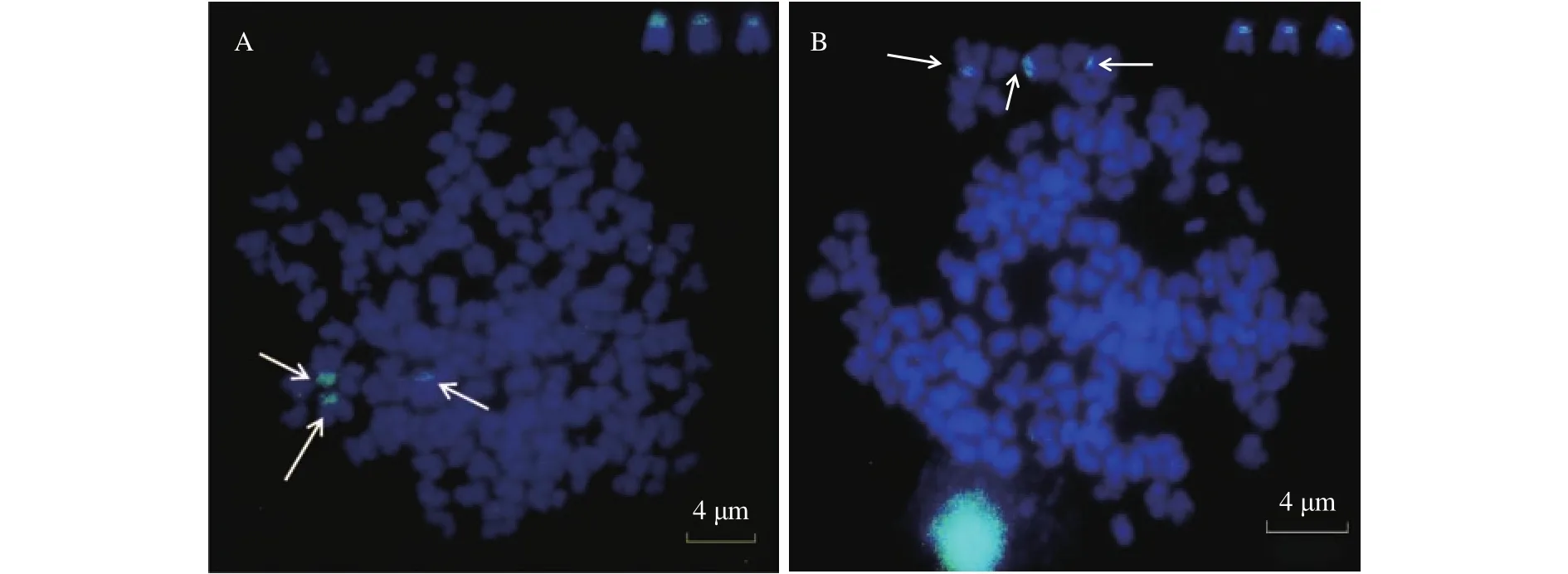
图3 地高辛标记的F782-BAC序列探针在银鲫有丝分裂中期相的荧光原位杂交Fig. 3 Fluorescence in situ hybridization of the DIG-labeled F782-BAC sequence on mitotic metaphases of gibel carp
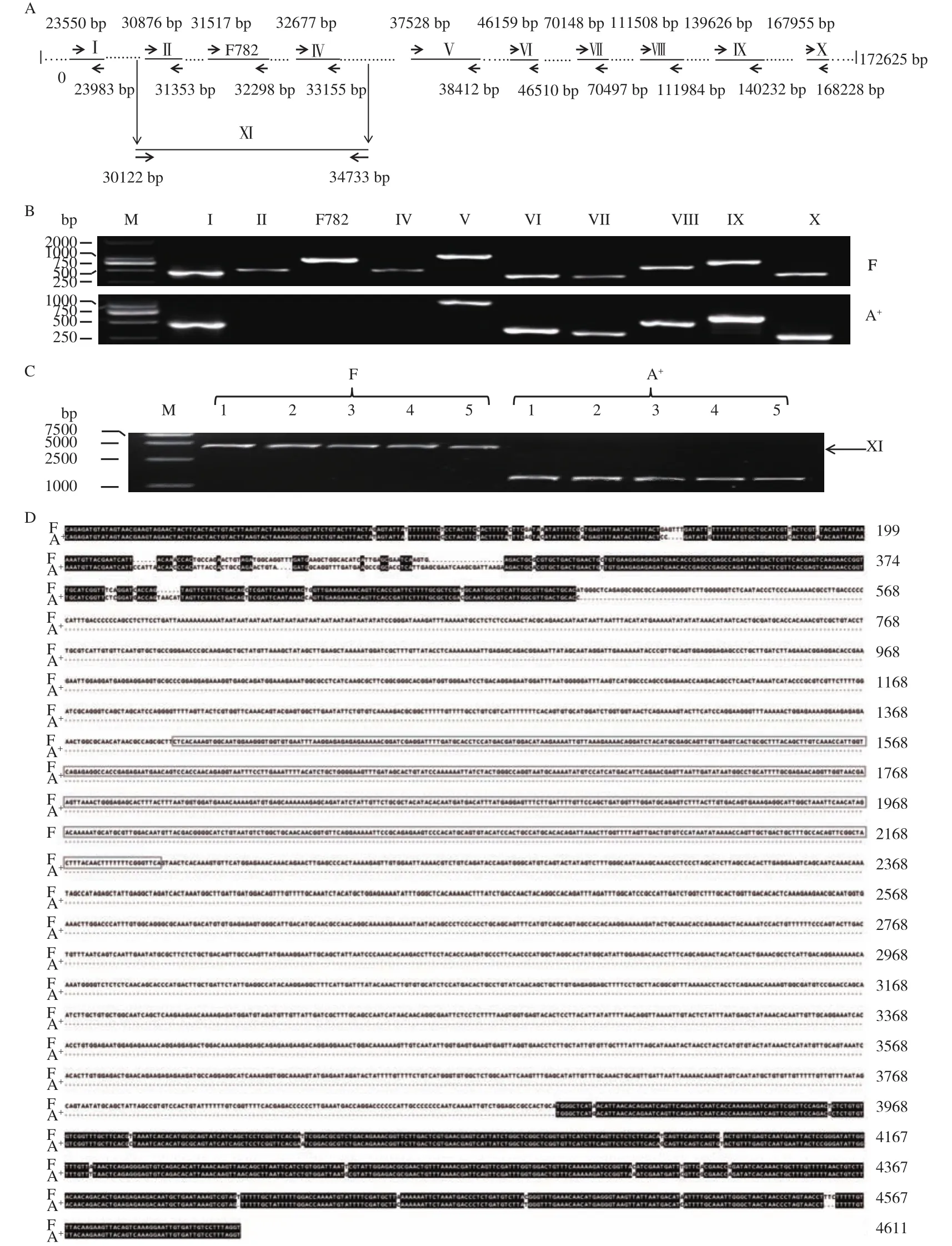
图4 F782-BAC序列在银鲫F系和A+系中的差异呈现Fig. 4 Differential screening of F782-BAC sequence between strain F and strain A+ of gibel carp
3 讨论
多倍体银鲫在其天然种群中存在着丰富的克隆多样性, 这些不同的克隆在形态学表型和遗传组成上都存在着显著的差异[1]。在前期研究中, 我们已经建立了血清转铁蛋白表型[24]、RAPD和SCAR标记[25,26]、微卫星DNA标记[27]、转铁蛋白等位基因多态性[26—28]以及mtDNA控制区序列[28]等多种遗传标记, 从我国银鲫天然种群中连续鉴别出众多不同的克隆, 并利用细胞工程育种技术和遗传标记辅助育种技术连续培育出三代异育银鲫品种, 推动了我国鲫鱼养殖业的快速持续发展[1]。为了揭示一个具有育种潜力的银鲫品系F系与主养品种“中科3号”的遗传差异以及解析F系的遗传特征, 本研究从基因组Survery分析获得的2个品系之间的差异序列中, 选取了一个存在于银鲫F系而不存在于A+系的差异片段进行分析, 经系列比较研究, 筛选获得了可用于区分银鲫F系和A+系的SCAR标记。
在鱼类的进化历程中, 多倍化是一个重要的且频繁发生的现象, 对其进化具有重要意义[19]。多倍化为基因功能的歧化和功能创新提供了原始材料[29], 增加了物种的适应性, 促进了物种的分化[30],是一种重要的进化动力[1]。相对于二倍化的四倍体鲤科鱼类, 银鲫被认为具有更高的倍性, 是一个正在二倍化的六倍体[1]。与银鲫5S rDNA染色体定位和单个染色体的描绘结果相同[18], F782-BAC克隆的信号位于3个相同大小的近端部着丝粒染色体上,而且它们的位置一致, 都是位于短臂的近着丝粒区域(图4)。上述的结果表明银鲫在其基因组或整套染色体组中的同源染色体为3个。对鲤鱼全基因组序列的解析证实了其异源四倍体特征和独特的全基因组复制事件[31]。本实验室对两个歧化的银鲫Dmrt1基因的染色体定位及系统发生分析, 揭示了银鲫的两轮多倍化起源历程。大约在1849万年前,一次早期的多倍化形成了银鲫、鲫和鲤的共同四倍体祖先; 随后大约在51万年前, 在鲫这个分支上发生了另一次近期的多倍化事件, 形成了现在的六倍体银鲫[11]。银鲫可能与鲤类似, 存在着两套不同来源的染色体组, 这两套染色体组之间虽然有一定的同源性, 但仍有十分明显的序列差异[11]。因此,当以单个染色体的进行描绘[21]或以F782-BAC克隆序列为探针进行染色体荧光原位杂交实验时, 只检测到3个荧光信号。
F782-BAC克隆序列上富含转座子序列(图2)。转座子是一类可以在基因组上及基因组之间移动的具有一定重复性的DNA片段[32,33]。根据转座机制, 转座子可分为DNA转座子和反转录转座子两类[34]。目前, 在真核生物基因组中, 已发现至少17个DNA转座子超家族[35]。转座子能够导致剪切位点的缺失, 插入位点特异序列的扩增[14,35,36], 最终增加等位基因的遗传多态性[37], 改变基因功能,甚至会产生新的基因或者使基因功能丧失[12]。转座子在移动过程中, 有时会造成剪切位点染色体断裂, 引起染色体重排[14,35,36]。转座子的活动能加速物种的进化速率, 被视为生物基因组进化的内在驱动[38]。银鲫目前正处在近期多倍化之后的二倍化过程之中, 且生殖方式也正在由雌核生殖向两性生殖转变[2,3]。在二倍化过程中, 多倍体物种的基因组会发生染色体缺失、复制和重组等巨大的变化[12,13], 而这些染色体重排现象通常是转座子活动的结果[14]。F782-BAC序列上含有丰富的转座子序列, 而与F系相比, A+系缺失了该克隆上一段大小为3393 bp的片段, 该缺失片段可能与转座子的活动相关联。Tc1-like转座子在硬骨鱼类基因组中占有很高的比例。在鲤中,Tc1-like转座子约占到基因组比例的近1.7%, 仅次于hAT转座子超家族(约2.3%)[31,39]。因此, 全面和深入地分析银鲫的转座子有助于我们了解银鲫复杂基因组的组成和基因组进化的内在驱动机制。
[1]Gui J F, Zhou L. Genetic basis and breeding application of clonal diversity and dual reproduction modes in polyploidCarassius auratus gibelio[J].SCIENCE CHINA Life Sciences, 2010, 42(2): 92—103 [桂建芳, 周莉. 多倍体银鲫克隆多样性和双重生殖方式的遗传基础和育种应用. 中国科学: 生命科学, 2010, 42(2): 92—103]
[2]Zhang J, Sun M, Zhou L,et al. Meiosis completion and various sperm responses lead to unisexual and sexual reproduction modes in one clone of polyploidCarassius gibelio[J].Scientific Reports, 2015, 5: 10898
[3]Li X Y, Zhang Q Y, Zhang J,et al. Extra microchromosomes play male determination role in polyploid gibel carp [J].Genetics, 2016, 115: 185843
[4]Rylkova K, Kalous L, Slechtova V,et al. Many branches,one root: First evidence for a monophyly of the morphologically highly diverse goldfish (Carassius auratus) [J].Aquaculture, 2010, 302(1-2): 36—41
[5]Liu W, Li S Z, Li Z,et al. Complete depletion of primordial germ cells in an all-female fish leads to Sex-biased gene expression alteration and sterile all-male occurrence[J].BMC Genomics, 2015, 16(1): 971
[6]Zhou L, Wang Y, Gui J F. Genetic evidence for gonochoristic reproduction in gynogenetic silver crucian carp(Carassius auratus gibeliobloch) as revealed by RAPD assays [J].Journal of Molecular Evolution, 2000, 51(5):498—506
[7]Jiang F F, Wang Z W, Zhou L,et al. High male incidence and evolutionary implications of triploid form in northeast AsiaCarassius auratuscomplex [J].Molecular Phylogenetics and Evolution, 2013, 66(1): 350—359
[8]Abramenko M I, Nadtoka E V, Makhotkin M A,et al.Distribution and cytogenetic features of triploid male goldfish in Azov basin [J].Ontogenez, 2004, 35(5):375—386
[9]Jakovlic I, Gui J F. Recent invasion and low level of divergence between diploid and triploid forms ofCarassius auratuscomplex in Croatia [J].Genetica, 2011, 139(6):789—804
[10]Zhou L, Gui J F. Karyotypic diversity in polyploid gibel carp,Carassius auratus gibelioBloch [J].Genetica, 2002,115(2): 223—232
[11]Li X Y, Zhang X J, Li Z,et al. Evolutionary history of two divergentDmrt1genes reveals two rounds of polyploidy origins in gibel carp [J].Molecular Phylogenetics and Evolution, 2014, 78(1): 96—104
[12]Chen Z J, Ni Z F. Mechanisms of genomic rearrangements and gene expression changes in plant polyploids[J].Bioessays, 2006, 28(3): 240—252
[13]Tate J A, Joshi P, Soltis K A,et al. On the road to diploidization? Homoeolog loss in independently formed populations of the allopolyploidTragopogon miscellus(Asteraceae) [J].BMC Plant Biology, 2009, 9(1): 80
[14]López M M, Pérez L G. DNA Transposons: Nature and applications in Genomics [J].Current Genomics, 2010,11(2): 115—128
[15]Wang Z W, Zhu H P, Wang D,et al. A novel nucleocytoplasmic hybrid clone formed via androgenesis in polyploid gibel carp [J].BMC Research Notes, 2011,4(1): 1—13
[16]Mei J, Gui J F. Genetic basis and biotechnological manipulation of sexual dimorphism and sex determination in fish[J].SCIENCE CHINA Life Sciences, 2014, 44(12):1198—1212 [梅洁, 桂建芳. 鱼类性别异形和性别决定的遗传基础及其生物技术操控. 中国科学: 生命科学,2014, 44(12): 1198—1212]
[17]Yi M S, Li Y Q, Liu J D,et al. Molecular cytogenetic detection of paternal chromosome fragments in allogynogenetic gibel carp,Carassius auratus gibelioBloch [J].Chromosome Research, 2003, 11(7): 665—671
[18]Dan C, Mei J, Wang D,et al. Genetic Differentiation and Efficient Sex specific Marker Development of a Pair of Y- and X-linked Markers in Yellow Catfish [J].International Journal of Biological Sciences, 2013, 9(10):1043—1049
[19]Zhu H P, Ma D M, Gui J F. Triploid origin of the gibel carp as revealed by 5S rDNA localization and chromosome painting [J].Chromosome Research, 2006, 14(7):767—776
[20]Peng J X, Xie J L, Zhou L,et al. Evolutionary conservation ofDazlgenomic organization and its continuous and dynamic distribution throughout germline development in gynogenetic gibel carp [J].Journal of Experimental Zoology Part B Molecular amp; Developmental Evolution, 2009,312B(8): 855—871
[21]Feschotte C, Pritham E J. DNA Transposons and the Evolution of Eukaryotic Genomes [J].The Annual Review of Genetics, 2007, 41: 331—368
[22]Levin H L, Moran J V. Dynamic interactions between transposable elements and their hosts [J].Nature Reviews,2011, 12(9): 615—627
[23]Shao H, Tu Z. Expanding the diversity of theIS630-Tc1-mariner superfamily: discovery of a unique DD37E transposon and reclassification of the DD37D and DD39D transposons [J].Genetics, 2001, 159(3): 1103—1115
[24]Yang L, Yang S T, Wei X H,et al. Genetic diversity among different clones of the gynogenetic silver crucian carp,Carassius auratus gibelio, revealed by transferring and isozyme markers [J].Biochemical Genetic, 2001,39(5—6): 214—225
[25]Zhou L, Wang Y, Gui J F. Analysis of genetic heterogeneity among five gynogenetic clones of silver crucian carp,Carassius auratus gibelioBloch, based on detection of RAPD molecular markers [J].Cytogenetic amp; Cell Genetics, 2000, 88(1-2): 129—133
[26]Yang L, Gui J F. Positive selection on multiple antique allelic lineages of transferrin in the polyploidCarassius auratus[J].Molecular Biology Evoutionl, 2004, 21(7):1264—1277
[27]Yang L, Zhou L, Gui J F. Molecular basis of transferrin polymorphism in goldfish (Carassius auratus) [J].Genetica, 2004, 121(3): 303—313
[28]Li F B, Gui J F. Clonal diversity and genealogical relationships of gibel carp in four hatcheries [J].Animal Genetics, 2008, 39(1): 28—33
[29]Liu S, Li Z, Gui J F. Fish-specific duplicatedDmrt2bcontributes to a divergent function through Hedgehog pathway and maintains left-right asymmetry establishment function [J].PLoS One, 2009, 4(9): 761—768
[30]Parisod C, Holderegger R, Brochmann C. Evolutionary consequences of autopolyploidy [J].New Phytologist,2010, 186(1): 5—17
[31]Xu P, Li J T, Xu J. Genome sequence and genetic diversity of the common carp,Cyprinus carpio[J].Nature Genetics, 2014, 46(11): 1212—1219
[32]Van O T, Camilli A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms[J].Nature Reviews, 2013, 11(7): 435—442
[33]Tian H X. Research Progress of transposable elements in genome and gene evolution [J].Journal of Anhui Agricultural Sciences, 2011, 39(20): 12018—12020 [田海霞. 转座子在基因组和基因进化方面的研究进展. 安徽农业科学, 2011, 39(20): 12018—12020]
[34]Lerat E. Identifying repeats and transposable elements in sequenced genomes: how to find your way through the dense forest of programs [J].Heredity, 2010, 104(6):520—533
[35]Guo X M, Zhang Q Q, Sun Y W,et al.Tc1-like TransposaseThm3of Silver Carp (Hypophthalmichthys molitrix) Can Mediate Gene Transposition in the Genome of Blunt Snout Bream (Megalobrama amblycephala) [J].Heart Rhythm, 2015, 5(12): 464—471
[36]Lohe A R, Moriyama E N, Lidholm D A,et al. Horizontal transmission, vertical inactivation, and stochastic loss ofmariner-like transposable elements [J].Molecular Biology amp; Evolution, 1995, 12(1): 62—72
[37]Subramanian R A, Akala O O, Adejinmi J O,et al.Topi,anIS630/Tc1/mariner-type transposable element in the African malaria mosquito,Anopheles gambiae[J].Gene,2008, 423(1): 63—71
[38]Liu D, Tang W Q, Yang J Q,et al. Recent advancements inTc1-Like transposons [J].SCIENCE CHINA Life Sciences, 2011, 41(2): 87—96 [刘东, 唐文乔, 杨金权, 等. 类Tc1转座子研究进展. 中国科学: 生命科学, 2011, 41(2):87—96]
[39]Wang Q L, Ji P F, Xu P,et al. Identification and characterization of a novelTc1-like transposon in theCyprinus carpiogenome [J].Journal of Fishery Sciences of China,2011, 18(6): 1392—1398 [王全乐, 冀培丰, 徐鹏, 等. 鲤基因组中1个Tc1类转座的发现与鉴定. 中国水产科学,2011, 18(6): 1392—1398]
CHARACTERIZATION, CHROMOSOME LOCALIZATION, AND SCAR MARKER SCREENING OF A BAC SEQUENCE FOR DISCRIMINATING STRAIN F FROM STRAIN A+IN GIBEL CARP
HONG Wei1,2, LI Xi-Yin1, ZHOU Li1, WANG Yang1, LI Zhi1and GUI Jian-Fang1
(1. State Key Laboratory of Freshwater Ecology and Biotechnology, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
Gibel carp (Carassius gibelioBloch) has been widely used for basic and applied studies on developmental genetics and genetic engineering because of the polyploid background and multiple reproduction modes. In this study,we isolated a positive BAC clone with the fragment (F782-BAC) from a BAC library of F strain based on a 782 bp fragment (F782) specific to F strain revealed by comparative genome surveys between F and A+strains. Shotgun sequencing revealed the complete sequence of the F782-BAC that is composed of 172625 bp containing a completeTc1-like transposon and six incomplete transposons such astransposase-like transposon,PIF/Harbingertransposon, targetprimed reversed transcription (TPRT) transposon, and transponX-elementtransposon. Using the DIG-labeled F782-BAC sequence as a probe, three positive green fluorescence signals were clearly detected on short arms of three homologous acrocentric chromosomes in mitotic metaphases of both F and A+strains. Moreover, differential screening and sequence analysis of the homologus F782-BAC sequence were performed on the A+strain, and a deleted fragment of 3395 bp was identified in the corresponding F782-BAC sequence of A+strain, in which includes the F strain-specific fragment F782. Furthermore, we developed valuable SCAR markers for discriminating strain F from strain A+in gibel carp. Therefore, the current data provide genetic evidence for polyploidy origin in gibel carp and the identified SCAR markers for the marker-assisted selective breeding in gibel carp.
Polyploid gibel carp; Transposon;Tc1-like; BAC; SCAR marker
Q344+.1
A
1000-3207(2017)06-1169-08
10.7541/2017.145
2016-04-20;
2017-01-17
中国科学院战略性先导专项(XDA08030201); 现代农业产业技术体系(NYCYTX-49)资助 [Supported by the Strategic Priority Research Program of the Chinese Academy of Sciences (XDA08030201); Modern Agro-industry Technology Research System(NYCYTX-49)]
洪玮(1992—), 女, 江西南昌人; 硕士研究生; 主要从事发育遗传学研究。E-mail: 353849317@qq.com
桂建芳, E-mail: jfgui@ihb.ac.cn
