苹果YABBY基因家族的鉴定、进化及表达分析
2017-08-07邵红霞陈鸿飞吴海芹赵彩平韩明玉
邵红霞,陈鸿飞,张 东,吴海芹,赵彩平,韩明玉
(西北农林科技大学 园艺学院,陕西 杨凌 712100)
苹果YABBY基因家族的鉴定、进化及表达分析
邵红霞,陈鸿飞,张 东,吴海芹,赵彩平,韩明玉*
(西北农林科技大学 园艺学院,陕西 杨凌 712100)
从苹果全基因组范围内共鉴定出13个YABBY基因,通过聚类分析将其分为YAB1/YAB3、YAB2、INO、YAB5和CRC 5个亚家族。MdYABBY基因分布在苹果的9条染色体上,在第16条染色体上分布最多。MdYABBY蛋白长度介于60~538个氨基酸,等电点为4.84~9.81,所有MdYABBY蛋白都包含有锌指结构和YABBY 两个保守结构域,同一个亚家族内的成员表现出相似的基因结构和蛋白保守基序分布;基因顺式作用元件分析发现,MdYABBY基因包含许多与抗逆和激素响应有关的作用元件。基因表达分析发现,MdYABBY基因在花、果和叶中的表达量较高,5个MdYABBY基因在果实发育的3个时期均明显地上调表达。
苹果;YABBY基因;聚类分析;进化;基因表达
转录因子能够特异识别并结合于靶基因上游启动子区域的特异DNA序列模体,使其转录激活或抑制,从而调节植物的生长发育[1-2]。根据转录因子蛋白质序列中特异DNA结合域的不同,可以将转录因子分为不同的家族[3-4]。一些转录因子同时存在于植物和动物,如HB(同源异型盒蛋白)、HSF(热激转录因子)等;也有一些转录因子是植物特有的,比如WRKY转录因子[5]、YABBY转录因子[6-7]。其中,YABBY转录因子包含2个保守结构域:N端的锌指结构域和C端的YABBY结构域[8];除锌指结构域和YABBY结构域以外,不同亚家族基因编码的蛋白通常还具有以该亚家族为特点的其他保守结构域[9]。在拟南芥、西红柿等植物中,YABBY基因家族包含5个亚族,分别为CRABS CLAW(CRC)、FILAMENTOUS FLOWER(FIL)/YABBY3(YAB3)、INNER NO OUTER(INO)、YABBY2(YAB2)和YABBY5(YAB5),而水稻中只包含4个亚族,没有YABBY5(YAB5)亚家族。同一亚族内YABBY基因在进化过程中往往发生功能分化[10-12]。此外,拟南芥、水稻和番茄等被子植物中许多YABBY基因的发现为研究YABBY基因家族的进化提供了重要参考价值[12]。
大量的研究表明,YABBY转录因子能够决定植物侧生器官远轴面细胞命运[13],并与叶和花等器官的形态建成有关[6,14]。拟南芥中共鉴定出6个YABBY基因家族成员,分别为FILAMENTOUSFLOWER(FIL)、YABBY3、CRABSCLAW(CRC)、INNERNOOUTER(INO)、YABBY2和YABBY5[15]。其中,FIL在植物花器官发育过程中发挥重要作用[16-18],CRC跟蜜腺大小和心皮极性有关[19-20],INO参与外珠被的发育[21],YAB2和YAB3在侧生器官的远轴面表达[22]。
苹果被誉为温带水果之王,与人们生活和国民经济密不可分。然而,YABBY蛋白作为一类影响植物生长发育的重要调控因子,在苹果中研究还比较少。本研究中,在苹果全基因组范围内共鉴定出13个YABBY基因家族成员,并对它们的基因复制、基因结构、蛋白保守基序、顺式作用元件、组织特异性和在果实发育中的表达特性等做了研究,以期为进一步深入探索苹果YABBY基因家族的功能提供借鉴。
1 材料与方法
1.1 苹果YABBY基因的鉴定
在Pfam数据库(http://pfam.xfam.org/)下载包含YABBY结构域的多重序列比对文件(PF04690),并以此文件构建HMM模型(隐马尔科夫模型)。从GDR(Genome Database for Rosaceae)数据库(http://www.phytozome.net/apple)下载金冠苹果全基因组序列[23]。然后用HMMER 2.0[24]软件包在苹果全基因组范围内进行YABBY家族成员的搜索(e≥0.01)。为了进一步确保搜索的准确性,对得到的所有苹果YABBY家族候选成员用Pfam(http://pfam.xfam.org/search/sequence)和NCBI Conserved Domains Database(NCBI CDD, http://www.ncbi.nlm.nih.gov/Structure/cdd/)数据库进行YABBY蛋白保守结构域预测(e≥0.01)[25]。最后,在苹果全基因组范围内共鉴定出13个YABBY基因。
1.2 苹果YABBY基因的系统进化分析
依据前人对拟南芥、玉米、水稻和西红柿YABBY基因的鉴定结果[14,26,11,27],分别在The Arabidopsis Information Resource(TAIR,http://www.Arabidopsis.org/index.jsp)、V3版玉米自交系B73参考基因组数据(http: //plants. Ensembl.org /Zea_mays /Info /Index)、National Center for Biotechnology Information(NCBI,https://www.ncbi.nlm.nih.gov/)和Sol Genomics Network (https://solgenomics.net/)下载拟南芥、玉米、水稻和西红柿的YABBY蛋白序列,然后利用MAGE 5.0软件对来自5个物种的共49个YABBYA蛋白构建系统发育树。生成算法采用邻接法(Neighbor-Joining,NJ),校验参数采用Bootstrap重复1 000次。
1.3 苹果YABBY基因的复制和染色体位置
从AppleGFDB(http://www.applegene.org/)下载苹果YABBY基因的染色体位置信息,用MapInspect软件作图。利用DNAMAN软件[28]分别对13个苹果YABBY的基因和蛋白序列进行多重序列比对,依据比对结果鉴定苹果YABBY基因旁系同源对。旁系同源对基因须满足以下2个条件:(1)两基因序列对齐之后,短基因序列比长基因序列的覆盖率大于70%。(2)两基因蛋白序列的一致性大于70%[29-30]。
1.4 苹果YABBY基因的结构、保守基序分析
利用在线Gene Structure Display Server(GSDS, http://gsds.cbi.pku.edu.cn/)工具对苹果YABBY基因结构进行分析。利用在线MEME 4.11.1(http://meme-suite.org/)工具对苹果YABBY蛋白序列进行保守基序搜索,除最大保守基序数量为10个以外,其他参数均为默认设置。
1.5 苹果YABBY蛋白的理化性质和多重序列比对分析
用ExPaSy提供的在线Protparam(http://web.expasy.org/protparam/)软件进行蛋白长度、分子量、理论等电点、不稳定系数、脂肪族氨基酸指数和蛋白质疏水性分析。利用DNAMAN软件[28]对13个苹果YABBY蛋白和6个拟南芥YABBY蛋白进行多重序列比对,参数采用系统默认数值。
1.6 苹果YABBY基因的顺式作用元件分析
从金冠苹果全基因组序列中获取YABBY基因起始密码子(ATG)上游1 500 bp的DNA序列,利用在线PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行分析,采用默认参数设置。
1.7 苹果YABBY基因的表达特性分析
从The European Bioinformatics Institute(EBI,http://www.ebi.ac.uk/)芯片数据库下载金冠以及不同苹果杂交种不同器官的基因表达数据(E-GEOD-42873),提取MdYABBY基因表达量;同样在EBI芯片数据库下载果实发育芯片数据(E-GEOD-24523)及其探针序列(A-GEOD-11164),构建探针序列的本地blast数据库,然后利用苹果YABBYCDS序列进行比对,选取e=0且DNAMAN软件比对之后完全匹配的探针代表该YABBY基因,提取MdYABBY基因表达量。最后,采用Mev_4_9_0软件进行聚类并作图分析。
2 结果与分析
2.1 苹果YABBY基因的鉴定
通过生物信息学分析,本研究共获得13个苹果YABBY基因家族成员(表1),根据它们的染色体位置信息,将得到的13个苹果YABBY基因依次命名为MdYABBY1 ~MdYABBY13。对MdYABBY基因和MdYABBY蛋白的理化性质进行分析后发现,MdYABBY基因的CDS序列长度介于183(MdYABBY11)~1 617 bp(MdYABBY6);MdYABBY蛋白包含60(MdYABBY11)~538(MdYABBY6)个氨基酸,分子量为6 691.5(MdYABBY11)~59 846.3 u(MdYABBY6),等电点介于4.84(MdYABBY13)~9.81 (MdYABBY11)。进一步研究发现,所有预测的MdYABBY蛋白的GRAVY值均为负值,即MdYABBY蛋白都是亲水蛋白,只是亲水程度不同。13个MdYABBY蛋白成员中只有MdYABBY1,MdYABBY10和MdYABBY11的不稳定
表1 苹果YABBY基因家族
Table 1 YABBY gene family in apple
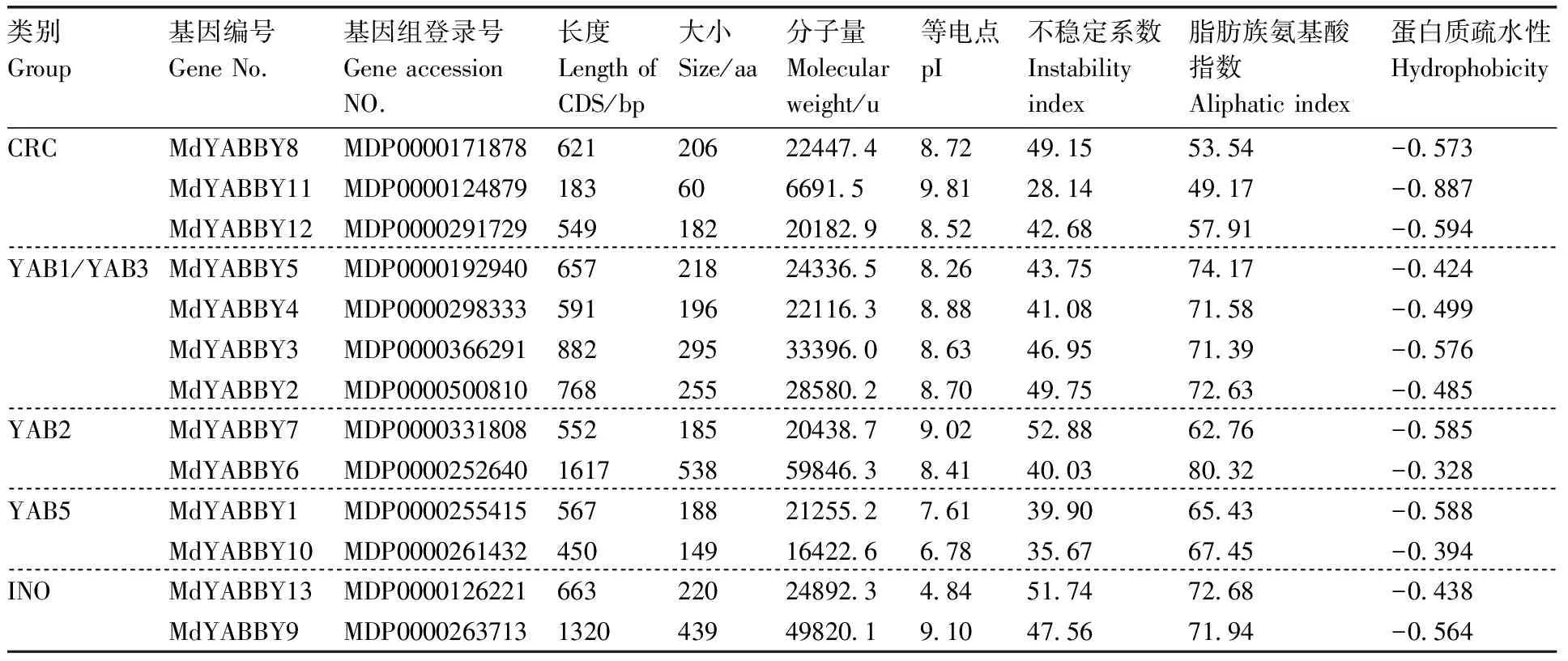
类别Group基因编号GeneNo.基因组登录号GeneaccessionNO.长度LengthofCDS/bp大小Size/aa分子量Molecularweight/u等电点pI不稳定系数Instabilityindex脂肪族氨基酸指数Aliphaticindex蛋白质疏水性HydrophobicityCRCMdYABBY8MDP000017187862120622447487249155354-0573MdYABBY11MDP0000124879183606691598128144917-0887MdYABBY12MDP000029172954918220182985242685791-0594YAB1/YAB3MdYABBY5MDP000019294065721824336582643757417-0424MdYABBY4MDP000029833359119622116388841087158-0499MdYABBY3MDP000036629188229533396086346957139-0576MdYABBY2MDP000050081076825528580287049757263-0485YAB2MdYABBY7MDP000033180855218520438790252886276-0585MdYABBY6MDP0000252640161753859846384140038032-0328YAB5MdYABBY1MDP000025541556718821255276139906543-0588MdYABBY10MDP000026143245014916422667835676745-0394INOMdYABBY13MDP000012622166322024892348451747268-0438MdYABBY9MDP0000263713132043949820191047567194-0564
系数小于40,证明大部分MdYABBY蛋白都是不稳定蛋白。这些分析结果可以进一步为MdYABBY蛋白的提纯、活性和功能研究提供理论基础。
2.2 苹果YABBY基因的复制和染色体定位分析
根据13个MdYABBY基因在染色体上的位置信息,利用MapInspect工具进行染色体定位作图。所有的MdYABBY基因都能找到相匹配染色体定位信息,共分布在9条染色体上(分别为染色体2、5、6、9、10、11、13、15和16)(图1)。其中,16号染色体分布3个,是富集MdYABBY基因最多的染色体,在第2和13号染色体上均分布2个MdYABBY基因,其他染色体分别仅有1个MdYABBY基因分布。另外,共鉴定出3组基因复制对(MdYABBY1/MdYABBY10、MdYABBY8/MdYABBY12、MdYABBY4/MdYABBY5)。
2.3YABBY基因系统进化树的构建和蛋白多重序列比对
为了研究MdYABBY基因的系统进化关系,利用MAGE 5.0软件构建了一个包含拟南芥、番茄、苹果、水稻和玉米5个物种YABBY基因的系统发育树(图2)。发现苹果YABBY家族与拟南芥、玉米、水稻和番茄的相似性较低,而苹果YABBY家族成员之间有明显的对应关系,比如,MdYABBY9与MdYABBY13,MdYABBY8与MdYABBY11、MdYABBY12,MdYABBY1与MdYABBY10等。根据它们的系统进化关系,并结合前人对拟南芥、玉米和水稻YABBY基因的系统进化分析[31,12],将13个MdYABBY基因分为5个亚家族,分别命名为YAB1/YAB3、YAB2、CRC、INO和YAB5。其中,YAB1/YAB3包含的家族成员数量最多,有4个家族成员(MdYABBY2、MdYABBY3、MdYABBY4和MdYABBY5),CRC包含有3个家族成员(MdYABBY8、MdYABBY11和MdYABBY12);YAB2、YAB5和INO都分别只包含2个家族成员。
为了研究YABBY蛋白序列的特征,用DNAMAN软件对苹果和拟南芥所有的YABBY蛋白的氨基酸进行多重序列比对分析,结果表明,所有的序列都具有2个相对保守的结构域,即位于N端的锌指结构域和C端的YABBY结构域(图3),这说明YABBY蛋白在不同物种间具有高度保守性。
2.4MdYABBY基因结构与蛋白保守基序分析
利用在线基因结构显示系统(GSDS)对所有YABBY基因的结构分析发现,MdYABBY基因的外显子个数从3(MdYABBY12)到11(MdYABBY6)不等。利用MEME工具对所有YABBY家族成员的保守基序进行搜索鉴定,共鉴定出10个保守基序,其中Motif 2和Motif 3代表C2C2型锌指结构域,Motif 1代表YABBY结构域。同一个亚家族的成员表现出相似的基因结构和蛋白保守基序分布,比如亚家族YAB1/YAB3的成员都含有7个外显子和Motif 4、Motif 5,且都是由第4~6个外显子编码YABBY结构域。亚家族INO中所有成员的YABBY结构域都是由自3′端向5′端为顺序的第2、3和4这3个外显子所编码,且都包含motif 9和motif 10。进一步分析发现,除MdYABBY10以外,所有成员的YABBY结构域均由3个外显子编码;所有编码YABBY结构域的外显子均位于基因片段的3′端,所有编码锌指结构域的外显子位于基因片段的5′端。上述结果表明,无论是基因结构还是蛋白保守基序,MdYABBY基因家族都有一定的保守性。
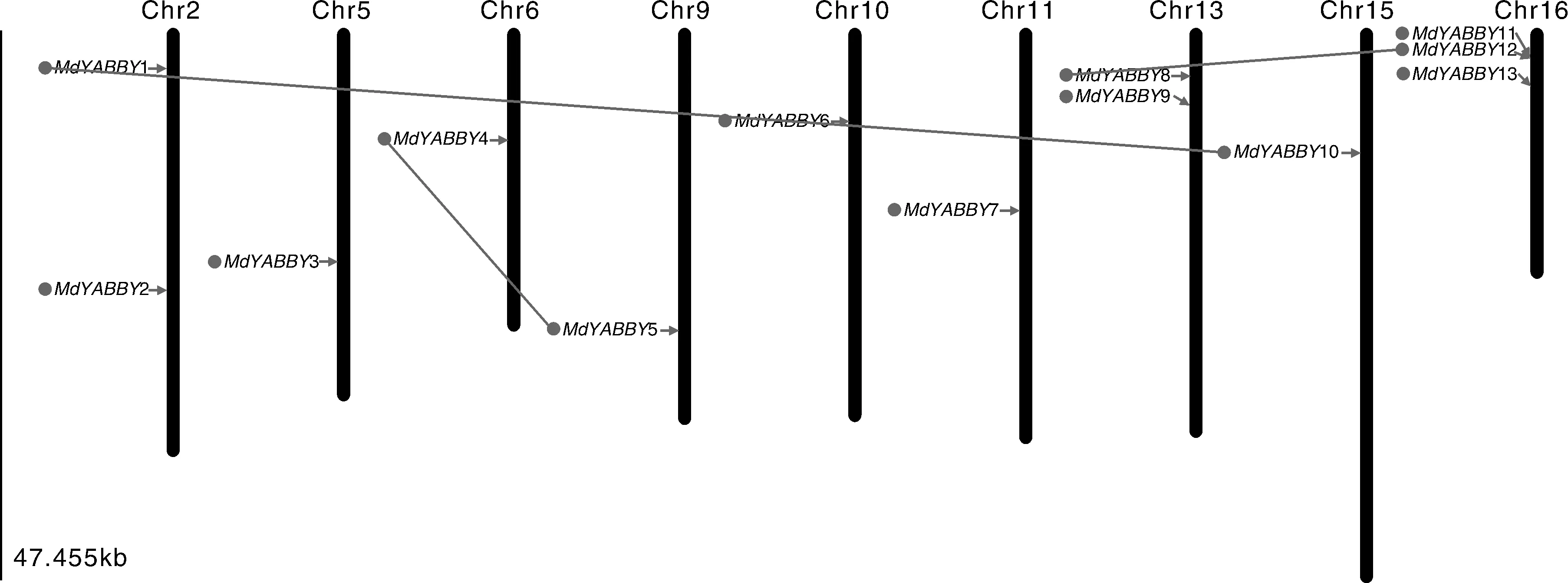
图1 MdYABBY基因在染色体上的位置Fig.1 Chromosomal locations of MdYABBY genes
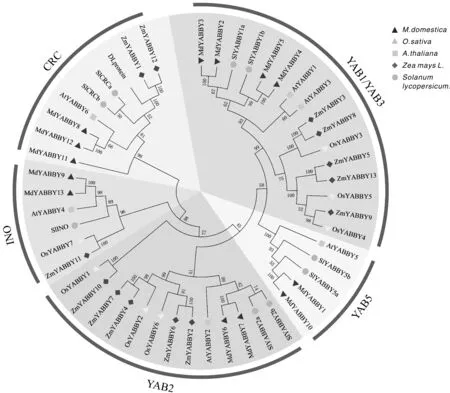
图2 苹果、水稻、玉米、番茄和拟南芥YABBY转录因子的系统发育树Fig.2 An unrooted phylogenetic tree of YABBY transcription factors in apple, rice, maize, tomato and Arabidopsis

图3 苹果和拟南芥YABBY蛋白保守结构域氨基酸序列比对Fig.3 Amino acid sequence alignment of YABBY protein conserved domains in apple and Arabidopsis
2.5MdYABBY基因的顺式作用元件分析
基因的表达依赖于顺式作用元件和反式作用因子的相互作用,通过分析启动子及其上游序列的顺式作用元件能够预测基因的功能。对13个苹果YABBY基因的顺式作用元件进行分析发现,YABBY基因含有大量与光响应和胚乳表达相关的顺式作用元件,其次,含有许多与抗逆(干旱和低温响应等)和激素响应(脱落酸和赤霉素诱导等)有关的作用元件(图5)。

图4 MdYABBY基因结构与蛋白基序分配Fig.4 Gene structures of MdYABBY genes and schematic diagram of conserved motifs of MdYABBY proteins
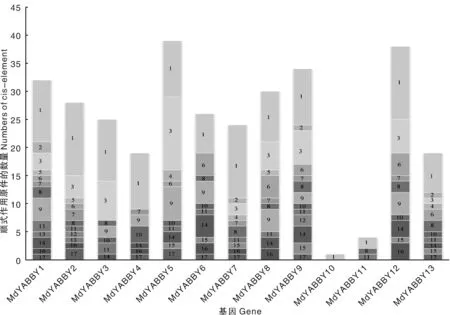
1, 光响应;2, 分生组织表达与激活;3, 胚乳表达;4, 种子特异性调控;5, 芽特异表达和光响应;6, 抗病和胁迫诱导;7, 玉米醇溶蛋白代谢调控;8, 脱落酸诱导;9, 茉莉酸甲酯诱导;10, 水杨酸诱导;11, 赤霉素诱导;12, 生长素诱导;13, 乙烯响应;14, 干旱诱导;15, 低温响应;16, 热胁迫响应;17, 昼夜节律调控1, light-responsiveness; 2, meristem expression and specific activation; 3, endosperm expression; 4, Seed-specific regulation; 5, shoot-specific expression and light responsiveness; 6, defense and stress responsiveness; 7, zein metabolism regulation; 8, abscisic acid responsiveness; 9, MeJA-responsiveness; 10, salicylic acid responsiveness; 11, gibberellin-responsiveness; 12, auxin-responsiveness; 13, ethylene-responsiveness; 14, drought-inducibility; 15, low-temperature responsiveness; 16, heat stress responsiveness; 17, circadian control图5 MdYABBY基因顺式作用元件Fig.5 The cis-acting elements of MdYABBY genes
2.6MdYABBY基因在不同品种苹果中的表达
为了研究MdYABBY基因在不同组织中的表达特性,从EBI芯片数据库下载MdYABBY基因在金冠以及不同杂交种苹果不同器官的表达数据[32],并作图(图6)。分析发现,MdYABBY7在花、叶以及不同成熟期的果实中表达量均较高,MdYABBY1和MdYABBY10在不同杂交种苹果花和叶中的表达量较高,MdYABBY8、MdYABBY11和MdYABBY12在不同杂交种苹果花和果实中的表达量明显高于其他器官。总体来看,苹果YABBY家族基因在花、果和叶中的表达量相对较高。
2.7MdYABBY基因在不同果实发育时期的表达
为了进一步研究MdYABBY基因在苹果果实发育过程中的作用,从EBI芯片数据库获取果实发育芯片数据,表达结果如图7所示。图中从左到右依次分别代表苹果品种Honey Crisp(HC)和Crispps Pink(CP)在果实发育过程的3个时期:果实成熟期(week-0)、果实成熟之前2周(week-2)和果实成熟之前4周(week-4)MdYABBY基因的表达情况。结果表明,MdYABBY1、MdYABBY8和MdYABBY10在果实发育的3个时期表达量一直较低,而MdYABBY2、MdYABBY3、MdYABBY6、MdYABBY7和MdYABBY9在不同品种果实发育的3个时期表达量均比较高。
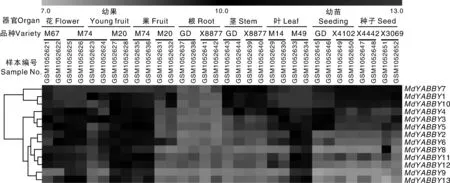
GD, 金冠;MM67、M74、M20、X8877、M14、M49、X4102、X4442和X3069为不同苹果杂交种GD, Golden Delicious; M67, M74, M20, X8877, M14, M49, X4102, X4442 and X3069 are various apple hybrids图6 MdYABBY基因在不同品种苹果不同器官中的表达Fig.6 The expresstion levels of MdYABBY genes in various apple organ systems
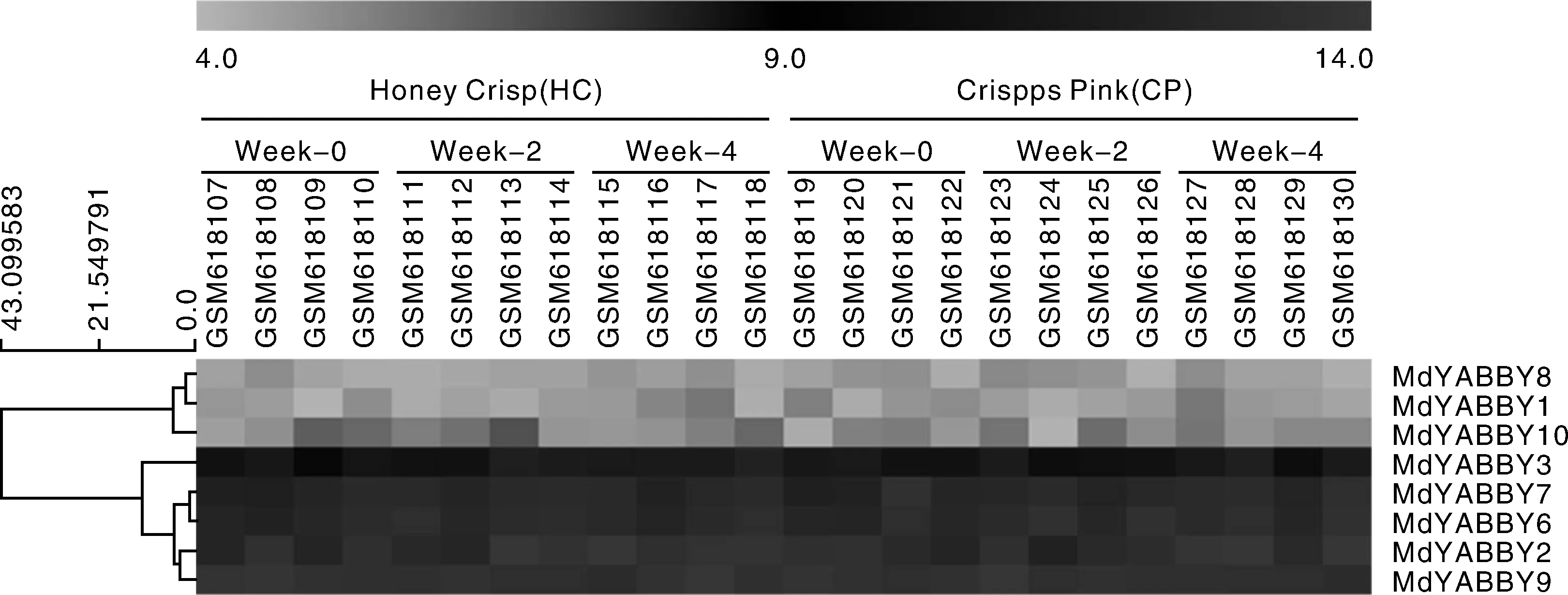
图7 苹果YABBY家族基因在果实成熟过程中的表达Fig.7 Expression profile of the MdYABBY family genes during fruit development in apple
3 讨论
YABBY蛋白是植物特有的一类转录因子,其家族成员已在不同物种中被鉴定出来。其中,在拟南芥、水稻、大白菜、玉米和西红柿中分别鉴定出6[33]、8[12]、11[34]、13[26,31]、9个[35]。本研究利用生物信息学方法,首次在苹果全基因组范围内鉴定出13个YABBY基因,发现YABBY基因家族成员在不同物种中的数量较少。根据13个MdYABBY基因的系统进化关系将其分为5个亚家族,其中,YAB1/YAB3亚家族包含的MdYABBY基因数量最多,这些与水稻、玉米等其他物种中的结论一致。除此之外,无论单纯在苹果这一物种内的横向比较,还是在苹果和拟南芥2个物种间的纵向比较,同一个亚家族内基因成员的基因结构以及蛋白保守基序的分布都具有一定的保守性。
大量的研究表明,YABBY基因在植物侧生器官发育过程中具有重要的作用[36-38]。例如,在拟南芥和金鱼草的研究中发现,YABBY基因涉及一个产生于侧生器官并能够影响分生组织活动的信号[39-41];在水稻中发现,YABBY家族成员在种子、花器官和叶发育过程中均有表达[12];在西红柿中,YABBY家族成员在花、营养组织和果实前期发育中均有表达[35]。此外,YABBY转录因子还能够分别与LEUNIG(LUG)和SEUSS(SEU)互作,从而形成抑制因子复合体(LUG是拟南芥花同源异型基因的阻遏物,SEU是LUG的辅调节蛋白)[41-42]。
本研究发现,YABBY基因含有大量的激素诱导和植物逆境响应相关顺式作用元件,表明MdYABBY基因可能与植物激素信号转导和植物逆境响应密切相关,特别是赤霉素、脱落酸和乙烯等激素诱导相关作用元件和胚乳表达有关作用元件的发现,推测MdYABBY基因可能与苹果花发育和果实有关。对13个MdYABBY基因在金冠以及不同杂交种苹果不同器官的表达特性作了系统分析,结果发现,MdYABBY基因明显地在花、果和叶等侧生器官中上调表达。这进一步暗示了MdYABBY基因与拟南芥YABBY基因类似,它们在侧生器官比如花、果和叶等发育过程中发挥着一定的作用。值得一提的是,MdYABBY基因在不同的苹果杂交种相同的器官中表达有差异,比如MdYABBY基因在杂交种M20幼果中的表达量较成熟果高,而在M74中的表达情况却恰恰相反,但总体上,MdYABBY基因在苹果果实中的表达量比较高;基于此,关于YABBY基因在苹果某一品种或杂交种中具体的功能还需做专门的、更具针对性的深入研究。为了初步验证我们的猜想,从EBI芯片数据库下载果实不同发育期芯片数据,可以发现5个基因(MdYABBY2、MdYABBY3、MdYABBY6、MdYABBY7和MdYABBY9)在不同品种果实发育过程的3个时期表达明显上调。有研究指出,SlYABBY1a在调控西红柿果实形状和大小中发挥着重要的作用,SlYABBY2a在西红柿果实驯化过程中能够调控极端的果实大小[27]。从系统发育树上看,苹果中与SlYABBY1a和SlYABBY2a同源关系最近的基因分别为MdGATA2、MdGATA3以及MdGATA6、MdGATA7,这4个基因在苹果果实发育的3个阶段都有较高的表达,推测这些基因也会发挥类似的功能。这些结果初步证明了MdYABBY基因在苹果果实发育过程中可发挥一定的作用。进一步确认和深入分析MdYABBY基因如何在苹果果实发育过程中发挥功能,还需大量系统深入的分子生物学试验验证和结果分析,MdYABBY基因能否在花和叶等其他侧生器官发育的不同时期发挥不同的作用也需要进一步深入探索。
[1] LIU L, WHITE M J, MACRAE T H. Transcription factors and their genes in higher plants[J].EuropeanJournalofBiochemistry, 1999, 262(2): 247-257.
[2] QU L J, ZHU Y X. Transcription factor families inArabidopsis: major progress and outstanding issues for future research[J].CurrentOpinioninPlantBiology, 2006, 9(5): 544-549.
[3] BOGGON T J, SHAN W S, SANTAGATA S, et al. Implication of tubby proteins as transcription factors by structure-based functional analysis[J].Science, 1999, 286(5447): 2119-2125.
[4] TATUSOV R L, KOONIN E V, LIPMAN D J. A genomic perspective on protein families[J].Science, 1997, 278(5338): 631-637.
[5] 蒋明, 苗立祥, 张豫超, 等. 大白菜WRKY转录因子的全基因组鉴定与分析[J]. 浙江农业学报, 2013, 25(5): 980-986. JIANG M, MIAO L X, ZHANG Y C, et al. Genome-wide identification and analysis of WRKY transcription factors in Chinese cabbage[J].ActaAgriculturaeZhejiangensis, 2013, 25(5): 980-986. (in Chinese with English abstract)
[6] ECKARDT N A.YABBYgenes and the development and origin of seed plant leaves[J].PlantCell, 2010, 22(7): 2103-2103.
[7] YAMADA T, YOKOTA S, HIRAYAMA Y, et al. Ancestral expression patterns and evolutionary diversification ofYABBYgenes in angiosperms[J].PlantJournalforCell&MolecularBiology, 2011, 67(1): 26-36.
[8] KANAYA E, NAKAJIMA N, OKADA K. Non-sequence-specific DNA binding by the FILAMENTOUS FLOWER protein fromArabidopsisthalianais reduced by EDTA[J].JournalofBiologicalChemistry, 2002, 277(14): 11957-11964.
[9] BARTHOLMES C, HIDALGO O, GLEISSBERG S. Evolution of the YABBY gene family with emphasis on the basal eudicotEschscholziacalifornica(Papaveraceae)[J].PlantBiology, 2012, 14(1): 11-23.
[10] BOWMAN J L. The YABBY gene family and abaxial cell fate[J].CurrentOpinioninPlantBiology, 2000, 3(1): 17-22.
[11] LEE B, HENDERSON D A, ZHU J K. TheArabidopsiscold-responsive transcriptome and its regulation by ICE1[J].PlantCell, 2005, 17(11): 3155-3175.
[12] TORIBA T, HARADA K, TAKAMURA A, et al. Molecular characterization the YABBY gene family inOryzasativaand expression analysis ofOsYABBY1[J].MolecularGeneticsandGenomics, 2007, 277(5): 457-468.
[13] GOLZ J F, HUDSON A. Plant development: YABBYs claw to the fore[J].CurrentBiology, 1999, 9(22): 861-863.
[14] REINHARDT D, FRENZ M, MANDEL T, et al. Microsurgical and laser ablation analysis of leaf positioning and dorsoventral patterning in tomato[J].Development, 2005, 132(1): 15-26.
[15] BOWMAN J L, SMYTH D R.CRABSCLAW, a gene that regulates carpel and nectary development inArabidopsis, encodes a novel protein with zinc finger and helix-loop-helix domains[J].Development, 1991, 126(11): 2387.
[16] CHEN Q, ATKINSON A, OTSUGA D, et al. TheArabidopsisFILAMENTOUS FLOWER gene is required for flower formation[J].Development, 1999, 126(12): 2715-2726.
[17] SAWA S, ITO T, SHIMURA Y, ET AL. FILAMENTOUS FLOWER controls the formation and development ofArabidopsisinflorescences and floral meristems[J].PlantCell, 1999, 11(1): 69-86.
[18] SAWA S, WATANABE K, GOTO K, et al. FILAMENTOUS FLOWER, a meristem and organ identity gene ofArabidopsis, encodes a protein with a zinc finger and HMG-related domains[J].Genes&Development, 1999, 13(9): 1079-1088.
[19] ALVAREZ J, SMYTH D R. CRABS CLAW and SPATULA, twoArabidopsisgenes that control carpel development in parallel with AGAMOUS[J].Development, 1999, 126(11): 2377-2386.
[20] ESHED Y, BAUM S F, BOWMAN J L. Distinct mechanisms promote polarity establishment in carpels ofArabidopsis[J].Cell, 1999, 99(2): 199-209.
[21] VILLANUEVA J M, BROADHVEST J, HAUSER B A, et al. INNER NO OUTER regulates abaxial-adaxial patterning inArabidopsisovules[J].Genes&Development, 1999, 13(23): 3160-3169.
[22] SIEGFRIED K R, ESHED Y, BAUM S F, et al. Members of the YABBY gene family specify abaxial cell fate inArabidopsis[J].Development, 1999, 126(18): 4117-4128.
[23] JUNG K H, CHU K, LEE S T, et al. Circulating endothelial progenitor cells as a pathogenetic marker of moyamoya disease[J].JournalofCerebralBloodFlow&MetabolismOfficialJournaloftheInternationalSocietyofCerebralBloodFlow&Metabolism, 2008, 28(11): 1795-1803.
[24] FINN R D, CLEMENTS J, EDDY S R. HMMER web server: interactive sequence similarity searching[J].NucleicAcidsResearch, 2011, 39(Web Server issue): 29-37.
[25] WANG W, PENG W, YING L, et al. Genome-wide analysis and expression patterns of ZF-HD transcription factors under different developmental tissues and abiotic stresses in Chinese cabbage[J].MolecularGeneticsandGenomics, 2016, 291(3): 1451-1464.
[26] 葛敏, 吕远大, 张体付, 等. 玉米YABBY基因家族的全基因组鉴定与分析[J]. 江苏农业学报, 2014, 30(6): 1267-1272. GE M, LYU Y D, ZHANG T F, et al. Genome-wide identification and analysis of YABBY gene family in maize[J].JiangsuJournalofAgriculturalSciences, 2014, 30(6): 1267-1272. (in Chinese with English abstract)
[27] HAN H Q, LIU Y, JIANG M M, et al. Identification and expression analysis of YABBY family genes associated with fruit shape in tomato (SolanumlycopersicumL.).[J].Genetics&MolecularResearchGmr, 2015, 14(2): 7079-7091.
[28] ZHANG H X, JIN J H, HE Y M, et al. Genome-wide identification and analysis of the SBP-Box family genes under phytophthora capsici stress in pepper (CapsicumannuumL.)[J].FrontiersinPlantScience, 2016 (7): 1-14.
[29] YANG S, ZHANG X, YUE J X, et al. Recent duplications dominate NBS-encoding gene expansion in two woody species[J].MolecularGeneticsandGenomics, 2008, 280(3): 187.
[30] ZHOU T, WANG Y, CHEN J Q, et al. Genome-wide identification ofNBSgenes in japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes[J].MolecularGeneticsandGenomics, 2004, 271(4): 402-415.
[31] 曹宇, 郎志宏, 王磊. 玉米YABBY转录因子家族的特征及表达分析[J]. 中国农业科技导报, 2015, 17(1): 32-41. CAO Y, LANG Z H, WANG L. Characteristics and expression analysis of transcription factor YABBY family in maize[J].JournalofAgriculturalScienceandTechnology, 2015, 17(1): 32-41. (in Chinese with English abstract)
[32] 陈鸿飞, 邵红霞, 樊胜, 等. 苹果全基因组多聚半乳糖醛酸酶基因家族的鉴定及进化分析[J]. 园艺学报, 2016, 43(10): 1863-1877. CHEN H F, SHAO H X, FAN S, et al. The identification and phylogenetic analysis of polygalacturonase gene family in apple[J].ActaHorticulturaeSinica, 2016, 43(10): 1863-1877. (in Chinese with English abstract)
[33] BOWMAN J L. The YABBY gene family and abaxial cell fate[J].CurrentOpinioninPlantBiology, 2000, 3(1): 17-22.
[34] 李晓波, 杨翠翠, 邱念伟. 拟南芥和大白菜YABBY蛋白家族的生物信息学分析[J]. 山东农业科学, 2012, 44(12): 1-6. LI X B, YANG C C, QIU N W. Bioinformatic analysis of YABBY protein family inArabidopsisand Chinese cabbage[J].ShandongAgriculturalSciences, 2012, 44(12):1-6. (in Chinese with English abstract))
[35] HUANG Z, VAN H J, GONZALEZ G, et al. Genome-wide identification, phylogeny and expression analysis of SUN, OFP and YABBY gene family in tomato[J].MolecularGeneticsandGenomics, 2013, 288(3): 111-129.
[36] ESHED Y, IZHAKI A, BAUM S F, et al. Asymmetric leaf development and blade expansion inArabidopsisare mediated by KANADI and YABBY activities[J].Development, 2004, 131(12): 2997-3006.
[37] TANAKA W, TORIBA T, OHMORI Y, et al. The YABBY gene TONGARI-BOUSHI1 is involved in lateral organ development and maintenance of meristem organization in the rice spikelet[J].PlantCell, 2012, 24(1): 80-95.
[38] BOWMAN J L, ESHED Y, BAUM S F. Establishment of polarity in angiosperm lateral organs[J].TrendsinGeneticsTig, 2002, 18(3): 134-141.
[39] GOLZ J F, ROCCARO M, KUZOFF R, et al. GRAMINIFOLIA promotes growth and polarity ofAntirrhinumleaves[J].Development, 2004, 131(15): 3661-3670.
[40] GOLDSHMIDT A, ALVAREZ J P, BOWMAN J L, et al. Signals derived fromYABBYgene activities in organ primordia regulate growth and partitioning of Arabidopsis shoot apical meristems[J].PlantCell, 2008, 20(5): 1217-1230.
[41] STAHLE M I, KUEHLICH J, STARON L, et al. YABBYs and the transcriptional corepressors LEUNIG and LEUNIG_HOMOLOG maintain leaf polarity and meristem activity inArabidopsis[J].PlantCell, 2009, 21(10): 3105-3118.
[42] LIU Z, KARMARKAR V. Groucho/Tup1 family co-repressors in plant development[J].TrendsinPlantScience, 2008, 13(3): 137.
(责任编辑 侯春晓)
Identification, evolution and expression analysis of the YABBY gene family in apple (Malus×domesticaBorkh.)
SHAO Hongxia, CHEN Hongfei, ZHANG Dong, WU Haiqin, ZHAO Caiping, HAN Mingyu*
(CollegeofHorticulture,NorthwestA&FUniversity,Yangling712100,China)
13Malus×domesticaYABBYgenes were characterized and divided into five groups (YAB1/YAB3, YAB2, INO, YAB5 and CRC). All theM.domesticaYABBYgenes distributed on nine chromosomes in which the sixteenth contained the maximum amount of members. The length and isoelectric points ofM.domesticaYABBY proteins were from 60 to 538 amino acids and 4.84 to 9.81, respectively. All the 13M.domesticaYABBY proteins had a zinc finger-like domain in the N terminus and a YABBY domain in the C terminus, and the members of one subgroup present similar gene structure and conserved protein motifs, then thecis-elements analysis found that there were manycis-elements related to stress and hormone response. Gene expression analysis indicated that most of theM.domesticaYABBYgenes were highly expressed in flower, fruits and leaves. FiveMdYABBYgenes were all greatly up-regulated during three fruit development.
apple;YABBYgene; cluster analysis; evolution; gene expression
http://www.zjnyxb.cn邵红霞, 陈鸿飞, 张东, 等. 苹果YABBY基因家族的鉴定、进化及表达分析[J]. 浙江农业学报, 2017, 29(7): 1129-1138.
10.3969/j.issn.1004-1524.2017.07.10
2017-02-27
国家科技支撑计划项目(2013BAD20B03);国家星火计划项目(2014GA850002);国家现代农业产业技术体系项目(CARS-28),国家苹果改良中心杨凌分中心,陕西省农业科技创新与攻关项目(2015NY114);陕西省果业发展协同中心和陕西果业发展项目
邵红霞(1991—),女,陕西定边人,硕士研究生,研究方向为苹果花芽分化机制。E-mail: xnyyshx@163.com
*通信作者,韩明玉,E-mail: hanmy@nwsuaf.edu.cn
S661.1
A
1004-1524(2017)07-1129-10
浙江农业学报ActaAgriculturaeZhejiangensis, 2017,29(7): 1129-1138
