鼠伤寒沙门菌小RNA GcvB 靶基因筛选和验证分析
2020-11-05段世宇
潘 永,段世宇,杨 阳,杨 琦,3*
(1. 贵州大学动物科学学院,贵州 贵阳 550025;2. 贵州大学动物疫病研究所,贵州 贵阳 550025;3. 贵州省动物疫病与兽医公共卫生重点实验室,贵州 贵阳 550025)
鼠伤寒沙门菌(Salmonella typhimurium LT2,STM LT2)是引起人类和牲畜食源性疾病的主要病原之一,人类感染沙门菌会引起发烧、肠胃炎、败血症等,对社会经济和人类健康造成了很大影响[1]。目前细菌种群向耐药表型快速进化导致缺乏抗击耐药菌株的新型药物[2-3],给该病防控带来很大困难。
小RNA(small RNA,sRNA)是调控转录后基因表达的非编码RNA,sRNA 分子通常长40~400 个核苷酸[4-5]。sRNA 可以通过与mRNA 翻译起始靶位点结合。从而隔离核糖体备用结合位点或通过改变靶mRNA 二级结构暴露核糖核酸酶作用位点促进mRNA 降解来抑制的翻译。其还可以通过暴露螯合的核糖体结合位点或通过掩盖核糖核酸酶的切割位点来保护mRNA,从而激活其翻译[6-8]。此外,最近的研究表明,大约一半的mRNA 受sRNA 调控[9]。鉴于此,基于sRNA 介导的广泛调控促使合成sRNA 被作为新型抗菌药物来研究。研究表明,sRNA GcvB通过调控抗生素吸收所需基因来调节大肠杆菌抗生素抗性[4]。而STM LT2 与大肠杆菌中的gcvB 基因序列具有较高的同源性,其直接或间接影响STM LT2至少20%的基因表达,经验证其直接调控的靶mRNA 近30 个,其中大部分与STM LT2 氨基酸的摄取有关, 普遍认为GcvB 与细菌在高营养环境下避免多余能量的消耗相关[9]。研究表明,STM GcvB 存在3 个不同的功能基序,R1(TGTGATGTTGTGTTGTTGTGTTTGCAA)、R2(ACTTCCTGTA)和R3(TACC CTGTCTGTCCATAGTGATTAAT)[10-11],而受这些功能基序直接调控的靶mRNA 均能与R1、R2 或R3 产生完全或部分的碱基互补配对[10-11]。另外,GcvB 在STM LT2 对数早期表达量最高,之后逐渐降低,稳定期时几乎检测不到。
因此,本研究针对对数生长早期的STM LT2 进行研究[12],利用TARGET2 程序分别预测STM LT2 中能与GcvB R1、R2 或R3 功能基序产生碱基互补配对的靶基因,并进一步通过荧光定量PCR 检测STM LT2 经预测靶基因在其匹配功能基序敲除菌株、gcvB 基因敲除菌株以及野生型菌株中转录水平的变化,旨在探索更多STM LT2 sRNA GcvB 直接调控靶标,为进一步研究STM LT2 致病机制提供参考依据。
1 材料与方法
1.1 主要实验材料野生型STM LT2 株MA3409[13]、STM LT2/pKD46 株MA7455[13]、 STM LT2 ΔgcvB 株MA10241[14]、pKD3[15]和pKD46[15]质粒均由法国国家科学研究中心(CNRS)分子遗传学Bossi 实验室惠赠;pfu 和Taq DNA 聚合酶均购自北京索莱宝科技有限公司;PCR 产物纯化试剂盒和RNA 提取试剂TRIzol 均购自生工生物工程(上海)股份有限公司;胶回收试剂盒、反转录试剂盒HiScript II Q Select RT SuperMix for qPCR(+gDNA wiper)以及荧光染料AceQ Universal SYBR qPCR Master Mix 均购自南京诺唯赞生物科技有限公司;细菌总DNA 提取试剂盒购自天根生化科技有限公司。
1.2 STM LT2 GcvB 靶基因的预测登陆网站(http://cs.wellesley.edu/~btjaden/TargetRNA2/),输入分析对象(Salmonella enterica subsp.enterica serovar Typhimurium str.LT2),分别输入GcvB R1 功能基序(TGTGATGTTGTGTTGTTGTGTTTGCAA)、R2 功能基序(ACTT CCTGTA)和R3 功能基序(TACCCTGTCTGTCCATAGTGATTAAT)[10-11], 其它各项指标均按程序默认值设定,mRNA 匹配区域为-80~+20 nt,sRNA 窗口大小为20,最小杂交种子为7,p值最大阈值为0.05,过滤尺寸为400,点击搜索并导出预测与GcvB R1、R2 或R3功能基序所匹配的靶基因结果[16]。
1.3 引物的设计与合成如表1 所示,参照Gene-Bank 中相关序列,利用primerselect 软件设计引物,单横线表示碱基序列与MA3409 同源,双横线表示碱基与pKD3 质粒中氯霉素抗性基因序列同源,FgcvB-R1、FgcvB-R2 和FgcvB-R3 引物已分别删除gcvB 基因R1、R2 和R3 功能基序,所有引物均由上海生工生物工程技术服务有限公司合成。
1.4 STM LT2 gcvBΔR1、STM LT2gcvBΔR2 和STM LT2 gcvBΔR3 菌株的构建将氯霉素抗性基因克隆至gcvB 基因下游,为后续实验提供抗性筛选标记[15]。以ppF/ppR 为上下游引物,pKD3 为模板,PCR 扩增氯霉素抗性基因片段并经纯化回收产物。取5 μL 产物与50 μL MA7455 感受态细胞混合并进行电转化试验,次日挑取单个重组菌落以t1 为引物进行PCR 扩增及测序鉴定,鉴定正确的菌株命名为STM LT2 gcvB::cat。
以STM LT2 gcvB::cat 菌株总DNA 为模板,分别以FgcvB-R1/ppR、FgcvB-R2/ppR 和FgcvB-R3/ppR 为引物PCR 扩增同源重组基因片段并经纯化后回收产物。采用上述方法,将不同的同源重组基因片段分别电转化至MA7455 感受态细胞。次日挑取单个重组菌落以t1 为引物进行PCR 扩增及测序鉴定,鉴定正确的菌株分别命名为STM LT2 gcvBΔR1、STM LT2 gcvBΔR2 和STM LT2 gcvBΔR3。
1.5 预测靶基因转录水平的荧光定量PCR 检测根据TRIzol 说明书分别提取对数早期(OD600nm≈0.4)的MA3409、MA10241、STM LT2 gcvBΔR1、STM LT2 gcvBΔR2 和STM LT2 gcvBΔR3 菌株总RNA,并将其反转录为cDNA。检测经预测靶基因荧光定量PCR 引物特异性后,通过荧光定量PCR分别检测靶基因在其匹配功能基序敲除菌株(STM LT2 gcvBΔR1、STM LT2 gcvBΔR2 或STM LT2 gcvBΔR3 菌 株)、MA3409 和MA10241 中转录水平的变化,扩增程序为95 ℃15 s;60 ℃1 min;95 ℃15 s。每个样设3 个重复, 以16S rRNA 作为内参基因,结果根据2-ΔΔCt法计算[17]。
2 结 果
2.1 STM LT2 GcvB 靶基因的预测结果利用TargetRNA2 程序预测能与STM LT2 GcvB R1、R2 或R3功能基序碱基互补配对的靶基因。结果显示,共预测到6 个能与GcvB R1 功能基序形成至少10 个连续碱基互补配对的靶基因,5 个能与GcvB R2 功能基序形成至少7 个连续碱基互补配对的靶基因,5 个能与GcvB R3 功能基序形成至少7 个连续碱基互补配对的靶基因,且所有预测结果p 值均小于0.05,均符合预设值标准(表2)。以上结果表明,本研究共预测到GcvB 靶基因16 个,包括与GcvB R1 功能基序匹配的6个靶基因STM1856、metF、mltC、sbp、yhhT 和yaeC;与GcvB R2 功能基序匹配的5 个靶基因narY、sgbE、ybbP、speG和ybbA;以及与GcvB R3功能基序匹配的5个靶基因gshA、STM0849、yihU、ampH和aroF。
2.2 STM LT2 gcvBΔR1、STM LT2gcvBΔR2 和STM LT2gcvBΔR3 菌株的构建结果通过同源重组将氯霉素抗性基因克隆至STM LT2 gcvB 基因下游并进行PCR 及测序鉴定。PCR 鉴定结果显示,STM LT2 gcvB::cat 重组菌株出现与预期相符的目的条带(图1A);测序结果显示,STM LT2 gcvB::cat 重组菌株的目的条带大小为1 910 bp,与预期大小完全相符。表明STM LT2 gcvB::cat 菌株已正确构建。
以STM LT2 gcvB::cat 菌株总DNA 为模板分别扩增STM LT2 gcvB 基因R1、R2 和R3 基序缺失基因片段,通过同源重组构建STM LT2 GcvB 不同基序敲除菌株并进行PCR 及测序鉴定。PCR 鉴定结果显示,STM LT2 gcvBΔR1、STM LT2 gcvBΔR2 和STM LT2 gcvBΔR3 重组菌株均出现与预期相符的目的条带(图1B),测序结果显示,STM LT2 gcvBΔR1、STM LT2 gcvBΔR2 和STM LT2 gcvBΔR3 重组菌株的目的条带大小分别为1 886 bp、1 901 bp 和1 884 bp,与预期完 全 相 符。 表 明STM LT2 gcvBΔR1、 STM LT2 gcvBΔR2 和STM LT2 gcvBΔR3 菌株均已正确构建。

表1 本研究所用引物Table 1 Primers used in the paper
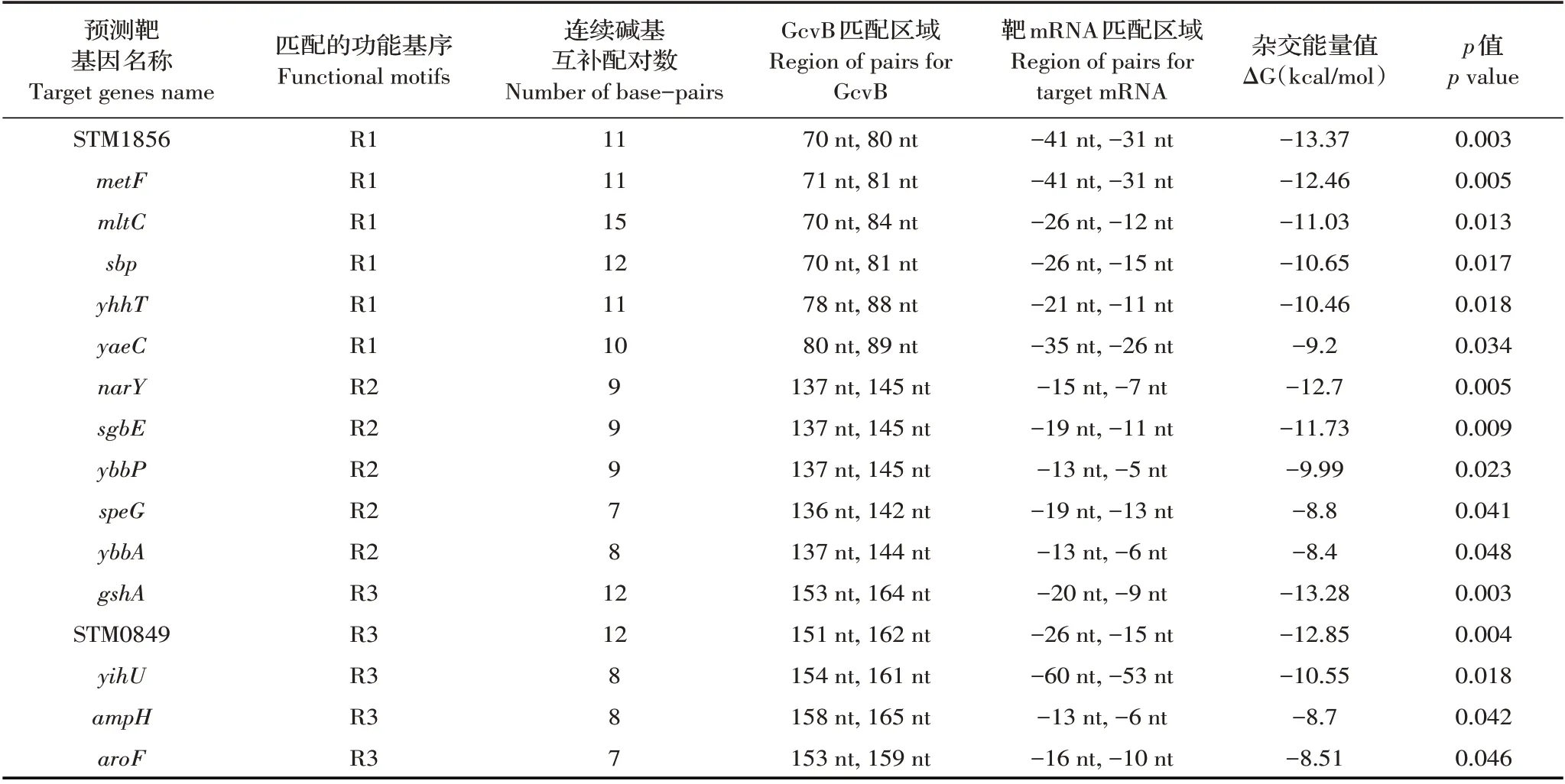
表2 TargetRNA2 程序预测能与GcvB 3 个功能基序碱基配对的靶基因Table 2 Predicted target genes base-paired to the three functional motifs of GcvB using the TargetRNA2 program

图1 重组菌株的PCR 鉴定结果Fig.1 PCR identification of recombinant strain
2.3 预测靶基因转录水平的荧光定量PCR 检测结果通过荧光定量PCR 分别检测预测靶基因在其匹配功能基序敲除菌株(STM LT2 gcvBΔR1、STM LT2 gcvBΔR2 或STM LT2 gcvBΔR3 菌 株)、MA3409 和MA10241 中转录水平的变化。结果显示,经预测能与GcvB R1 功能基序相互作用的6 个靶基因中,STM1856、metF、mltC 和sbp 基 因 在gcvB 基 因 或gcvB R1 功能基序被敲除后转录水平相对野生型菌株下调均超过200%(图2A);经预测能与GcvB R2 功能基序相互作用的5 个靶基因中,ybbP 和speG 基因在gcvB 基因或gcvB R2 功能基序被敲除后转录水平相对野生型菌株下调均超过200%(图2B);预测能与GcvB R3 功能基序相互作用的5 个靶基因中,ampH、gshA 和aroF 基因在gcvB 基因或gcvB R3 功能基序被敲除情况后转录水平相对野生型菌株下调均超过200%(图2C)。另外,MA10241 未检测到gcvB 基因转录,STM LT2 gcvBΔR1、STM LT2 gcvBΔR2 和STM LT2 gcvBΔR3 菌株均检测到gcvB 基因转录且转录水平与MA3409 相比无显著差异。结果表明,经预测靶 基 因STM1856、metF、mltC、sbp、ybbP、speG、ampH、gshA 和aroF 在转录水平受sRNA GcvB 的正调控,其中对STM1856、metF、mltC 和sbp 基因的调控与GcvB R1 基序存在显著关联(p<0.05);对ybbP和speG 基因的调控与GcvB R2 基序存在显著关联(p<0.05);对ampH、gshA 和aroF 基 因 的 调 控 与GcvB R3 基序存在显著关联(p<0.05)。
本研究预测并验证的基因STM1856、metF、mltC、sbp、ybbP、speG、ampH、gshA 和aroF 分别编码推定的胞质蛋白、5, 10-亚甲基四氢叶酸还原酶、膜结合的溶血性尿素转糖基化酶C、硫酸盐转运蛋白、推定的内膜蛋白、亚精胺N1-乙酰基转移酶、青霉素结合蛋白、gamma-谷氨酸-半胱氨酸连接酶以及3-脱氧-D-阿拉伯庚基庚酸酯-7-磷酸盐合酶,这些基因均显著受GcvB 正调控。
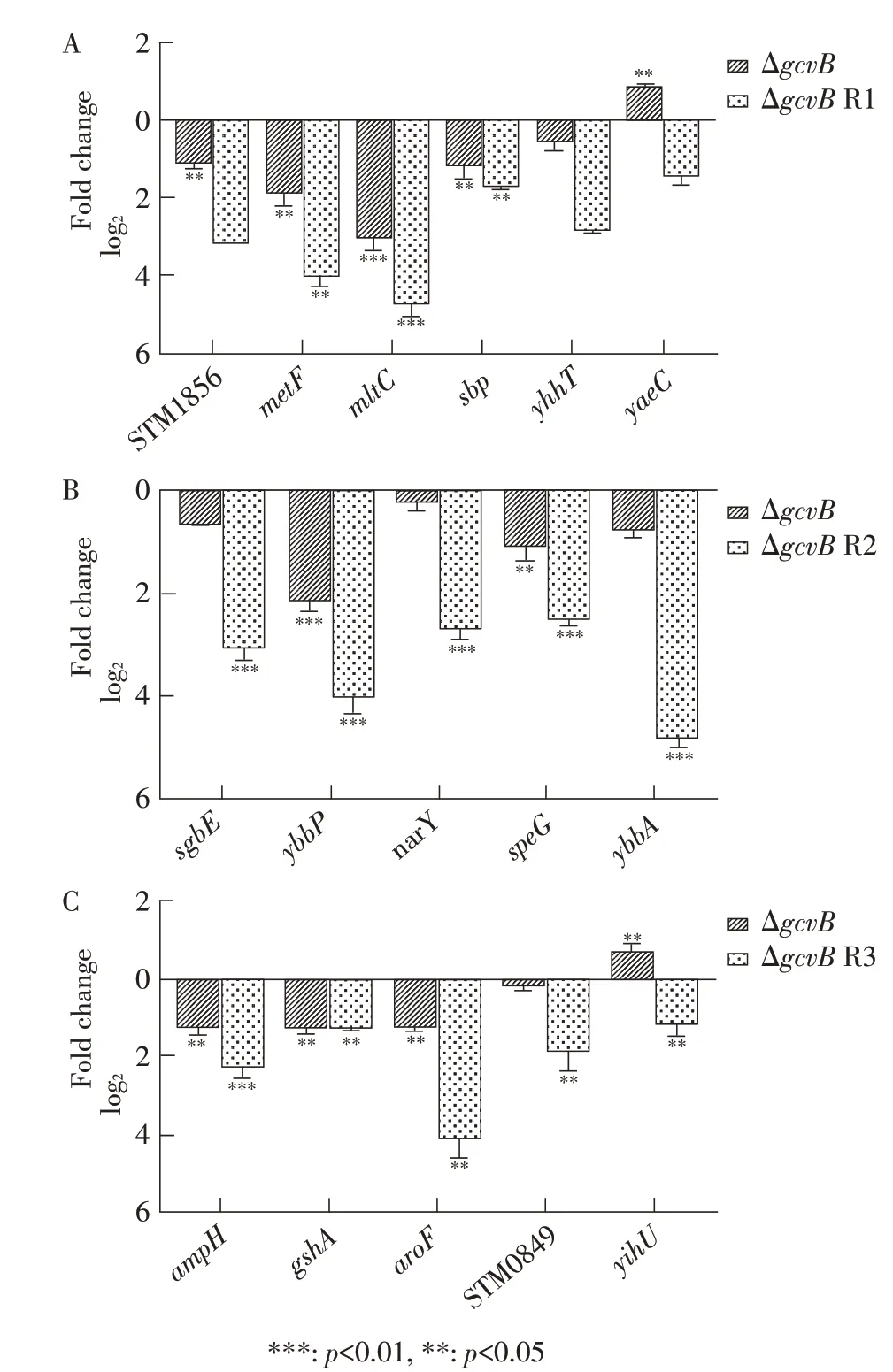
图2 预测靶基因转录水平的荧光定量PCR 检测结果Fig.2 Real-time qPCR analysis detection results of predicted target gene transcription levels
3 讨 论
以往研究主要通过对比和分析STM LT2 敲除hfq或gcvB 基因后转录水平或蛋白水平的变化,以此为基础筛选出差异表达较明显的基因。虽然基于该方法已经鉴定了许多GcvB 靶基因,但仍存在一些不足,如筛选基因总数太大,对GcvB 直接调控靶基因的验证存在很大的不确定性等。而通过大量的研究表明,GcvB 主要通过3 个功能基序与靶基因作用,并且许多靶基因与GcvB 的作用方式存在相同或类似的特点。本研究针对GcvB 3 个不同的功能基序,采用TargetRNA2 程序预测到16 个靶基因,其中9 个基因的研究结果表明,GcvB 可能直接调控这些基因。显然,这种针对研究对象特性并结合软件预测的研究方法有较高的筛选效率。
尽管荧光定量PCR 实验结果显示部分预测靶基因在不同的两种重组菌株中差异表达趋势相同,但也同时出现一些值得探究的现象。3 组荧光定量PCR 结果中,经预测靶基因在GcvB 不同功能基序的单敲除菌株中的差异表达程度总是强于完整gcvB 基因敲除菌株。除此之外,相同经预测靶基因在gcvB基因和GcvB 不同功能基序的单敲除菌株中出现不同的差异表达趋势,如yaeC 和yihU 基因。这些现象提示,GcvB 不同功能基序存在各自的调控网络,而不同的调控网络可能拥有密切的联系。Pulvermacher等对大肠杆菌GcvB 靶基因cycA 的研究中发现,GcvB可能通过多个冗余序列与靶基因结合[18]。尽管目前为止尚不清楚其中机制,但这提示GcvB 功能基序的作用机制较为复杂,有待深入研究。另外,Hfq 在不存在sRNA 的情况下仍能调控靶基因的表达,上述现象的产生或许与Hfq 的干扰有关,有待进一步确认。
GcvB 的负调控机制通常通过借助伴侣蛋白Hfq与靶mRNA 碱基配对来抑制基因表达,与mRNA 的作用区域通常位于核糖体结合位点或邻近上游富含ACA 的增强子序列[19-20]。GcvB 的正调控机制鲜有报道,目前主要存在两种通用机制,第一种机制通过sRNA 与mRNA 碱基互补配对将固有的翻译抑制结构展开,结果使核糖体与mRNA 顺利结合而增强翻译;另一种机制最为常见,sRNA 通过与mRNA 碱基配对相互作用干扰核糖核酸酶对mRNA 的降解,这种相互作用主要发生在mRNA Shine-Dalgarno(SD)序列到开放阅读框(ORF)中的第5 个密码子[21-22]。TargetRNA2 程序对STM1856、metF、mltC、sbp、ybbP、speG、ampH、gshA 和aroF 基因的预测结果显示各基因均存在与至少一个GcvB 功能基序碱基互补配对的区域,并且相互作用区域均位于mRNA Shine-Dalgarno(SD)序列到起始密码子之间,因此,推测GcvB 对STM1856、metF、mltC、sbp、ybbP、speG、ampH、gshA 和aroF 基因的正调控可能是基于干扰核糖核酸酶对mRNA 的降解而产生的,这是已报道最普遍的GcvB 正向调节靶基因的作用机理。
本研究预测并验证了16 个GcvB 靶基因,其中STM1856、metF、mltC、sbp、ybbP、speG、ampH、gshA 和aroF 基因均受GcvB 的正向调控,为进一步阐述GcvB 在STM LT2 中的调控机制奠定了基础。
