大麦ARF基因家族的全基因组分析
2016-12-29郭宝健袁泽宸张新忠许如根
郭宝健,李 赢,袁泽宸,吕 超,张新忠,许如根
(江苏省作物遗传生理国家重点实验室培育点/粮食作物现代产业技术协同创新中心/教育部植物功能基因组学重点实验室/扬州大学大麦研究所,江苏扬州 225009)
大麦ARF基因家族的全基因组分析
郭宝健,李 赢,袁泽宸,吕 超,张新忠,许如根
(江苏省作物遗传生理国家重点实验室培育点/粮食作物现代产业技术协同创新中心/教育部植物功能基因组学重点实验室/扬州大学大麦研究所,江苏扬州 225009)
生长素响应因子(auxin response factor,ARF)基因家族是植物特有的转录因子家族,在调控植物生长发育过程中起到重要作用。而关于大麦ARF基因家族全基因组分析的研究尚未见报道。为进一步探讨大麦ARF基因的功能,以公布的大麦栽培品种Morex的基因组数据为基础,采用生物信息学方法鉴定了大麦ARF基因,并对其结构、染色体分布、蛋白保守结构域、系统进化树及表达谱进行了分析。结果表明,共鉴定出了17个大麦ARF基因,除4号染色体外,其余6条染色体上均有分布,片段复制事件是大麦ARF基因家族的主要扩张方式;HvARF蛋白均具有保守的B3结构域和Auxin_resp结构域,而Aux/IAA结构域仅存在于12个HvARF蛋白中,且不同蛋白所含该结构域1~2个不等;不同植物中ARF基因在功能上具有保守性;不同组织器官中HvARF基因的表达存在明显的差异。
大麦;生长素响应因子;进化树;表达谱
生长素响应因子(auxin response factor,ARF)基因家族是植物特有的转录因子家族,调控生长素响应的基因表达[1-3]。绝大多数的ARF蛋白具有3个保守的结构域,即N-末端B3 DNA结合域(DNA-binding domain,DBD),具有DNA结合活性,能直接与生长素调控的下游基因启动子区的AuxREs结合;中间区域(Auxin_resp结构域),具有激活结构域(AD)或抑制结构域(RD)活性,具有激活或抑制转录子活性的作用;C末端二聚化结构域(CTD),即AUX/IAA结构域,与Aux/IAAs蛋白C-末端的结合域Ⅲ和Ⅳ高度保守,介导Aux/IAAs和ARFs的同源二聚化和异源二聚化的区域,调节ARFs与AuxREs的结合并调控下游生长素响应基因的表达[2,4-5]。
目前,植物中已克隆到大量的ARF基因,部分基因的功能也得到鉴定[3,6-7]。 AtARF5(MONOPTEROS)是植物中第一个通过图位克隆方法鉴定的ARF基因,该基因在胚模式建成、维管组织正常形成过程中起重要作用[6]。 AtARF7和 AtARF19是报道最多的ARF基因。拟南芥中, arf7突变体植株主要表现为下胚轴的向光性和对生长素运输抑制剂NPA的抗性, arf19突变体植株相对于野生型的表型没有明显的变化, arf7arf19双突变除了表现出单突变的表型外,侧根数目严重减少,即 AtARF7与 AtARF19在功能上有一定的冗余,在拟南芥侧根发生过程中起重要作用[7-9]。 AtIAA14和 AtLBD16/29与 AtARF7/19共同参与了拟南芥的侧根发育的调控网络[10-11]。Attia等[12]研究表明,降低 OsARF1基因的表达量,转基因水稻株系表现出种子活力低、发育迟缓、叶片短而卷曲和不育,表明 OsARF1对营养器官和生殖器官的发育都起着重要作用。
许多植物的ARF基因家族已得到系统分析,如拟南芥中有23个ARF基因、玉米中有31个ARF基因、水稻中有25个ARF基因、谷子中有24个ARF基因[7,13-17]。大麦是世界上第四大禾谷类作物,具有广泛的适应性和用途,栽培历史悠久。2012年,大麦全基因组测序完成,为大麦分子生物学研究奠定了良好的基础[18]。目前,大麦中还没有ARF基因家族分析的报道,也缺少对该基因家族的生物信息学分析和表达分析,因此有必要在大麦基因组测序完成的基础上对ARF基因家族进行系统分析。本研究利用生物信息学方法在全基因组水平上鉴定大麦ARF基因,并对其基因结构、染色体分布、蛋白保守结构域、分子进化及表达谱进行分析,以期为进一步探讨大麦ARF基因的功能奠定基础。
1 材料与方法
1.1 大麦ARF基因家族成员的鉴定及其编码蛋白的理化性质分析
利用国际大麦基因组测序联盟公布的大麦基因组数据[18],以已报道的拟南芥[7]、水稻[13]和玉米[14-16]的ARF蛋白序列作为引用数据,在IPK (http://planttfdb.cbi.pku.edu.cn/)和Gramene((http://gramene.org/)数据库中进行BLASTP比对(e-value<1e-5),搜索这些序列的同源蛋白,删除重复序列。大麦ARF候选基因的蛋白质序列利用在线软件Pfam (http://www.sanger.ac.uk/Software/Pfam/search.shtml)进行保守结构域分析[19],筛选含有 B3结构域、Auxin_resp结构域和AUX_IAA 结构域的序列,剔除冗余蛋白。借助Expasy (http://www.expasy.org/)在线软件计算大麦ARF蛋白的等电点和分子量[20]。
1.2 大麦ARF基因的结构、染色体定位和启动子序列分析
从Gramene数据库中检索大麦ARF基因的信息,包括内含子、外显子、染色体上的物理位置信息和启动子序列。采用GSDS (http://gsds.cbi.pku.edu.cn/)分析大麦ARF基因的内含子和外显子结构[21]。利用MapDraw软件构建染色体定位图[22]。按照大麦ARF基因在染色体上的排列顺序进行基因命名。在PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/)中输入大麦ARF基因的启动子序列,预测生长素响应元件[24]。
1.3 分子进化树的构建
利用Muscle软件对大麦、玉米、水稻和拟南芥ARF蛋白序列进行多序列比对,将比对结果输入MEGA 6.0,采用邻接法(neighbor-joining,NJ) 分别构建大麦ARF基因家族进化树及大麦、玉米、水稻和拟南芥的系统进化树,其中,校验参数(bootstrap)设置为1 000,其余均为默认参数[25]。片段复制事件参照Maher等[23]的方法,查找旁系同源基因上游和下游各10个编码蛋白的基因,如在ARF基因的两侧还存在旁系同源基因对,则表明该ARF旁系同源基因对起源于片段复制事件。
1.4 大麦ARF基因的表达谱分析
利用已公布的大麦RNA-seq数据,检索大麦ARF基因的表达谱(http://apex.ipk-gatersleben.de/apex/f?p=284:10:6281639160219::NO)[18]。数据库中提供了大麦栽培品种Morex的8个组织器官的表达数据,包括种子萌发后4 d的胚、幼苗的根和嫩芽、幼穗(5 mm和1~1.5 cm)、六叶期的分蘖、开花后5 d和15 d的籽粒(去稃壳),获取数据库中8个组织器官的FPKM(fragments per kilobase of transcript per million mapped reads)值,作为组织表达谱数据,构建基因表达热图。图中用绿色和红色表示基因表达的强度,绿色越亮表示基因表达量越低,红色越亮表示基因表达量越高。
2 结果与分析
2.1 大麦ARF基因的染色体定位及理化性质
利用已报道的31个玉米ARF基因、25个水稻ARF基因和23个拟南芥ARF基因序列在IPK (http://planttfdb.cbi.pku.edu.cn/)和Gramene (http://gramene.org/)数据库中进行BLASTP比对,得到35个大麦候选ARF基因。利用Pfam (http://www.sanger.ac.uk/Software/Pfam/search.shtml) 对获得的大麦候选ARF基因编码蛋白质序列进行保守结构域分析,剔除不含有典型ARF结构域的冗余蛋白后,获得17个大麦ARF基因。这17个大麦ARF基因分布于除4H染色体外的其余6条染色体上,3H和7H上含有的基因数目最多,均为4个,其次为2H和6H,均为3个,1H和5H上含有的ARF基因数目最少,分别为2个和1个(图1)。按照大麦ARF基因在染色体上的排列顺序命名为 HvARF1~ HvARF17(图1)。序列分析显示,17个HvARF基因编码区长度在1 356~3 387 bp之间,编码长度为452~1 129个氨基酸的蛋白质,分子量为50.35~125.38 kDa,等电点范围为5.77~8.92(表1)。
2.2 大麦ARF基因的结构和系统进化树
利用本研究中大麦ARF全长蛋白序列构建大麦系统进化树,结果(图2A)显示,在17个编码大麦ARF蛋白的基因中检测到了5个旁系同源基因对,即 HvARF1和 HvARF16、 HvARF2和 HvARF7、 HvARF8和 HvARF9、 HvARF11和 HvARF15、 HvARF12和 HvARF17。基因结构分析表明,不同大麦ARF基因所含外显子差异很大,其中, HvARF3、 HvARF5、 HvARF6、 HvARF9、 HvARF10和 HvARF11均含有14个外显子, HvARF4、 HvARF14和 HvARF17均含有13个外显子, HvARF8、 HvARF12和 HvARF15均含有12个外显子,而 HvARF1、HvARF13和 HvARF16则均含有3个外显子, HvARF2和 HvARF7则均含有2个外显子(图2B)。旁系同源基因对的基因步长值为100的基因对( HvARF2和 HvARF7、 HvARF8和 HvARF9、 HvARF11和 HvARF15)含有相似数目的外显子(图2B)。片段复制和串联重复是家族基因扩张的主要模式,分析发现, HvARF1和 HvARF6、 HvARF4和 HvARF5基因对存在片段复制事件;在Gramene数据库中通过Gbrowse查看成簇分布的ARF基因家族成员与相邻蛋白质编码基因的位置关系,未发现大麦ARF基因家族成员位于同一个或相邻的基因间区域,即大麦中ARF基因家族的扩张不存在串联重复方式,表明片段复制是大麦ARF基因家族扩张的主要方式。大麦(17个)、玉米(31个)、水稻(25个)及拟南芥(23个)ARF基因的系统进化树分析表明,来自4个植物的96个ARF基因分为6个类别(ClassⅠ~Ⅵ)[26],而本研究中得到的17个大麦ARF基因归于ClassⅠ~ClassⅤ类别(图3)。

图1 大麦ARF基因的染色体定位
表1 大麦ARF家族基因的基本信息
Table 1 The basic information of ARF gene family in barley

基因名称Genename基因号GeneID染色体Chromosome物理位置Physicalposition编码区长度Codingsequencelength/bp氨基酸长度Aminoacidlength/aa分子量MW/Da等电点pIHvARF1MLOC_64795.11H127102527~127107560206468874246.737.59HvARF2MLOC_38232.11H419398338~419403571180060065937.558.77HvARF3MLOC_64596.12H508740133~508745733197465873058.305.81HvARF4MLOC_55345.62H589588539~5895952242871957105593.965.79HvARF5MLOC_51932.12H615547380~615555915250883692948.875.85HvARF6MLOC_65945.13H141906589~141910998228676284195.966.24HvARF7MLOC_66439.13H435834578~435839922164154759634.608.84HvARF8MLOC_68907.13H523997002~524001458224174783941.846.27HvARF9MLOC_18401.13H529062935~529067687247882692329.446.03HvARF10MLOC_63938.15H22704804~22710863267389198194.835.77HvARF11MLOC_73144.46H69851096~6985889633871129125382.045.98HvARF12MLOC_66152.26H124383735~124387564135645250346.196.90HvARF13MLOC_77438.26H247346568~247349350210970376028.868.92HvARF14MLOC_14584.17H202204080~20221199932491083120278.156.00HvARF15MLOC_63193.27H533232921~53324135633031101123570.716.20HvARF16MLOC_69988.17H545375651~545378650209169775629.948.13HvARF17MLOC_58330.27H561567257~5615740832814938103351.366.14
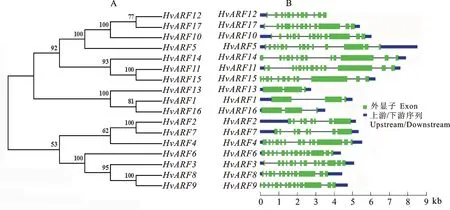
图2 大麦ARF基因家族的系统进化树(A)和基因结构(B)
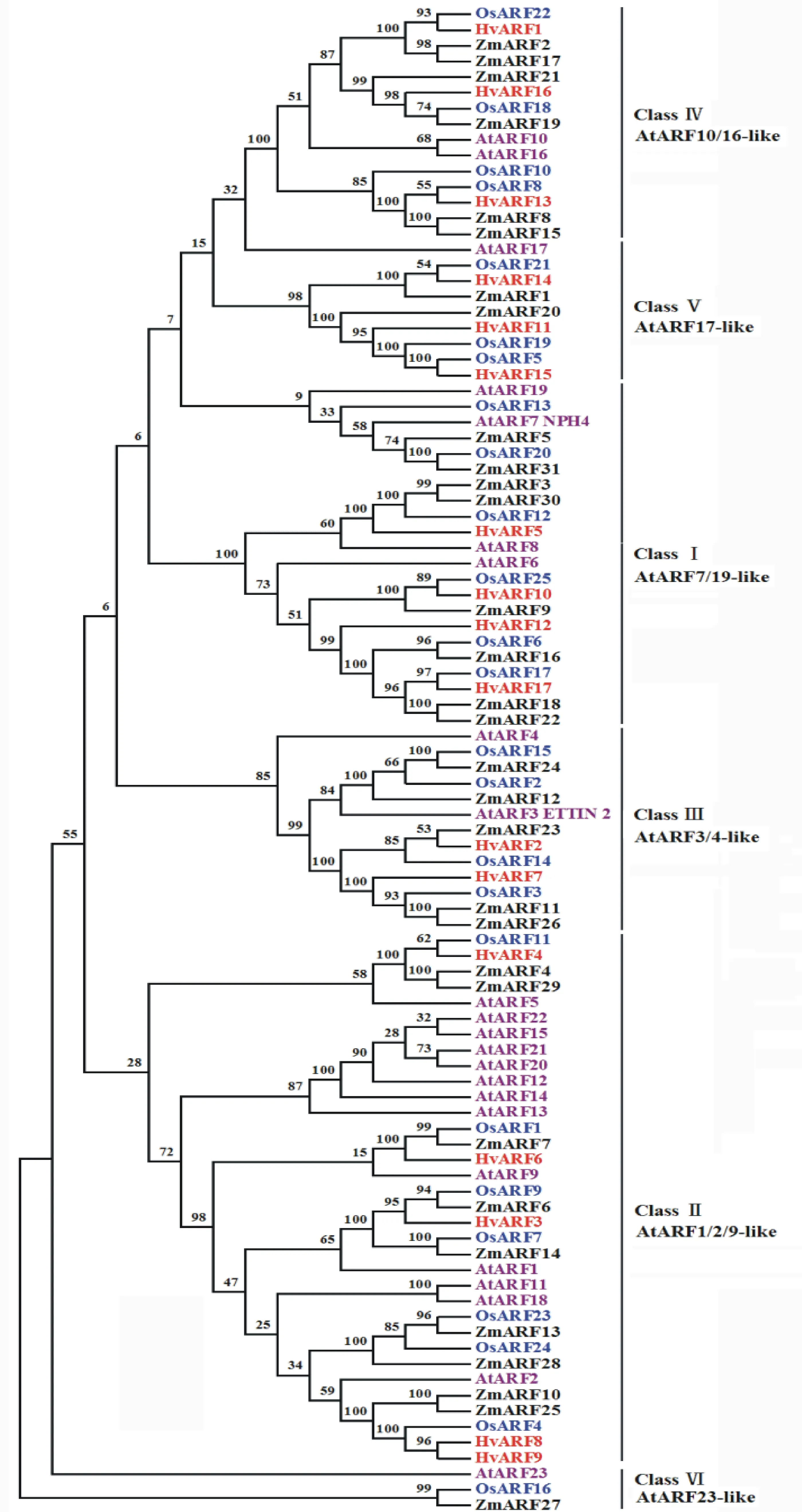
图3 大麦、玉米、水稻和拟南芥ARF基因的进化树分析
2.3 大麦ARF基因的启动子元件预测结果
利用PlantCARE软件对HvARF基因起始密码子上游2 kb的DNA序列分析表明,6个HvARF基因含有生长素响应元件(TGA-element,AACGAC),其中, HvARF16含有两个生长素响应元件,分别位于启动子正义链(1 130 bp)和反义链(-1 341 bp); HvARF1、 HvARF7和 HvARF13基因的生长素响应元件均位于正义链(1 523 bp、1 340 bp、1 249 bp); HvARF2和 HvARF15基因的生长素响应元件均位于反义链(-1 427 bp、-1 887 bp)。
2.4 大麦ARF蛋白的保守结构域
利用Pfam软件分析了大麦ARF蛋白的结构域,结果(图4)表明,所有大麦ARF蛋白都含有N-端DNA结合域(B3结构域),并具有激活或抑制活性的中间区域(Auxin_resp结构域),12个ARF蛋白含有C末端二聚化结构域(AUX/IAA结构域),其中HvARF8和HvARF9分别含有2个AUX/IAA结构域。

图4 大麦ARF蛋白的保守结构域
2.5 大麦ARF基因的表达谱
栽培品种Morex 8个组织器官的RNA-seq数据分析结果显示,17个HvARF基因中有12个在8个组织器官中检测到了转录本。如图5所示, HvARF3、 HvARF5、 HvARF10、 HvARF12和 HvARF17在幼穗(5 mm和1~1.5 cm)和六叶期的分蘖中具有较高的表达量,此外, HvARF3在幼苗期的根和嫩芽中也表现出了较高的表达量; HvARF1基因的转录本主要集中在六叶期的分蘖和开花后5 d的籽粒中,且随着成熟度的提高,在籽粒中的表达量下降; HvARF11、 HvARF15和 HvARF16基因主要在六叶期的分蘖中表达; HvARF4基因的转录本主要集中在幼穗中; HvARF6和 HvARF13基因为组成型表达基因,并且在 8个组织器官中均表现出低表达模式。

1:萌发后4天的胚;2:幼苗期的根;3:幼苗期的嫩芽;4:幼穗(5 mm);5:幼穗(1~1.5 cm);6:六叶期的分蘖;7:去稃壳籽粒(开花后5 d);8:去稃壳籽粒(开花后15 d)。
1:Embryo four-day after germination;2:Roots at seedling stage;3:Shoots at seedling stage;4:Young inflorescences (5 mm);5:Young inflorescences (1~1.5 cm);6:Tillers at six-leaf stage;7:Developing grain with hulls removed (5 DPA);8:Developing grain with hulls removed (15 DPA).
图5 大麦ARF基因在不同组织器官中的表达谱
Fig.5 Expression profile ofARFgenes in eight tissues of barley
3 讨 论
本研究在大麦全基因组水平上共检测到17个编码ARF蛋白的基因,少于已报道的拟南芥(23个)、水稻(25个)、玉米(31个)、谷子(24个)等植物ARF基因家族成员数目[7,13-17]。片段复制是大麦ARF基因扩张的唯一方式,可能限制了大麦ARF基因家族的扩张。
ARF蛋白的系统进化树分析表明,4个HvARF蛋白归于ClassⅠ,其中 HvARF5、 HvARF10和HvARF17蛋白含有B3结构域、转录激活活性的Auxin_resp结构域和AUX/IAA结构域三个保守的结构域,同时编码这三个蛋白的基因具有相似的表达模式,拟南芥中ClassⅠ的ARF蛋白同时含有三个保守的结构域,其中AtARF7和AtARF19作为转录激活子介导了拟南芥侧根形成过程[7,26],暗示 HvARF5、 HvARF10和 HvARF17基因可能存在相似的功能。本研究中5个HvARF蛋白归于ClassⅡ,该类基因数目最多,除含有B3结构域和AUX/IAA结构域外,还含有转录抑制活性的Auxin_resp结构域,仅有 HvARF3、 HvARF4和 HvARF9在八个组织器官中检测到转录本,且表达模式存在明显的差异,可能存在功能分化。同类的 AtARF3和 AtARF4编码了两个功能冗余的生长素响应因子, arf3arf4双突变体所有侧生器官背腹结构受到影响[27]。 HvARF2和 HvARF7基因与 AtARF3和 AtARF4基因同属于Class Ⅲ,且 HvARF2与 AtARF3均不含有AUX/IAA结构域[4],表明这类基因在植物生长发育过程中具有不同的功能。ClassⅣ和ClassⅤ的B3结构中含有额外的32~36个氨基酸,其余的保守结构域分别与ClassⅡ和ClassⅢ相似, AtARF10、 AtARF16和 AtARF17基因的单突变体没有明显的表型,而 arf10arf16双突变体根冠和根系向地性生长发生异常[7,28],大麦中共有5个HvARF基因属于此类别,但是具体的功能还有待于进一步的探讨。
本研究利用生物信息学方法在全基因组水平上分析了大麦ARF基因家族,并对其基因结构、染色体分布、蛋白保守结构域、分子进化及表达谱进行了分析,为进一步探讨大麦ARF基因的功能奠定一定的基础,但是关于大麦ARF基因如何调控生长素响应的基因表达参与大麦生长发育还需要进一步的探讨。
[1]GUILFOYLE T J,HAGEN G.Auxin response factors [J].JournalofPlantGrowthRegulation,2001,10:281.
[2]TIWARI S B,HAGEN G,GUILFOYLE T J.The roles of auxin response factor domains in auxin-responsive transcription [J].PlantCell,2003,15(2):533.
[3]ELLIS C M,NAGPAL P,YOUNG J C,etal.AUXINRESPONSEFACTOR1 andAUXINRESPONSEFACTOR2 regulate senescence and floral organ abscission inArabidopsisthaliana[J].Development,2005,132(20):4563.
[4]GUILFOYLE T J,HAGEN G.Auxin response factors [J].CurrentOpinioninPlantBiology,2007,10(5):453.
[5]ABEL S,OELLER P W,THEOLOGIS A.Early auxin induced genes encode short-lived nuclear proteins [J].ProceedingsoftheNationalAcademyofSciences,1994,91:326.
[6]HARDTKE C S,BERLETH T.TheArabidopsisgeneMONOPTEROSencodes a transcription factor mediating embryo axis formation and vascular development [J].EMBOJournal,1998,17(5):1405.
[7]OKUSHIMA Y,OVERVOORDE P J,ARIMA K,etal.Functional genomic analysis of the AUXIN RESPONSE FACTOR gene family members inArabidopsisthaliana:unique and overlapping functions of ARF7 and ARF19 [J].PlantCell,2005,17(2):444.
[8]LISCUM E,BRIGGS W R.Mutations in theNPH1 locus ofArabidopsisdisrupt the perception of phototropic stimuli [J].PlantCell,1995,7(4):473.
[9]STOWE-EVANS E L,HARPER R M,MOTCHOULSKI A V,etal.NPH4,a conditional modulator of auxin-dependent differential growth responses inArabidopsis[J].PlantPhysiology,1998,118(4):1265.
[10]FUKAKI H,NAKAO Y,OKUSHIMA Y,etal.Tissue-specific expression of stabilized SOLITARY-ROOT/IAA14 alters lateral root development inArabidopsis[J].PlantJournal,2005,44:382.
[11]OKUSHIMA Y,FUKAKI H,ONODA M,etal. ARF7 and ARF19 regulate lateral root formation via direct activation ofLBD/ASLgenes inArabidopsis[J].PlantCell,2007,19(1):118.
[12]ATTIA K A,ABDELKHALIK A F,AMMAR M H,etal.Antisense phenotypes reveal a functional expression of OsARF1,an auxin response factor,in transgenic rice [J].CurrentIssuesinMolecularBiology,2009,11:29.
[13]WANG D,PEI K,FU Y,etal.Genome-wide analysis of the auxin response factors (ARF) gene family in rice (Oryzasativa) [J].Gene,2007,394(1):13.
[14]XING H Y,PUDAKE R N,GUO G G,etal.Genome-wide identification and expression profiling of auxin response factor(ARF) gene family in maize [J].BMCGenomics,2011,12:178.[15]LIU Y,JIANG H Y,CHEN W,etal.Genome-wide analysis of the auxin response factor(ARF) gene family in maize(Zeamays) [J].PlantGrowthRegulation,2011,63(3):225.[16]WANG Y J,DENG D X,SHI Y T,etal.Diversification,phylogeny and evolution of auxin response factor (ARF) family:insights gained from analyzing maizeARFgenes [J].MolecularBiologyReports,2012,39(3):2401.
[17] 赵 艳,瓮巧云,马海莲,等.谷子ARF基因家族的鉴定与生物信息学分析[J].植物遗传资源学报,2016,17(3):547.
ZHAO Y,WENG Q Y,MA H L,etal.Genome-wide identification and bioinformatics analysis of ARF gene family in foxtail milletSetariaitalic[J].JournalofPlantGeneticResources,2016,17(3):547.
[18]The International Barley Genome Sequencing Consortium.A physical,genetic and functional sequence assembly of the barley genome [J].Nature,2012,491:711.
[19]PUNTA M,COGGILL P C,EBERHARDT R Y,etal.The Pfam protein families database [J].NucleicAcidsResearch,2012,40:D290.
[20]ARTIMO P,JONNALAGEDDA M,ARNOLD K,etal.Expasy:sib bioinformatics resource portal [J].NucleicAcidsResearch,2012,40(w1):W597.
[21]HU B,JIN J,GUO A Y,etal.GSDS 2.0:an upgraded gene feature visualization server [J].Bioinformatics,2014,31(8):1296.
[22]LIU R H,MENG J L.MapDraw:a microsoft excel macro for drawing genetic linkage maps based on given genetic linkage data [J].Hereditas,2003,25(3):317.
[23]MAHER C,STEIN L,WARE D.Evolution ofArabidopsismicroRNA families through duplication events [J].GenomeResearch,2006,16(4):510.
[24]LESCOT M,DÉHAIS P,THIJS G,etal.PlantCARE,a database of plantcis-acting regulatory elements and a portal to tools forinsilicoanalysis of promoter sequences [J].NucleicAcidsResearch,2002,30:325.
[25]TAMURA K,STECHER G,PETERSON D,etal.MEGA6:molecular evolutionary genetics analysis version 6.0 [J].MolecularBiologyandEvolution,2013,30(4):2725.
[26]FINET C,BERNE-DEDIEU A,SCUTT C P,etal.Evolution of the ARF gene family in land plants:old domains,new tricks [J].MolecularBiologyandEvolution,2013,30(1):45.
[27]PEKKER I,ALVAREZ J P,ESHED Y.Auxin response factors mediateArabidopsisorgan asymmetry via modulation of KANADI activity [J].PlantCell,2005,17(11):2899.
[28]WANG J W,WANG L J,MAO Y B,etal.Control of root cap formation by microRNA-targeted auxin response factors inArabidopsis[J].PlantCell,2005,17(8):2204.
Genome-Wide Analysis of Auxin Response Factor (ARF) Family in Barley
GUO Baojian,LI Ying,YUAN Zechen,LÜ Chao,ZHANG Xinzhong,XU Rugen
(Jiangsu Key Laboratory of Crop Genetics and Physiology/Co-Innovation Center for Modern Production Technology of Grain Crops/Key Laboratory of Plant Functional Genomics of the Ministry of Education/Barley Research Institution of Yangzhou University,Yangzhou,Jiangsu 225009,China)
Auxin response factor (ARF) gene family is the specific transcription factor in plant,and plays an important role in regulation of plant organ development.However,genome-wide analysis of the ARF gene family has not been reported in barley.In this study,HvARFgenes were identified based on the public genomic database of barley cultivar Morex by using bioinformatics methods.A comprehensive overview of theHvARFgenes was undertaken,including gene structure,chromosome position,conserved domain of proteins,phylogenetic tree and gene expression profiling.The results showed thatHvARFgenes located on all chromosomes except for 4H,and segmental duplication contributed to the expansion of the ARF gene family in barley;all HvARF proteins contain a conserved B3 domain and Auxin_resp domain,and twelve HvARF proteins contain one or two Aux/IAA domains;phylogenetic classification showed that the function ofARFgenes were conserved in different species;and gene expression analysis showed that the expression levels ofHvARFgenes were obviously different at different development stages in barley.
Barley;Auxin response factor;Phylogenetic tree;Expression profiling
时间:2016-11-04
2016-05-16
2016-10-13
国家自然科学基金项目(31401370;31571648);国家大麦青稞产业技术体系建设专项(CARS-05);江苏省农业科技自主创新基金项目(CX(14)2123);江苏高校优势学科建设工程资助项目
E-mail:bjguo@yzu.edu.cn
许如根(E-mail:rgxu@yzu.edu.cn)
S512.3;S330
A
1009-1041(2016)11-1426-07
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20161104.0924.004.html
