基于微卫星的泥蚶5个地理群体遗传多样性分析
2015-12-02程雪艳滕爽爽肖国强邵艳卿张炯明柴雪良
程雪艳, 滕爽爽 , 肖国强 , 邵艳卿 , 张炯明 , 方 军 , 柴雪良
(1. 温州医科大学 检验医学院 生命科学学院, 浙江 温州 325035; 2. 浙江省海洋水产养殖研究所, 浙江 温州 325005; 3. 浙江省近岸水域生物资源开发和保护重点实验室 浙江 温州 325001)
泥蚶(Tegillarca granosa)属软体动物门(Mollusca)、双壳纲(Bivalvia)、蚶目(Arcoida)、蚶科(Arcidae), 是一种栖息于沿海滩涂的广温广盐性双壳贝类, 在我国主要分布于山东以南沿海[1], 是我国主要的养殖经济贝类之一, 具有很高的经济价值。但近年来, 过度釆捕和生态环境的改变对泥蚶的自然资源造成严重的破坏。人工养殖的泥蚶种质大多是未经遗传改良的野生型, 在经过多年累代养殖后,已出现种质退化等制约泥蚶养殖业持续健康发展的现象[2]。因此, 有必要对泥蚶遗传背景进行深入调查研究, 为泥蚶自然资源的恢复和遗传改良奠定理论基础。
目前, 对于泥蚶群体的遗传多样性的研究涉及形态学水平[1,3]、同工酶水平[4-5], DNA分子水平[3,6-8]等方面。微卫星标记(microsatellite)广泛分布于真核生物基因组中, 由 2~6个核苷酸的串联重复片段构成。又称为简单重复序列(simple sequence repeats)或短串联重复序列(simple tandem repeats, STRs)[9]。由于具有数量丰富、多态性丰富、共显性标记、实验重复性好以及结果可靠等优点[10], 被广泛应用于物种的种群遗传结构和遗传多样性分析。在水产动物种群遗传结构的分析中已被广泛使用, 如海湾扇贝(Argopecten irradias)[11]、虾夷扇贝(Patinopecten yessoensis)[12]、文蛤(Meretrix meretrix)[13]、长牡蛎(Crassostrea gigas)[14]、缢蛏(Sinonovacula constricta)[15-16]、马氏珠母贝(Pinctada fucata)[17]、斧文蛤(Meretrix lamarckii)[18]等。
本研究采集了韩国以及我国沿海地区 5个地理群体泥蚶, 利用本实验室开发的泥蚶 SSR引物对 5个群体进行比较分析, 对泥蚶的遗传多样性水平进行评估, 为更好地开发利用该物种生物资源提供理论基础。
1 材料与方法
1.1 样品采集
泥蚶(T. granosa)的 5个地理群体分别取自浙江乐清(ZJ), 广西企沙(GX), 海南海口(HN), 山东日照(SD),韩国釜山(KR)等地海域, 5个地理群体均为自然群体,数量分别为 24、23、23、26、24个。样品采集后活体运回实验室, –70℃超低温冰箱保存备用。
1.2 基因组DNA提取
将所采集样品分别解剖, 取闭壳肌组织。基因组DNA提取采用酚-氯仿法抽提[19], 对所提取DNA进行纯度、浓度以及完整性检测后, 4℃保存备用。
1.3 PCR扩增与电泳检测
本研究所用引物由本课题组开发设计而成[20],引物序列及退火温度见表1, 引物由上海生工生物工程有限公司合成。在Eppendorf扩增仪上进行PCR反应, 反应体系: 10×PCR buffer(Mg2+plus)1.5 μl,2.5 mmol/L dNTP 1.2 μl, 10 μmol/L 上下游引物各 1.5 μl,Taq 酶(5 U/μl)0.075 μl, DNA 模板 100 ng, 灭菌水补足至15 μl。PCR反应条件设置: 94℃预变性5 min;94℃变性30 s, 50℃退火30 s, 72℃延伸45 s, 30个循环; 最后72℃延伸7 min。PCR结束后取4 μl扩于2%琼脂糖凝胶电泳检测(1×TBE, 5 V/cm 恒压),DuGreen染色, 凝胶成像系统观察和拍照。PCR 扩增结束后将产物于 4℃保存, 扩增产物用 6%的变性聚丙烯酰胺凝胶电泳检测。

表1 泥蚶17个微卫星位点的特性Tab.1 Characteristics of 17 microsatellites for T. granosa
1.4 数据统计

2 结果
2.1 泥蚶5个地理群体遗传参数分析
使用17对微卫星引物对泥蚶5个不同地理群体共120个样品进行了DNA检测, 17对引物在5个泥蚶群体中分别检测到 102(山东)、99(广西)、91(海南)、86(韩国)、99个(浙江)等位基因, 由于不同群体检出的等位基因不一致, 共获得115个不同等位基因。不同位点检测的等位基因数为2~12个不等, 其中位点Teg39等位基因数最多, 为12个等位基因; Teg12次之, 为11个等位基因; Teg18最少, 为2个等位基因。其中Teg28在其他群体中为2~3个等位基因, 在韩国群体中为1个等位基因。
通过基因产生的频率计算17个位点的PIC(表2),17个位点的平均多态性信息含量为0.142~0.857。其中Teg18最低, 为0.142; Teg28、Teg24、Teg8的多态性信息含量为0.176、0.273、0.377, 10个位点的多态性信息含量在0.5以上, 表现出高度多态性。5个群体的PIC为0.525~0.608, 说明泥蚶5个群体的遗传多样性处于中等偏上水平。
5个地理群体的平均观测杂合度 Ho为 0.430~0.516, 平均期望杂合度He为0.573~0.656, 其中山东群体的Ho和He均为最高。
在85个群体位点中, Teg28在韩国群体中的位点为单态的, 对84个多态性的群体位点进行分析发现,41个群体位点符合Hardy-Weinberg平衡(P>0.05), 而其余 43个(51.2%)群体位点由于杂合子缺失导致偏离Hardy-Weinberg平衡(P<0.05)。在 17个微卫星位点中,Teg12、Teg17、Teg18、Teg19四个位点在5个群体中均没有偏离Hardy-Weinberg平衡, Teg29、Teg37、Teg39三个位点在5个群体中均偏离Hardy- Weinberg平衡。
2.2 不同群体间遗传结构
使用Popgene 32(Version 1.31)软件分析泥蚶5个地理群体遗传距离及遗传相似性指数。各群体间遗传相似性指数以广西群体和山东群体之间最大(0.936 5), 其次为浙江群体和韩国群体之间(0.015 9),广西群体和韩国群体之间的遗传相似性最小(0.842 8);相应的, 各群体之间的遗传距离以广西群体和韩国群体之间最大(0.171 1), 以广西群体和山东群体(0.065 6)、浙江群体和韩国群体之间(0.087 9)相对较小(表3)。利用MEGA6.0软件对泥蚶 5个群体进行聚类分析中, 获得UPGMA聚类图(图1)。浙江群体和韩国群体亲缘关系最近首先聚在一起, 然后与海南群体聚合; 广西群体和山东群体亲缘关系也较近,聚为一支, 然后与上面3个群体聚合。

图1 基于泥蚶5个群体遗传距离构建的UPGMA聚类图Fig.1 UPGMA dendrogram based on genetic distances of five populations of T. granosa
对5个泥蚶群体进行了F-统计量以及基因流分析(表 4)。5个泥蚶群体中 Teg17、Teg19、Teg34位点的近交系数 FIS为负值, 其余位点的 FIS为正值;FIS值为–0.009 9~0.738 2, 平均值为0.217 4。各位点的总群体近交系数为 0.000 7~0.774 9, 平均值为0.264 2。度量群体间遗传差异程度的FST为0.014 2~0.164 4, 平均值为 0.059 8, 基因流 Nm为 1.271~17.343 9, 平均值为3.927 5, 不同位点的基因流值变化波动较大。
利用Arlequin (version 3.1)软件对泥蚶5个群体之间的FST分析, 结果如表5。泥蚶5个群体间的FST值在 0.012和 0.062之间, 其中韩国群体-山东群体(0.062)的FST值最高, 韩国群体-广西群体(0.060), 海南群体-山东群体(0.051)的FST值仅次于韩国群体-山东群体。韩国群体-海南群体(0.012)、山东群体-广西群体(0.016)的 FST值最低, 韩国群体-海南群体与山东群体-广西群体之外的其他群体间的 FST值均达到显著性差异。对 5个地理种群的泥蚶进行 AMOVA分析, 结果见表 6, 发现种群间变异占 3.59%, 种群个体间变异占 96.41%, 结果表明引起种群总体变异的主要因素还是种群内的遗传变异。

表2 17个微卫星位点在泥蚶5个地理群体上的遗传多样性分析Tab.2 Parameters of genetic diversity analyzed by seventeen loci in T. granosa five geographic populations
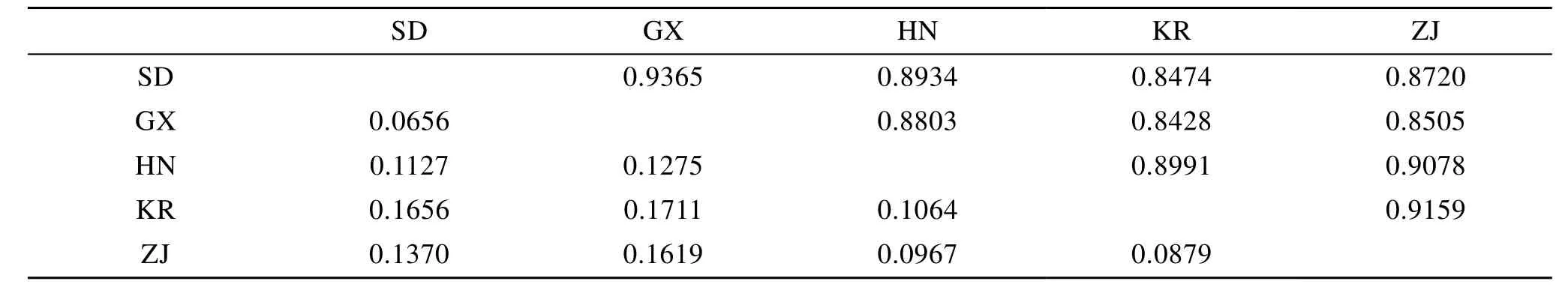
表3 泥蚶5个地理群体间的Nei’s遗传相似性(上三角)和遗传距离(下三角)Tab.3 Nei’s genetic identity(above diagonal) and genetic distance(below diagonal) among five populations of T. granosa
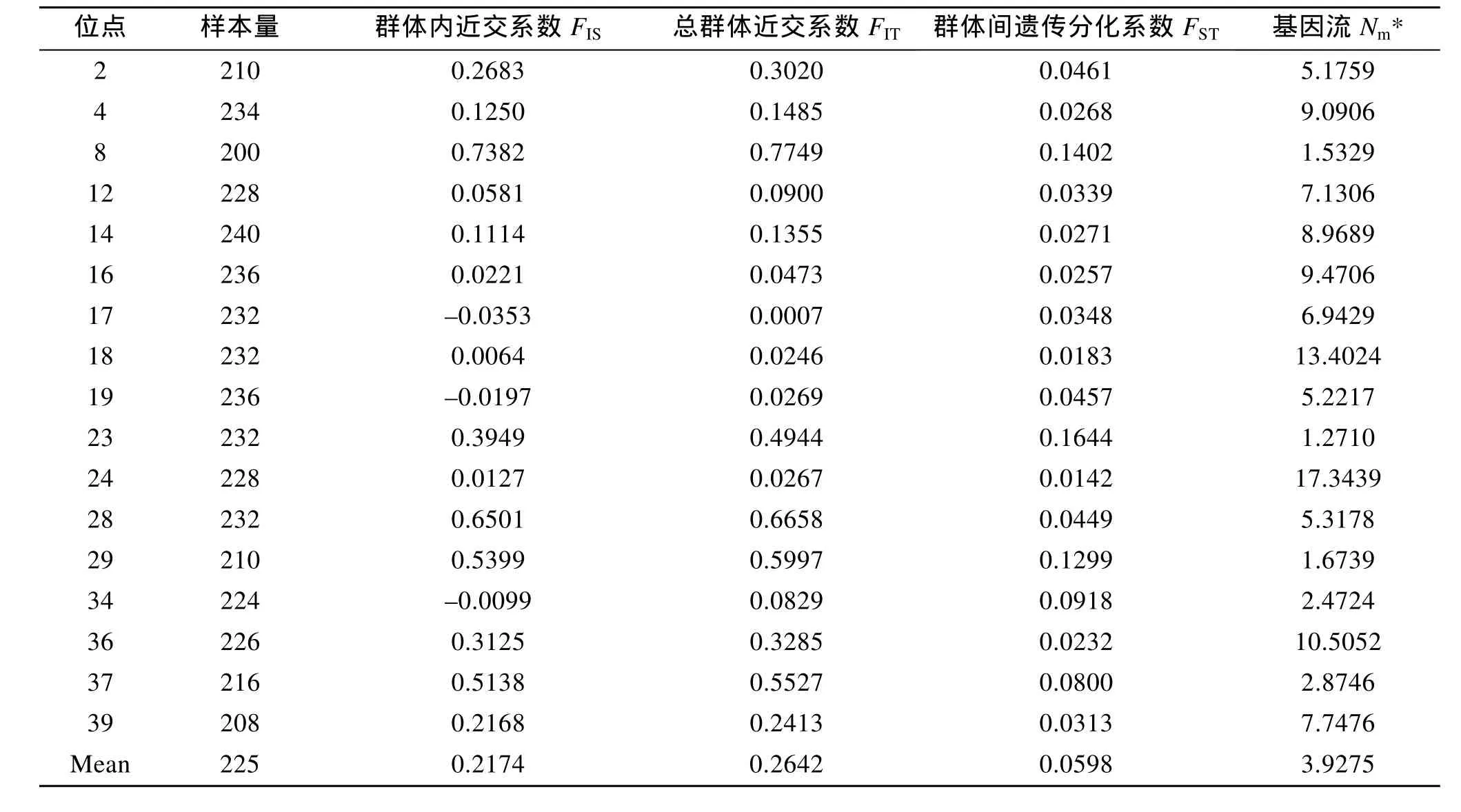
表4 17个微卫星位点的固定指数及基因流Tab.4 Fixation index and gene flow of 17 polymorphic loci

表5 泥蚶5个群体样本的遗传分化系数FSTTab.5 The FST value among 5 populations of T. granosa
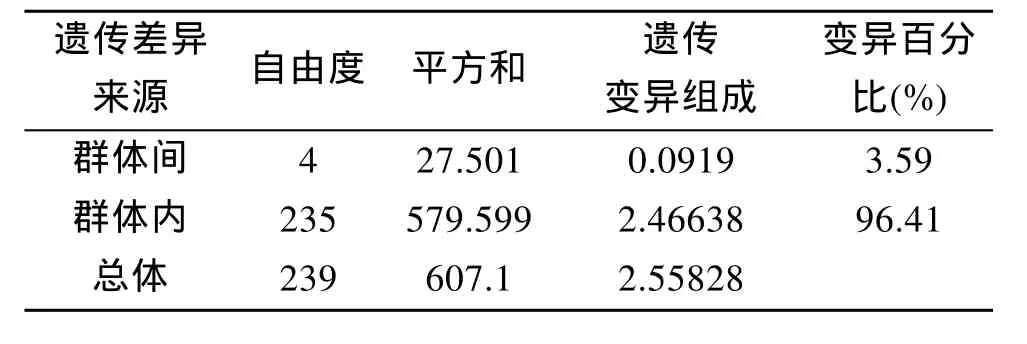
表6 泥蚶群体之间的AMOVA分析结果Tab.6 Analysis of AMOVA of T. granosa populations
用 IBDWS 3.23对泥蚶群体间的地理距离和遗传距离的关系进行分析, 结果如图2所示, 发现泥蚶5个群体间的遗传分化程度和地理距离之间没有显著的相关性。
3 讨论
3.1 泥蚶不同地理群体的遗传多样性分析

图2 地理距离和遗传距离的关系图Fig.2 Diagram of the relationship between geographic distance and genetic distance
遗传多样性是物种和某个群体长期进化的产物,是衡量一个种群种质资源质量的重要标准[22]。一个种群的遗传多样性越高, 其对生存环境的适应能力就越强, 进化的潜力也越大[23]。
目前, 对于泥蚶群体的遗传多样性已有较多研究, 喻子牛等[24]采用水平淀粉凝胶电泳技术研究了三个泥蚶群体的等位基因酶遗传变异, 在12种等位基因酶中共检测到了27个基因位点, 平均观察杂合度为0.075, 平均有效等位基因数为1.372; 李太武等[6]采用随机扩增多态性DNA(RAPD)技术对5个泥蚶群体的遗传多样性进行研究, 其平均期望杂合度为0.260 7; 吕振明等[4]采用垂直聚丙烯酰胺同工酶电泳技术对 7个泥蚶群体的遗传多样性进行分析, 不同群体间位点数为 21~28, 平均期望杂合度为0.086~0.186, 平均观测杂合度为 0.035~0.083。本实验采用微卫星标记技术对 5个泥蚶群体的遗传多样性进行分析, 共检测到115个不同等位基因, 5个群体的He为0.573~0.656, Ho为0.430~0.516, 明显高于其他标记的等位基因数和杂合度, 这与姚韩韩等[3]采用相同技术对 2个泥蚶群体的遗传多样性的分析结果相一致。较高的等位基因数和杂合度, 说明微卫星标记技术相对于其他标记技术可以揭示出较高水平的遗传多样性, 是研究物种遗传多样性的理想工具, 也有可能是因为本实验中选择的17个位点均有较高的的理论多态信息含量。
17对微卫星引物的多态信息含量为0.142~0.857,依据Botstein等[21]的划分标准, 本研究的17个位点中, 有5个位点为中度多态位点, 10个位点为高度多态位点, 这些微卫星位点在泥蚶遗传多样性分析中可提供有效的信息。Teg28和Teg18的多态信息含量最低, 其中Teg28在韩国群体中为单态位点, 分析原因可能是样品数量不够充足或者位点本身多态信息含量低, 可能在韩国群体中为保守位点。5个泥蚶群体的平均多态信息含量介于 0.525~0.608, 其中山东群体的平均多态信息含量最高, 为0.608; 5个群体的平均多态信息含量均处于中度偏上水平, 具有较高的遗传多样性水平。
群体内Ho和He之间的平衡关系是由FIS反映的。当FIS<0时, 说明杂合子过度; FIS>0时, 说明杂合子缺失, FIS越接近0, 说明基因型的分布越接近平衡状态[25]。本实验中, FIS除了 3个微卫星位点外, 其余14个位点均大于0, FIS的平均值为0.217 4, 说明了泥蚶群体中杂合子缺失, 海洋贝类的杂合子缺失现象在很多贝类中都有相关报道[26,27]。由于杂合子缺失导致 43个(51.2%)群体位点偏离 Hardy-Weinberg平衡, Yu等[27]认为主要原因可能是无效等位基因的存在; 喻子牛等[24]认为可能是由于近交衰退、Wahlund效应、电泳图谱的阅读、自然选择等原因造成的。
3.2 泥蚶群体的遗传分化
遗传距离是用来估计不同种群之间遗传分化程度的一个指标, 根据其大小可以确定群体间亲缘关系的远近。群体间的遗传距离越大, 则基因型差异越大, 表明群体间的亲缘关系越远, 遗传相似性越小;反之亦然[28]。本研究采用17个微卫星位点对5个泥蚶群体进行遗传多样性分析, 根据Nei's指数法对泥蚶 5个群体的遗传结构进行遗传距离和遗传相似性统计分析, 结果表明广西-山东群体遗传相似度最大(0.936 5), 遗传距离最小(0.065), 聚为一支。浙江-韩国群体遗传相似度(0.915 9)仅次于广西-山东群体,也聚为一支。海南群体与上述两支群体间的遗传相似度比较接近, 所以处于两支群体之间。其中浙江-韩国群体遗传距离比较接近, 在很多其他的研究中均有此结论[6,29], 5个群体间遗传分化的水平的高低并不完全符合地理分布的特征, 分析原因可能是由于在泥蚶大规模养殖育种过程中, 不同地理群体之间互相引种[1,6], 造成不同地理群体之间交流频繁,尤其是泥蚶养殖业比较发达的地区, 致使群体间的遗传分化程度并不完全符合地理分布特征。
遗传分化系数 FST(F-statistic)和基因流(gene flow)是反映种群间的遗传分化程度和各种群间基因交流情况的重要指标[30]。当0 郑文娟等[7]利用 COI标记对泥蚶地理群体进行种群结构分析, 得到了我国沿海泥蚶已分化形成福建以南和以北两大类群的结论; Ni等[8]利用COI、ITS标记对中国东部海域和南海泥蚶类群进行种群遗传结构分析, 提出了两个进化显著单元的观点; 邵艳卿等[32]利用 AFLP对泥蚶种群结构进行分析, 提出未发现有效的南北间基因流屏障的结论; 刘春芳[33]利用 COI标记对泥蚶 7个地理群体进行分析, 发现泥蚶群体的遗传分化主要来自于群体内部, 与之前学者提出的南北分化且群体间的遗传距离大于群体内遗传距离的观点不甚相同。所以, 目前关于我国沿海泥蚶的南北区系分化的说法依然存在争议, 分析原因可能是由于取样的时间不同, 而且取样数量均较少, 不能代表整个地理群体的种质资源情况; 而且不同类型的标记方法特点不同, 都具有各自的优势和局限性, 检测基因组的部位也不同, 可能会导致结果不一样。 本研究利用17种微卫星标记对泥蚶5个地理群体进行遗传多样性分析, 结果显示泥蚶 5个群体的遗传多样性水平较高, 种质资源良好, 对泥蚶养殖产业的良种选育、种质资源保护等工作具有指导意义。但是, 本研究中采集的泥蚶的群体数和个体数有限, 为了更好地保护泥蚶的种质资源, 我们应加强对泥蚶群体资源的遗传学研究, 制定一套健康科学的保护措施, 保证泥蚶种质资源得到合理和可持续性的利用。 [1] 张永普, 林志华, 应雪萍, 等.不同地理种群泥蚶的形态差异与判别分析[J]. 水产学报, 2004, 28(3):339-342. [2] 姚韩韩, 董迎辉, 林志华, 等. 泥蚶 4个快速生长家系的遗传变异分析[J]. 水产学报, 2011, 35(3):340-347. [3] 姚韩韩, 林志华, 周小龙, 等. 泥蚶奉化种群与韩国种群形态特征和遗传结构的差异分析[J]. 江苏农业科学, 2014, 42(3): 183-186. [4] 吕振明, 李太武, 苏秀榕, 等. 泥蚶遗传多样性的研究[J]. 高科技通讯, 2005, 15(12): 104-110. [5] 王日昕, 李太武, 吕振明, 等. 泥蚶不同地理居群同工酶变异及遗传分化的研究[J]. 海洋与湖沼, 2005,36(3): 228-234. [6] 李太武, 李成华, 宋林生, 等. 5个泥蚶群体遗传多样性的RAPD分析[J].生物多样性, 2003, 11(2): 118-124. [7] 郑文娟, 朱世华, 沈锡权, 等. 基于线粒体 COI基因序列探讨泥蚶的遗传分化[J]. 动物学研究, 2009,30(1): 17-23. [8] Ni G, Li Q, Kong LF, et al. Phylogeography of the bivalve Tegillarca granosa in coastal China:implications for management and conservation[J].Marine Ecology Progress Series, 2012, 452: 119-130. [9] 刘华, 贾继增. 指纹图谱在作物品种鉴定中的应用[J]. 作物品种资源, 1997, (2): 45-48. [10] Powell W, Machray G C, Provan J. Polymorphism revealed by simple sequence repeats[J]. Trends Plant Sci, 1996, 1: 215-222. [11] 李宏俊, 刘晓, 杜雪地, 等. 海湾扇贝微卫星标记开发及其分离方式分析[J]. 海洋科学, 2009, 33(12):4-8. [12] 韩斐斐, 张继彪, 李莉, 等. 虾夷扇贝养殖群体及其子代的遗传多样性分析[J]. 海洋科学, 2012, 36(9):1-8. [13] Lu X, Wang H X, Liu B Z, et al. Microsatellite-based genetic and growth analysis for a diallel mating design of two stocks of the clam, Meretrix meretrix[J].Aquaculture research, 2012, 43(2): 260-270. [14] 张琳琳, 李莉, 张国范. 长牡蛎(Crassostrea gigas)EST 串联重复序列的组成和分布[J]. 海洋科学,2011, 35(4): 9-14. [15] 刘博, 邵艳卿, 滕爽爽, 等. 乐清湾养殖缢蛏群体遗传结构的微卫星标记分析[J]. 中国农学通报, 2012,28(2): 69-73. [16] 刘博, 邵艳卿, 王侃, 等. 4 个缢蛏群体遗传多样性和系统发生关系的微卫星分析[J]. 海洋科学, 2013,37(8): 96-102. [17] 汤健, 管云雁, 刘文广, 等. 马氏珠母贝家系遗传结构的微卫星分析[J]. 海洋科学, 2013, 37(8): 35-41. [18] 滕爽爽, 方军, 金文辉, 等. 4个斧文蛤群体微卫星标记的遗传多样性分析[J]. 中国水产科学, 2015, 22(3):580-587. [19] Sambrook J, Fritsch E, Maniatis T. Molecular Cloning:a Laboratory Manual[M]. third ed. New York : Cold Spring Harbor Laboratory Press, 2001. [20] Liu B, Teng S S, Shao Y Q, et al. Isolation and characterization of 39 novel polymorphic EST-SSR loci for the blood clam, Tegillarca granosa[J]. Conservation Genet Resour, 2012, 4(2): 375-378. [21] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet,1980, 32(3): 314-331. [22] 姚韩韩. 泥蚶野生群体与选育群体的分子遗传基础研究[D].上海: 上海海洋大学, 2011. [23] 邓春兴. 基于 COI基因序列的中国东南沿海四指马鲅属鱼类的遗传多样性分析[D]. 广州: 暨南大学,2014. [24] 喻子牛, 孔晓瑜, 杨锐, 等. 泥蚶等位基因酶遗传变异研究[J]. 中国水产科学, 1997, 4(5): 5-21. [25] Weir B S, Cockerham C C. Estimation F-statistics for the analysis of population structure[J].Evolution, 1984,38: 1358-1370. [26] 李刚, 金启增, 姜卫国, 等. 合浦珠母贝和长耳珠母贝的生化遗传变异[J]. 遗传学报, 1985, 12(3):204-212. [27] Yu H, Li Q. Genetic variation of wild and hatchery populations of the Pacific oyster Crassostrea gigas assessed by microsatellite markers[J]. Journal of Genetics and Genomics 2007, 34(12): 1114-1122. [28] 张爱兵, 王正军, 谭声江, 等. 分子生态学重要概念-遗传距离及其测定的理论研究概况[J]. 生态学报,2002, 22(6): 943-949. [29] 李成华, 李太武, 宋林生, 等. 福建南北泥蚶种内分化的RAPD分析[J]. 动物学研究, 2003, 24(5): 362-366. [30] Allendorf F W. Isolation, gene flow and genetic differentiation among populations[J]. Genetic and Conservation, 1983, 5l-65. [31] Weight S. Evolution and the genetics of population[M],Volume 4: variability within and among Natural Population Chicago. Chicago: university of Chicago press, 1987. [32] 邵艳卿, 柴雪良, 董迎辉等.地理距离对基因流的影响: 6个泥蚶群体的AFLP分析[C]//中国动物学会、中国海洋湖沼. 中国海洋湖沼学会贝类学会分会第十四次学会研讨会论文集.青岛: 中国海洋湖沼学会,2009: 95. [33] 刘春芳.中国近海蚶总科(Arcoidea)贝类的系统发育及泥蚶(Tegillarca granosa)不同群体遗传多样性研究[D]. 青岛: 中国科学院海洋研究所, 2014.
